老面,又称为老面酵头、酵子、面肥、起子、老酵面、传统酸面团等,是我国发酵面食中用到的一种传统发酵剂[1-2]。老面发酵技术是将上次制作面食制品留下的一块面团作为发酵剂用于下次面团的发酵,这样的过程称为传代发酵处理[3-4]。相比较于市售的单一酵母纯种发酵,老面发酵面团的优势在于其主要是以酵母菌、乳酸菌为主的多菌发酵体系,混合协同发酵不仅能够产生更加丰富的风味和口感,同时乳酸菌发酵过程中产生的有机酸还能够抑制病原微生物生长,延长发酵面食的保质期[5-6]。
老面发酵技术作为一种自然、有效的食品加工技术在改善面食制品品质方面的应用得到了越来越多的关注[7-9]。老面中的微生物菌群会被多种因素影响,如不同原料、地域环境和发酵工艺等[10-12]。MICHEL E等[13]通过高通量测序技术和实时定量聚合酶链式反应(polymerase chainreaction,PCR)法检测16个法国发酵面团样品中乳酸菌的相对丰度结果表明,旧金山乳杆菌(Lactobacillus sanfranciscensis)是主要的优势菌种;高静等[14]采用高通量技术分析老面中的菌群组成发现,河北、山西、山东样品中以片球菌属(Pediococcus claussen)为主,而云南样品中以乳杆菌属(Lactobacillus)为主;李晓敏等[15]对山西、河北、青海、甘肃和河南5个地区酸面团样品中的细菌菌群结构进行分析发现,优势菌群为乳杆菌属、乳球菌属(Lactococcus)和魏斯氏菌属(Weissella);闫博文[16]采用传统分离手段结合高通量测序技术对山东和河南2个地区的15个老面酵头样品的菌群多样性进行分析发现,山东老面酵头中主要是旧金山乳杆菌、食窦魏斯氏菌(Weissella cibaria),而河南酵头主要由食窦魏斯氏菌、植物乳杆菌(Lactobacillus plantarum)和戊糖片球菌(Pediococcus pentosaceus)构成。目前对我国不同区域老面样品及发酵不同时期的菌群结构差异的研究报道较多,对于老面传代发酵过程中的菌群结构变化的研究报道较少。老面中的微生物菌群结构是保障发酵面食产品质量和风味的前提与基础[17]。老面发酵因其菌群结构复杂,目前主要局限于家庭自用和小作坊生产。探究老面传代发酵过程的细菌多样性和菌群结构的演替规律,对于老面的工业化生产尤为重要。
本研究采用Illumina MiSeq高通量测序技术考察了传代发酵过程(1代、2代和4代)中老面样品中细菌菌群结构,对高通量测序结果进行α多样性及β多样性分析,并对其细菌菌群进行京都基因与基因组百科全书(Kyoto Encyclopedia of Genes and Genomes,KEGG)富集分析,以期揭示老面传代发酵过程中细菌群落结构的动态变化,为传统老面发酵剂的工业化生产提供一定理论基础。
1 材料与方法
1.1 材料与试剂
实验用老面(sourdough,SD)(即每次面团发酵完成后留取的部分面团,循环使用):来自于实验室保存;小麦粉(塞北雪):市售。Power Soil 脱氧核糖核酸(deoxyribonucleic acid,DNA)试剂盒:上海天根生化科技有限公司,2×Taq Plus Master Mix:美国KAPA公司;甲醇、乙腈、氨水、醋酸铵、2-氯苯丙氨酸(均为色谱纯):麦克林生化科技有限公司。
1.2 仪器与设备
高通量二代测序仪:美国Illumina公司;Agilent 2100生物分析仪:德国Agilent公司;Labchip GX生物大分子分析仪:美国PerkinElmer公司;ABI 9700 PCR仪:美国ABI公司;Nanodrop2000 超微量分光光度计:美国Thermo Scientific公司。
1.3 方法
1.3.1 传代发酵面团的制备
以老面50 g、小麦粉200 g、蒸馏水105 mL于UKOEO全自动揉面机智能模式下揉面15 min,取出覆保鲜膜在室温条件下(25~28 ℃)自然发酵至16 h,命名为第一代,取出50 g老面面团继续上述步骤,连续传代4代,取第1代、第2代、第4代老面发酵面团的样品分别命名为SD1、SD2和SD3[3]。每代发酵时期平行取3个发酵面团样本,编号分别为1、2和3。
1.3.2 Illumina MiSeq测序和数据分析[18]
根据Power Soil DNA试剂盒说明书,以老面发酵面团微生物总DNA为模板,采用338F(5'-ACTCCTACGGGAGGCAGCAG-3')和806R(5'-GGACTACHVGGGTWTCTAAT-3')为上下游引物扩增细菌16S rRNA的V3-V4区域。25 μL PCR扩增体系:DNA样品3 μL,2×Taq Plus Master Mix 12.5 μL,5 μmol/L 338F和806R各1 μL,双蒸水(ddH2O)7.5 μL。PCR扩增程序为:94 ℃预变性5 min;94 ℃变性30 s,55 ℃退火30 s,72 ℃延伸60 s,28个循环;72 ℃延伸7 min。扩增完成后,以3 μL PCR产物点样1.0%琼脂糖凝胶电泳确认PCR产物质量,以500 bp左右处出现清晰的条带为准。最后将PCR产物送至北京奥维森基因科技有限公司Illumina MiSeq平台测序,使用软件UPARSE进行质量控制,以97%相似性划分操作分类单元(operational taxonomic units,OTUs),并提交到奥维森云分析平台进行多样性分析(http://cloud.allwegene.com)。
1.3.3 功能预测分析[19]
使用PICRUSt软件进行微生物群落的代谢功能预测,参照京都基因与基因组百科全书(KEGG)数据库进行功能注释。采用Excel 2010软件进行数据整理和相关图表制作。
2 结果与分析
2.1 高通量测序结果和Alpha多样性分析
通过Illumina MiSeq高通量测序,9份老面样本共获得538 981条优化序列,平均长度为429 bp,各样本抽平OTU数和Alpha多样性指数见表1。由表1可知,所有序列均按97%相似度归为OTU,9个样品共聚合了495个OTU。经OTU分析,这些序列归属于6个门、14个纲、20个目、28个科、39个属、15个种。随着传代次数的增加,总OTUs、Phylum(门)、Class(纲)、Order(目)、Family(科)、Genus(属)、Species(种)的OTU数量,呈先减少后增加趋势。老面传代发酵过程中,四代(SD3)的总OTU数、属水平的OTU数量高于一代(SD1)和二代(SD2)。
表1 老面样品微生物菌群的操作分类单元数和Alpha多样性指数分析
Table 1 Operational taxonomic units number and alpha diversity index analysis of microbial community in sourdough samples
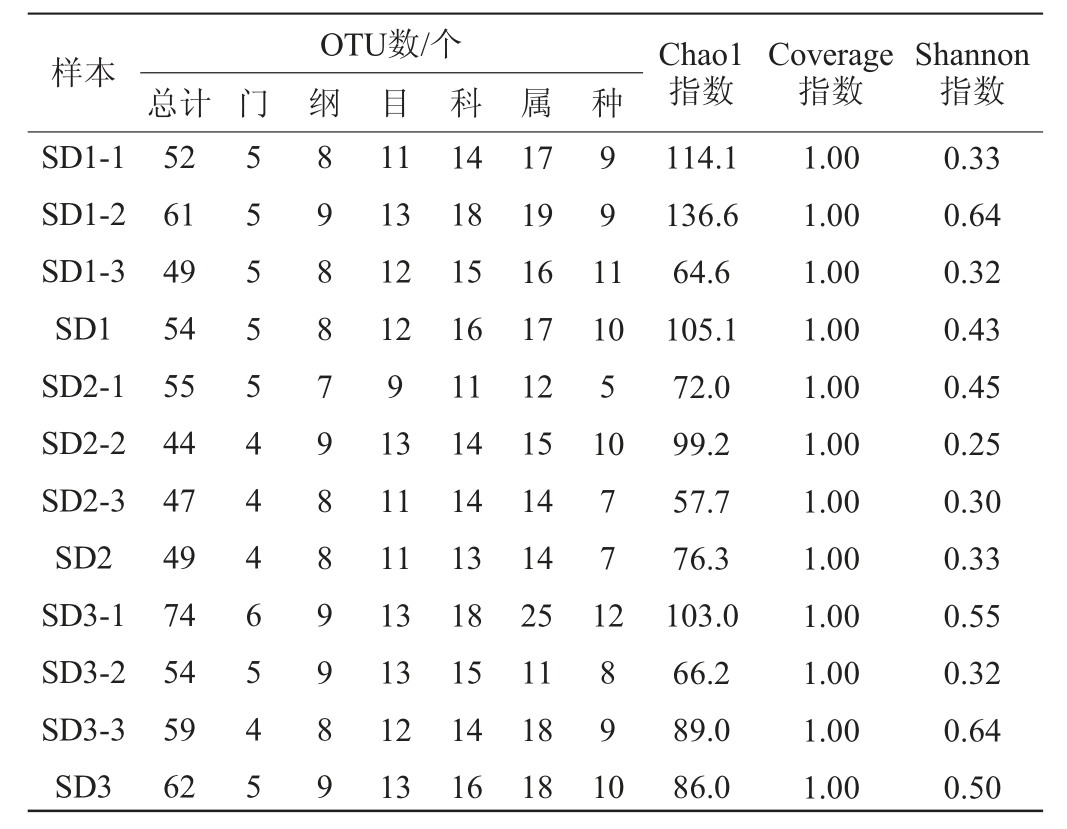
样本 Chao1指数OTU数/个总计 门 纲 目 科 属 种Coverage指数Shannon指数SD1-1 SD1-2 SD1-3 SD1 SD2-1 SD2-2 SD2-3 SD2 SD3-1 SD3-2 SD3-3 SD3 52 61 49 54 55 44 47 49 74 54 59 62 5 555544465 45 898879889989 11 13 12 12 9 13 11 11 13 13 12 13 14 18 15 16 11 14 14 13 18 15 14 16 17 19 16 17 12 15 14 14 25 11 18 18 991 1 10 5 10 771 2891 0 114.1 136.6 64.6 105.1 72.0 99.2 57.7 76.3 103.0 66.2 89.0 86.0 1.00 1.00 1.00 1.00 1.00 1.00 1.00 1.00 1.00 1.00 1.00 1.00 0.33 0.64 0.32 0.43 0.45 0.25 0.30 0.33 0.55 0.32 0.64 0.50
Shannon指数、Chao1指数是基于97%相似度水平下的OTU信息以评估样品微生物多样性和丰富度。Coverage指数表示测序深度,本研究中样品的Coverage指数均为1.00,表明测序深度良好,能够真实反映样品的微生物信息。Shannon指数和Coverage指数反映群落的多样性,Chao 1指数反映群落的丰富度。Shannon指数越大,说明群落多样性越高,Chao 1指数逐渐降低,说明微生物的丰富度逐渐降低[20]。由表1可知,Chao 1指数和Shannon指数变化趋势相同,均呈先降低后增加趋势,SD2样品的Chao 1指数和Shannon指数均明显低于SD1和SD3样品,说明老面传代发酵至第二代时,物种丰富度和群落多样性最低。
2.2 老面微生物群落结构分析
2.2.1 基于门水平细菌群落多样性分析
基于门水平不同老面样品的细菌群落结构见图1。由图1可知,在门分类水平上,主要优势菌门为厚壁菌门(Firmicutes),平均相对丰度呈先增加后减少趋势,SD1、SD2和SD3的平均相对丰度依次为93.62%、95.58%和92.38%,其次为蓝藻菌门(Cyanobacteria)和变形菌门(Proteobacteria),变化趋势与厚壁菌门相反,呈先减少后增加趋势,平均相对丰度分别为SD3(4.43%)>SD1(3.48%)>SD2(1.98%)、SD3(3.17%)>SD1(2.90%)>SD2(2.43%),拟杆菌门(Bacteroidetes)、疣微菌门(Verrucomicrobia)、放线菌门(Actinobacteria)的相对丰度极低(0.04%)。研究表明,厚壁菌门广泛存在于发酵食品中[21];高静等[14]采集河北、山西、山东、云南、青海、甘肃和河南酸面团地区的传统酸面团样品的高通量测序结果表明,厚壁菌门在所有样品中都占有绝对优势。因此,在门水平上,各地老面酵头的优势菌群有高度的一致性,厚壁菌门是最普遍、最丰富的存在。
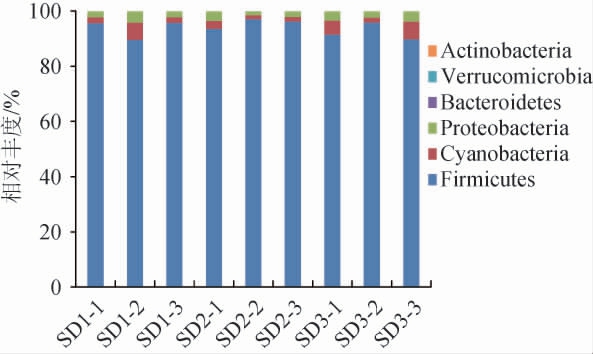
图1 基于门水平不同老面样品的细菌群落结构
Fig. 1 Bacterial community structure of sourdough samples based on the phylum level
2.2.2 基于属水平、种水平细菌群落多样性分析
基于属、种水平不同老面样品的细菌群落结构见图2。
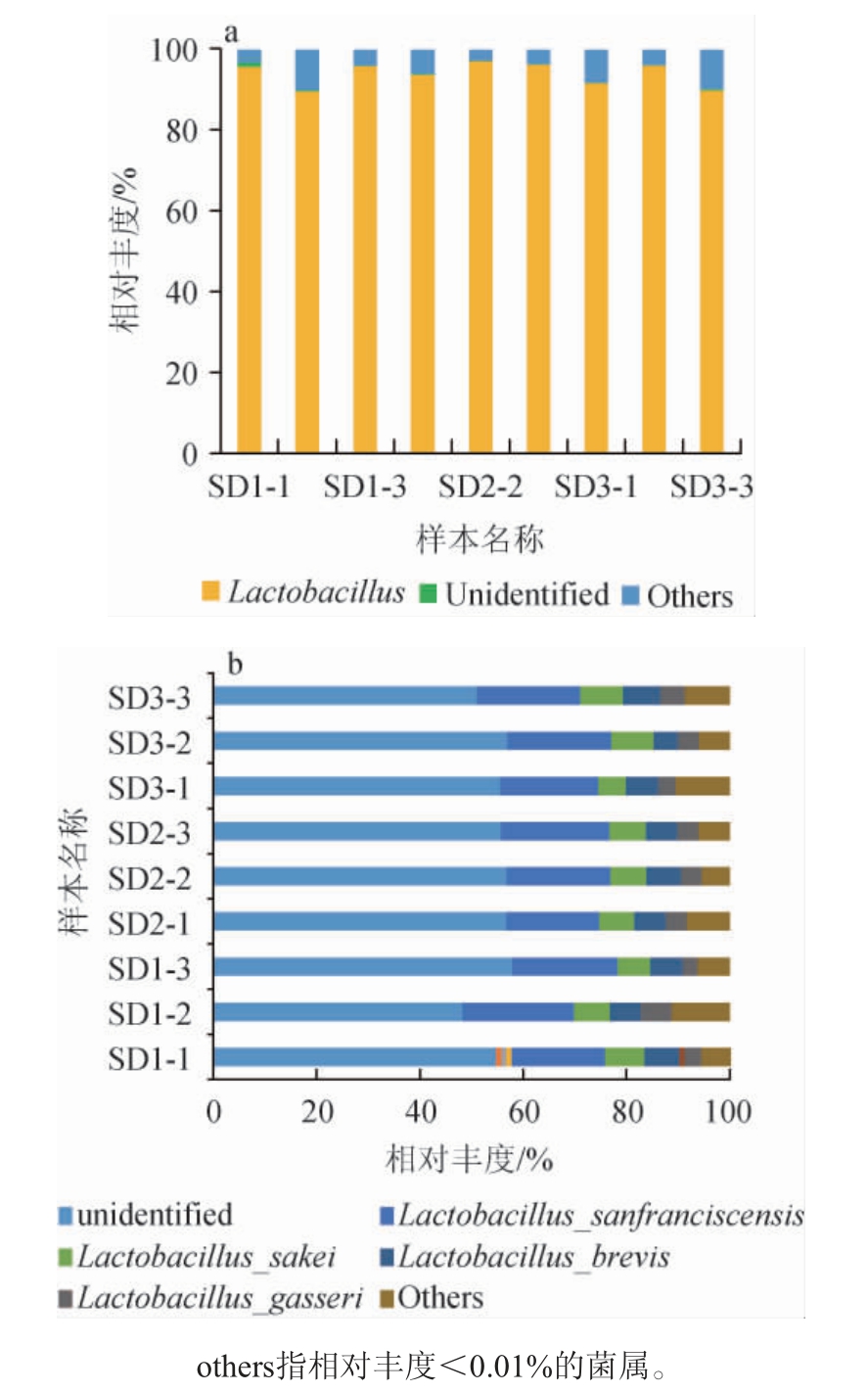
图2 基于属(a)、种(b)水平不同老面样品的细菌群落结构
Fig. 2 Bacterial community structure of sourdough samples based on the genus (a) and species (b) level
由图2a可知,老面传代样品发酵过程中,主要菌属(平均相对丰度>4%)为乳杆菌属(Lactobacillus),其属于厚壁菌门(Firmicutes)。乳杆菌属(Lactobacillus)的相对丰度呈先增加后减少趋势,其在SD1、SD2和SD3样品中的平均相对丰度依次为93.59%、95.54%和92.33%。
由图2b可知,除了未能鉴定的种属外,老面传代发酵中的优势种(平均相对丰度>4%)为旧金山乳杆菌(L.sanfranciscensis)、干酪乳杆菌(L.sakei)、短乳杆菌(L.brevis)和加氏乳杆菌(L.gasseri),它们均属于乳杆菌属(Lactobacillus)。其中旧金山乳杆菌(L.sanfranciscensis)和干酪乳杆菌(L.sakei)的平均相对丰度变化为先减少后增加,平均相对丰度分别为SD1(20.02%)>SD3(19.73%)>SD2(19.68%)和SD3(7.27%)>SD1(7.00%)>SD2(6.93%)。短乳杆菌(L.brevis)的平均相对丰度变化为一直减少,即SD1(6.35%)>SD2(6.33%)>SD3(6.11%),加氏乳杆菌(L.gasseri)的平均相对丰度无变化,均为4.01%。
研究表明,老面面团在连续发酵过程中,在发酵初期的优势菌主要是肠球菌属、乳球菌属和明串珠菌属;经过一段时间发酵后,面团中的乳酸杆菌属、片球菌属和魏氏菌属逐渐增多[22];发酵完成后,老面面团中以适应性专性异型发酵乳酸菌为主导,如旧金山乳杆菌、发酵乳杆菌、植物乳杆菌等乳酸杆菌属,而肠杆菌科、假单胞菌和霉菌等会在发酵过程中逐渐减少至无法检测[23-24]。产生这种变化的原因就在于老面面团中的乳酸菌能够发酵碳水化合物产生乳酸和乙酸,进而使面团的pH值不断降低,而乳酸菌对面团中的酸性环境适应性较强,逐渐成为其中的优势菌群,同时较低的pH抑制了肠杆菌科的生长[25]。同时老面发酵属于传统的自然发酵,以连续传代、连续发酵的方式生产,不需要额外添加菌种,发酵温度低(30 ℃以下),所以发酵完成后主要以短乳杆菌、植物乳杆菌、旧金山乳杆菌等适应低温发酵的乳酸杆菌为主导[1]。
本研究老面传代发酵过程中,主要是以厚壁菌门的乳杆菌属为主,在连续传代过程中,老面传代发酵中的优势种为乳杆菌属(Lactobacillus)的旧金山乳杆菌(L.sanfranciscensis)、干酪乳杆菌(L. sakei)、短乳杆菌(L. brevis)和加氏乳杆菌(L.gasseri)。在传代过程中,微生物多样性虽有变化,但相对含量变化不显著(P>0.05)。这也再次验证了前人的研究结论,说明老面发酵最终会形成一个趋于稳定、成熟的微生物区系,能够适应外界环境的变化[16,25]。
2.3 β多样性分析
2.3.1 聚类分析
为研究老面传代发酵过程中不同发酵阶段群落结构的相似性或差异关系,基于unifrac样本距离对3个发酵阶段样品细菌菌群的β多样性指数进行聚类分析,结果见图3。
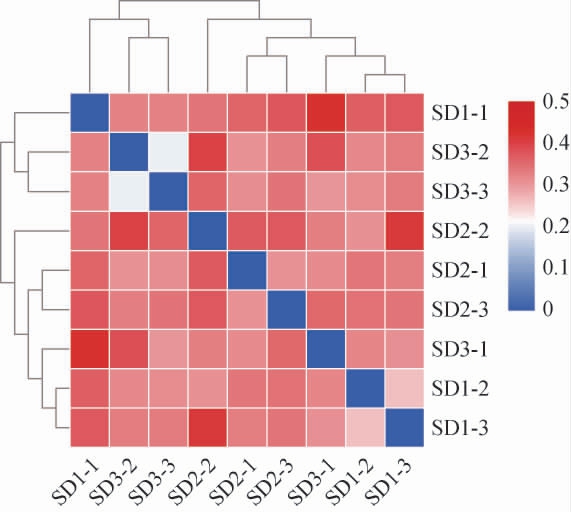
图3 基于属水平老面样品细菌菌群的β多样性指数聚类分析热图
Fig. 3 Heat map for β-diversity index cluster analysis of sourdough samples based on the genus level
由图3可知,3个发酵阶段可聚为3类,SD1-3、SD1-2、SD3-1聚为一类,SD2-1、SD2-2、SD2-3聚为一类,SD1-1、SD3-2、SD3-3聚为一类。由属水平细菌群落结构(图2a)可知,其中一代(SD1)、二代(SD2)、四代(SD3)的优势菌属均为乳杆菌属(Lactobacillus),平均相对丰度依次为93.59%、95.54%、92.33%。总体而言,老面传代发酵过程中在细菌群落结构较为相似,丰度变化不显著(P>0.05),这与细菌群落在属水平上相对丰度分析结果一致。
2.3.2 主坐标分析
主坐标分析(principal co-ordinates analysis,PCoA),是通过分析不同样本OTU(97%相似性)组成可以反映样本间的差异和距离来研究多个样品间数据的多样性、差异性或相似性的一种方法,通过PCoA图可以分析老面传代发酵过程中样品细菌群落的差异。
由图4可知,PC1的方差贡献率为94.59%,PC2的方差贡献率为4.36%,累计方差贡献率为98.95%,能很好地区分样品的细菌群落分布差异情况。在PCA中,各个点之间的距离越大,表明它们之间的菌群差异越大[26]。反之,样本点越接近,表明两样本物种组成则越相似[27]。在三个平行样本中,SD1相较于SD2和SD3其样本之间距离较远,表明1代老面样品组内的差异较大,可能是老面刚接入新鲜面粉中发酵,菌属分布不均匀所致。从整体来看,SD1-1、SD2-2、SD2-3、SD3-2分布于第一象限,SD1-2、SD3-1、SD3-3分布于第二象限,分布于同一象限的两两之间距离最近且聚集度较高,说明其细菌群落结构较为相似。而SD1、SD2和SD3在一、二象限均有分布且距离较近,因此,SD1、SD2和SD3在微生物群落组成和结构上也存在相似之处,这与样本聚类分析结果相同。
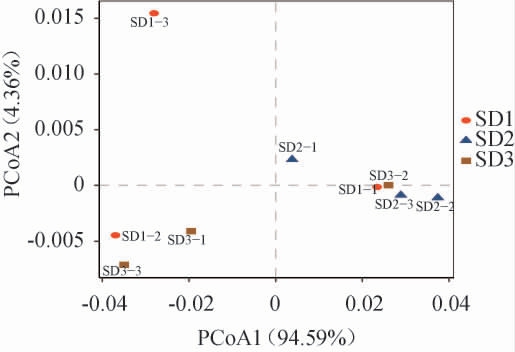
图4 老面样品在属水平上细菌菌群的主坐标分析
Fig. 4 Principal coordinates analysis of sourdough samples at the genus level
2.4 老面传代发酵过程中细菌菌群的代谢功能预测分析
老面传代发酵过程中细菌菌群KEGG代谢功能预测结果见图5。由图5可知,KEGG在一级水平上细菌群落功能包括有机系统、细胞进程、人类疾病、基因处理和代谢进程。二级水平上共14种代谢通路,关于代谢途径序列的相对丰度解释了90.44%。细菌群落功能大多集中在碳水化合物代谢(41.95%~43.57%)、维生素及辅酶因子代谢(15.25%~15.87%)、分子排序、结构折叠与降解(14.12%~14.67%)、氨基酸代谢(10.91%~11.19%)等功能分类中。碳水化合物代谢、维生素及辅酶因子代谢、能量代谢、萜类及聚酮化合物代谢等能为细菌的生长繁殖提供必需的营养物质;面团发酵过程中,碳水化合物的代谢主要是通过糖酵解途径和三羧酸循环进行。葡萄糖经糖酵解和三羧酸循环产生的α-酮酸,例如丙酮酸、乙酰辅酶A、α-酮戊二酸等,是参与氨基酸代谢及合成多种风味物质的重要前提物[16,28]。分子排序、结构折叠与降解、翻译是微生物自身合成遗传物质的主要代谢通路;氨基酸代谢在微生物演替中的作用主要包括两个方面,一方面是氨基酸的合成代谢,用于合成自身独特的蛋白质、多肽和其他含氮物质,以产生相应的氮源为微生物利用;另一方面是氨基酸的分解代谢,通过脱氨基、转氨基、联合脱氨基的方式分解为α-酮酸、胺类和二氧化碳,显著影响发酵食品风味的形成[29]。
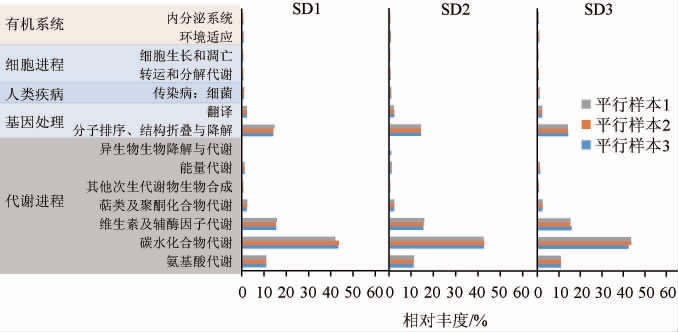
图5 老面传代发酵过程中细菌菌群KEGG代谢功能预测结果
Fig. 5 Prediction results of KEGG metabolic function of bacterial community in sourdough during generation fermentation
一代至四代老面样品碳水化合物的相对丰度先增加后减少;氨基酸代谢、维生素及辅酶因子代谢、萜类及聚酮化合物代谢、能量代谢、分子排序、结构折叠与降解的相对丰度先减少后增加。但老面酵头传代发酵过程中,细菌群落的代谢通路变化不显著(P>0.05),最主要代谢通路即碳水化合物代谢等功能成分的变化与乳杆菌属物种丰度指数变化趋势基本一致,即从一代到四代过程中,基因数量先减少,然后逐渐恢复,这说明群落结构的变化是其整体代谢功能发生变化的重要原因。
相对丰度排名前20的老面传代发酵过程中细菌菌群3级KEGG同源功能预测结果见图6。由图6可知,3级KEGG同源功能预测结果显示,在排名前20的代谢途径中,磷酸戊糖途径的相对丰度远远高于其他代谢途径。磷酸戊糖途径是异型乳酸菌发酵的主要代谢途径,在老面酵头传代发酵过程中具有重要作用。磷酸戊糖途径属于二级代谢途径中碳水化合物代谢途径,这再次验证了碳水化合物代谢途径在老面酵头发酵过程中有重要作用。发酵面制食品重要风味物质主要受乳酸菌影响,大约40%的风味物质如醇类、酯类、酮类、酸类、醛类等都和乳酸菌发酵相关[19]。旧金山乳杆菌的生长代谢的适宜温度和pH值与酸面团发酵过程中温度和pH值的变化相匹配,因此更利于它们的生长代谢[30];同时乳酸菌具有多种应激反应机制来克服酸性环境、高温或低温环境、高渗透压或脱水环境以及缺乏可利用底物环境等劣势条件[31]。

图6 老面传代发酵过程中细菌菌群3级KEGG同源功能预测结果
Fig. 6 Prediction results of 3rd grade KEGG homology function of sourdough bacterial community during generation fermentation
3 结论
利用Illumina Miseq高通量测序技术对老面传代发酵过程中的细菌菌群多样性进行解析,并对其细菌菌群进行京都基因与基因组百科全书(KEGG)富集分析。结果表明,从一代(SD1)到四代(SD3)老面样品中的优势菌门、属和种的相对丰度无显著变化(P>0.05),优势菌门为厚壁菌门(Firmicutes)、蓝藻菌门(Cyanobacteria)和变形菌门;优势菌属均为乳杆菌属(Lactobacillus);优势菌种为旧金山乳杆菌(L.sanfranciscensis),其次为干酪乳杆菌(L.sakei)、短乳杆菌(L.brevis)和加氏乳杆菌(L.gasseri),其相对丰度变化同群落丰度变化相同,不同传代间的老面样品样品的平均相对丰度变化不显著(P>0.05)。从SD1到SD3代老面样品中细菌菌群主要功能为碳水化合物代谢、其次为维生素及辅酶因子代谢、分子排序、结构折叠与降解、氨基酸代谢;磷酸戊糖途径是最主要的三级代谢功能。老面在发酵过程中,会形成复杂而稳定的微生物生态环境,传代处理对其群组成和动态变化影响较低,这不仅反映出对面团特定生境的适应潜力,也展示出乳酸菌的种内异质性。本研究可为推动老面发酵剂的工业化生产提供依据。
[1]苏珊,王玉婷,吴洁,等.酸面团中的优势微生物及其相互作用研究进展[J].食品与发酵工业,2023,49(7):316-323.
[2]刘建利,孙敏,曹晓虹,等.利用高通量测序技术分析民间面引子中的真菌多样性[J].食品科学,2018,39(22):186-194.
[3]杨小萍,卜晓苑,吴庆,等.基于非靶向代谢组学分析老面酵头传代发酵过程中的代谢差异[J].中国酿造,2022,41(9):111-117.
[4]刘同杰.传统酸面团中微生物多样性及其风味物质代谢研究[D].杭州:浙江大学,2017.
[5]于静,许倩,李芬,等.新疆7地区馕用酸面团微生物多样性分析[J].食品研究与开发,2023,44(2):183-189.
[6]张海燕,柳陈坚,刘祥祥,等.基于高通量测序分析老面细菌多样性[J].食品工业科技,2016,37(10):145-153.
[7]LIU T J,LI Y,WU S R,et al.Isolation and identification of bacteria and yeast from Chinese traditional sourdough[J]. Mod Food Sci Technol,2014,30(9):114-120.
[8]张国华,王伟,涂建,等.基于宏转录组学技术解析传统酸面团中微生物代谢机理[J].中国粮油学报,2019,34(11):10-16.
[9]吴庆,卜晓苑,辛世华,等.传统酸面团菌群结构及其风味物质分析[J].中国粮油学报,2023,38(4):43-50.
[10]邢小龙.河南地区老酵面团菌群结构及优势菌种复配研究[D].郑州:河南农业大学,2020.
[11] DE VUYST L, KERREBROECK S V, LEROY F. Microbial ecology and process technology of sourdough fermentation[J].Adv Appl Microbiol,2017,100:49-160.
[12] ARORA K, AMEUR H, POLO A, et al. Thirty years of knowledge on sourdough fermentation:A systematic review[J].Trends Food Sci Tech,2021,108:71-83.
[13] MICHEL E, MONFORT C, DEFFRASNES M, et al. Characterization of relative abundance of lactic acid bacteria species in French organic sour-dough by cultural,q PCR and MiSeq high-throughput sequencing methods[J].Int J Food Microbiol,2016,239:35-43.
[14]高静,刘敏,吴丹丹,等.基于高通量测序分析老面中菌群多样性及其对馒头品质的影响[J].中国粮油学报,2019,34(7):13-19.
[15]李晓敏,韩伟,黎琪,等.传统酸面团菌群结构及其酵制馒头风味物质分析[J].食品科学,2021,42(10):162-170.
[16]闫博文.老面酵头微生物菌群多样性差异分析及其对馒头风味特性的影响[D].无锡:江南大学,2019.
[17]李梁,薛蓓,罗章,等.基于高通量测序分析冻干老面与新鲜老面细菌群落结构[J].中国食品添加剂,2018(10):126-131.
[18]阿尔菲娅·安尼瓦尔,伊萨克·阿卜杜热合曼,等.吐鲁番传统馕饼酸面团中微生物多样性及挥发性香气成分的分[J].微生物学通报,2017,44(8):1908-1917.
[19]WILKINSON T J,HUWS S A,EDWARDS J E,et al.CowPI: a rumen microbiome focussed version of the PICRUSt functional inference software[J].Front Microbiol,2018,9:1095.
[20]吴进菊,李宇昂,王梓杭,等.襄阳大头菜发酵过程中细菌的多样性[J].食品科学,2020,41(4):112-117.
[21]马上超,程超,田成,等.基于高通量测序技术分析恩施州淡豆豉的细菌多样性[J].食品科学,2022,43(10):156-161.
[22]HARTH H,KERREBROECK S V,VUYST L D.Community dynamics and metabolite target analysis of spontaneous,backslopped barley sourdough fermentations under laboratory and bakery conditions[J]. Int J Food Microbiol,2016,228(2):22-32.
[23] MAIDANA S D, FICOSECO C A, BASSI D, et al. Biodiversity and technological-functional potential of lactic acid bacteria isolated from spontaneously fermented chia sourdough[J].Int J Food Microbiol,2020,316:108425.
[24] OSHIRO M, MOMODA R, TANAKA M, et al. Dense tracking of the dynamics of the microbial community and chemicals constituents in spontaneous wheat sourdough during two months of backslopping[J].J Biosci Bioeng,2019,128(2):170-176.
[25] DINARDO F R, MINERVINI F, DE ANGELIS M, et al. Dynamics of Enterobacteriaceae and Lactobacilli in model sourdoughs are driven by pH and concentrations of sucrose and ferulic acid[J]. LWT-Food Sci Technol,2019,114:108394.
[26]贺成虎,赵海珍,陆兆新,等.高通量测序分析麦麸发酵过程中微生物群落结构的变化[J].食品科学,2020,41(24):102-109.
[27]谢芳,谢华德,唐振华,等.基于16S rDNA高通量测序分析水牛初乳与常乳中细菌多样性[J].食品工业科技,2021,42(13):125-132.
[28]郭淑文,石晶红.基于高通量测序分析内蒙古地区传统酸面团中细菌菌群的多样性[J].中国酿造,2022,41(12):86-90.
[29]张楚楚.自然发酵面团细菌菌群结构对馒头挥发性风味物质影响的研究[D].无锡:江南大学,2017.
[30]傅莉莉.西北地区酸面团中的微生物区系变化研究[D].北京:北京林业大学,2020.
[31]PETEL C,ONNO B,PROST C.Sourdough volatile compounds and their contribution to bread:A review[J].Trends Food Sci Technol,2017,59:105-124.