酱油是起源于我国的传统酿造调味品,并传播到日本、韩国、东南亚等国家地区,其独特的鲜香风味广受东亚及东南亚人民喜爱,成为了东方美食不可或缺的调味品。中国与日本是世界上两大酱油生产国与消费国,两者在悠久的历史中发展出符合自身饮食文化的独特工艺及风味,但酱油的酿造依旧高度依赖于米曲霉(Aspergillus oryzae)的发酵及其酶系的作用。米曲霉作为酱油酿造的主发酵菌,提供了丰富的酶类用于水解、转化原料中的蛋白质[1],形成氨基酸、肽、糖、有机酸及众多复杂的生化反应产物,赋予了酱油独特的色、香、味、体等特征[2-3]。因此,优良的米曲霉菌种是酱油酿造行业不断追求的核心技术,其中米曲霉沪酿3.042自被诱变选育出来后就成为我国众多酱油生产企业使用的酱油生产菌株,该菌株酶活旺盛,原料的利用率显著提高[4]。为了进一步提高原料利用率和发酵效率,研究者通过多种育种方式筛选蛋白酶活力更高的菌株[5-7],并证明了高蛋白酶活力菌株具有良好的发酵性能和氨基酸态氮提升效果。
酱油的独特风味不仅来源于氨基酸,肽类物质对风味同样具有显著影响,如苦味肽、抑咸肽、浓厚肽、鲜味肽等[8-10],这些呈味肽的种类与含量差异会引起酱油风味的差异,而酱油曲中蛋白酶的种类及酶活直接影响了呈味肽的组成。过去在米曲霉育种工作中,大部分研究以提升蛋白酶活力为目的,缺少对产蛋白酶系菌株更精细的定向选育,各种蛋白酶对风味的影响也缺乏系统的研究。
随着现代生物技术的发展,人们对米曲霉的认识逐步深入,育种工作深入到米曲霉蛋白分子、基因等微观层面,使得更精准的定向选育成为可能。本文从米曲霉基因组数据角度分析米曲霉蛋白酶资源基因挖掘现状,蛋白酶(分泌型内肽酶、分泌型外肽酶)的酶学特性研究进展,以期了解米曲霉的主要蛋白酶组成、特性以及对呈味物质形成的作用,推动基于特定风味物质形成的米曲霉菌种定向选育及酶系优化工艺开发,促进我国酱油发酵菌种的迭代升级,提升我国酱油产品品质。
1 米曲霉蛋白酶基因的挖掘
2005年由日本、英国和美国共26个研究机构合作率先完成了对Aspergillus oryzae RIB40的全基因组测序,首次揭示了米曲霉的基因组信息[11],我国科学家2012年也完成了对沪酿3.042的全基因组测序[12]。KOBAYASHI T等[13]通过对Aspergillus oryzae RIB40的基因进行分析,挖掘出134个蛋白酶基因,其中69个为外肽酶基因,65个为内肽酶基因,占米曲霉基因总数的1%以上,并发现有较多可在酸性pH环境中作用的蛋白酶。随着研究人员对米曲霉基因组数据的不断分析,有更多蛋白酶基因和同源物被发现。MEROPS数据库(https://www.ebi.ac.uk/merops)显示,不同种属曲霉的肽酶及非肽酶同源基因数量存在明显差异,如表1所示,米曲霉基因组中已知和潜在的肽酶基因有335个,非肽酶类的同源基因(即基因只在碱基序列上与对应家族蛋白酶具有同源性,但其表达的蛋白质为无活性酶蛋白或非蛋白酶活性蛋白)有206个,高于黑曲霉、构巢曲霉、黄曲霉、烟曲霉、白曲霉等曲霉属真菌[14]。
表1 不同曲霉的肽酶及非肽酶同源基因数量差异
Table 1 Differences in the number of peptidase and non-peptidase homologous genes in different Aspergillus

注:AP代表天冬氨酸蛋白酶、CP代表半胱氨酸蛋白酶、GP代表谷氨酸蛋白酶、MP代表金属蛋白酶、SP代表丝氨酸蛋白酶、TP代表苏氨酸蛋白酶。
种属 参考菌株 肽酶基因数 非肽酶同源基因数 AP CP GP MP SP TP米曲霉(Aspergillus oryzae)黑曲霉(Aspergillus niger)构巢曲霉(Aspergillus nidulans)黄曲霉(Aspergillus flavus)烟曲霉(Aspergillus fumigatus)白曲霉(Aspergillus kawachii)RIB40 ATCC 1015 FGSC A4 NRRL3357 A1163 IFO 4308 335 209 280 173 252 100 206 94 227 62 204 14 19 22 18 17 9 13 82 87 68 63 72 53 572535 123 113 91 91 93 64 212 183 143 114 121 59 24 22 21 22 19 18
尽管米曲霉的蛋白酶基因数更高,但大部分为胞内或膜结合蛋白酶。根据MEROPS数据库对不同种属曲霉的分泌型胞外蛋白酶基因家族分布进行汇总,如表2所示。
表2 不同曲霉的分泌型蛋白酶基因家族分布
Table 2 Distribution of secreted protease gene families in different Aspergillus
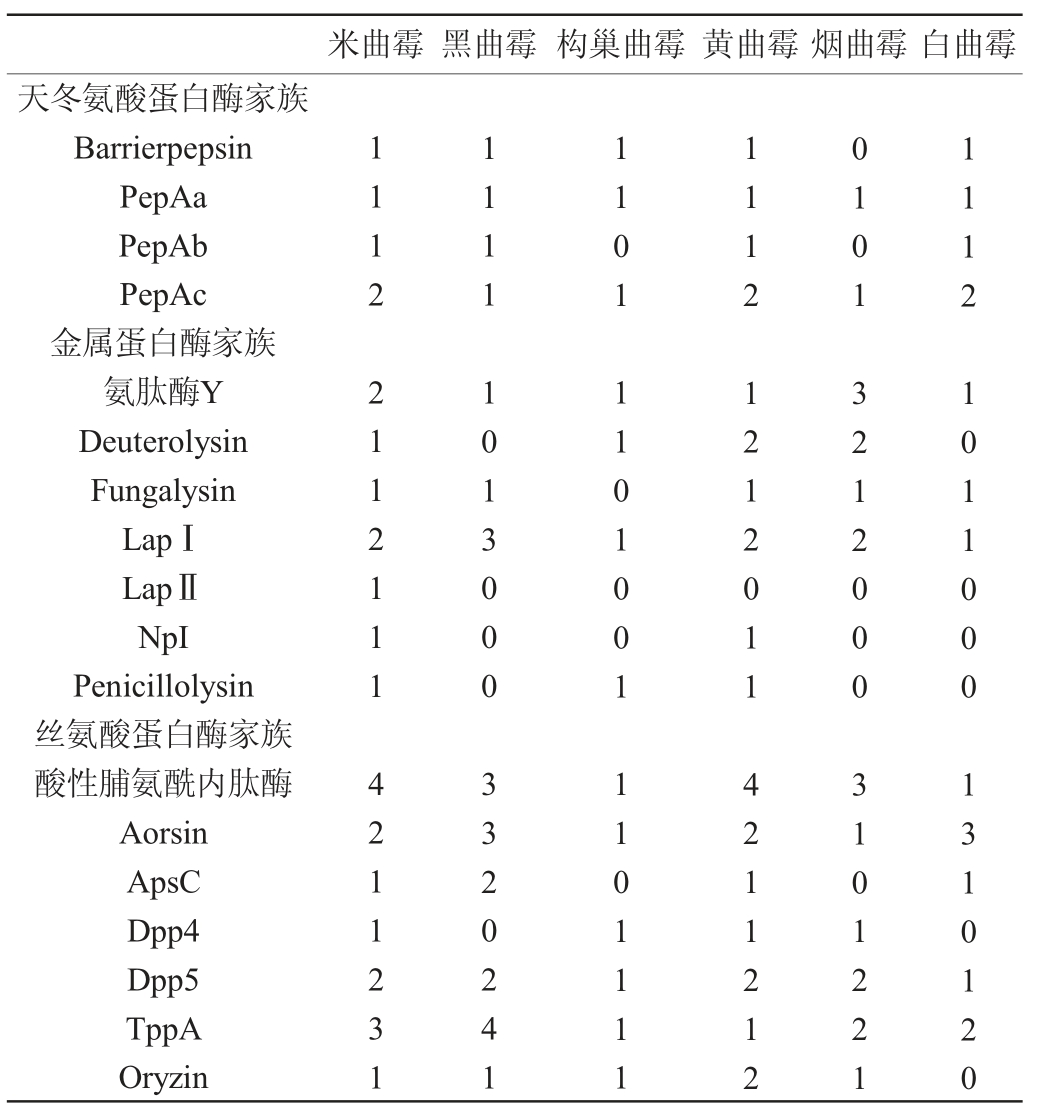
米曲霉 黑曲霉 构巢曲霉 黄曲霉烟曲霉 白曲霉天冬氨酸蛋白酶家族Barrierpepsin PepAa PepAb PepAc金属蛋白酶家族氨肽酶Y Deuterolysin Fungalysin LapⅠLapⅡNpI Penicillolysin丝氨酸蛋白酶家族酸性脯氨酰内肽酶Aorsin ApsC Dpp4 Dpp5 TppA Oryzin 1112 2112111 4211231 1111 1013000 3320241 1101 1101001 1101111 1112 1212011 4211212 0101 3212000 3101221 1112 1011000 1310120
分泌型胞外蛋白酶分别隶属于天冬氨酸蛋白酶、金属蛋白酶和丝氨酸蛋白酶三个家族,对应了酱油酿造行业关注米曲霉的酸性蛋白酶、中性蛋白酶和碱性蛋白酶。与其他类型曲霉相比,米曲霉在三种主要的分泌型胞外蛋白酶家族中均有丰富的基因分布,其中天冬氨酸蛋白酶均为胃蛋白酶样的酸性蛋白酶,金属蛋白酶包括中性蛋白酶I(NpI)、中性蛋白酶II(Deuterolysin、Penicillolysin)、中性蛋白酶III(Fungalysin)和氨肽酶(氨肽酶Y、亮氨酰氨肽酶1、亮氨酰氨肽酶2),丝氨酸蛋白酶包括碱性蛋白酶(Oryzin)、酸性脯氨酰内肽酶、胰蛋白酶样蛋白酶(Aorsin)、二肽酶(ApsC、Dpp4、Dpp5)和三肽酶(TppA)。天冬氨酸蛋白酶家族适合在低pH环境中发挥水解作用,米曲霉含有与黑曲霉、白曲霉等耐酸真菌相同的蛋白酶基因,说明米曲霉具有良好的产酸性蛋白酶的能力,该类型蛋白酶适合在弱酸性酱油发酵体系中催化,对于提升酱醪发酵过程中的原料利用率至关重要。与黑曲霉、白曲霉相比,米曲霉同时含有更丰富的胞外金属蛋白酶家族基因,该家族蛋白酶具有良好的热稳定性,涵盖了中性蛋白酶和多种氨肽酶,这两类蛋白酶对于原料蛋白的水解以及游离氨基酸的释放具有重要意义,是提升酱油氨基酸的重要贡献者。在丝氨酸蛋白酶家族方面,米曲霉与黄曲霉具有显著的酶系优势,该家族蛋白通常具有耐高pH特点,包括了具有广泛酶切位点特异性的碱性蛋白酶,同时含有丰富的二肽酶与三肽酶,对于将大分子蛋白质水解成小分子寡肽具有重要意义,而小分子寡肽是呈味肽的重要来源以及氨肽酶催化形成游离氨基酸的重要底物,因此对于酱油风味的形成至关重要。
天冬氨酰氨肽酶DapA在米曲霉液态发酵状态下以胞内酶形式存在[15],但在米曲霉固态发酵时却以胞外酶形式分泌[16],并对鲜味氨基酸的释放具有重要作用。酱油酿造过程中米曲霉蛋白酶系水解蛋白质的机制见图1[17]。由图1可知,大豆和小麦原料中的蛋白质在碱性蛋白酶、中性蛋白酶和酸性蛋白酶的共同作用下,酶解释放出可溶性的大分子肽,随后由各种肽酶作用进行进一步切割释放出氨基酸和寡肽,其中亮氨酰氨肽酶(Lap)负责从肽分子的N端释放疏水氨基酸达到脱苦效果,二肽酶4(Dpp4)负责切割并释放N端的Xaa-Pro二肽解除脯氨酸对酶解的限制作用,天冬氨酰氨肽酶(Dap)负责从N端释放酸性氨基酸从而达到提升酱油鲜味的作用,游离谷氨酰胺以及肽分子中的谷氨酰胺分别在谷氨酰胺酶和蛋白质谷氨酰胺酶的催化下水解成谷氨酸,后者在Lap的催化下进一步被转化成游离态,进一步提升鲜味氨基酸的含量。由此可见,米曲霉蛋白酶的协同作用是蛋白质转化及风味形成的关键。尽管不同米曲霉在酶系上十分相近,但各自酿造的酱油在呈味肽种类及含量上都有显著差异,一些对风味具有修饰作用的蛋白酶尚未被充分认识,因此,挖掘米曲霉中更多未知的蛋白酶对于深入研究米曲霉在原料蛋白降解和风味的形成中的作用具有重要意义。
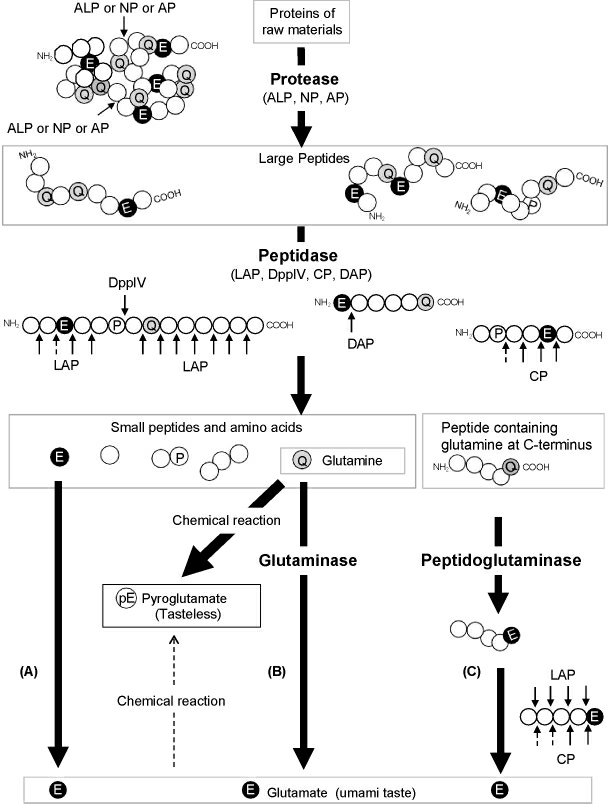
图1 酱油酿造过程中米曲霉蛋白酶系水解蛋白质的机制
Fig. 1 Mechanism of protein hydrolysis by proteases from Aspergillus oryzae during soy sauce brewing process
2 米曲霉源蛋白酶酶学特性
根据水解位点的不同,米曲霉的蛋白酶可被分为内肽酶与外肽酶,其中内肽酶包括酸性蛋白酶、中性蛋白酶、碱性蛋白酶、脯氨酰内肽酶等,外肽酶包括亮氨酰氨肽酶、天冬氨酰氨肽酶、氨基酰脯氨酰氨肽酶、二肽酶、三肽酶等。在酱油酿造过程中,来源于大豆和小麦的蛋白首先通过内肽酶的作用被转化为可溶性蛋白,然后被进一步切割为分子质量更小的肽分子,同时外肽酶作用于肽分子的末端,释放出氨基酸、二肽和三肽,产生大量呈味氨基酸和呈味肽。米曲霉的中性蛋白酶[18]、碱性蛋白酶[19]、氨肽酶[20-21]对于酱油氨基酸态氮和全氮的提升具有重要作用,因此是过去米曲霉育种工作中重点评估的生化指标。根据现有的研究报道,综合MEROPS蛋白酶数据库[14]、BRENDA酶数据库[22](https://www.brenda-enzymes.org/index.php)、Uniprot蛋 白质数据库[23](https://www.uniprot.org/)的信息,对米曲霉主要的分泌型内肽酶和外肽酶进行整理和介绍。
2.1 分泌型内肽酶
2.1.1 中性蛋白酶
目前已发现米曲霉可分泌至少三种中性蛋白酶,分别为中性蛋白酶I和中性蛋白酶II,均为含Zn2+的金属蛋白酶。中性蛋白酶普遍具有较高的热稳定性,如属于丝氨酸蛋白酶的碱性蛋白酶最适酶活温度为40 ℃[24],中性蛋白酶I的最适酶活温度则为55 ℃[25],中性蛋白酶II的最适温度甚至可以达到60~70 ℃,但在65~75 ℃容易发生自溶而失活[26]。
中性蛋白酶I由npI基因编码,毕赤酵母表达沪酿3.042来源的中性蛋白酶I进行酶学特性分析,显示其最适pH为7.0~8.0,在5.0~9.0范围内依旧保持稳定,胰岛素B链酶切位点分析显示,中性蛋白酶I偏好于识别并水解P1'位为亮氨酸(Leu)或苯丙氨酸(Phe)的肽键[24],因此会释放出N端含疏水氨基酸的肽类,这些肽类往往具有苦味,需要与亮氨酰氨肽酶协作达到脱苦的效果[27]。中性蛋白酶I在发酵中主要发挥将原料蛋白转化为可溶性蛋白以及将大分子蛋白质进行有限度的切割的作用,因此提升中性蛋白酶活力有助于总氮含量的提升,提高原料蛋白的利用率,但其自身无法产生小分子肽或氨基酸,对氨基酸态氮提升不明显[15]。
中性蛋白酶II又称为Deuterolysin,由TATSUMI H等[28]于1991年从Aspergillus oryzae ATCC20386中发现,随后HIROSHI M等[26]利用Aspergillus oryzae RIB40的基因组数据进行挖掘,发现了另外三个与中性蛋白酶II同源的基因(deuA、deuB、deuC),通过异源表达与酶学特性分析,证明DeuA与过去报道的NpII一致,该蛋白酶具有极高的耐热性,但与中性蛋白酶I相比,其底物特异性较强,对大豆蛋白的水解能力较弱,但对组蛋白、鱼精蛋白等碱性蛋白的特异性较高。在酶切位点特异性上,偏好切割P1'位为疏水氨基酸的肽键,与中性蛋白酶I类似。DeuB与DeuA类似[29],但热稳定性较差,DeuC的特性及功能目前尚不明确。
中性蛋白酶III又称为Fungalysin,2005年发布的米曲霉全基因组测序及分析证明了该蛋白酶编码基因的存在,MEROPS数据库显示,胰岛素B链酶切实验证明Fungalysin对P1'位为Leu或Phe的肽键具有显著的偏好性,与中性蛋白酶I相似。目前该蛋白酶在米曲霉生长发酵过程中的作用尚不明确,在烟曲霉中Fungalysin可能作为一种毒力因子,具有水解细胞外基质中的弹性蛋白和胶原蛋白功能,破坏细胞屏障从而促进感染发生[30]。
2.1.2 碱性蛋白酶
米曲霉来源的碱性蛋白酶又称为Oryzin,由alpA基因编码,属于丝氨酸蛋白酶,在pH 7.0~10.0范围内具有高水解活性[31-32]。与中性蛋白酶类似,碱性蛋白酶参与了原料蛋白转化为可溶性全氮的过程,并对可溶性蛋白进一步水解释放出多肽,但氨基酸释放能力很弱。与中性蛋白酶相比,碱性蛋白酶在酶切位点上更加广泛,对于疏水氨基酸、芳香族氨基酸和碱性氨基酸的羧基端肽键有较高的特异性[33],因此会释放出苦味肽,同时具有酯酶活力,能水解由氨基酸或肽类羧基端与各种醇类形成的酯键,这种酯酶活力在其他丝氨酸蛋白酶中也较为常见。
WATANABE J[34]从Aspergillus oryzae RIB40基因组中发现了与该基因同源的的alpB(AO090020000517)基因,并利用基因工程手段构建出ΔalpB菌株,该基因敲除菌株在曲汁培养基上表现为菌落发白,分生孢子形成显著减少的性状。Uniprot数据库显示,基因AlpB参与了细胞的自噬过程,该基因的缺失会导致菌株自噬进行受阻,进而影响细胞的分化与孢子的形成,此外,敲除菌株的总蛋白酶活力提升至原始菌株的1.1倍,进一步提高了菌株的实用性,这可能与基因Alpb缺失导致米曲霉孢子分化受阻,延长了米曲霉菌丝体形态的存在时间,而菌丝体是米曲霉胞外水解酶分泌的关键阶段,从而提升了蛋白酶的总活力。
2.1.3 酸性蛋白酶
酸性蛋白酶是一种分泌型的天冬氨酸蛋白酶,通过活性位点中的两个天冬氨酸残基激活亲核水分子攻击肽键实现水解功能。酸性蛋白酶全酶中存在一段含57个氨基酸的前肽,在pH<4.6时前肽会被切除形成成熟的酸性蛋白酶,成熟的酸性蛋白酶在pH 3.0~6.0范围内可以保持稳定,pH<2.0或>7.0时蛋白酶会失活。酸性蛋白酶偏好于切割P1位和P1'位均为疏水氨基酸的肽键。TADANOBU N等[35]利用从Aspergillus oryzae 460菌株中分离的酸性蛋白酶水解大豆蛋白,发现在水解达到稳定后共释放出11%的原料全氮,水解产物中含有7~9个氨基酸组成的肽分子,但未检测出氨基酸,说明酸性氨基酸的主要功能是提升可溶性全氮,对氨基酸态氮影响较小。
酸性蛋白酶由pepA基因编码[36],不同菌株在酸性蛋白酶的基因数目上可能有所不同,如RANDY M B等[37]从米曲霉的基因组分析中发现,pepO酸性蛋白酶编码基因,在酱油曲霉中也含有额外且独特的酸性氨基酸基因序列。值得注意的是,酸性蛋白酶基因只能在固态发酵时表达,且受培养温度影响较大[38],由此证明制曲工艺的水分与温度可能影响酸性蛋白酶的表达量,进而影响酶系比例、原料水解与风味的形成。
2.2 分泌型外肽酶
2.2.1 二肽酶IV
二肽酶IV又称为氨基酰脯氨酰二肽酶,是一种分泌型的丝氨酸蛋白酶[39],其最适pH作用范围为7.0~7.5,能够特异性识别多肽N端的Xaa-Pro序列,并以Xaa-Pro为单位水解与脯氨酸(Pro)残基羧基端连接的肽键,释放出Xaa-Pro二肽,当二肽C端连接的氨基酸为Pro时(Xaa-Pro-Pro-)肽键无法被水解[40]。由于脯氨酸独特的分子刚性结构造成的空间位阻,大部分氨肽酶无法从N端释放Xaa-Pro-序列,导致多肽的N端水解被终止,二肽酶IV的作用在于将Xaa-Pro-切除暴露出其他氨肽酶可切割位点,从而提升多肽的水解程度,有助于提升氨基酸态氮。研究表明,缺少二肽酶IV活力的酶解产物会大量积累分子质量在1 000 Da左右的多肽[41]。由此可证明,氨基酰脯氨酰二肽酶对于多肽的降解、氨基酸态氮的提升、鲜味氨基酸的释放具有重要作用,同时也解释了酱油中含有大量Xaa-Pro的原因。
2.2.2 亮氨酰氨肽酶
米曲霉基因组中含有两种亮氨酰氨肽酶基因,分别为lapI和lapII,均为外切型的金属蛋白酶。LapI的全酶序列中含有一段79个氨基酸的前肽,切除后形成成熟的酶蛋白,其最适作用pH范围在7.0~8.5,能够选择性从蛋白质或多肽的N端逐个释放氨基酸,其中底物多肽N端为亮氨酸时具有很高的水解活性,因此可以释放由中性蛋白酶和碱性蛋白酶水解产生的苦味肽的N端疏水氨基酸,从而显著降低酶解产物的苦味,该酶是风味蛋白酶具有脱苦作用的关键。此外,亮氨酰氨肽酶也能水解N端为其他氨基酸的多肽,如水解Phe-pNA、Met-pNA、Lys-pNA和Arg-pNA的活性分别为水解Leu-pNA活性的39%、29.6%、24.5%和16.5%,对Ala-pNA、Pro-pNA、Ile-pNA、Val-pNA、Gly-pNA和Glu-pNA的水解能力很弱,无法水解Asp-pNA[42-43]。由此可以推测,亮氨酰氨肽酶主要发挥脱苦和提升氨基酸态氮的作用,但对于鲜味及甜味氨基酸的释放作用较小。此外,LapI只能作用于氨基酸数目>3个的肽分子,因此不会破坏普遍具有呈味特性的二肽和三肽。LapII在制曲过程中的表达水平低于lapI,LapII最适pH范围为9.5~10.0,且底物特异性较LapI更低,对于所有类型的氨基酰对硝基苯胺(Xaa-pNA)都有水解作用,但无法作用于与Pro连接的N端氨基酸肽键[35]。
2.2.3 天冬氨酰氨肽酶
天冬氨酰氨肽酶是一种外切型金属蛋白酶,由dapA基因编码,其最适作用pH范围为7.0~8.0,在pH 5.0~8.0范围内可以保持稳定,对多肽N端的酸性氨基酸,尤其是天冬氨酸具有很强的特异性,同时能够水解释放出N端的谷氨酸,但不作用于其他类型的氨基酸[44]。虽然天冬氨酰氨肽酶在Uniprot数据库中显示不含有信号肽结构,但KUSUMOTO K I等[45]构建了一株过表达天冬氨酰氨肽酶的米曲霉,在进行液体培养时,天冬氨酰氨肽酶以胞内酶的形式存在,而在米曲中培养则以胞外酶的形式存在,说明固态发酵有利于天冬氨酰氨肽酶的分泌。此外天冬氨酰氨肽酶具有一定的耐盐性,JUN W等[44]利用大肠杆菌对米曲霉来源的天冬氨酰氨肽酶进行异源表达,通过酶学特性分析,证明该酶在15%NaCl溶液中可以保持40%的活力,在20%NaCl溶液中保持30%的活力,进一步将纯酶作用于脱脂大豆部分水解产物中,发现谷氨酸含量提升至对照组的1.12倍,由此推测,天冬氨酰氨肽酶独特的酶切特异性及中度耐盐性可能对于酱油发酵体系中鲜味氨基酸的提升至关重要。
2.2.4 氨基酰脯氨酰氨肽酶
氨基酰脯氨酰氨肽酶,又称为Prolidase或氨肽酶P,是一种以Mn2+为辅因子的金属蛋白酶,具有水解Xaa-Pro二肽肽键的能力,同时可以直接作用于多肽N端的Xaa-Pro-二肽序列,直接从多肽N端释放氨基酸,但无法水解Gly-Pro,该酶与氨基酰脯氨酰二肽酶协同作用有助于提升酱油的氨基酸态氮含量。该酶的最适作用pH为8.5~9.0,在pH 3.5~12.0的范围内均能保持稳定,同时具有很强的耐盐性,在3 mol/L的NaCl溶液中依旧保持原有活力[46],因此,在酱油高盐体系中发挥着重要的作用。
2.2.5 羧肽酶
羧肽酶为外切型丝氨酸蛋白酶,能够从多肽的C端释放出氨基酸。HIROTO M等[47-49]从Aspergillus oryzae RIB40的基因组数据中挖掘出5个潜在的羧肽酶基因,分别为cpI、ocpA、ocpB、ocpC、ocpO,通过异源表达获得重组酶并进行酶学特性分析,证明5个基因均能编码羧肽酶,且均在较低的pH体系中有最优活性,特异性底物酶解实验显示,羧肽酶无法水解C端为甘氨酸和脯氨酸的多肽。根据数据库的检索结果显示,cpI与ocpB基因编码的羧肽酶虽然具有信号肽,但可能是位于溶酶体中的水解酶,ocpA基因编码的羧肽酶与其他曲霉中的羧肽酶S1同源,同样具有信号肽结构,但功能尚不明确。目前认为羧肽酶对于提升酱油的氨基酸态氮有一定作用,但其作用效果较氨肽酶弱。
3 结论与展望
米曲霉由于蛋白酶系丰富且酶切位点全面,成为了酱油酿造的理想菌种。米曲霉通过中性蛋白酶、碱性蛋白酶和酸性蛋白酶的作用,将大豆和小麦中不可溶的蛋白质切断为可溶性的多肽分子,并进一步将可溶性的多肽切割成分子量更小的肽段,然后通过亮氨酰氨肽酶从N端逐渐将氨基酸水解释放,提升酱油的氨基酸态氮水平,同时降低苦味肽带来的不良风味,天冬氨酰氨肽酶则负责从N端释放出鲜味氨基酸,提升酱油的鲜味;当遇到Xaa-Pro-的片段时,由氨基酰脯氨酰二肽酶作用切除,使得其他氨肽酶可以继续发挥作用,释放出来的Xaa-Pro二肽由氨基酰脯氨酰氨肽酶水解,进一步提升氨基酸的含量,最终产生丰富的氨基酸以及难以被进一步水解的二肽和三肽,这些产物作为其他微生物生长发酵的原料以及其他生物和化学反应的底物,进一步形成酱油独特的色香味体。随着生物技术的发展,对于米曲霉蛋白酶系组成、各种酶蛋白对风味的影响、潜在风味相关蛋白酶的挖掘等研究逐步变得深入,对于进一步改良酿造菌种,深入挖掘米曲霉的蛋白酶资源具有重要意义。
[1]DEVANTHI P V P,GKATZIONIS K.Soy sauce fermentation: Microorganisms, aroma formation, and process modification[J]. Food Res Int,2019,120:364-374.
[2]RISA H,MASANOBU Y,KOTATO I,et al.Influence of yeast and lactic acid bacterium on the constituent profile of soy sauce during fermentation[J]. J Biosci Bioeng,2017,123(2):203-208.
[3] YANG Y, DENG Y, JIN Y L, et al. Dynamics of microbial community during the extremely long-term fermentation process of a traditional soy sauce[J].J Sci Food Agr,2017,97:3220-3227.
[4]张贺迎,武金霞,闫博.活性电泳分析米曲霉沪酿3.042蛋白酶组分[J].中国酿造,2010(12):162-164.
[5]樊嘉训,刘松,陆信曜,等.高产蛋白酶米曲霉菌株的选育及对酱油风味生成的影响[J].食品与发酵工业,2021,47(21):1-8.
[6]郭婷,王凤军,周新运,等.米曲霉盐胁迫对酱油发酵理化指标及微生物群落的影响[J].中国酿造,2023,42(6):97-102.
[7]范林旭.高产蛋白酶米曲霉的筛选及其在豆瓣酱中的应用[D].无锡:江南大学,2023.
[8]汪卓,陈楚锐,许立锵,等.pH值对酱油中呈味肽种类和呈味特性的影响[J].食品科学,2021,42(2):60-65.
[9]ZHUANG M Z,ZHAO M M,LIN L Z,et al.Macroporous resin purification of peptides with umami taste from soy sauce[J]. Food Chem,2016,190:338-344.
[10] MOTONAKA K, YUMIKO K, JUNKO Y, et al. Determination and quantification of the kokumi peptide,γ-glutamyl-valyl-glycine,in commercial soy sauce[J].Food Chem,2013,141(2):823-828.
[11]MASAYUKI M,KIYOSHI A,MOTOAKI S,et al.Genome sequencing and analysis of Aspergillus oryzae[J].Nature,2005,438(22):1157-1161.
[12]ZHAO G Z,YAO Y P,QI W,et al.Draft genome sequence of Aspergillus oryzae strain 3.042[J].Eukaryot Cell,2012,11(9):1178.
[13]KOBAYASHI T,ABE K,ASAI K,et al.Genomics of Aspergillus oryzae[J].Biosci Biotechnol Biochem,2007,71(3):646-670.
[14] RAWLING N D, BARRETT A J, THOMAS P D, et al. The MEROPS database of proteolytic enzymes,their substrates and inhibitors in 2017 and a comparison with peptidases in the PANTHER database[J]. Nucleic Acids Res,2018,46:624-632.
[15]KUSUMOTO K I,MATSUSHITA M M,FUTUKAWA I,et al.Efficient production and partial characterization of aspartyl aminopeptidase from Aspergillus oryzae[J].J Appl Microbiol,2008,105:1711-1719.
[16] JUN W,HISAKI T, TAKUMI A, et al. Characterization of Aspergillus oryzae aspartyl aminopeptidase expressed in Escherichia coli[J].Biosci Biotechnol Biochem,2007,71(10):2557-2560.
[17] KOTARO I, ASAHI M. Koji molds for Japanese soy sauce brewing:characteristics and key enzymes[J].J Fungi,2021,7:658.
[18]童星,彭勃.一株高酶活力米曲霉菌株的选育及其在酱油生产中的应用[J].中国酿造,2018,37(11):51-55.
[19]吕远平,姚开,贾冬英,等.高活力酸性蛋白酶菌株在酱油酿造中的应用[J].中国酿造,2002,21(3):10-11.
[20]孟广超.基于ARTP技术筛选高产氨肽酶米曲霉菌株及发酵和酶解条件优化的研究[D].郑州:郑州大学,2022.
[21] TIMO S, JACOB E, MICHAEL M, et al. A novel glutamyl (aspartyl)-specific aminopeptidase A from Lactobacillus delbrueckii with promising properties for application[J].Plos One,2016,11(3):e0152139.
[22]CHANG A,JESKE L,ULBRICH S,HOFMANN J,et al.BRENDA,the ELIXIR core data resource in 2021: new developments and updates[J].Nucleic Acids Res,2021,49:498-508.
[23]BATEMAN A,MARTIN M J,ORCHARD S,et al.Uniprot:the universal protein knowledge in 2023[J].Nucleic Acids Res,2023,51:523-531.
[24]GUO J P,MA Y.High-level expression,purification and characterization of recombinant Aspergillus oryzae alkaline protease in Pichia pastoris[J].Protein Expres Purif,2008,58:301-308.
[25] KE Y, HUANG W Q, LI J Z, et al. Enzymatic characteristics of a recombinant neutral protease I (rNpI) from Aspergillus oryzae expressed in Pichia pastoris[J].Agr Food Chem,2012,60:12164-12169.
[26] HIROSHI M, TORU K, DAISUKE S, et al. A novel non-thermostable deuterolysin from Aspergillus oryzae[J]. Biosci Biotechnol Biochem,2016,80(9):1813-1819.
[27]黄磊,周其洋.大豆水解蛋白中苦味肽的研究进展[J].现代食品,2022,28(21):22-24.
[28]TATSUMI H,MURAKAMI S,TSUJI R F,et al.Cloning and expression in yeast of a cDNA clone encoding Aspergillus oryzae neutral protease II,a unique metalloprotease[J].Mol Gen Genet,1991,228:97-103.
[29] NAKADAI T, NASUNO S, IGUCHI N. Purification and properties of neutral proteinase I from Aspergillus oryzae purification and properties of neutral proteinase II from Aspergillus oryzae[J]. Agr Biol Chem,2014,37(12):2695-2708.
[30] DANIEL F, SILVIA R, JOSEP V, et al. A functional and structural study of the major metalloprotease secreted by the pathogenic fungus Aspergillus fumigatus[J].Acta Crystallogr D,2013,69:1946-1957.
[31]柯野,陈丹,李家洲,等.米曲霉碱性蛋白酶基因的克隆表达及水解特性[J].华南理工大学学报(自然科学版),2012,40(8):88-94.
[32]冯颖杰.米曲霉蛋白酶的酶学性质及其应用研究[D].无锡:江南大学,2015.
[33]TATSUMI H,OHSAWA M,TSUJI R F,et al.Cloning and sequencing of the alkaline protease cDNA from Aspergillus oryzae[J].Agr Biol Chem,2016,52(7):1887-1888.
[34]WATANABE J.Aspergillus sp.lacking function of gene encoding Alpb and utilization thereof:Japan,5305353[P].2013-10-02.
[35]TADANOBU N,SEIICHI N.The action of acid proteinase from Aspergillus oryzae on soybean proteins[J].Agr Biol Chem,1977,41(2):409-410.
[36] KATSUYA G, KENJI A, NAOKATA K, et al. Cloning and nucleotide sequence of the acid protease-encoding gene (pepA) from Aspergillus oryzae[J].Biosci Biotechnol Biochem,1993,57(7):1095-1100.
[37]RANDY M B,CYNTHIA L C,KIRK J H,et al.Isolation and characterization of the Aspergillus oryzae gene encoding aspergillopepsin O[J].Gene,1993,125:195-198.
[38]SNYMAN C,THERON L W,DIVOL B.Understanding the regulation of extracellular protease gene expression in fungi:a key step towards their biotechnological applications[J]. Appl Microbiol Biotechnol, 2019,103:5517-5532.
[39]SATO K,MIYASAKA S,TSUJI A,et al.Isolation and characterization of peptides with dipeptidyl peptidase IV(DPPIV)inhibitory activity from natto using DPPIV from Aspergillus oryzae[J].Food Chem,2018,261:51-56.
[40]HIROSHI M,DAISUKE S,TAKUJI K,et al.Three extracellular dipeptidyl peptidases found in Aspergillus oryzae show varying substrate specificities[J].Appl Microbiol Biotechnol,2016,100(11):4947-4958.
[41]HIROSHI T.A role of X-prolyl-dipeptidyl-aminopeptidase in hydrolysis of proteins during a model system of soy sauce fermentation[J].J Brew Soc Jpn,1998,93:307-311.
[42]TADANOBU N,SEIICHI N,NOBUYOSHI I.Purification and properties of leucine aminopeptidase I from Aspergillus oryzae[J].Agr Biol Chem,1973,37(4):757-765.
[43] TADANOBU N, SEIICHI N. Purification and properties of leucine aminopeptidase IV from Aspergillus oryzae[J]. Agr Biol Chem,1977,41(9):1657-1666.
[44] JUN W, HISAKI T, TAKUMI A, et al. Characterization of Aspergillus oryzae aspartyl aminopeptidase expressed in Escherichia coli[J].Biosci Biotechnol Biochem,2007,71(10):2557-2560.
[45] KUSUMOTO K I, MATSUSHITA M M, FURUKAWA I, et al. Efficient production and partial characterization of aspartyl aminopeptidase from Aspergillus oryzae[J].J Appl Microbiol,2008,105:1711-1719.
[46]MAYUMI M M,SAWAKI T,SATOSHI S,et al.Enzymatic characterization of a novel Xaa-Pro aminopeptidase XpmA from Aspergillus oryzae expressed in Escherichia coli[J].J Biosci Bioeng,2017,124(5):534-541.
[47]HIROTO M,AYAKO O,YOHEI Y,et al.Heterologous expression and characterization of CpI, OcpA, and novel serine-type carboxypeptidase OcpB from Aspergillus oryzae[J].Appl Microbiol Biotechnol,2009,85:335-346.
[48] HIROTO, KEN-ICHI K, NORITAKA A, et al. Molecular cloning of ocpO encoding carboxypeptidase O of Aspergillus oryzae IAM2640[J].Biosci Biotechnol Biochem,2010,74(5):1000-1006.
[49] HIROTO M, HARUKA A, AYAKO O, et al. Enzymatic properties of the recombinant serine-type carboxypeptidase OcpC,which is unique to Aspergillus oryzae[J]. Biosci Biotechnol Biochem, 2011, 75(4): 662-668.