奶酪是生牛乳在发酵剂与凝乳酶的作用下经过一段时间成熟后制成的一种乳制品,其含有丰富的蛋白质、脂肪、维生素、矿物质和全部的必需氨基酸[1-2]。蒙古族奶酪,蒙古语称“胡乳达”,是蒙古族等少数民族喜欢食用的一种传统乳制品,其制作工艺已经被纳入内蒙古的非物质文化遗产[3]。随着产业化进程加快,使得传统奶酪品类和产量逐渐减少,其中宝贵的微生物资源也将面临遗失。因而,加快调查、挖掘和保护蒙古族传统发酵奶酪中的微生物资源,已迫在眉睫。张冬蕾等[4]通过实时荧光定量聚合酶链式反应(real-time fluorescent quantitative polymerase chain reaction,RT-fqPCR)技术对采集自内蒙古地区酸乳中的乳酸菌进行分析,发现瑞士乳杆菌(Lactobacillus helveticus)为优势乳酸菌;李伟程等[5]采用454焦磷酸测序技术对传统发酵乳制品进行分析,发现酸牛奶样品中的细菌以乳酸菌为主,真菌以酵母菌为主;焦晶凯等[6]对内蒙古奶酪的微生物进行研究,发现内蒙古传统奶酪微生物与西方奶酪发酵剂有较大差别。但是,目前关于奶酪自然发酵过程中微生物的研究鲜有报道,对奶酪成熟过程中微生物群落结构的复杂性、多样性以及动态变化情况等仍缺乏全面的认识。
近年来,高通量测序技术得到快速发展,已经应用于不同学科领域研究,尤其在测定传统发酵食品微生物群落的宏基因组研究中较为广泛[7-10]。与传统微生物分离培养技术、变性梯度凝胶电泳和时相温度梯度凝胶电泳等方法相比,高通量测序技术可一次性对样品中的几十万到几百万条脱氧核糖核酸(deoxyribonucleic acid,DNA)分子序列进行测定,快速确定其中微生物的种类和丰度[11],可以更准确且快速的揭示奶酪微生物群落多样性和演替规律[12-14],为研究微生物群落提供了良好的技术。
本研究以内蒙古牧区蒙古族木桶自然发酵生产奶酪为研究对象,采用Illumina MiSeq高通量测序技术分析发酵凝乳及不同成熟阶段奶酪样品微生物菌群多样性,客观地评价微生物种类、数量及优势菌群的变化规律,旨在为后期提高少数民族地区奶酪品质、菌株的精准筛选及风味调控提供参考。
1 材料与方法
1.1 材料与试剂
蒙古族传统奶酪:内蒙古锡林郭勒盟西乌珠穆沁旗奶酪作坊;E.Z.N.A.RSoil DNA Kit:美国Omega Bio-Tek公司;biowest agArose琼脂糖:西班牙Biowest公司;AxyPrep DNA Gel Extraction Kit:美国Axygen公司;MiSeq Reagent Kit v3/NovaSeq Reagent Kits:美国Illumina公司。其他试剂均为国产分析纯。
1.2 仪器与设备
ABI GeneAmpR9700型PCR仪:美国ABI公司;Illumina MiSeq测序仪:美国Illumina公司;Invitrogen Qubit3.0荧光计:美国Life公司;ZF-288凝胶成像系统:美国UVP公司。
1.3 方法
1.3.1 样品取样
到内蒙古锡林郭勒盟西乌珠穆沁旗牧民作坊,跟踪一个批次木桶发酵生产奶酪完整生产过程,分别采集发酵凝乳、成熟0 d、10 d、20 d和30 d的奶酪样品各3份,分别标记为Mni、M0d、M10d、M20d、M30d,样品采集方式为随机采样,采集得到的样品立即送往实验室-20 ℃保存。
1.3.2 样品微生物菌群基因组DNA的提取、PCR扩增及高通量测序
按照E.Z.N.A.Rsoil DNA Kit试剂盒说明提取样品的微生物群落总DNA,采用1%的琼脂糖凝胶电泳检测DNA的提取质量,并使用NanoDrop2000测定DNA浓度和纯度。以其为模板,细菌使用338F(5'-ACTCCTACGGGAGGCAGCAG-3')和806R(5'-GGACTACHVGGGTWTCTAAT-3')对16S rRNA基因V3-V4可变区进行PCR扩增,真菌使用ITS1F(5'-CTTGGTCATTTAGAGGAAGTAA-3')和ITS2R(5'-GCTGCGTTCTTCATCGATGC-3')对ITS基因进行PCR扩增。PCR扩增程序:95 ℃预变性3 min,27个循环(95 ℃变性30 s,55℃退火30 s,细菌72 ℃延伸30 s,真菌72 ℃延伸45 s),然后72 ℃稳定延伸10 min,在4 ℃进行保存。PCR扩增体系:5×TransStart FastPfu缓冲液4 μL,2.5 mmol/L脱氧核糖核苷三磷酸(deoxyribonucleoside triphosphates,dNTPs)2 μL,上游引物(5 μmol/L)0.8 μL,下游引物(5 μmol/L)0.8 μL,TransStart FastPfu DNA聚合酶0.4 μL,模板DNA 10 ng,双蒸水(ddH2O)补足至20 μL。每个样本3个重复,将3个重复的PCR扩增产物混合,使用2%琼脂糖凝胶电泳检测产物。使用AxyPrep DNA Gel Extraction Kit纯化PCR扩增产物后用QuantusTMFluorometer进行检测定量,按照每个样本的测序量要求,进行相应比例的混合。通过Illumina MiSeq PE300平台进行高通量测序[15]。
1.3.3 数据处理及生物信息学分析
利用上海美吉公司提供的生物云信息平台对测序数据进行分析,使用FLASH软件对测序数据进行拼接,使用UPARSE软件,依据97%的相似度对序列进行操作分类单元(operational taxonomic units,OTU)聚类并剔除嵌合体[16-17]。利用RDP classifier对每条序列进行物种分类注释[18],比对Silva 16S rRNA数据库,设置比对阈值为70%。利用Mothur进行多样性分析,利用R语言(version 3.3.1)进行主坐标分析(principal coordinate analysis,PCoA)和作图。采用Origin 9.0以及美吉云平台提供的绘图工具进行绘图。
2 结果与分析
2.1 发酵凝乳及奶酪成熟过程中微生物菌群高通量测序结果
发酵凝乳及奶酪成熟过程中微生物菌群的高通量测序结果见表1。由表1可知,5个样品中真菌菌群及细菌菌群的有效序列数均>50 000条,细菌OTU数为43~58个,真菌OTU数为30~47个。
表1 发酵凝乳及奶酪成熟过程中微生物菌群的高通量测序结果
Table 1 High-throughput sequencing results of fermented curd and cheese during ripening
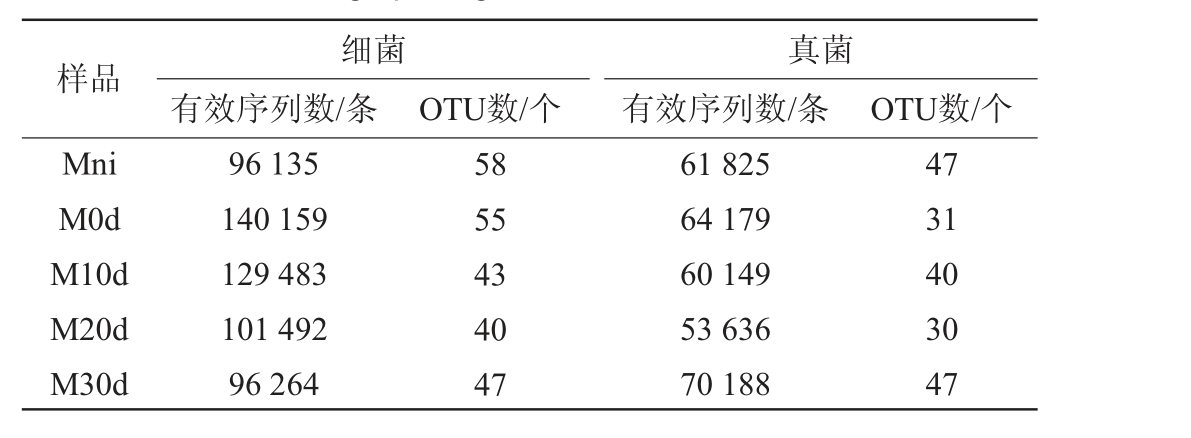
样品Mni M0d M10d M20d M30d细菌有效序列数/条OTU数/个真菌有效序列数/条 OTU数/个96 135 140 159 129 483 101 492 96 264 58 55 43 40 47 61 825 64 179 60 149 53 636 70 188 47 31 40 30 47
随机抽取一定的测序序列数,基于该测序序列划分的OTU和数据量构建稀释曲线,结果见图1。由图1可知,随着测序量的逐渐增多,5个样品中细菌及真菌菌群的稀释曲线均趋于平缓,达到平台期,说明测序数据量渐进合理,更多的数据量只会产生少量新的OTU以及新的注释物种。
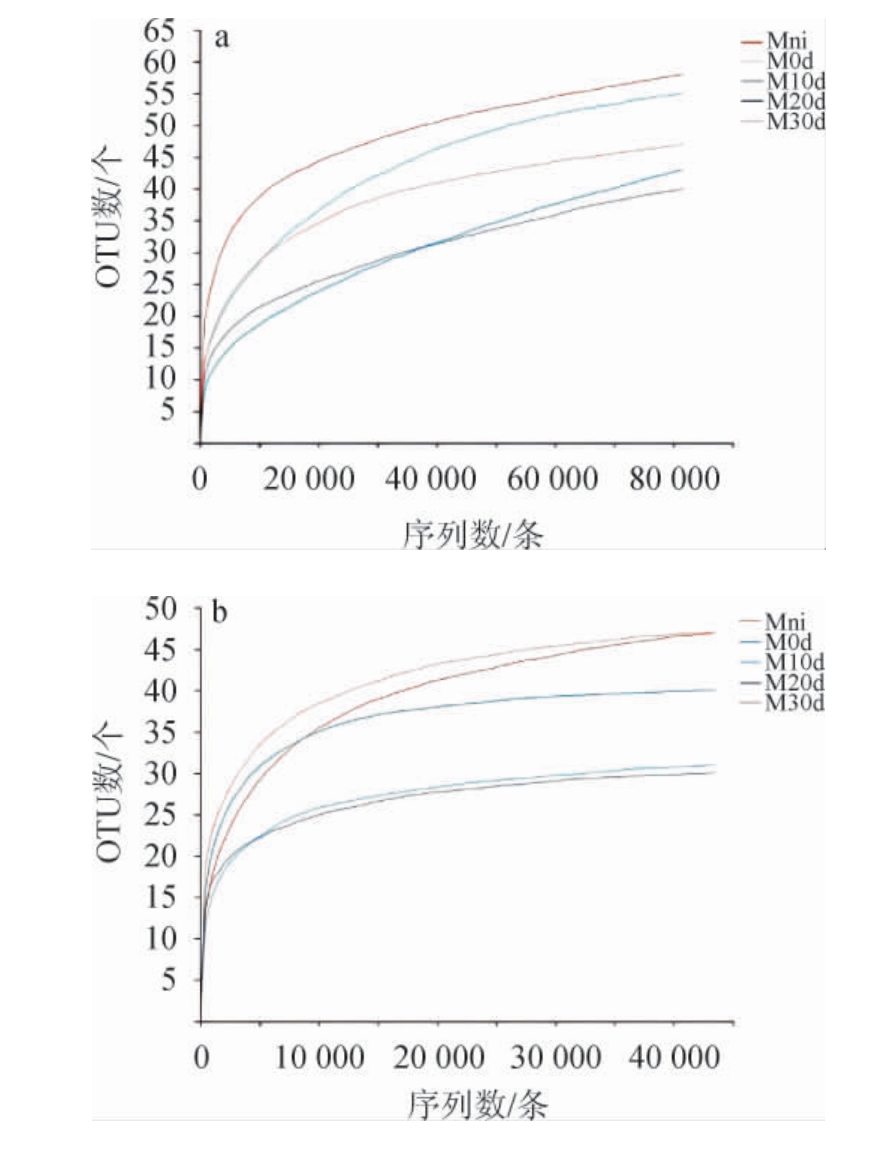
图1 发酵凝乳及奶酪成熟过程中细菌(a)及真菌(b)菌群的操作分类单元稀释性曲线
Fig.1 Operational taxonomic unit dilution curves of bacterial (a) and fungal (b) communities in fermented curd and cheese during ripening process
2.2 发酵凝乳及奶酪成熟过程中微生物菌群Alpha多样性分析
发酵凝乳及奶酪成熟过程中细菌及真菌菌群的Alpha多样性分析结果见表2和表3。由表2及表3可知,5个样本细菌和真菌菌群的Coverage值均>0.99%,表明奶酪样品中微生物群落信息可以由该测序结果进行解释。Chao1指数和ACE指数主要衡量物种丰富度[19],Shannon指数和Simpson指数代表物种多样性[20]。由表2可知,发酵凝乳的Shannon指数最大,Simpson指数最低,表明凝乳中细菌群落多样性最高,说明木桶奶酪发酵细菌菌群主要来自于凝乳,奶酪中细菌菌群受木桶环境影响较大。奶酪成熟10 d时,ACE指数和Chao1指数最高,细菌群落丰富度最高;成熟20 d时,细菌菌群丰富度和多样性均下降,可能是由于发酵时间的延长乳酸大量积累,导致pH值下降,部分耐酸性弱的细菌死亡所导致。成熟30 d时,细菌菌群多样性下降但丰富度上升,可能受环境微生物影响较大,与真菌产生互作有关。由表3可知,发酵凝乳中的真菌群落丰富度最高,但多样性指数最低,此时可能参与发酵的主要是一些优势酵母菌。奶酪成熟10 d时,真菌群落多样性指数达到最高,丰富度指数达到最低,可能受环境影响或与细菌互作有关。成熟20 d时,多样性指数下降,成熟30 d时,又逐渐增高,但丰富度均开始升高,表明成熟环境适合大多数真菌生长繁殖。综上,凝乳中细菌菌群多样性最高,成熟10 d时丰富度最高;奶酪成熟10 d时真菌菌群菌群多样性最高,凝乳中丰富度最高;说明采用木桶发酵加工的奶酪,其发酵细菌主要来源于木桶,细菌和真菌之间可能存在互作效应。木桶微生物菌群构成影响其发酵生产奶酪微生物菌群结构,与文献[21]报道相一致。
表2 发酵凝乳及奶酪成熟过程中细菌菌群α 多样性分析结果
Table 2 Result of α-diversity analysis of bacterial flora in fermented curd and cheese during ripening process
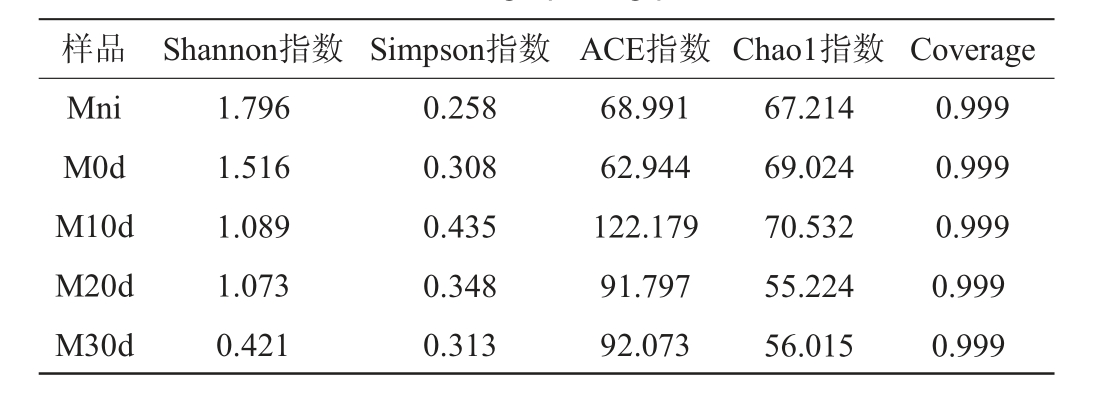
样品 Shannon指数 Simpson指数 ACE指数 Chao1指数 Coverage Mni M0d M10d M20d M30d 0.999 0.999 0.999 0.999 0.999 1.796 1.516 1.089 1.073 0.421 0.258 0.308 0.435 0.348 0.313 68.991 62.944 122.179 91.797 92.073 67.214 69.024 70.532 55.224 56.015
表3 发酵凝乳及奶酪成熟过程中真菌菌群α 多样性分析结果
Table 3 Result of α-diversity analysis of fungal flora in fermented curd and cheese during ripening process
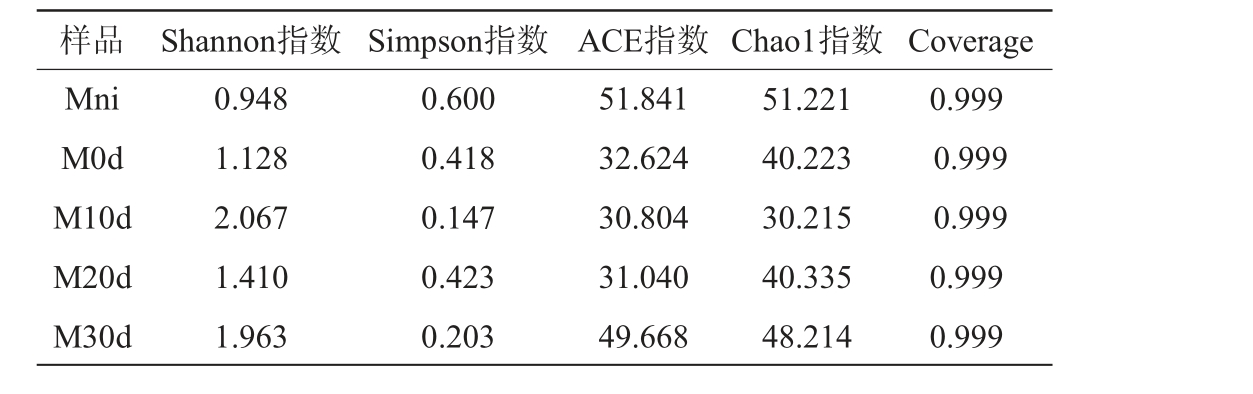
样品 Shannon指数 Simpson指数 ACE指数 Chao1指数 Coverage Mni M0d M10d M20d M30d 0.948 1.128 2.067 1.410 1.963 0.600 0.418 0.147 0.423 0.203 51.841 32.624 30.804 31.040 49.668 51.221 40.223 30.215 40.335 48.214 0.999 0.999 0.999 0.999 0.999
2.3 发酵凝乳及奶酪成熟过程中微生物群落结构分析
基于属水平,发酵凝乳及奶酪成熟过程中细菌和真菌菌群结构见图2。
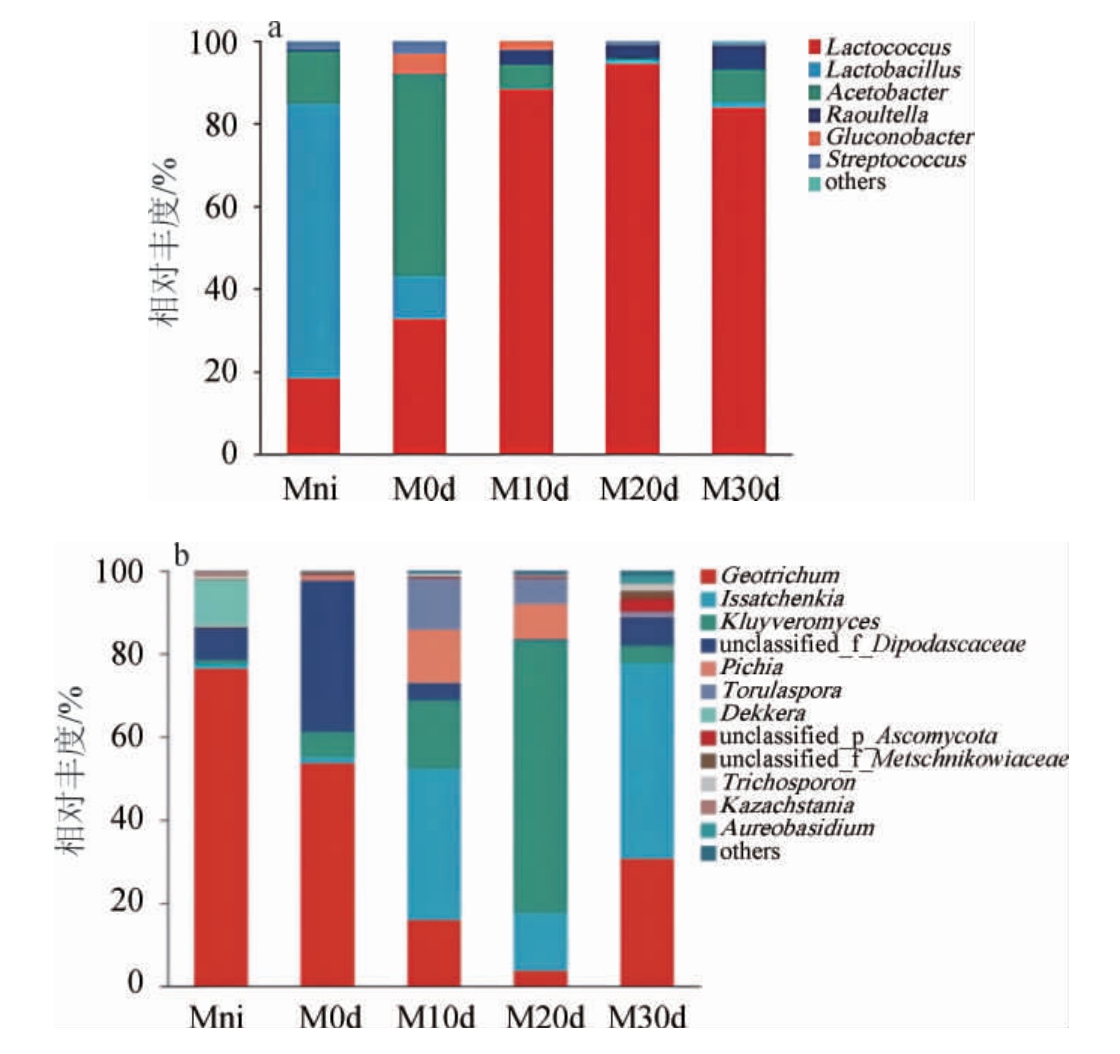
图2 基于属水平发酵凝乳及奶酪成熟过程中细菌(a)和真菌(b)群落组成
Fig.2 Composition of bacterial (a) and fungal (b) communities composition of fermented curd and cheese during ripening process based on genus level
others:相对丰度较低的其他菌属。
由图2a可知,在属水平上,从5个样品中共注释到6个优势细菌属(平均相对丰度>1%),分别为乳球菌属(Lactococcus)(63.53%)、乳杆菌属(Lactobacillus)(15.73%)、醋酸杆菌属(Acetobacter)(15.09%)、拉乌尔菌属(Raoultella)(2.65%)、葡萄糖杆菌属(Gluconobacter)(1.39%)、链球菌属(Streptococcus)(1.27%)。发酵凝乳中以乳杆菌属(Lactobacillus)为优势细菌属,在奶酪成熟过程中相对丰度由73.25%下降至6.58%(成熟20 d),在奶酪成熟10 d时相对丰度最低,该菌属与乳酸乙酯的形成具有密切关系,对于有机酸具有一定的耐受能力,能减弱酵母菌的好氧代谢速度,延长前发酵期,有利于发酵有益菌的生长,增加香味物质[22]。乳球菌属(Lactococcus)在发酵凝乳与奶酪成熟过程均有参与,是奶酪成熟过程中的优势菌属,其在发酵凝乳中相对丰度(22.48%)较低,随着奶酪成熟时间的延长,相对丰度发生变化,当奶酪成熟10 d时Lactococcus占明显优势,相对丰度达到88.33%;成熟20 d时,相对丰度达到94.42%,处于主导地位;成熟30 d时,相对丰度下降至83.82%。醋酸杆菌属(Acetobacter)在奶酪成熟0 d时相对丰度达到最高,为40.52%,之后开始下降。木桶发酵生产的奶酪以乳酸菌(乳球菌属和乳杆菌属)为主要发酵菌种,其来源可能是通过奶牛乳房、挤奶机器、奶牛生活环境、运输和加工厂以及蒙古族常用的木桶进入到奶酪中[23-24]。此外,在木桶发酵生产的奶酪样品中还首次发现了葡糖杆菌属(Gluconobacter)与拉乌尔菌属(Raoultella),其中葡糖杆菌属(Gluconobacter)对奶酪品质影响还未见报道,需进一步研究;值得注意的是,拉乌尔菌属(Raoultella)有可能会引起奶酪风味的变化,并对其安全性产生影响[25]。结果表明,木桶中发酵生产的奶酪受木桶中天然存在乳酸菌菌群影响较大,在奶酪成熟10 d菌群结构发生明显变化,并随着奶酪成熟时间的延长趋于稳定,可能是由于酸度降低引起菌群结构发生变化所致。
由图2b可知,在属水平上,从5个样品中共注释到7个优势真菌属(平均相对丰度>1%),分别为地霉属(Geotrichum)(36.07%)、伊萨酵母属(Issatchenkia)(19.93%)、克鲁维酵母属(Kluyveromyces)(18.56%)、未分类双足囊菌科(unclassified_f_Dipodascaceae)(11.30%)、毕赤酵母属(Pichia)(4.61%)、孢圆酵母属(Torulaspora)(3.96%)、德克酵母属(Dekkera)(2.18%)。地霉属(Geotrichum)在凝乳和奶酪成熟0 d时相对丰度较高,分别为76.42%和53.7%,并且在奶酪成熟过程中普遍存在,成熟20 d时相对丰度最低,为15.33%。有研究发现,地霉菌属(Geotrichum)在奶酪中具有可代谢乳酸、降低奶酪pH值的作用,有效改善奶酪风味和口感,对奶酪香味的形成起主要作用[26]。也有研究发现,酵母菌主要参与蛋白水解、脂类降解、乳糖降解以及乳酸和柠檬酸同化作用,对奶酪的风味形成起到很重要的作用[27]。Dekkera在发酵凝乳中相对丰度较高,为16.5%,是木桶中主要酵母菌。奶酪成熟10 d和20 d样品中Torulaspora、Pichia和Kluyveromyces的相对丰度较其他成熟阶段高,分别为12.4%和6.2%、19.8%和12.8%、26.8%和65.4%,Issatchenkia在奶熟成熟10 d~30 d样品中的相对丰度由13.9%上升至47%,在30 d时处于主导地位。以上结果表明,奶酪不同成熟阶段样品间真菌菌群构成存在明显差异。
为进一步探究发酵凝乳及奶酪成熟过程中微生物群落组成的差异性,在微生物属水平上,基于Bray-Curtis距离算法对所有样品进行PCoA,结果见图3。
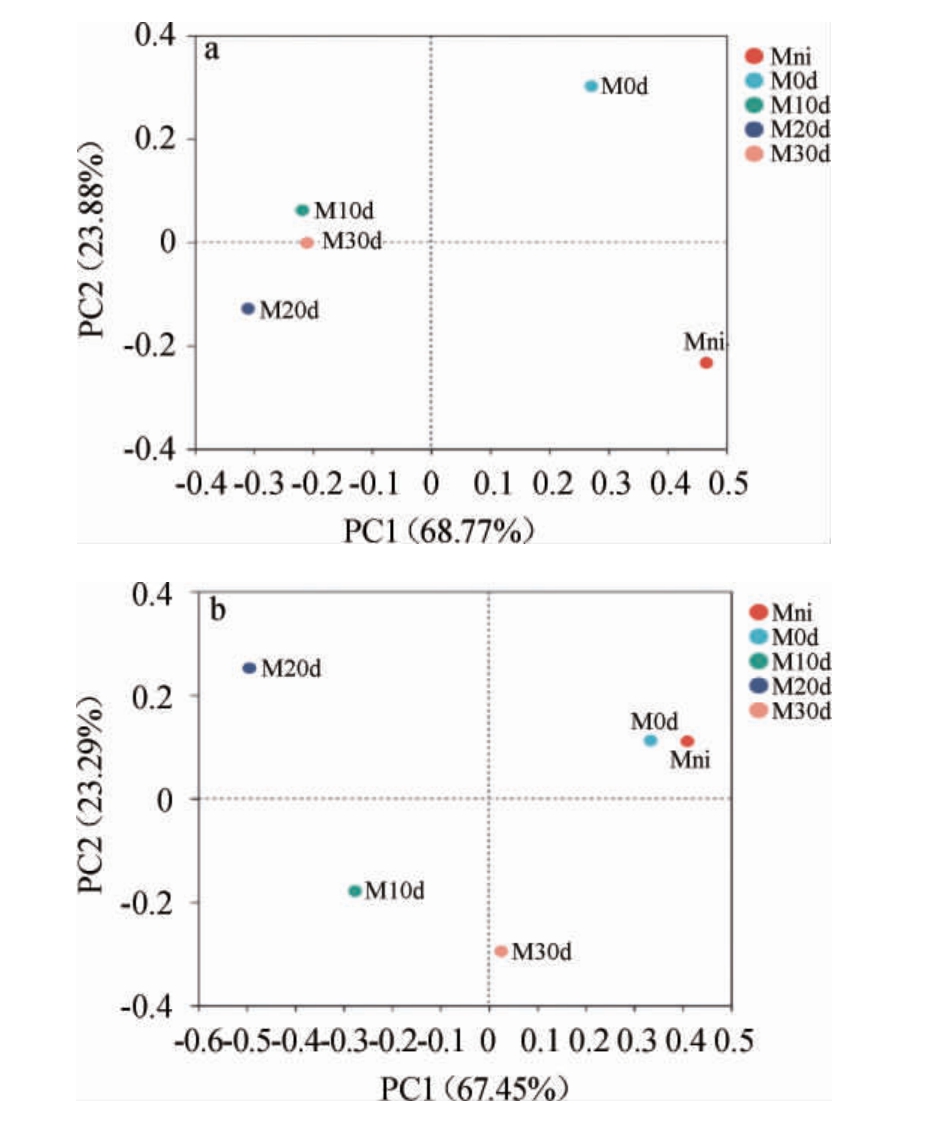
图3 基于属水平发酵凝乳及奶酪成熟过程中细菌(a)和真菌(b)群落的主坐标分析结果
Fig.3 Principal coordinate analysis results of bacterial (a) and fungal (b) communities of fermented curd and cheese during ripening process based on genus level
由图3可知,细菌菌群PC1和PC2的方差贡献率分别为68.77%和23.88%,累计方差贡献率为92.65%;真菌菌群PC1和PC2的方差贡献率分别为67.45%和23.29%,累计方差贡献率为90.74%,说明细菌菌群和真菌菌群的两个主成分均能解释原变量的绝大部分信息[28]。由图3a可知,成熟10 d、20 d和30 d奶酪样品的分布较为集中,说明这3种样品中的细菌群落结构较为相似。发酵凝乳和成熟0 d奶酪样品的分布较为分散,说明这2种样品的细菌群落结构差异较大。由图3b可知,发酵凝乳及成熟0 d奶酪样品的分布较为集中,说明这2种样品中的真菌群落结构较为相似,成熟10 d、20 d和30 d奶酪样品的分布较为分散,说明这3种样品的真菌群落结构差异较大。
3 结论
通过高通量测序技术分析蒙古族传统木桶自然发酵生产的奶酪不同成熟阶段细菌及真菌群落变化情况,结果发现,发酵凝乳中细菌菌群多样性最高,奶酪成熟10 d时细菌菌群丰富度及真菌菌群多样性最高,成熟30 d时真菌菌群丰富度最高。从发酵凝乳及奶酪成熟过程中共注释到5个优势细菌属(平均相对丰度>1%)和7个优势真菌属,优势细菌属为乳球菌属(Lactococcus)(63.53%)、乳杆菌属(Lactobacillus)(15.73%)、醋酸杆菌属(Acetobacter)(15.09%)、拉乌尔菌属(Raoultella)(2.65%)、葡萄糖杆菌属(Gluconobacter)(1.39%)、链球菌属(Streptococcus)(1.27%);优势真菌属为地霉属(Geotrichum)(36.07%)、伊萨酵母属(Issatchenkia)(19.93%)、克鲁维酵母属(Kluyveromyces)(18.56%)、未分类双足囊菌科(unclassified_f_Dipodascaceae)(11.30%)、毕赤酵母属(Pichia)(4.61%)、孢圆酵母属(Torulaspora)(3.96%)、德克酵母属(Dekkera)(2.18%)。PCoA结果表明,发酵凝乳和奶酪成熟0 d时细菌群落结构差异较大,真菌结构较为相似;奶酪成熟10 d、20 d和30 d时细菌群落结构较为相似,真菌群落结构差异较大。综上,发酵凝乳及奶酪不同成熟阶段的微生物群落存在差异,且成熟过程对微生物相对丰度具有明显影响,这为进一步研究奶酪的发酵代谢机理及菌株筛选奠定了基础。
[1]顾春华,刘煜,王建军,等.奶酪的营养价值及奶酪中生物活性肽的研究进展[J].食品安全导刊,2021(20):186-189.
[2]干佳俪,谭励,王蓓,等.切达奶酪挥发性特征风味物质的多典型相关融合分析[J].中国乳品工业,2021,49(9):12-18,27.
[3]马晓刚,李昊.粮仓实牧歌扬奶业兴[N].农民日报,2021-10-09(1).
[4]张冬蕾,陈红霞,德亮亮,等.内蒙古鄂尔多斯地区传统发酵乳制品中乳酸菌优势菌群q-PCR定量分析[J].乳业科学与技术,2015,38(6):1-5.
[5]李伟程,侯强川,于洁.传统发酵乳制品中微生物多样性研究[J].食品工业科技,2018,39(1):131-133.
[6]焦晶凯,莫蓓红.Illumina MiSeq平台高覆盖率测定干酪中的细菌微生物多样性[J].中国酿造,2014,33(5):34-38.
[7]LI X F,LI C,YE H,et al.Changes in the microbial communities in vacuumpackaged smoked bacon during storage[J].Food Microbiol, 2019, 77:26-37.
[8]李婕,黄琳翔,詹艺舒,等.双孢蘑菇贮藏保鲜过程中菇体表面的菌相变化[J].食品工业科技,2019,40(9):109-114.
[9] AREGBE A Y, MU T H, SUN H N.Effect of different pretreatment on the microbial diversity of fermented potato revealed by high-throughput sequencing[J].Food Chem,2019,290(3):125-134.
[10] LIU A, YANG X, GUO Q, et al.Microbial communities and flavor compounds during the fermentation of traditional Hong Qu glutinous rice wine[J].Foods,2022,11(8):1097.
[11] YUN J M, ZHAO F Q, ZHANG W W, et al.Monitoring the microbial community succession and diversity of Liangzhou fumigated vinegar during solid-state fermentation with next-generation sequencing[J].Annal Microbiol,2019,69(3):279-289.
[12]WOLFE B E,BUTTON J E,SANTARELLI M,et al.Cheese rind communities provide tractable systems for in situ and in vitro studies of microbial diversity[J].Cell,2014,158(2):422-433.
[13] DIMITRAKOPOULOU M E, PANTELELI E, VANTARAKIS A.Improved PCR-DGGE analysis by emulsion-PCR for the determination of food geographical origin: A case study on Greek PDO "avgotaracho Mesolonghiou"[J].Curr Res Food Sci,2021,4:746-751.
[14]董晓婉,李宝坤,李开雄,等.新疆蒙古族和哈萨克族传统乳制品中乳酸菌多样性的比较[J].食品工业科技,2013,34(21):162-166.
[15]于佳琦.酸马奶发酵过程中微生物群落结构与风味物质的相关性研究[D].呼和浩特:内蒙古农业大学,2021.
[16] MAGOC T, SALZBERG S L.FLASH: fast length adjustment of short reads to improve genome assemblies[J].Bioinformatics,2011,27(21):2957-2963.
[17]EDGAR R C.UPARSE:highly accurate OTU sequences from microbial amplicon reads[J].Nat Methods,2013,10(10):996-998.
[18]于佳琦,夏亚男,乔晓宏.锡林郭勒牧区鲜马奶风味物质与微生物多样性分析[J].中国乳品工业,2021,49(5):8-9.
[19]赵凤琴.凉州熏醋传统酿造过程主要功能微生物群落演替对四甲基吡嗪的影响[D].兰州:甘肃农业大学,2016.
[20]曾椿淋.内蒙古手工奶酪微生物多样性和功能性研究[D].镇江:江苏大学,2020.
[21]RIVAS M N,BURTON O T,WISE P,et al.A microbiota signature associated with experimental food allergy promotes allergic sensitization and anaphylaxis[J].J Allergy Clin Immun,2013,131(1):201-212.
[22]CARMEN PORTILLO M,MAS A.Analysis of microbial diversity and dynamics during wine fermentation of Grenache grape variety by highthroughput barcoding sequencing[J].LWT-Food Sci Technol, 2016,72:317-321.
[23] METZKER M L.Sequencing technologies-the next generation[J].Nat Rev Genet,2010,11(1):31-46.
[24]DE VUYST L,TSAKALIDOU E.Streptococcus macedonicus,a multifunctional and promising species for dairy fermentations[J].Int Dairy J,2008,18(5):476-485.
[25]王鸿,谢斯超,周广敏.拉乌尔菌Raoultella sp.Pan22x菌株群体感应抑制活性的培养基及发酵条件的优化[J].浙江工业大学学报,2018,46(1):101-103.
[26]高代微,王琳琳,张岩.相对湿度对牦牛毛霉霉菌奶酪成熟过程中品质的影响[J].食品工业科技,2020,41(5):23-25.
[27]MC SWEENEY P L H.Biochemistry of cheese ripening[J].Int Dairy J,2004,57(6):17-37.
[28]RIVAS M N,BURTON O T,WISE P,et al.A microbiota signature associated with experimental food allergy promotes allergic sensitizationand anaphylaxis[J].J Allergy Clin Immun,2013,131(1):201-212.