发酵食品由于原料类型、生产工艺和微生物组成等造就了其多样性和独特性。发酵微生物在利用食品原料生长、繁殖和代谢的过程中产生各种代谢产物,赋予发酵食品独特的风味和营养价值。中国有着悠久的食用发酵食品的历史,拥有独特的发酵食品种类,诞生了众多诸如白酒、豆豉、豆酱、酱油、腐乳等有特色的传统发酵食品。在发酵过程中,蕴藏着丰富多样的微生物群落,它们对发酵食品的营养价值、感官体验和品质起到决定性的作用,对于保证发酵食品的品质至关重要。发酵是一个动态变化的微生态环境体系,在发酵过程的不同阶段,微生物的多样性受到环境因素如营养状态、温度、湿度、pH等理化性质的影响发生显著的变化;同时也受到其他不同微生物种群间的作用,极大地影响微生物物种的生长、繁殖和演替。
噬菌体是一类可以感染细菌、放线菌、真菌和藻类等从原核生物到真核生物的所有生命形式的病毒总称,是病毒中最为普遍和分布最广的微生物群体[1]。噬菌体的结构非常简单,通常由单链或双链核酸以及保护核酸的蛋白质或脂蛋白衣壳组成。根据其溶原性,噬菌体又可以分为烈性噬菌体和温和噬菌体。烈性噬菌体通过粘附将核酸注入宿主细胞内,利用宿主提供的养分和能量,转录和翻译自身的噬菌体结构,最终裂解宿主完成释放[2]。温和噬菌体是将自己的遗传物质整合至宿主基因组,成为原噬菌体并处于休眠状态,通过宿主繁殖可以将自己的遗传物质稳定的传递给后代,一般不会造成宿主的破坏。但当环境发生剧烈变化或受到应激刺激时,前噬菌体会脱离宿主染色体,从而独立复制转变成烈性噬菌体[2]。
噬菌体广泛存在于自然环境中,具有调节微生物群落结构的作用。噬菌体与其他微生物的相互作用对微生物进化有着重要意义,其对微生物生态环境的塑造至关重要[3]。一方面,噬菌体可以直接调节细菌种群,影响宿主菌的代谢机制从而影响整个群落结构;另一方面,噬菌体介导的细菌裂解可为其他微生物提供物质基础,间接支持和促进其他微生物的生长和繁殖。此外,噬菌体还可驱动细菌进化,从而提高宿主的适应性[4-5]。发酵食品的生产过程受到周围环境等微生态的影响,一些自然环境中的噬菌体跟随发酵微生物被引入发酵体系,为其繁殖和发育提供了丰富的媒介。目前,噬菌体已在多种发酵食品的生产过程中被发现和分离,其宿主大多为发酵主要功能菌或腐败菌、致病菌等。有大量的研究表明,噬菌体会对发酵过程产生危害[5]。发酵主要功能菌噬菌体的存在通常会抑制宿主菌的正常生长或使宿主菌大量裂解,导致发酵异常甚至失败。此外,噬菌体能够特异性裂解致病菌或产生不良风味的菌,对发酵产生有利的影响,从而达到良好的生物防治效果[6]。也有研究发现,发酵菌噬菌体的某些序列能够帮助宿主菌自溶,释放胞内酶,改善发酵食品风味[16]。此外,通过对噬菌体的不敏感宿主进行筛选,可以在一定程度上避免发酵失败[9],还可影响微生物群落演替,起到帮助推进发酵过程的作用[20,29]。
本文总结了目前不同类型的发酵食品中噬菌体的产生和功能,阐述了噬菌体在发酵生产中的危害、影响及应用,讨论了在发酵食品中检测噬菌体的方法,以期更加全面了解噬菌体在发酵食品生产中的功能和作用,为更好的控制发酵生产提供参考。
1 食品发酵过程中噬菌体的产生及功能
目前在食品发酵过程中发现噬菌体的名称、分类、宿主及功能见表1。1935年,噬菌体首次被发现能够导致工业生产的乳制品发酵失败[32],此后,噬菌体在发酵香肠、发酵豆制品、泡菜等多种发酵食品中均被发现能够特异性感染和裂解发酵微生物[22,33-34],导致产品质量降低甚至发酵失败,从而给发酵工业带来巨大的经济损失。在干酪的生产过程中,大多数分离得到的噬菌体为乳酸菌噬菌体,例如长尾病毒科(Siphoviridae)和丝杆状病毒科(Inoviridae)噬菌体特异性感染费氏柠檬酸杆菌(Propionibacterium freudenreichii),造成干酪发酵失败[35]。在葡萄酒的生产中,发现了15种乳酸菌噬菌体,能够裂解苹果酸乳酸发酵菌—酒类酒球菌(Oenococcus oeni),导致葡萄酒香气、色泽异常[23]。在纳豆的生产过程中,枯草芽孢杆菌噬菌体是导致γ-聚谷氨酸(γ-polyglutamic acid,PGA)合成失败的重要因素,极大的降低了纳豆品质[8]。LU Z等[19,22]对德国酸菜和黄瓜发酵过程中的微生物体系进行了研究,对其中的优势乳酸菌噬菌体进行了分离和筛选。类似的,PARK E J等[36]对泡菜、德国酸菜等发酵蔬菜食品进行宏病毒组学分析,发现噬菌体是发酵食品中病毒的主要组成群体,尽管这些研究已经从发酵食品中分离出若干类型的噬菌体,但还未对其进行分析和注释,特别是噬菌体在发酵过程中的作用还未明确。在我国,食品发酵工业中同样发现了噬菌体[37],尤其是乳制品生产中经常出现乳酸菌噬菌体,严重影响乳酸菌发酵,通常是导致发酵异常的主要原因。因此,有必要对发酵食品中噬菌体的组成和功能做系统性梳理,为发酵食品中噬菌体的控制和利用提供参考。
表1 不同发酵食品中分离得到的噬菌体种类和功能
Table 1 Species and functions of phages isolated from different fermented foods

来源 噬菌体名称 分类(科) 宿主 功能 引文PM1纳豆ΦNIT1发酵豆类韩国豆酱BSP10 BCP1-1 BCP8-2 ΦD-10酱油喜马拉雅地区发酵豆制品Kinema酸奶蓝纹干酪(Blue cheese)Φ105,proCM3等发酵乳类FAGK1,FAGK2 R707,C19A和C19B等Q69,156模拟干酪未知长尾噬菌体科(Siphoviridae)肌病毒科(Myoviridae)肌病毒科(Myoviridae)肌病毒科(Myoviridae)长尾噬菌体科(Siphoviridae)长尾噬菌体科(Siphoviridae),肌病毒科(Myoviridae),短尾噬菌体科(Podoviridae)长尾噬菌体科(Siphoviridae)长尾噬菌体科(Siphoviridae)长尾噬菌体科(Siphoviridae),肌病毒科(Myoviridae)未知枯草芽孢杆菌(Bacillus subtilis)枯草芽孢杆菌(Bacillus subtilis)枯草芽孢杆菌(Bacillus subtilis)蜡样芽孢杆菌(Bacillus cereus)嗜盐四联球菌(Tetragenococcus halophila)巨大芽孢杆菌(Bacillus megaterium)枯草芽孢杆菌(Bacillus subtilis)植物乳杆菌(Lactobacillus plantarum)肠膜明串珠菌(Leuconostoc mesenteroides)粪肠球菌(Enterococcus faecalis)乳酸乳球菌(Lactococcus lactis)导致发酵异常,破坏纳豆生产导致发酵异常,破坏纳豆生产抑制宿主生长,降低大豆发酵品质抑制致病菌,用于生物防治帮助筛选噬菌体不敏感菌株,保证正常发酵未知导致发酵异常导致发酵异常作为生物技术工具,减少产生酪胺的细菌,抑制酪胺生成帮助启动细胞裂解,释放胞内乳酸脱氢酶,改善干酪风味并加速成熟[7][8-9][10][11][12][13][14][15][16-17][18]
续表

来源 噬菌体名称 分类(科) 宿主 功能 引文发酵蔬菜酸黄瓜泡菜德国酸菜Φ3.8.18,Φ3.8.43和Φ3.8.48等Φps05未知Φ1-A4,Φ1-F8和Φ3-A4等长尾噬菌体科(Siphoviridae),肌病毒科(Myoviridae)长尾噬菌体科(Siphoviridae)短尾噬菌体科(Podoviridae),长尾噬菌体科(Siphoviridae),肌病毒科(Myoviridae)长尾噬菌体科(Siphoviridae),肌病毒科(Myoviridae)短乳杆菌(Lactobacillus brevis)植物乳杆菌(Lactobacillus plantarum)类肠膜魏斯氏菌(Weissella paramesenteroides)食窦魏斯氏菌(Weissella cibaria)耐乙醇片球菌(Pediococcus ethanolidurans)片球菌(Pediococcus)乳酸杆菌(Lactobacillus)维塞拉菌(Weissella)明串珠菌(Leuconostoc)明串珠菌属(Leuconostoc)魏斯氏菌属(Weissella)乳酸杆菌属(Lactobacillus)裂解发酵菌,可能使宿主菌大量死亡裂解发酵菌,影响细菌演替鉴定原料来源影响不同属乳酸菌菌种的演替[19][20][21][22]发酵酒类葡萄酒啤酒白酒Og44,ΦS11和Φ10MC等温和噬菌体ΦOE33PA等裂性噬菌体GC1 SA-C12长尾噬菌体科(Siphoviridae)长尾噬菌体科(Siphoviridae)复层噬菌体科(Tectiviridae)肌病毒科(Myoviridae)未知类双生病毒科(Genomoviridae),拟菌病毒科(Mimiviridae),长尾噬菌体科(Siphoviridae),细小病毒科(Parvoviridae)酒类酒球菌(Oenococcus oeni)酒类酒球菌(Oenococcus oeni)蜡染葡糖杆菌(Gluconobacter cerinus)短乳杆菌(Lactobacillus brevis)乳酸杆菌属(Lactobacillus)肠杆菌属(Enterobacter)泛菌属(Pantoea)芽孢杆菌属(Bacillus)片球菌属(Pediococcus)葡萄球菌属(Staphylococcus)可能导致发酵异常导致发酵异常抑制腐败菌抑制腐败菌,用于生物防治促进微生物演替[23-25][26-28][29][30][31]
2 噬菌体对食品发酵的影响
2.1 噬菌体对乳制品发酵的影响
在噬菌体污染方面,发酵乳制品是被报道得最多的发酵食品,尤其在干酪和酸乳制品的生产中均发现了噬菌体的存在。已有的研究表明,噬菌体对乳酸菌的感染被认为是乳制品发酵失败的最主要原因[15],它们大量感染产风味的乳酸菌,导致风味成分减少[38]。在蓝纹干酪的生产过程中,研究人员在发酵失败的干酪中发现了肠膜明串珠菌(Leuconostoc mesenteroides)噬菌体,这是一种具有不可收缩尾部的长尾噬菌体[15,38],其来源有可能是生牛乳和乳清。在酸牛乳中,同样发现了长尾噬菌体科的噬菌体,对植物乳杆菌(Lactobacillus plantarum)的36个菌株造成潜在的影响[14]。研究者对工厂的环境表面和空气进行采样分析发现,环境空气中是可能导致噬菌体污染的主要来源[39]。另一方面,乳酸菌可能携带原噬菌体,加工环境改变对其刺激后可以转变为烈性噬菌体,最终导致乳酸菌大量死亡并导致发酵失败[40-41]。因此,需要加强对生产各个环节的监控,筛查噬菌体的存在。对于乳制品中噬菌体的防治,依靠热处理和调节盐离子浓度等方式,能有效减灭乳酸菌噬菌体[42],但是过高的温度容易导致蛋白变性。此外,通过化学方法例如次氯酸钠有效抑制噬菌体,然而次氯酸钠会导致化学物质残留[43]。因此探索低毒、有效、安全的噬菌体灭活方法是发酵生产需要重视的关键技术问题。此外,通过复配多种乳酸菌进行协同发酵,避免噬菌体对单一乳酸菌造成影响从而导致延迟发酵或发酵失败。通过定期更换对噬菌体不敏感的乳酸菌或寻找突变菌株也是可行的方法,但是往往这些菌株产酸能力较弱,很难达到发酵要求。未来,可以利用规律成簇的间隔短回文重复序列(clustered regularly interspaced short palindromic repeat,CRISPR)及其相关蛋白(CRISPER associated proteins,Cas)所构建的CRISPR/Cas系统等基因编辑技术实现工程菌的改造,通过基因编辑手段设计抗性菌株,并最大程度的保留其发酵能力。然而,噬菌体的变异和进化可以快速逃逸宿主的免疫系统,噬菌体的侵染与宿主菌的抵抗必然是个持续的共进化过程。
2.2 噬菌体对豆制品发酵的影响
纳豆发酵主要利用纳豆枯草芽孢杆菌,其在发酵过程中产生γ-PGA,该物质赋予纳豆独特的黏稠感。纳豆生产中断时,从未形成粘液的异常发酵大豆中主要分离到的噬菌体是PM1,是一种长尾病毒科噬菌体,其基因组由大约50 kb的线性双链脱氧核糖核酸(deoxyribonucleic acid,DNA)组成[7]。进一步研究发现,该噬菌体能产生PGA解聚酶,将PGA降解为双谷氨酸和三谷氨酸,从而降低纳豆的黏度[8]。另一项研究分离得到了ΦNIT1噬菌体,该噬菌体携带一个γ-聚谷氨酸水解酶基因(γ-PGA hydrolase gene,PghP)[9],导致该噬菌体感染的枯草芽孢杆菌不能积累PGA,致使纳豆发酵失败。噬菌体对纳豆的污染主要通过未加工的大豆或空气污染带进发酵过程[44]。进一步的调查显示,在工厂和污水中也发现了噬菌体的存在[44]。因此,发酵过程环境清洁控制对发酵食品至关重要。
在韩国豆酱(Cheonggukjang)中分离得到一株枯草芽孢杆菌噬菌体BSP10。该噬菌体属于肌尾噬菌体科(Myoviridae)。BSP10对韩国豆酱发酵过程中的发酵菌枯草芽孢杆菌的生长具有显著的抑制作用。在豆酱的发酵过程中,与对照组相比,100 PFU/g 噬菌体就可以将发酵菌的数量降低2个数量级。与纳豆污染类似,噬菌体感染在大豆发酵过程中极大地促进了PGA的降解,对豆酱的生产造成负面影响[10,45]。在日本酱油中,同样分离得到两种长尾噬菌体科噬菌体,它们均可以特异性侵染嗜盐链球菌[12],但是通过基因突变筛选找到了对噬菌体不敏感菌株,用于抵抗噬菌体对酱油发酵过程的危害。宏基因组分析还揭示了发酵豆制品(Kinema)中芽孢杆菌噬菌体的存在,这些噬菌体分属于长尾噬菌体科、肌尾噬菌体科和短尾噬菌体科[13]。综上可知,发酵豆类食品中大多以芽孢杆菌噬菌体为主,这可能与其独特的发酵类型有关,对于以霉菌为主要发酵微生物的我国传统豆类发酵食品中是否存在噬菌体,还缺乏相应的报道,噬菌体是否影响发酵菌的生长和繁殖还有待进一步确认。
2.3 噬菌体对蔬菜发酵的影响
KOT W等[46]在低盐酸黄瓜发酵中检测出57种噬菌体,大部分噬菌体的宿主以短乳杆菌(Lactobacillus brevis)和植物乳杆菌(Lactobacillus plantarum)为主,首次发现同一噬菌体可以同时感染多个细菌菌属。发酵过程中,共发现17种不同类型的噬菌体,并且噬菌体的丰度与乳酸菌的丰度呈正相关。进一步通过电子显微镜分析发现,发酵蔬菜中的噬菌体为有尾噬菌体,具有二十面体的头部,分别属于肌尾噬菌体科和长尾噬菌体科。此外,在发酵黄瓜的盐渍液中分离出了烈性噬菌体Φps05,对商业发酵菌种片球菌(Pediococcus sp.)LA0281产生特异性感染[20]。在另一项针对德国酸菜的研究中,研究人员共分离出171个噬菌体,分属于26种不同噬菌体类型。通过测序分析发现这些噬菌体的宿主为明串珠菌属(Leuconostoc)、魏斯氏菌属(Weissella)和乳酸杆菌属(Lactobacillus)等28种乳酸菌。同样,这些噬菌体分属于粘病毒科和长尾噬菌体科噬菌体[47]。有趣的是,JUNGM J等[21]通过分析不同地域的泡菜,发现来源地不同的泡菜中分离到的噬菌体有显著差异,揭示泡菜中的病毒群落与地理起源的相关性,其本质上是不同地域环境所蕴含的微生物种类和相应噬菌体类型差异造成的,该研究有助于深入了解发酵食品的微生态系统,对区分不同原料来源的发酵食品提供有价值的借鉴意义。
2.4 噬菌体对酒类发酵的影响
针对葡萄酒微生物噬菌体的研究早在20世纪就已经展开,目前从葡萄酒酿酒及环境中分离得到的噬菌体大多数是以酒类酒球菌(Oenococcus oeni)作为宿主。多数感染酒类酒球菌的噬菌体为温和噬菌体[46],例如Og44,phiS11和Φ10MC[24-25]。目前从葡萄酒中分离出来的大部分属于长尾噬菌体科,该噬菌体感染可能会引起酒类酒球菌裂解,从而造成苹果酸乳酸发酵(malolactic fermentation,MLF)失败,虽然温和噬菌体不会自发导致乳酸菌的裂解,但当环境发生改变,例如酒精浓度和pH发生变化,有可能诱发前噬菌体的从宿主基因组分离并转变成烈性噬菌体,引起乳酸菌溶解[23]。但是目前还没有直接的证据表明噬菌体会导致MLF失败。但是值得注意的是,从法国的波雅克产区生产的红酒中分离得到了烈性噬菌体ΦOE33PA,这种噬菌体丧失了溶原性,很有可能直接导致乳酸菌的大量死亡[26-28]。葡萄酒发酵过程中的噬菌体首先可能来自于葡萄的种植环境,并有可能通过环境中的溶原性细菌转运带入[47]。其次,在酿酒设备的表面,例如在管道、不锈钢桶和橡木桶等都会有潜在的噬菌体存在。此外,一些商业上的发酵剂也有可能遭受噬菌体的感染,从而被引入葡萄酒发酵过程中[48]。
KANG J等[31]首次对白酒的低、中、高温大曲进行病毒组分析,发现三种白酒大曲均存在噬菌体。通过病毒组学测序发现,这些噬菌体以乳杆菌噬菌体为主,还包括肠杆菌属(Enterobacter)、泛菌属(Pantoea)、芽孢杆菌属(Bacillus)、片球菌属(Pediococcus)和葡萄球菌属(Staphylococcus)噬菌体等,分别属于类双生病毒科,拟菌病毒科,长尾病毒科和细小病毒科噬菌体[31]。这些噬菌体的存在很有可能对白酒酿造微生物的生长和繁殖产生影响,从而影响发酵过程。然而,由于白酒酿造过程是混菌发酵,噬菌体对单一微生物的作用可能被其他微生物替代或者抵消,可能不会像单一菌种发酵造成剧烈影响,但噬菌体会造成某一类微生物的消亡,从而影响代谢产物的积累。噬菌体在白酒发酵中的功能和作用还有待进一步发掘。此外,由于气候、土壤环境和栽培方法不同,在不同条件下生长的原料其携带的微生物组成和理化性质截然不同,原料源噬菌体可能会影响白酒产区当地微生态系统,从而直接影响到酿酒品质。通过分析噬菌体类型、宿主和来源从而对酿酒原料进行溯源,有可能为精准控制原料品质和保护酿酒生态提供帮助[21]。
3 噬菌体对食品发酵有害菌的抑制
食品中存在的腐败菌如需氧型芽孢杆菌和厌氧型梭状芽孢杆菌,具有较强的热抵抗能力,通常是导致食品腐败和变质的主要微生物。有些腐败菌甚至产生生物毒素,对食品安全造成威胁。为控制这些食源性有害微生物的生长和繁殖,研究者已经利用噬菌体特异性清除这些有害微生物。目前,从食品生产到包装,噬菌体在食品加工过程中的各个环节都有应用。目前研究和应用最广泛的是针对单增生李斯特氏菌的噬菌体,已经应用在包括即食肉制品[49]、海鲜产品[50]、蔬菜水果[51]、奶制品[52-53]等食品包装和加工中。此外,利用噬菌体鸡尾酒作为食物防腐剂或制作成生物活性包装材料喷洒在食物表面,可以控制多种病原微生物的生长,延长食品货架期[54]。此外,这类产品在抑制其他食源性致病菌如大肠杆菌(Escherichia coli)O157:H7[55-56]、沙门氏菌(Salmonella)[57-58]、志贺氏菌(Shigella)[59]和金黄色葡萄球菌(Staphylococcus aureus)[60]等表现出了显著的抑制作用。除了通过直接造成宿主的裂解和死亡,一些噬菌体通过编码内溶素,能够间接地清除致病微生物及生物被膜[61]。
然而,在噬菌体的实际应用方面,工业中常用的一些消毒剂或环境因素,如pH值、温度等,可能会影响噬菌体的稳定性[62];而且,根据噬菌体—宿主进化动力学,噬菌体的存在不可避免地导致其宿主的进化,从而出现宿主产生抗性、基因转移等问题[63]。这些问题可通过CRISPR/Cas工程技术、使用不同的噬菌体混剂等方法进行解决[64-65]。总的来说,考虑到潜在安全问题的可能性,在开发噬菌体制剂作为新的抗菌策略时,应从产生噬菌体耐药细菌的可能性、宿主细菌的进化、噬菌体的水平基因转移以及噬菌体如何影响自然界中的微生物群落等多方面进行评估,验证噬菌体的安全性和环保性[62]。
值得注意的是,一些溶原性噬菌体可能携带毒性基因或抗生素抗性基因,也可能通过基因突变或水平基因转移的方式将毒性基因传递给宿主菌,因此,在选择噬菌体时,需要利用裂解噬菌体而非溶原性噬菌体作为生物控制剂,并对噬菌体予以安全性评价,通过全基因组测序,对其毒力因子、毒素、抗生素耐药性和潜在的过敏原基因进行比对,判定其是否对人体有害[66]。
一般而言,噬菌体具有自限性,随着噬菌体对靶向微生物的作用,宿主细菌数量逐渐下降,噬菌体的数量也会逐渐下降,最终在环境中降解[67]。这种特性避免了噬菌体在环境中长期积累。此外,噬菌体在环境中大量存在,包括土壤、海洋、动植物、人体内等,迄今为止,没有证据表明噬菌体对人类或动物造成有害的影响[68-69]。目前,少数关于噬菌体的食品安全应用的研究已经考虑了如何解决残留噬菌体活性的问题,可以通过添加“噬菌体中和剂”(引入植物提取液或热灭活细菌)、离心、过滤等方式降低噬菌体生物防治后噬菌体残留的问题[70]。另外,经过高温等处理后食品中的噬菌体可以被有效地清除和杀灭[71],因此,经噬菌体处理后的食物可以被认为是安全的。
目前,噬菌体作为食源性致病菌和腐败菌的抑制剂已在欧美国家得到了广泛的应用[72]。然而在我国,人们对噬菌体还缺乏足够认知,消费者对其应用在食品中的安全性存在担忧,限制了噬菌体在食品领域中的应用。因此,加强有益噬菌体的开发、安全性评价和宣传,将有助于噬菌体在食品工业中的应用。
发酵过程中的噬菌体动力学是保证微生物良好演替的关键,使发酵过程中各菌群的生物量保持平衡。在酸菜发酵的生态学研究中,细菌的演替与发酵不同阶段发现的噬菌体数量有关[22]。微生物数量在发酵第3~7天发生显著变化,这与噬菌体的种类变化呈显著相关性。在前3天,主要的噬菌体以明串珠菌和魏瑟拉菌噬菌体为主,而在第7天之后,则以乳杆菌噬菌体为主[22],因此乳酸菌在发酵中的演替过程受到噬菌体的调控。由于环境中的微生物的污染,蜡样芽孢杆菌作为一种致病菌和腐败菌经常出现在大豆发酵食品中,蜡样芽孢杆菌噬菌体BCP1-1显著降低大豆发酵食品中蜡样芽孢杆菌的数量,而没有影响发酵微生物枯草芽孢杆菌[11]。因此,噬菌体具有在不干扰其他共生微生物的同时,选择性地靶向和杀死特定宿主菌的能力。这种高度特异性降低了对有益细菌或非靶向微生物造成伤害的风险,因此提高了其安全性。此外,发酵食品是良好的分离有益噬菌体的资源,通过对发酵食品中噬菌体的分离方法的建立和优化,可以发掘出更多独特的抗致病菌和腐败菌的生物控制方法。
噬菌体除了可以调控宿主微生物的数量,还可以降低乳酸菌在发酵食品中释放的有毒代谢产物。噬菌体Q69可以有效减少酪胺在干酪中的积累[16]。类似的,在干酪模拟生产中,研究者发现肌尾噬菌体科的粪肠球菌噬菌体156可以降低干酪中生物胺的含量[17]。此外,原噬菌体造成的乳酸菌裂解可以释放其中的胞内酶,促进干酪风味物质产生[18]。在葡萄酒酿造过程中,一些葡萄酒腐败细菌如醋酸杆菌和葡萄糖酸杆菌可以将乙醇转化为醋酸,它们的存在会产生不愉悦的风味物质[73-74],对葡萄酒质量产生危害。研究人员发现了一种可以感染蜡状葡糖杆菌(Gluconobacter cerinus)的噬菌体,该噬菌体具有无尾二十面体头部的复层噬菌体科噬菌体,该噬菌体的存在有效降低了葡萄酒中醋酸的含量[29],这项研究结果为葡萄酒发酵管理提供了指导依据。此外,DEASY T等[30]分离出了感染短乳杆菌(Lactobacillus brevis)的噬菌体SA-C12 并鉴定了其形态学特征。通过啤酒酿酒试验,该噬菌体在啤酒环境中稳定存在,并显著减少啤酒中的腐败菌的数量。综上,发酵过程中的噬菌体并非均导致发酵异常,只有深入了解噬菌体的功能和特性,才有助于控制有害噬菌体并维持有益噬菌体,提高发酵性能。
4 发酵食品中噬菌体的检测方法
如图1所示,目前噬菌体的检测方法多采用直接法和间接法进行研究。直接法多利用双层平板法对噬菌体进行培养,适用于对烈性噬菌体的直接观察和计数,但对于一些温和噬菌体无法进行判断和分析。此外可以通过采用超速离心的方法,直接分离出噬菌体并用透射电子显微镜或扫描透射电子显微镜进行形态观察[75-76],或通过流式细胞术[77]、纳米颗粒跟踪[78]、落射荧光显微镜和原子力显微镜[79]等方法进行计数。但是,对有些不可培养微生物的噬菌体无法进行有效的分离。随着研究技术的发展与进步,噬菌体的研究同时结合表型观测与基因组分析技术,充分了解不同食品发酵体系中的噬菌体组成和分布特征。目前,随着测序技术的兴起和迭代,宏基因组测序分析技术也逐渐被用于噬菌体分析。宏基因组测序无需进行病毒分离,可通过直接提取环境样品中全部微生物的基因组并进行高通量测序以反映样品所有的遗传信息[36]。研究人员利用Illumina的二代测序(next generation sequencing,NGS)技术分析了Kinema中的噬菌体[13],比较了中国和韩国泡菜的乳酸菌群落和噬菌体群落,发现利用噬菌体比细菌可以更好的区分发酵食品的地域来源[21]。该方法可以宏观地反映环境中所有微生物的信息,并且解决了绝大部分微生物难以培养从而无法识别的问题,因此在发酵食品微生物学研究中具有广泛的应用前景。随着对噬菌体宏基因组测序的研究越来越深入以及测序技术的进步,利用宏病毒组测序技术可以进一步有针对性的对病毒乃至噬菌体进行提纯和分析,对发酵食品样品中病毒富集并提取DNA样本[36],之后进行文库构建进行测序分析。对干酪表面样品病毒浓缩之前添加过滤步骤,采用密度梯度超速离心替代纯化所得到的浓缩产物可被用于更好地解析病毒在干酪微生物生态系统中的重要性。通过宏病毒组学实现了在白酒大曲中对噬菌体种类的分析,并对噬菌体的宿主做了注释和预测[31]。但该方法对食品基质的保存、取样方法、噬菌体分离和核酸物质提取具有较高的要求,会直接影响提取噬菌体的质量和丰度。因此,在复杂食品基质体系中探索并优化提取噬菌体基因组的方法是决定测序结果准确性的关键,针对发酵食品中噬菌体组的特异性提取方法将有待进一步被优化。
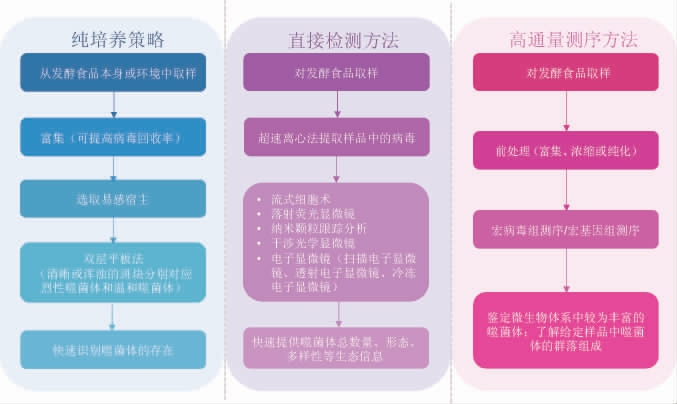
图1 发酵食品中噬菌体的检测方法
Fig.1 Detection methods of phages in fermented foods
5 总结与展望
由于噬菌体不直接参与发酵,其在发酵食品中的作用经常被忽视,但它通过调节微生物群落结构,间接影响发酵微生物的增殖和演替。通过了解噬菌体在发酵体系中的功能,才能更好的控制和利用噬菌体,进而控制发酵过程。尽管利用下一代测序技术为研究发酵食品中的噬菌体带来了便利,但目前该方法仍受限于噬菌体基因库的不完善,因此需要进一步完善噬菌体基因组的提取方法和噬菌体基因库的构建,利用更先进的方法研究噬菌体在发酵微生物群落中的关键作用,将为发酵食品的标准化生产奠定基础。
[1]HYMAN P,ABEDON S T.Bacteriophage host range and bacterial resistance[J].Adv Appl Microbiol,2010,70:217-248.
[2]CAMPBELL A.The future of bacteriophage biology[J].Nat Rev Genet,2003,4(6):471-477.
[3] KOSKELLA B, BROCKHURST M A.Bacteria-phage coevolution as a driver of ecological and evolutionary processes in microbial communities[J].FEMS Microbiol Rev,2014,38(5):916-931.
[4]CHEVALLEREAU A,PONS B J,VAN HOUTE S,et al.Interactions between bacterial and phage communities in natural environments[J].Nat Rev Microbiol,2022,20(1):49-62.
[5] PAILLET T, DUGAT-BONY E J C O I F S.Bacteriophage ecology of fermented foods:anything new under the sun?[J].Curr Opin Food Sci,2021,40:102-111.
[6] HAGENS S, LOESSNER M J.Application of bacteriophages for detection and control of foodborne pathogens[J].Appl Microbiol Biotechnol,2007,76(3):513-519.
[7] UMENE K, SHIRAISHI A.Complete nucleotide sequence of Bacillus subtilis(natto)bacteriophage PM1,a phage associated with disruption of food production[J].Virus Genes,2013,46(3):524-534.
[8]KIMURA K,ITOH Y.Characterization of poly-gamma-glutamate hydrolase encoded by a bacteriophage genome:possible role in phage infection of Bacillus subtilis encapsulated with poly-gamma-glutamate[J].Appl Environ Microbiol,2003,69(5):2491-2497.
[9]OZAKI T,ABE N,KIMURA K,et al.Genomic analysis of Bacillus subtilis lytic bacteriophage varphiNIT1 capable of obstructing natto fermentation carrying genes for the capsule-lytic soluble enzymes poly-gammaglutamate hydrolase and levanase[J].Biosci Biotechnol Biochem,2017,81(1):135-146.
[10]GHOSH K,SENEVIRATHNE A,KANG H S,et al.Complete nucleotide sequence analysis of a novel Bacillus subtilis-infecting bacteriophage BSP10 and its effect on poly-gamma-glutamic acid degradation[J].Viruses,2018,10(5):240.
[11]BANDARA N,JO J,RYU S,et al.Bacteriophages BCP1-1 and BCP8-2 require divalent cations for efficient control of Bacillus cereus in fermented foods[J].Food Microbiol,2012,31(1):9-16.
[12] HIGUCHI T, UCHIDA K, ABE K.Preparation of phage-insensitive strains of Tetragenococcus halophila and its application for soy sauce fermentation[J].Biosci Biotechnol Biochem,1999,63(2):415-417.
[13]KUMAR J,SHARMA N,KAUSHAL G,et al.Metagenomic insights into the taxonomic and functional features of kinema,a traditional fermented soybean product of Sikkim Himalaya[J].Front Microbiol, 2019, 10:1744.
[14] DE ANTONI G, ZAGO M, VASEK O, et al. Lactobacillus plantarum bacteriophages isolated from Kefir grains: phenotypic and molecular characterization[J].J Dairy Res,2010,77(1):7-12.
[15]PUJATO S A,GUGLIELMOTTI D M,ACKERMANN H W,et al.Leuconostoc bacteriophages from blue cheese manufacture: long-term survival, resistance to thermal treatments, high pressure homogenization and chemical biocides of industrial application[J].Int J Food Microbiol,2014,177:81-88.
[16]LADERO V,GOMEZ-SORDO C,SANCHEZ-LLANA E,et al.Q69(an E. faecalis-infecting bacteriophage) as a biocontrol agent for reducing tyramine in dairy products[J].Front Microbiol,2016,7:445.
[17]DEL RIO B,SANCHEZ-LLANA E,REDRUELLO B,et al.Enterococcus faecalis bacteriophage 156 is an effective biotechnological tool for reducing the presence of tyramine and putrescine in an experimental cheese model[J].Front Microbiol,2019,10:566.
[18]O'SULLIVAN D,ROSS R P,FITZGERALD G F,et al.Investigation of the relationship between lysogeny and lysis of Lactococcus lactis in cheese using prophage-targeted PCR[J].Appl Environ Microbiol,2000,66(5):2192-2198.
[19]LU Z,PEREZ-DIAZ I M,HAYES J S,et al.Bacteriophage ecology in a commercial cucumber fermentation[J].Appl Environ Microbiol,2012,78(24):8571-8578.
[20] YOON S S, BARRANGOU-POUEYS R, BREIDT F, et al.Detection and characterization of a lytic Pediococcus bacteriophage from the fermenting cucumber brine[J].J Microbiol Biotechnol,2007,17(2):262-270.
[21]JUNG M J,KIM M S,YUN J H,et al.Viral community predicts the geographical origin of fermented vegetable foods more precisely than bacterial community[J].Food Microbiol,2018,76:319-327.
[22]LU Z,BREIDT F,PLENGVIDHYA V,et al.Bacteriophage ecology in commercial sauerkraut fermentations[J].Appl Environ Microbiol,2003,69(6):3192-3202.
[23] COSTANTINI A, DORIA F, SAIZ J C, et al.Phage-host interactions analysis of newly characterized Oenococcus oeni bacteriophages:Implications for malolactic fermentation in wine[J].Int J Food Microbiol,2017,246:12-19.
[24]GINDREAU E,TORLOIS S,LONVAUD-FUNEL A.Identification and sequence analysis of the region encoding the site-specific integration system from Leuconostoc oenos(Oenococcus oeni) temperate bacteriophage phi 10MC[J].FEMS Microbiol Lett,1997,147(2):279-285.
[25]SAO-JOSE C,SANTOS S,NASCIMENTO J,et al.Diversity in the lysis-integration region of oenophage genomes and evidence for multiple tRNA loci, as targets for prophage integration in Oenococcus oeni[J].Virology,2004,325(1):82-95.
[26]PHILIPPE C,CHAIB A,JAOMANJAKA F,et al.Characterization of the first virulent phage infecting Oenococcus oeni, the queen of the cellars[J].Front Microbiol,2020,11:596541.
[27]PHILIPPE C,CHAIB A,JAOMANJAKA F,et al.Wine phenolic compounds differently affect the host-killing activity of two lytic bacteriophages infecting the lactic acid bacterium Oenococcus oeni[J].Viruses,2020,12(11):1316.
[28]JAOMANJAKA F,CLAISSE O,PHILIPPE C,et al.Complete genome sequence of lytic Oenococcus oeni bacteriophage OE33PA[J].Microbiol Resour Announc,2018,7(6):e00818-00818.
[29] PHILIPPE C, KRUPOVIC M, JAOMANJAKA F, et al.Bacteriophage GC1,a novel Tectivirus infecting Gluconobacter cerinus,an acetic acid bacterium associated with wine-making[J].Viruses,2018,10(1):39.
[30] DEASY T, MAHONY J, NEVE H, et al.Isolation of a virulent Lactobacillus brevis phage and its application in the control of beer spoilage[J].J Food Prot,2011,74(12):2157-2161.
[31] KANG J,CHEN X,HAN B Z, et al.Insights into the bacterial, fungal,and phage communities and volatile profiles in different types of Daqu[J].Food Res Int,2022,158:111488.
[32] WU Q, LI L, XIANG P, et al.Phages in fermented foods: Interactions and applications[J].Fermention,2023,9(3):201.
[33] OH H, SEO D J, JEON S B, et al.Isolation and characterization of Bacillus cereus bacteriophages from foods and soil[J].Food Environ Virol,2017,9(3):260-269.
[34]秦松跃,赵莉,狄亚心,等.乳酸菌噬菌体的分离及生物学特性鉴定[J].中国畜牧兽医,2021,48(6):2140-2149.
[35] CHENG L, MARINELLI L J, GROSSET N, et al.Complete genomic sequences of Propionibacterium freudenreichii phages from Swiss Cheese reveal greater diversity than Cutibacterium (formerly Propionibacterium)acnes phages[J].BMC Microbiol,2018,18(1):19.
[36]PARK E J,KIM K H,ABELL G C,et al.Metagenomic analysis of the viral communities in fermented foods[J].Appl Environ Microbiol,2011,77(4):1284-1291.
[37]李海涛,王慕华,赵玉明,等.酸奶生产中噬菌体污染的判断及防治[J].中国乳品工业,2018,46(3):13-15.
[38] KLEPPEN H P, NES I F, HOLO H.Characterization of a Leuconostoc bacteriophage infecting flavor producers of cheese starter cultures[J].Appl Environ Microbiol,2012,78(18):6769-6772.
[39]VERREAULT D,GENDRON L,ROUSSEAU G M,et al.Detection of airborne lactococcal bacteriophages in cheese manufacturing plants[J].Appl Environ Microbiol,2011,77(2):491-497.
[40] LUNDE M, AASTVEIT A H, BLATNY J M, et al.Effects of diverse environmental conditions on φLC3 prophage stability in Lactococcus lactis[J].Appl Environ Microbiol,2005,71(2):721-727.
[41]MADERA C,GARCIA P,RODRIGUEZ A,et al.Prophage induction in Lactococcus lactis by the bacteriocin Lactococcin 972[J].Int J Food Microbiol,2009,129(1):99-102.
[42]于晓丽,赵莉,程溪,等.发酵过程中不同理化因素对乳酸菌噬菌体的影响[J].中国乳品工业,2021,49(6):17-22.
[43] GUGLIELMOTTI D M, MERCANTI D J, REINHEIMER J A, et al.Review:efficiency of physical and chemical treatments on the inactivation of dairy bacteriophages[J].Front Microbiol,2011,2:282.
[44] CHUKEATIROTE E, PHONGTANG W, KIM J, et al.Significance of bacteriophages in fermented soybeans:A review[J].Biomol Concepts,2018,9(1):131-142.
[45]GHOSH K,KANG H S,HYUN W B,et al.High prevalence of Bacillus subtilis-infecting bacteriophages in soybean-based fermented foods and its detrimental effects on the process and quality of Cheonggukjang[J].Food Microbiol,2018,76:196-203.
[46]KOT W,NEVE H,HELLER K J,et al.Bacteriophages of Leuconostoc,Oenococcus,and Weissella[J].Front Microbiol,2014,5:186.
[47]PHILIPPE C,JAOMANJAKA F,CLAISSE O,et al.A survey of oenophages during wine making reveals a novel group with unusual genomic characteristics[J].Int J Food Microbiol,2017,257:138-147.
[48]LEDORMAND P,DESMASURES N,DALMASSO M.Phage community involvement in fermented beverages:an open door to technological advances?[J].Crit Rev Food Sci Nutr,2021,61(17):2911-2920.
[49]BIGOT B,LEE W J,MCINTYRE L,et al.Control of Listeria monocytogenes growth in a ready-to-eat poultry product using a bacteriophage[J].Food Microbiol,2011,28(8):1448-1452.
[50]SONI K A,NANNAPANENI R,HAGENS S.Reduction of Listeria monocytogenes on the surface of fresh channel catfish fillets by bacteriophage Listex P100[J].Foodborne Pathog Dis,2010,7(4):427-434.
[51] OLIVEIRA M, VINAS I, COLAS P, et al.Effectiveness of a bacteriophage in reducing Listeria monocytogenes on fresh-cut fruits and fruit juices[J].Food Microbiol,2014,38:137-142.
[52] GUENTHER S,LOESSNER M J.Bacteriophage biocontrol of Listeria monocytogenes on soft ripened white mold and red-smear cheeses[J].Bacteriophage,2011,1(2):94-100.
[53]SILVA E N,FIGUEIREDO A C,MIRANDA F A,et al.Control of Listeria monocytogenes growth in soft cheeses by bacteriophage P100[J].Braz J Microbiol,2014,45(1):11-16.
[54]SUKUMARAN A T,NANNAPANENI R,KIESS A,et al.Reduction of Salmonella on chicken breast fillets stored under aerobic or modified atmosphere packaging by the application of lytic bacteriophage preparation SalmoFreshTM[J].Poult Sci,2016,95(3):668-675.
[55]BOYACIOGLU O,SHARMA M,SULAKVELIDZE A,et al.Biocontrol of Escherichia coli O157:H7 on fresh-cut leafy greens[J].Bacteriophage,2013,3(1):e24620.
[56] MCLEAN S K, DUNN L A, PALOMBO E A.Phage inhibition of Escherichia coli in ultrahigh-temperature-treated and raw milk[J].Foodborne Pathog Dis,2013,10(11):956-962.
[57]GUENTHER S,HERZIG O,FIESELER L,et al.Biocontrol of Salmonella typhimurium in RTE foods with the virulent bacteriophage FO1-E2[J].Int J Food Microbiol,2012,154(1-2):66-72.
[58]KANG H W,KIM J W,JUNG T S,et al.Wksl3,a new biocontrol agent for Salmonella enterica serovars enteritidis and typhimurium in foods:characterization, application, sequence analysis, and oral acute toxicity study[J].Appl Environ Microbiol,2013,79(6):1956-1968.
[59]ZHANG H,WANG R,BAO H.Phage inactivation of foodborne Shigella on ready-to-eat spiced chicken[J].Poult Sci,2013,92(1):211-217.
[60] BUENO E, GARCIA P, MARTINEZ B, et al.Phage inactivation of Staphylococcus aureus in fresh and hard-type cheeses[J].Int J Food Microbiol,2012,158(1):23-27.
[61] LINDEN S B, ZHANG H, HESELPOTH R D, et al.Biochemical and biophysical characterization of PlyGRCS,a bacteriophage endolysin active against methicillin-resistant Staphylococcus aureus[J].Appl Microbiol Biotechnol,2015,99(2):741-752.
[62] FERNANDEZ L, GUTIERREZ D, RODRIGUEZ A, et al.Application of bacteriophages in the agro-food sector: A long way toward approval[J].Front Cell Infect Microbiol,2018,8:296.
[63]PHILIPPE C,CORNUAULT J K,DEMELO A G,et al.The never-ending battle between lactic acid bacteria and their phages[J].FEMS Microbiol Rev,2023,47(4):035.
[64] BARI S M N, WALKER F C, CATER K, et al.Strategies for editing virulent staphylococcal phages using CRISPR-Cas10[J].ACS Synth Biol,2017,6(12):2316-2325.
[65] OECHSLIN F.Resistance development to bacteriophages occurring during bacteriophage therapy[J].Viruses,2018,10(7):351.
[66] VIKRAM A, WOOLSTON J, SULAKVELIDZE A.Phage biocontrol applications in food production and processing[J].Curr Issues Mol Biol,2021,40:267-302.
[67] OLUWARINDE B O, AJOSE D J, ABOLARINWA T O, et al.Safety properties of Escherichia coli O157:H7 specific bacteriophages: Recent advances for food safety[J].Foods,2023,12(21):3989.
[68]ENDERSEN L,O'MAHONY J,HILL C,et al.Phage therapy in the food industry[J].Annu Rev Food Sci Technol,2014,5:327-349.
[69]SARKER S A,BERGER B,DENG Y,et al.Oral application of Escherichia coli bacteriophage: safety tests in healthy and diarrheal children from Bangladesh[J].Environ Microbiol,2017,19(1):237-250.
[70] DHOWLAGHAR N, DENES T G, NUTRITION.Control of residual phage in the evaluation of phage-based food safety applications[J].Crit Rev Food Sci Nutr,2023:1-7.
[71] SAMSON J E, MOINEAU S.Bacteriophages in food fermentations:new frontiers in a continuous arms race[J].Annu Rev Food Sci Technol,2013,4:347-368.
[72]GARCÍA P,MARTÍNEZ B,OBESO J M,et al.Bacteriophages and their application in food safety[J].Lett Appl Microbiol, 2008, 47(6): 479-485.
[73] BARTOWSKY E J,HENSCHKE P A.Acetic acid bacteria spoilage of bottled red wine—a review[J].Int J Food Microbiol,2008,125(1):60-70.
[74]BARTOWSKY E J,XIA D,GIBSON R L,et al.Spoilage of bottled red wine by acetic acid bacteria[J].Lett Appl Microbiol,2003,36(5):307-314.
[75]DUGAT-BONY E,LOSSOUARN J,DE PAEPE M,et al.Viral metagenomic analysis of the cheese surface:A comparative study of rapid procedures for extracting viral particles[J].Food Microbiol, 2020, 85:103278.
[76] LINDEROTH N A, SIMON M N, RUSSEL M.The filamentous phage pIV multimer visualized by scanning transmission electron microscopy[J].Science,1997,278(5343):1635-1638.
[77]OLIVEIRA J,MAHONY J,HANEMAAIJER L,et al.Detecting Lactococcus lactis prophages by Mitomycin C-mediated induction coupled to flow cytometry analysis[J].Front Microbiol,2017,8:1343.
[78] XU H, SHEN J, YANG C T, et al.Naked-eye counting of pathogenic viruses by phage-gold nanobiomaterials as probes[J].Mater Today Adv,2021,10:100122.
[79] ZAGO M, SCALTRITI E, FORNASARI M E, et al.Epifluorescence and atomic force microscopy:Two innovative applications for studying phage-host interactions in Lactobacillus helveticus[J].J Microbiol Methods,2012,88(1):41-46.