中国白酒是具有民族特色的酒精饮料,通常以不同的谷物为原料,以大曲、麸曲等为糖化发酵剂,经蒸煮、发酵、蒸馏、贮存和勾兑组合而成[1]。不同的自然环境、酿造原料、制曲工艺、酿造工艺、气候条件等决定了白酒的风格,目前已形成了各具特色的12种白酒香型,其中浓香、清香和酱香型深受国内外市场的喜爱。许多研究和生产实践表明,复杂而独特的微生物贯穿酿造全生命周期,对白酒的风味特征起着重要作用。酿造微生物来源是多途径的,原辅料、糖化发酵剂、发酵容器、生产场地和工具等都会提供不同种类的微生物,进而形成复杂多样的微生物生态系统。因此,全面解析酿造微生物的群落组成特征、代谢通路、代谢产物等对明晰白酒特征风味物质、改善酒体品质、推进智能化酿造方面具有重要意义。
长期以来,研究人员一直利用传统的微生物学技术研究复杂的酿造微生物体系。随着分子技术及测序技术的进步,一些组学手段(如基因组学、转录组学、蛋白质学和代谢组学等)已经开始被运用于酿造微生物研究,为全面解析酿造功能菌、功能基因和代谢通路、酶活性和代谢产物及其互作研究提供了技术支持[2-4]。例如,目前已通过基因组学和代谢组学技术挖掘到多个细菌(如乳酸菌、芽孢杆菌、葡萄球菌和梭菌)、真菌(如镰刀菌、毕赤酵母和根霉)等微生物与酒体风味形成相关[5-10],这些微生物有望成为提升白酒风味的媒介。本文对基因组学、转录组学、蛋白质学和代谢组学技术在白酒酿造微生物的应用进展进行综述,并对组学技术在未来酿造微生物的研究方向提出了思考与展望,旨在为后续探索微生物协同作用、风味形成机理、质量控制等相关领域的研究提供理论指导。
1 基因组学技术在白酒酿造微生物中的应用
基因组几乎包含了生物体所有的遗传信息,是微生物特定功能和表型形成的根源。因此,全基因组测序为在基因水平揭示白酒酿造微生物群落结构、功能基因、代谢机制等信息提供技术手段。一代测序以Sanger双链终止法为代表,具有测序读段长(可达1 000 bp),准确性高(可达99.999%)等特点,但存在测序成本高、测序通量低等缺点。二代测序技术以Roche公司的454技术、Illumina公司的Solexa/Hiseq技术和ABI公司的Solid技术为代表,具有测序成本低、测序速度快、准确性高等特点。三代测序以PacBio公司的单分子实时测序技术和ONT公司的单分子纳米孔测序技术为代表,具有准确性高、读长超长、检测简便等优势。目前,全基因组测序主要借助于二代和三代测序技术,这些技术在解析酿造微生物的群落结构及多样性特征、功能基因及通路、溯源酿造微生物等方面得到了广泛应用(表1)。
表1 基因组学技术在酿造微生物中的应用
Table 1 Application of genomics technology in brewing microorganism

白酒香型 研究对象 基因组学技术 研究内容 参考文献豉香凤香复合香馥郁香兼香酱香酱香酱香酱香酱香酱香老白干老白干米香浓香浓香浓香浓香清香清香芝麻香其他酒曲微生物酒醅微生物粮醅微生物酒曲微生物酒醅微生物酒醅微生物酒曲微生物酒曲微生物环境微生物环境微生物大曲微生物酒醅微生物酒曲微生物酒曲微生物窖泥微生物酒曲微生物酒曲微生物环境微生物酒醅微生物酒曲微生物酒曲微生物酒曲微生物二代测序二代测序二代测序二代测序二代测序三代测序二代+三代测序二代测序二代测序三代测序二代测序二代测序二代测序二代测序二代测序二代+三代测序三代测序二代测序二代测序二代+三代测序二代测序三代测序微生物多样性和演替规律发酵过程中微生物群落结构及演替规律不同发酵方式的微生物群落结构冬季和夏季微生物群落动态的变化微生物菌落结构与代谢通路解析耐酒精菌株的功能放线菌基因组装及功能基因挖掘大曲微生物菌群结构及功能组成微生物结构演替及与环境交互作用发酵过程微生物与环境交互作用溯源酿造过程产物酿造过程微生物菌群多样性制曲过程微生物菌群多样性细菌菌群结构和功能多样性微生物结构、空间分布和功能多样性解析新产蛋白酶菌的功能解析发酵新菌株的功能溯源酿造过程微生物菌群结构随发酵时间的变化规律微生物多样性和演替规律微生物群落结构和演替规律解析分散型泛菌的功能[11][12][13][14][15][16][17][18-19][20-21][22][23][24][25][26-27][28-31][32][33][34][35][36][37][38]
1.1 二代测序技术
目前,二代测序技术在白酒酿造微生物群落方面已取得诸多成果。例如,在酱香型白酒中,ZHU C T等[18-19]研究发现,温度、湿度和酸度影响曲块微生物群落结构和演变规律;无色杆菌属(Achromobacter)、寡养单胞菌属(Stentrophomonas)和代尔夫特菌属(Delftia)是翻曲过程中的优势菌,且一次翻曲操作有助于风味功能微生物的富集,二次翻曲有利于酯化功能微生物的富集,该研究对翻曲操作进行科学解释。在浓香型白酒中,WANG X J等[28-29]研究发现,在同一窖池中,不同空间位置、不同发酵原料的窖泥微生物群落在丰度和结构组成上具有显著差异,其中古菌在窖泥中丰度最高,其次是细菌和真菌;并且发现微生物的丰富度和多样性与窖泥年限呈正相关。在酿造环境方面,ZHU Q等[39-40]对茅台酿造核心产区、和义兴优势产区和习酒产区环境空气的微生物群落解析,发现茅台酿造产区的微生物结构与另外两个产区显著不同;进一步对茅台酒酿造环境、酿造过程、周边环境以及其他区域共100余株酿酒酵母基因组研究,发现茅台核心产区长期的酿造活动筛选进化出独特的酿酒酵母(Saccharomyces cerevisiae),为茅台酒的酿造提供了优质酵母资源。
1.2 三代测序技术
近年来,三代测序技术在酿造微生物的新基因组装与挖掘、生物学功能注释等方面研究逐渐增多。例如,XU Y Q等[38]对从酒曲中筛选出的分散型泛菌(Pantoea dispersa)BJQ0007进行测序,获得了其基因组的完整图。该菌的基因组大小为4.3 Mbp,鸟嘌呤(guanine,G)胞嘧啶(cytosine,C)含量为57.9%,共有4 285个基因,其中有4 176编码基因;通过基因注释发现,超三分之一的编码基因与碳水化合物、氨基酸等代谢功能相关;进一步的代谢通路分析表明,一些与邻苯二甲酸酯降解相关的基因在该菌中显著富集,而邻苯二甲酸酯是白酒中常见的塑化剂。该研究加强了对微生物降解邻苯二甲酸酯的认识,也为白酒食品安全管控提供新思路。由此可见,三代测序技术能获得目标菌株的完整基因组信息,并对其基因功能进行深入解析。
1.3 二代与三代测序技术联合
二代与三代测序技术逐渐被联合用于酿造微生物研究,主要研究思路是先对较大批量的菌株进行二代测序,然后挑选代表性菌株进行三代测序,并整合二代和三代测序数据来分析基因组特征。田浩杰等[17]对新筛选的酱香型高温大曲糖莱斯氏菌(Laceyella sacchari)FBKL4.014的基因组结构和功能进行深度解析,发现该菌株具备碳水化合物和氨基酸代谢潜力。LIU Y B等[32]对新分离纯化的高产蛋白酶的贝莱斯芽孢杆菌(Bacillus velezensis)DW-7进行蛋白编码基因预测、代谢通路富集分析,共注释出3 662个蛋白编码基因,其中有196个高产蛋白酶基因主要富集于丙氨酸、天冬氨酸、谷氨酸等代谢通路。
综上可知,在白酒酿造微生物研究中,基因组测序主要用于微生物群落结构和演变规律解析。二代测序虽然在数据分析方面存在难度,但因其通量高、成本低、精度高等优势被运用得最为广泛。三代测序虽然读段长,但目前测序错误率高,且测序错误随机出现。因此,二代测序与三代测序的联合,将在提高测序精度的同时,又能全面注释和解析新基因的功能。
2 转录组学技术在白酒酿造微生物中的应用
转录组学技术可以对特定时期、特定环境生物体的基因转录情况以及调控规律进行研究。随着分子生物技术的快速发展,相继出现了多种转录组学技术。基因表达谱芯片技术是最早应用于微生物领域的转录组学技术,其具有效率高、成本低、可以同时测序多个样本等优点;但也存在精度较低、对低表达的基因敏感度较低、只能测已知序列等缺点。近年来,具有高分辨率、高灵敏度、速度快、通量大等优点的核糖核酸测序(ribonucleicacidsequencing,RNA-seq)技术成为主流。此外,一些新兴测序技术,如单细胞转录组学技术、空间转录组学技术等,由于成本高、通量低、操作要求高等缺点还尚未在微生物领域中得到广泛应用。目前,转录组学技术在解析酿造微生物群落中活性物种的精细组成及表征对应功能基因和通路的表达水平中得到较广泛应用(表2)。
表2 转录组学技术在酿造微生物研究中的应用
Table 2 Application of transcriptomics technology in brewing microorganism

白酒香型 转录组学技术 研究对象 研究内容 参考文献酱香酱香浓香浓香浓香浓香浓香浓香浓香浓香清香其他基因表达谱芯片RNA-seq RNA-seq RNA-seq RNA-seq RNA-seq RNA-seq RNA-seq RNA-seq RNA-seq RNA-seq基因表达谱芯片酒曲微生物酒曲微生物窖泥微生物窖泥微生物酒醅微生物酒醅微生物酒醅微生物酒醅微生物酒醅微生物酒曲微生物酒醅微生物酿造微生物胁迫条件下基因表达差异研究活性微生物组成和功能性基因活性微生物组成、基因表达及关键代谢通路活性关键微生物和基因表达差异活性微生物的功能基因和通路活性微生物组成、功能特征及代谢通路功能微生物组成及代谢通路活性微生物的空间异质性活性微生物组成和关键转录本鉴定关键基因表达活性微生物组成、驱动因素及其作用胁迫条件下基因表达差异研究[41][42][43][44][45][46][47][48][49][50][51][52]
2.1 基因表达谱芯片技术
基因表达谱芯片采用互补脱氧核糖核酸(complementary deoxyribonucleic acid,cDNA)或寡核苷酸片段作探针,用基于核酸探针杂交的原理检测基因表达水平的变化,在酿造微生物领域运用极少。叶燕锐等[52]为研究酿酒酵母(Saccharomyces cerevisiae)对数生长期后期的代谢重构现象,采用表达谱芯片研究了酿酒酵母的全基因组表达谱变化。研究发现,在酿酒酵母对数生长期后期,许多与氨基酸合成和代谢相关的基因表达上调,提示酵母的代谢转向三羧酸循环(tricarboxylic acid cycle,TCA)和乙醛酸循环,进而导致酒精的生产速率降低,该研究为深入解析酒精发酵机制提供理论基础。
彭素琴等[41]以产酱香地衣芽孢杆菌(Bacillus licheniformis)CGMCC 3963为研究对象,从转录组学角度系统分析该菌在多重胁迫条件下的耐受机制。研究发现,在高温条件下,该菌与耐高渗、耐酸、耐乙醇相关的基因表达异常;进一步分析表明,高温刺激能更强地诱导与其他耐受相关的特异性基因的表达,进而导致CGMCC 3963的环境适应能力更强,该研究为后续筛选和富集酿造过程中高耐受能力菌株提供了思路。
2.2 RNA-seq技术
经文献检索发现,RNA-seq技术在浓香型白酒的研究中运用较为广泛,在其他白酒香型中应用较少。ZHOU W等[53]应用RNA-seq技术对浓香型白酒窖底和窖四周的窖泥微生物功能及影响浓香型白酒质量的关键微生物和基因进行解析。结果表明,古菌、梭状芽孢杆菌和一些嗜热微生物对老窖泥功能有显著影响,且碳水化合物和氨基酸在老窖泥中能代谢产生更多的有机酸,进一步研究发现有机酸代谢通路有8种关键酶表达活跃,进而表明有机酸代谢相关的关键基因对浓香型白酒生产质量有重要影响。王康丽[46]对浓香型白酒酿造过程中不同发酵节点酒醅样品中的微生物进行研究,发现菌群主要富集在代谢相关通路,且主要为碳水化合物代谢通路;同时也注释出在酒醅发酵过程中参与调控细胞代谢和生理过程的基因,该研究为揭示浓香型白酒发酵机理及发酵过程调控提供理论基础。
综上可知,转录组学技术可以表征微生物的基因表达水平和代谢通路激活情况。随着现有转录组学技术的不断成熟,新兴转录组学技术如单细胞组学技术等的快速发展,将有助于了解微生物在不同情况下的转录表达及调控信息,转录组学技术也将更广泛的被应用在酿造微生物研究。
3 蛋白组学技术在白酒酿造微生物中的应用
蛋白质组学技术可以对特定时期、特定环境下生物体的蛋白质组成特征、表达水平、变化规律及调控机制进行研究。目前主流的蛋白质组学技术有双向凝胶电泳(twodimensional electrophoresis,2-DE),蛋白质非标记定量(label-free quantification,LFQ)、同位素标记相对和绝对定量(isobaric tags for relative and absolute quantitation,iTRAQ)、串联质谱标签(tandem mass tags,TMT)和数据非依赖性采集(data-independent acquisition,DIA)技术等。目前,蛋白组学技术已被成功用于酿造微生物蛋白图谱构建、差异蛋白研究及功能蛋白发掘(表3)。
表3 蛋白组学技术在酿造微生物研究中的应用
Table 3 Application of proteomics technology in brewing microorganism
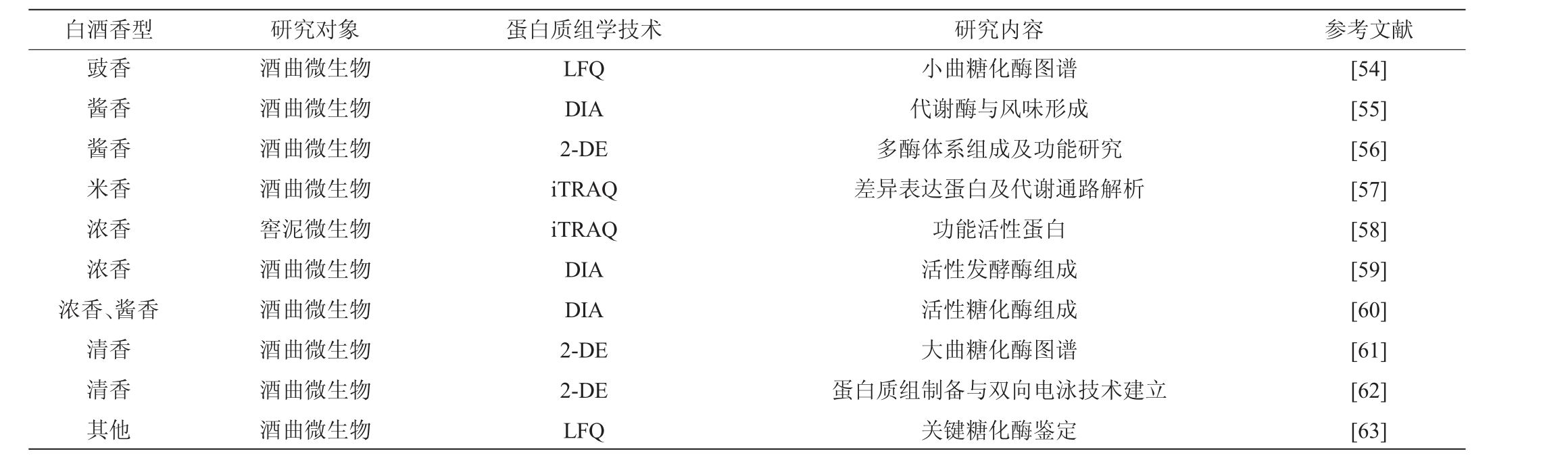
白酒香型 研究对象 蛋白质组学技术 研究内容 参考文献豉香酱香酱香米香浓香浓香浓香、酱香清香清香其他酒曲微生物酒曲微生物酒曲微生物酒曲微生物窖泥微生物酒曲微生物酒曲微生物酒曲微生物酒曲微生物酒曲微生物LFQ DIA 2-DE iTRAQ iTRAQ DIA DIA 2-DE 2-DE LFQ小曲糖化酶图谱代谢酶与风味形成多酶体系组成及功能研究差异表达蛋白及代谢通路解析功能活性蛋白活性发酵酶组成活性糖化酶组成大曲糖化酶图谱蛋白质组制备与双向电泳技术建立关键糖化酶鉴定[54][55][56][57][58][59][60][61][62][63]
3.1 2-DE技术
2-DE技术建立于1975年,其原理根据蛋白质等电点和相对分子质量的不同,在两个维度对复杂的蛋白质样品进行分离,是蛋白质组学领域中应用最早且应用范围最广的核心技术之一,但该技术存在重复性差、灵敏度低、覆盖率低等缺点。近年来,该技术与质谱技术的结合可用于蛋白质定性和鉴定差异蛋白。刘龙山[56]为了解酱香高温大曲中多酶系的组成和功能,采用2-DE技术结合质谱分析对普通高温大曲和强化高温大曲中的蛋白质组成进行鉴定和比较。研究首先在强化高温大曲和普通高温大曲中分别鉴定出185种和165个蛋白,进一步的差异分析发现,强化高温大曲相比普通高温大曲共有24个蛋白表达显著上调,4个蛋白表达显著下调,这些蛋白主要与蛋白水解酶、糖代谢、次生代谢产物合成等相关。
3.2 LFQ技术
LFQ是一种不依赖于同位素标记的非标记蛋白组定量技术,只需分析大规模鉴定蛋白质时所产生的质谱数据,从而对肽段对应的蛋白质进行相对定量,该技术具有实验灵活性强、成本低等优点。目前,LFQ技术主要应用于酒曲糖化酶鉴定及图谱构建方向。如,WANG B W等[63]利用LFQ技术对白酒发酵过程的糖化酶及其功能进行解析,结果共鉴定出51种与白酒发酵相关的碳水化合物水解酶,且80%的碳水化合物水解酶是由酒曲提供的;其中,来自曲霉属(Aspergillus)和根霉属(Rhizopus)产生的α-淀粉酶和葡萄糖淀粉酶是白酒发酵的关键糖化酶,该研究揭示了白酒发酵过程中多种糖化酶对乙醇产量的协同作用。
3.3 iTRAQ技术
iTRAQ是由美国AB Sciex公司研发的多肽体外标记定量技术,该技术通过采用不同数量同位素标签特异性标记多肽氨基酸基团,即可实现不同样本中蛋白质的相对定量。该技术具有灵敏度高、准确性高、通量高、鉴定深度高等优势,是近年来定量蛋白质组学常用的高通量筛选技术。目前,iTRAQ技术主要应用于酿造微生物的功能蛋白筛选及差异蛋白表征方向。如在米香型白酒中,HUANG G D等[57]应用iTRAQ技术探索了酒曲微生物的蛋白质组学变化,研究共鉴定出4 062个蛋白,其中上调表达蛋白123个,下调表达蛋白88个;代谢通路分析发现,这些差异表达蛋白显著富集于碳代谢、乙醛酸和二羧酸代谢、丙酮酸代谢、碳水化合物代谢通路等;进一步的差异蛋白调控趋势和碳代谢通路分析提示,铵代偿促进了葡萄糖消耗,导致本应进入高级醇合成途径的碳流被逆转到三羧酸循环(TCA),从而降低了高级醇的含量。因此,基于iTRAQ技术可高效鉴定出不同样本间的差异蛋白,并对其影响的生物学通路进行研究。
3.4 DIA技术
DIA技术是将质谱整个全扫描范围分为若干个小窗口,然后高速、循环地对每个窗口中所有离子进行选择、碎裂、检测,从而获得样本中所有离子的全部碎片信息,对数据的进一步生物信息学分析即可对蛋白进行鉴定、定量及差异蛋白分析等。该技术具有高覆盖率、超高准确率、高通量、高稳定等优势。刘龙山[56]以酱香大曲为研究对象,采用DIA技术对大曲酶系在苯丙氨酸代谢中的功能进行解析。结果发现,在酱香大曲中共鉴定到1 965个蛋白,其中参与苯丙氨酸代谢的蛋白有31个,涉及11种酶,包括醛脱氢酶、酰胺酶、转氨酶等;进一步的功能分析发现,在这些酶和一些微生物的作用下苯丙氨酸可代谢合成芳香族化合物苯乙酸、苯乙醛,这些化合物为酱香大曲提供花果香,该研究为酱香型白酒风味的形成提供理论基础。
综上可知,在众多蛋白质组学研究方法中,2-DE技术虽然经典但因分离能力有限、操作程序复杂等原因已逐渐被淘汰;LFQ成本较低,但只可以检测到低丰度和高丰度蛋白质;iTRAQ适合样本量且需要精度较高的研究,样本量大且需要高覆盖率则DIA更为合适。因此,iTRAQ和DIA等定量蛋白质组学技术已被常用于解析酿造微生物功能蛋白和绘制糖化酶图谱,为系统研究微生物的代谢调控提供技术支持。
4 代谢组学技术在白酒酿造微生物中的应用
代谢组学技术是对生物体在特定时期或特定状态下的低分子质量(<1 500 Da)代谢产物、激素和信号分子的规律进行分析。根据研究目的的不同,代谢组学可进一步分为非靶向(无偏向性的检测所有小分子代谢物)和靶向代谢组学(针对特定一类代谢物)。根据分析手段的不同,代谢组学技术可分为气相色谱-质谱联用(gas chromatographymass spectrometer,GC-MS)、液相色谱-质谱联用(liquid chromatograph-mass spectrometer,LC-MS)、全二维气相色谱-飞行时间质谱(comprehensive two dimensional gas chromatography-time-of-flight mass spectrometry,GC×GC-TOF/MS)等。这些技术已经在酿造微生物代谢化合物组成与变化、代谢化合物与风味有着广泛的应用(表4),在一定程度上推动了白酒酿造机理的探索进程。
表4 代谢组学技术在酿造微生物中的应用
Table 4 Application of metabolomics technology in brewing microorganism

白酒香型 代谢组学技术 研究内容 参考文献凤香凤香酱香酱香酱香清香清香、浓香、酱香6种香型6种香型LC-MS GC-MS GC×GC-TOF/MS LC-MS LC-MS GC×GC-TOF/MS GC×GC-TOF/MS GC×GC-TOF/MS GC-MS白酒老熟过程中的代谢化合物解析年份基酒差异化合物解析大曲代谢化合物解析酿酒酵母和乳杆菌的代谢相互作用不同类型大曲的代谢化合物差异白酒在老熟过程中挥发化合物变化中国白酒与西方白酒成分异同分析不同地区的白酒特征风味化合物筛选不同香型香气化合物鉴定及预测模型构建[64][65][66][67][68][69][70][71][72-74]
4.1 GC-MS技术
GC-MS技术采用气相色谱分离样品,根据色谱保留时间和离子质荷比进行定性,基于色谱峰面积进行定量。该技术被广泛应用于挥发性香气成分的分析,其具有色谱的高分离度、高通量及质谱的高灵敏度和特异性,但存在样本前处理比较复杂、产生的数据难以充分利用的缺点。目前,GC-MS在微生物代谢组学的研究运用得较为广泛。例如,杨辉等[65]为研究凤香型基酒储存过程中化合物的变化规律,采用GC-MS对6组不同年份凤香型基酒的化合物进行检测。结果发现,在新酒中检测到的化合物共有51种,是所有年份基酒中化合物种类最少的,而经过2年以上酒海贮存的基酒中化合物种类增多到了59种,说明贮存时间越长,化合物种类和含量越多。进一步的变量重要性分析筛选出包括有机酸、氨基酸、糖类等共20种差异化合物,这些物质在贮存过程中含量发生显著变化,该研究为深入解析白酒老熟机理提供一定的理论指导。
4.2 LC-MS技术
LC-MS技术主要原理是采用液相色谱分离样品,根据色谱保留时间、母离子质荷比和二级碎片离子图谱进行定性,采用一级质谱获得的母离子峰面积进行定量。该技术被广泛用于非挥发性香气成分的分析,具有快速、高效、高灵敏度和高置信等优势;但存在基质效应,导致目标物的离子化效率降低或增强,引起响应降低或增高。LUO S等[68]为研究酱香黑曲的形成机制,采用LC-MS对从发酵仓随机采集39份大曲样本的代谢物进行检测和比较分析。结果发现,从所有样品中共鉴定出1 062种相对丰度不同的代谢化合物;差异分析发现,相比于白曲和黄曲,黑曲中有251种代谢物含量上调;进一步的代谢通路功能分析表明,这些上调的代谢化合物显著富集于酪氨酸代谢通路,而该通路与酶促褐变和黑色素生成相关。该研究为控制黑曲的形成和提高白酒品质提供理论指导。
4.3 GC×GC-TOF/MS技术
GC×GC-TOF/MS可以检测到更多一维方法无法分离的色谱峰,相比于常规气质联用具有高容量、高分离度和高灵敏度等显著优势,是解析复杂物质与检测未知物质的重要技术手段。该技术存在的主要缺点是在全扫描模式下灵敏度低,对于检测复杂基质中痕量成分时存在误差。在酱香型白酒中,YANG L等[66]为研究高温黄曲、白曲和黑曲三种类型的代谢物组成不明确、发酵性能不清晰等问题,采用GC×GC-TOF/MS技术对高温大曲中的化合物进行解析,研究共发现高温大曲有647种化合物,其中有401种挥发性化合物和246种难挥发化合物;同时对大曲物质形成的代谢通路进行分析,发现有21条通路与大曲代谢表型差异相关。进一步通过多元统计分析筛查出48个可用于区分不同类型大曲的生物标志物,主要为醇类、芳香族化合物及难挥发的酸类化合物,该研究对揭示大曲代谢机制提供了理论基础,也促进了不同类型高温大曲的有效应用及酱香型白酒的高质量酿造。
综上可知,代谢组学技术在解析酿造微生物代谢化合物领域运用广泛。尽管目前代谢组学技术还在仪器分析范围存在局限、数据分析繁琐、数据库不完整等挑战,但随着技术的不断发展以及数据库的不断完善,代谢组学技术在未来的酿造微生物代谢研究中仍将占主导地位。
5 多组学技术在白酒酿造微生物中的应用
白酒特征风味的形成是由多个代谢通路协调作用的复杂调控过程,仅从群落组成、基因表达、蛋白质或代谢物等的任一层次都无法全面揭示其中的机制。多组学分析是指将两种或两种以上由组学技术产生的数据进行有效整合,从而实现不同层次数据的互补,有助于增强对微生物与微生物、微生物与环境之间复杂相互作用解析。目前,多组学分析已被用于解析微生物群落多样性与风味物质形成机理、功能活性及代谢特征之间的关联机制等方面(表5)。
表5 多组学技术在酿造微生物研究中的应用
Table 5 Application of multi-omics technology in brewing microorganism

白酒香型 研究对象 多组学技术 研究内容 参考文献酱香酱香酱香酱香老白干米香浓香浓香浓香浓香浓香清香清香清香、浓香、酱香其他其他酒醅微生物酒醅微生物酒醅微生物酒曲微生物酒曲微生物酒醅微生物原料微生物窖泥微生物窖泥微生物窖泥微生物酒曲微生物酒曲微生物酒曲微生物酒醅微生物原料微生物酒曲微生物基因组学+转录组学+代谢组学基因组学+转录组学基因组学+代谢组学转录组学+代谢组学基因组学+代谢组学基因组学+代谢组学基因组学+代谢组学基因组学+蛋白组学+代谢组学基因组学+代谢组学基因组学+代谢组学基因组学+代谢组学基因组学+转录组学基因组学+代谢组学基因组学+转录组学+代谢组学基因组学+转录组学基因组学+蛋白组学微生物多样性、功能活性及代谢特征微生物菌落组成及转录活性微生物多样性与风味物质形成酶活性、挥发性代谢物和菌群特征微生物多样性与风味物质形成微生物多样性与风味物质形成理化性质、微生物群落和挥发性化合物的动态变化微生物组成、蛋白质和代谢物丰度的变化微生物多样性与风味物质形成细菌群落的空间分布及与挥发性化合物积累的关系理化性质、微生物群落和挥发性化合物的动态变化微生物菌落组成及转录活性微生物多样性风味物质形成不同地域的群落、风味、代谢特征微生物菌落组成及转录活性糖化关键菌群及酒曲品质提升[4][75][6,76][77][10][78-79][80][3][5,8][81][82-83][84][7,9][85][86][87]
基因组学和代谢组学技术的联合分析在酿造微生物研究中尤为常见。例如,MU Y等[82]探讨了太空诱变和生物合成的两种生物扰动对浓香大曲微生物群落、理化性质和代谢特征的影响。研究发现,经生物扰动后乳酸菌和曲霉菌数量显著减少,而酶的活性都升高;此外,在鉴定出的14种芳香活性挥发物和51种差异代谢物中,大多数在生物扰动后含量都升高,如吡嗪类、醇类、小肽类和碳水化合物;进一步相关性分析和代谢通路分析表明,大曲风味品质的差异主要与细菌群落有关,其中与芽孢杆菌(Bacillus)等相关性最为显著,该研究对阐明大曲风味形成机理和推动生物扰动技术在传统发酵工业中的应用提供了参考。
基因组学、转录组和代谢组学技术的联合分析也逐渐开始流行。如TAN Y W等[85]为了评估白酒风味化合物和微生物生态的地理特征,从全中国9个代表性酒厂收集了403个发酵样品,通过联合基因组学、转录组学、代谢组学和可培养验证等技术手段进行分析。结果发现,中国白酒不同香型的风味特征差异主要体现在20~40个不同的特征风味化合物的组成上,而微生物菌群的区域差异微生物生态主要是48~156个核心微生物的共同作用,进而形成了不同香型白酒的产区特征优势;进一步功能分析发现,基于真菌-细菌相互作用与菌群组装模式,可能与白酒发酵优质率、发酵时长特征相关,该研究为阐明中国白酒产区的地域微生态酿造优势提供了新证据。
综上可知,在解析酿造微生态研究方面,多组学技术联合比单一组学技术更具优势。例如,基因组数据和转录组数据的联合分析,可以实现不同微生物群落差异表达基因和代谢通路表达分析,有助于鉴定群落的特征基因、核心代谢通路,系统解析酿造微生物的功能机制。因此,多组学联合分析有助于全面了解白酒酿造过程的复杂性和整体性,加深对酿造微生物的多层次认识,未来多组学整合将是白酒酿造微生物研究中的大趋势。
6 结论与展望
白酒风味形成主要由酿造体系内菌系、酶系和物系的共同作用。越来越多的研究表明,使用单一的组学技术并不能系统解析白酒发酵过程。多组学技术可以从基因水平到转录水平到蛋白水平到代谢水平对白酒酿造微生物的多样性和风味成分进行系统研究。但目前多组学技术联合应用价格较贵,且产生的数据维度高、异质性高、价值密度稀疏等特点,导致算法模型的精确度和灵敏度受到影响。此外,现有的实验方法尚不能对数据分析产生的结果进行完整验证,导致从研究数据指导实际生产还存在困难。当然,随着近年来人工智能和机器学习的不断发展,将有助于挖掘多组学数据中隐含的知识和规律。因此,研究人员要与时俱进,积极将大数据和人工智能领域的新兴技术引用到酿造微生物领域来,加强与多组学数据的联动,全面解析酿造微生物机理,实现白酒生产科学化和标准化,助推中国白酒高质量生产。
[1]ZHENG X W,HAN B Z.Baijiu(白酒),Chinese liquor:History,classification and manufacture[J].J Ethnic Foods,2016,3(1):19-25.
[2]ZHUANSUN W W,XU J,LI Z Q,et al.Dynamic changes in the microbial community,flavour components in jiupei of a novel Maotai-Luzhouflavoured liquor under various daqu blending modes and their correlation analysis[J].LWT-Food Sci Technol,2022,172:114167.
[3]LIU M K,TANG Y M,GUO X J,et al.Structural and functional changes in prokaryotic communities in artificial pit mud during Chinese Baijiu production[J].Msystems,2020,5(2):e00829-19.
[4] ZHANG H X,TAN Y W,WEI J L, et al.Fungal interactions strengthen the diversity-functioning relationship of solid-state fermentation systems[J].Msystems,2022,7(4):e00401-22.
[5]GAO J J,LIU G Y,LI A J,et al.Domination of pit mud microbes in the formation of diverse flavour compounds during Chinese strong aromatype Baijiu fermentation[J].LWT-Food Sci Technol,2021,137:110442.
[6]ZHANG H X,WANG L,WANG H Y,et al.Effects of initial temperature on microbial community succession rate and volatile flavors during Baijiu fermentation process[J].Food Res Int,2021,141(3):109887.
[7] TANG J, LIU Y C, LIN B, et al.Effects of ultra-long fermentation time on the microbial community and flavor components of light-flavor Xiaoqu Baijiu based on fermentation tanks[J].World J Microb Biot, 2022,38(1):3.
[8]XU S S,ZHANG M Z,XU B Y,et al.Microbial communities and flavor formation in the fermentation of Chinese strong-flavor Baijiu produced from old and new Zaopei[J].Food Res Int,2022,156(6):111162.
[9] LIN B, TANG J, YANG Q, et al.Microbial succession and its effect on key aroma components during light-aroma-type Xiaoqu Baijiu brewing process[J].World J Microb Biot,2022,38(10):166.
[10]MA B T,FAN E D,LI Z X,et al.Study on relationship between brewing microflora and microconstituents of Laobaigan-flavor Baijiu[J].J Food Sci Tech,2020,38(2):20-33.
[11]ZHAO W H,LIANG Z,QIAN M,et al.Evolution of microbial communities during fermentation of Chi-flavor type Baijiu as determined by high-throughput sequencing[J].LWT-Food Sci Technol, 2022, 170:114102.
[12]陈雪,张永利,闫宗科,等.凤香型酒醅微生物群落演替及其与理化指标的相关性分析[J].食品科学,2020,41(22):200-205.
[13]程伟,陈兴杰,周端,等.复合香型白酒粮醅机械化和传统堆积发酵过程中微生物群落结构的对比分析[J].食品科学,2023,44(14):152-161.
[14]KANG J M,ZHENG X W,YANG X,et al.Contrasting summer versus winter dynamic microbial communities and their environmental driving factors in the solid-state saccharification process of Fuyu-flavor Baijiu[J].Food Res Int,2022,154(4):111008.
[15]范培文,刘蒲临,张明春,等.浓酱兼香型白酒发酵酒醅微生物群落及理化指标对正丙醇产出的影响[J].中国酿造,2022,41(7):32-37.
[16]WANG J L,LU C S,XU Q,et al.Genomic analysis of Lactiplantibacillus pentosus LTJ12,a novel strain with high alcohol tolerance isolated from Chinese Baijiu[J].Foods,2023,12:35.
[17]田浩杰,李豆南,邱树毅,等.酱香大曲中高温放线菌的筛选及基因组解析[J].食品科学,2023,44(18):68-76.
[18]ZHU C T,CHENG Y X,ZUO Q C,et al.Exploring the impacts of traditional crafts on microbial community succession in Jiang-flavored Daqu[J].Food Res Int,2022,158(8):111568.
[19] JIANG Q E, WU X Y, XU Y Q, et al.Microbial composition and dynamic succession during the Daqu production process of Northern Jiangflavored liquor in China[J].3 Biotech,2021,11(4):1-15.
[20]LI Y Y,CHENG Y X,WANG H,et al.Diverse structure and characteristics of the fungal community during the different rounds of Jiangflavoured Baijiu production in Moutai town[J].LWT-Food Sci Technol,2022,161:113313.
[21] WANG L, HUANG Y G, HU X X, et al.The impact of environmental factors on the environmental bacterial diversity and composition in the Jiang-flavoured Baijiu production region[J].LWT-Food Sci Technol,2021,149:111784.
[22]LIU S P,JIANG Z F,MA D N,et al.Distance decay pattern of fermentedrelated microorganisms in the sauce-flavor Baijiu producing region[J].Food Biosci,2023,51(2):102305.
[23]LI D N,HUANG W,WANG C X,et al.The complete genome sequence of the thermophilic bacterium Laceyella sacchari FBKL4.010 reveals the basis for tetramethylpyrazine biosynthesis in Moutai-flavor Daqu[J].Microbiologyopen,2019,8(12):e922.
[24]马冰涛,范恩帝,李泽霞,等.老白干香型白酒酿造微生物菌群及其与微量成分的关系[J].食品科学技术学报,2020,38(2):20-33.
[25]马冰涛,范恩帝,李泽霞,等.老白干香型白酒制曲过程中微生物多样性及其与风味成分的关系[J].食品与发酵工业,2020,46(16):7-16.
[26]HU Y L,LEI X Y,ZHANG X M,et al.Characteristics of the microbial community in the production of Chinese rice-flavor Baijiu and comparisons with the microflora of other flavors of Baijiu[J].Front Microbiol,2021,12(4):673670.
[27]ZHAO X X,WANG Y R,CAI W C,et al.High-throughput sequencingbased analysis of microbial diversity in rice wine koji from different areas[J].Curr Microbiol,2020,77(1):882-889.
[28] WANG X J, ZHU H M, REN Z Q, et al.Characterization of microbial diversity and community structure in fermentation pit mud of different ages for production of strong-aroma Baijiu[J].Pol J Microbiol,2020,69(2):151-164.
[29]CAI W C,XUE Y A,TANG F X,et al.The depth-depended fungal diversity and non-depth-depended aroma profiles of pit mud for strong-flavor Baijiu[J].Front Microbiol,2022,12(1):789845.
[30]FU J X,CHEN L,YANG S Z,et al.Metagenome and analysis of metabolic potential of the microbial community in pit mud used for Chinese strongflavor liquor production[J].Food Res Int,2021,143(5):110294.
[31]LI J J,SUN H Z,WANG Q,et al.Microbial community spatial structures in Luzhou-flavored liquor pit muds with different brewing materials[J].Peer J,2022,10:e12987.
[32] LIU Y B, FU J Y, WANG L L, et al.Isolation, identification, and whole-genome sequencing of high-yield protease bacteria from Daqu of ZhangGong Laojiu[J].Plos One,2022,17(4):e0264677.
[33]XU Y Q,WU M Q,NIU J L,et al.Clostridium btbubcensis BJN0001,a potentially new species isolated from the cellar mud of Chinese strongflavor Baijiu,produces ethanol,acetic acid and butyric acid from glucose[J].3 Biotech,2022,12(9):203.
[34]WANG X S,DU H,XU Y.Source tracking of prokaryotic communities in fermented grain of Chinese strong-flavor liquor[J].Int J Food Microbiol,2017,244(3):27-35.
[35]赵欣怡,甄攀,赵鑫锐,等.清香型白酒立醅期酒醅中主体酸和细菌菌群结构动态解析[J].食品与发酵工业,2022,48(9):26-33.
[36]WANG Q,WANG C Y,XIANG X Q,et al.Analysis of microbial diversity and succession during Xiaoqu Baijiu fermentation using high-throughput sequencing technology[J].Eng Life Sci,2022,22(7):495-504.
[37]孙思佳,翟磊,于学健,等.扣囊复膜孢酵母CICC33077对芝麻香白酒高温大曲微生物群落结构和功能特性影响的研究[J].食品与发酵工业,2023,49(7):99-105.
[38] XU Y Q, ZHAO J R, HUANG H Q, et al.Biodegradation of phthalate esters by Pantoeadispersa BJQ0007 isolated from Baijiu[J].J Food Compos Ana,2022,105(1):104201.
[39]ZHU Q,CHEN L Q,PENG Z,et al.Analysis of environmental driving factors on core functional community during Daqu fermentation[J].Food Res Int,2022,157(7):111286.
[40]HAO F,TAN Y W,LV X B,et al.Microbial community succession and its environment driving factors during initial fermentation of Maotaiflavor Baijiu[J].Front Microbiol,2021,12(5):669201.
[41]彭素琴,吴群,徐岩.产酱香地衣芽孢杆菌CGMCC3963耐受特征及基于转录组学的耐受机制分析[J].微生物学通报,2014,41(12):2395-403.
[42] YI Z L, JIN Y L, XIAO Y, et al.Unraveling the contribution of high temperature stage to Jiang-flavor Daqu,a liquor starter for production of Chinese Jiang-flavor Baijiu, with special reference to metatranscriptomics[J].Front Microbiol,2019,10(3):472.
[43]ZHOU W,LIAO Z M,WU Z Y,et al.Analysis of the difference between aged and degenerated pit mud microbiome in fermentation cellars for Chinese Luzhou-flavor Baijiu by metatranscriptomics[J].J Sci Food Agr,2021,101(11):4621-4631.
[44] BASHIARDES S, ZILBERMAN-SCHAPIRA G, ELINAV E.Use of metatranscriptomics in microbiome research[J].Bioinformatics Biol Insights,2016,10:19-25.
[45]田瑞杰,张勇,冯大鸿,等.基于宏转录组学的浓香型白酒酒醅活性微生物群落空间异质性研究[J].轻工学报,2022,37(1):1-11.
[46]王康丽.基于宏转录组学的浓香型酒醅微生物群落多样性及代谢特征研究[D].郑州:郑州轻工业大学,2021.
[47]田源,孔小勇,方芳.浓香型白酒发酵过程微生物合成正丙醇途径解析[J].微生物学报,2020,60(7):1421-1432.
[48]TIAN R J,ZHANG Y,FENG D H,et al.Study on spatial heterogeneity of active microbial community in strong-flavor Baijiu fermented grains based on metatranscriptome[J].J Light Ind,2022,37(1):1-11.
[49] HUANG Y H, YI Z L, JIN Y L, et al.Metatranscriptomics reveals the functions and enzyme profiles of the microbial community in Chinese nong-flavor liquor starter[J].Front Microbiol,2017,8(9):1747.
[50]ALI B,YI Z L,FANG Y,et al.Characterization of a fungal thermostable endoglucanase from Chinese Nong-flavor daqu by metatranscriptomic method[J].Int J Biol Macromol,2019,121:183-190.
[51] PAN Y Y, WANG Y, HAO W J, et al.Metatranscriptomics unravel composition, drivers, and functions of the active microorganisms in Light-flavor liquor fermentation[J].Microbiol Spectrum,2022,10(3):e02151-21.
[52]叶燕锐,唐语谦,陈宏运,等.酿酒酵母对数生长后期代谢重构的全基因组表达谱芯片分析[J].生物工程学报,2008(6):962-967.
[53] ZHOU W, LIAO Z M, WU Z Y, et al.Analysis of the difference between aged and degenerated pit mud microbiome in fermentation cellars for Chinese Luzhou-flavor Baijiu by metatranscriptomics[J].J Sci Food Agr,2021,101(11):4621-4631.
[54]王柏文.小曲糖化酶谱形成机制及其对白酒发酵过程的影响[D].无锡:江南大学,2020.
[55]柳习月,朱琪,杨帆,等.蛋白组学揭示苯丙氨酸代谢对酱香大曲风味影响[J].食品科技,2021,46(8):1-6.
[56]刘龙山.酱香型白酒高温大曲的宏蛋白组学研究[D].福州:福建师范大学,2016.
[57] HUANG G D, REN H, WANG A L, et al.iTRAQ-based proteomic analysis reveals the molecule mechanism of reducing higher alcohols in Chinese rice wine by nitrogen compensation[J].Ann Microbiol,2021,71(1):1-13.
[58]ZHENG Q,LIN B R,WANG Y B,et al.Proteomic and high-throughput analysis of protein expression and microbial diversity of microbes from 30-and 300-year pit muds of Chinese Luzhou-flavor liquor[J].Food Res Int,2015,75(9):305-314.
[59]FAN W Y,ZHAO X R,DU G C,et al.Metaproteomic analysis of enzymatic composition in Baobaoqu fermentation starter for Wuliangye Baijiu[J].Int J Food Sci Tech,2021,56(8):4170-4181.
[60]XIA Y,ZHU M,DU Y K,et al.Metaproteomics reveals protein composition of multiple saccharifying enzymes in nongxiangxing daqu and jiangxiangxing daqu under different thermophilic temperatures[J].Int J Food Sci Tech,2022,57(8):5102-5113.
[61]张武斌.清香大曲糖化酶的提取及宏蛋白质组学分析[D].太原:山西师范大学,2014.
[62]张武斌,张秀红,段江燕.汾酒大曲宏蛋白质组的制备与双向电泳技术的建立[J].生物学杂志,2013,30(6):50-53.
[63]WANG B W,WU Q,XU Y,et al.Synergistic effect of multiple saccharifying enzymes on alcoholic fermentation for Chinese Baijiu production[J].Appl Environ Microbiol,2020,86(8):e00013-20.
[64] JIA W, LI Y L, DU A, et al.Foodomics analysis of natural aging and gamma irradiation maturation in Chinese distilled Baijiu by UPLC-Orbitrap-MS/MS[J].Food Chem,2020,315:126308.
[65]杨辉,刘丽丽,荆雄,等.基于衍生化GC-MS代谢组学方法分析凤香型年份基酒的差异[J].现代食品科技,2021,37(11):295-303.
[66] YANG L, FAN W L, XU Y.GC×GC-TOF/MS and UPLC-Q-TOF/MS based untargeted metabolomics coupled with physicochemical properties to reveal the characteristics of different type daqus for making soy sauce aroma and flavor type Baijiu[J].LWT-Food Sci Technol,2021,146:111416.
[67]LIU Y F,WAN B,YANG F,et al.Metabolomics-Driven elucidation of interactions between Saccharomyces cerevisiae and Lactobacillus panis from Chinese Baijiu fermentation microbiome[J].Fermentation,2022,8(1):33.
[68]LUO S,ZHANG Q L,YANG F,et al.Analysis of the formation of sauceflavored Daqu using non-targeted metabolomics[J].Front Microbiol,2022,13:857966.
[69]WANG X L,SONG X B,ZHU L,et al.Unraveling the acetals as ageing markers of Chinese Highland Qingke Baijiu using comprehensive twodimensional gas chromatography-time-of-flight mass spectrometry combined with metabolomics approach[J].Food Qual Saf,2021,5:1-8.
[70]FANG C,DU H,JIA W,et al.Compositional differences and similarities between typical Chinese Baijiu and western liquor as revealed by mass spectrometry-based metabolomics[J].Metabolites,2018,9(1):2.
[71]SONG X B,JING S,ZHU L,et al.Untargeted and targeted metabolomics strategy for the classification of strong aroma-type Baijiu(liquor)according to geographical origin using comprehensive two-dimensional gas chromatography-time-of-flight mass spectrometry[J].Food Chem,2020,314:126098.
[72] XI H, JELEN′ H H.Comprehensive two-dimensional gas chromatography-time of flight mass spectrometry(GC×GC-TOFMS)in conventional and reversed column configuration for the investigation of Baijiu aroma types and regional origin[J].J Chromatogr A,2021,1636:461774.
[73]XI H,HUANG Y M,GORSKA-HORCZYCZAK E,et al.Rapid analysis of Baijiu volatile compounds fingerprint for their aroma and regional origin authenticity assessment[J].Food Chem,2021,337:128002.
[74]HE Y X,LIU Z P,QIAN M,et al.Unraveling the chemosensory characteristics of strong-aroma type Baijiu from different regions using comprehensive two-dimensional gas chromatography-time-of-flight mass spectrometry and descriptive sensory analysis[J].Food Chem,2020,331:127335.
[75]HU X L,WANG K L,CHEN M E,et al.Profiling the composition and metabolic activities of microbial community in fermented grain for the Chinese strong-flavor Baijiu production by using the metatranscriptome,high-throughput 16S rRNA and ITS gene sequencings[J].Food Res Int,2020,138(12):109765.
[76]XU Y Q,WU M Q,NIU J L,et al.Characteristics and correlation of the microbial communities and flavor compounds during the first three rounds of fermentation in Chinese sauce-flavor Baijiu[J].Foods, 2023,12(1):207.
[77]SHI W,CHAI L J,FANG G Y,et al.Spatial heterogeneity of the microbiome and metabolome profiles of high-temperature Daqu in the same workshop[J].FoodResInt,2022,156(6):111298.
[78]ZHAO C,SU W,MU Y C,et al.Correlations between microbiota with physicochemical properties and volatile flavor components in black glutinous rice wine fermentation[J].Food Res Int,2020,138:109800.
[79]XIAO R,CHEN S Q,WANG X Q,et al.Microbial community starters affect the profiles of volatile compounds in traditional Chinese Xiaoqu rice wine:Assement via high-throughput sequencing and gas chromatography-ion mobility spectrometry[J].LWT-Food Sci Technol,2022,170:114000.
[80] HU X L, TIAN R J, WANG K L, et al.The prokaryotic community,physicochemical properties and flavors dynamics and their correlations in fermented grains for Chinese strong-flavor Baijiu production[J].Food Res Int,2021,148:110626.
[81]YAN S B,JIA Y L,ZHANG Q,et al.Bacterial diversity associated with volatile compound accumulation in pit mud of Chinese strong-flavor Baijiu pit[J].AMB Express,2023,13(1):1-13.
[82] MU Y, HUANG J, ZHOU R Q, et al.Comprehensive analysis for the bioturbation effect of space mutation and biofortification on strong-flavor Daqu by high-throughput sequencing,volatile analysis and metabolomics[J].Food Chem,2023,403:134440.
[83]ZHANG Q M,DU G C,CHEN J,et al.Systematic analysis of Baobaoqu fermentation starter for Wuliangye Baijiu by the combination of metagenomics and metabolomics[J].Front Microbiol,2022,13:1062547.
[84] HU Y L, YANG Q, CHEN D, et al.Study on microbial communities and higher alcohol formations in the fermentation of Chinese Xiaoqu Baijiu produced by traditional and new mechanical technologies[J].Food Res Int,2021,140(2):109876.
[85] TAN Y W, DU H, ZHANG H X, et al.Geographically associated fungus-bacterium interactions contribute to the formation of geography-dependent flavor during high-complexity spontaneous fermentation[J].Microbiol Spectrum,2022,10(5):e01844-22.
[86] DU R B, WU Q, XU Y.Chinese liquor fermentation: identification of key flavor-producing Lactobacillus spp.by quantitative profiling with indigenous internal standards[J].Appl Environ Microbiol,2020,86(12):e00456-20.
[87] WANG B W, WU Q, XU Y, et al.Specific volumetric weight-driven shift in microbiota compositions with saccharifying activity change in starter for Chinese Baijiu fermentation[J].Front Microbiol, 2018, 9:2349.