魔芋(Amorphophallus konjac)为天南星科(Araceae)魔芋属(Amorphophallus B1.ex Decne.)多年生草本植物的块茎,是目前自然界唯一能大量提供葡甘聚糖的重要经济作物[1]。葡甘聚糖是一种高分子优质膳食纤维,具有凝胶性、增稠性、持水性和成膜性等独特的性质,被广泛应用于生物医学、食品和化工等领域[2-3]。虽然葡甘聚糖得到了广泛的应用,但由于其相对分子质量较大、黏度大,因此在食品工业的应用上受到了限制[4]。因此将魔芋中的大分子降解为小分子,即魔芋低聚糖,不仅可以大大降低其黏度,还可以提高其在食品中的应用范围[5]。据报道,魔芋低聚糖具有促进有益微生物特别是耐氧双歧杆菌生长[6-7]、提高机体抗氧化能力[8]、降血脂及护肝作用[9-10]、排毒与增强免疫功能的作用[11]。
魔芋低聚糖是一系列葡甘露低聚糖的混合物,是由β-甘露聚糖酶水解甘露聚糖中的β-1,4-D-甘露糖苷键得到的产物[12]。β-甘露聚糖酶的活性决定了魔芋低聚糖的水解程度[13]。目前,β-甘露聚糖酶主要来源于微生物[14-15],细菌来源的常见于芽孢杆菌(Bacillus)、假单胞菌(Pseudomonas)及产酸细菌等[16-18]。虽然β-甘露聚糖酶来源广泛、数量众多,但由于饲料固体发酵中发酵温度不易控制和底物复杂性的影响,β-甘露聚糖酶的应用受到了限制[19-20]。因此,获得底物利用广泛,温度适应区间大,活性高的β-甘露聚糖酶是解决饲料发酵中的重点[21-22]。本实验拟以昆明、宁德和南阳的多年种植魔芋区域的根际土壤为研究对象,通过Illumina MiSeq高通量测序技术对3地区魔芋根际土壤中细菌种类和群落结构多样性进行分析,旨在获得魔芋根际土壤中更加准确、全面的细菌多样性信息,为后续β-甘露聚糖酶的筛选及开发利用奠定研究基础。
1 材料与方法
1.1 材料与试剂
1.1.1 材料
魔芋根际土壤:分别采自云南昆明、福建宁德、河南南阳。
1.1.2 化学试剂
E.Z.N.A.R Soil 脱氧核糖核酸(deoxyribonucleic acid,DNA)Kit:美国Omega Bio-Tek公司;琼脂糖:西班牙Biowest公司;FastPfu 聚合酶:北京全式金生物技术(TransGen Biotech)有限公司;AxyPrep DNA Gel Extraction Kit:美国Axygen公司;NEXTFLEXR Rapid DNA-Seq Kit:美国Bioo Scientific公司;MiSeq Reagent Kit v3:美国llumina公司。
1.2 仪器与设备
N13462C移液器、5430R小型离心机、5424R高速台式冷冻离心机:德国Eppendorf公司;NanoDrop2000超微量分光光度计:美国Thermo Fisher Scientific公司;BioTek ELx800酶标仪:美国Biotek公司;QuantiFluorTM-ST微型荧光计:美国Promega公司;ABI GeneAmpR 9700型聚合酶链式反应(polymerase chain reaction,PCR)仪:美国ABI公司;Illumina Miseq测序仪:美国Illumina公司。
1.3 方法
1.3.1 样品采集
试验样品采自云南昆明、福建宁德、河南南阳的魔芋根际土壤,分别编号为ND(种植时间3年,海拔1 992 m,东经118°88′,北纬27°08′)、KM(种植时间1年,海拔996 m,东经103°47′、北纬25°40′)、NY(种植时间半年,海拔949 m,东经111°15′,北纬33°38′)。采用随机取样法,在魔芋种植区域随机选取3个样方(1 m×1 m),每个样方中随机选取3个魔芋植株。取样时铲去表层土壤(0~5 cm),取5~20 cm附着于魔芋根际及块茎上的土壤装入无菌自封袋后充分混匀,将3个取样点采集的土壤混合为一个样品。每个取样地点采集样品3个,-20 ℃保存,备用。
1.3.2 样品总DNA提取和PCR扩增及高通量测序
采用磁珠法土壤和粪便基因组DNA提取试剂盒提取DNA,使用NanoDrop ND-2000分光光度计和0.8%琼脂糖凝胶电泳检测合格后,送至上海美吉生物医药科技有限公司进行PCR扩增和高通量测序。选择细菌16S rRNA通用引物338F和806R对细菌16S rRNA序列的V3~V4可变区进行扩增。引物序列为338F:5'-ACTCCTACGGGAGGCAGCAG-3',806R:5'-GGACTACHVGGGTWTCTAAT-3'。PCR扩增体系:4 μL 5×FastPfu Buffer,0.4 μL FastPfu聚合酶,0.2 μL 牛血清蛋白(bovine serum albumin,BSA),上下游引物(5 μmol/L)各0.8 μL,2 μL 脱氧核糖核苷三磷酸(deoxyribonucleoside triphosphates,dNTPs)(2.5 mmol/L),1 μL DNA模板(10 ng/μL),双蒸水(ddH2O)补至20 μL。PCR扩增条件:95 ℃、3 min;95 ℃、30 s;55 ℃、30 s;72 ℃、45 s,30个循环;72 ℃、10 min;4 ℃保存。PCR扩增产物经2%琼脂糖凝胶检测合格,纯化后构建PE文库,使用Illumina公司的Miseq台式测序仪进行高通量测序。
1.3.3 高通量测序数据处理与分析
原始测序序列使用Trimmomatic软件进行质控后,采用Flash软件进行拼接。过滤质控后50 bp以下的reads,去除含N碱基的reads,根据PE reads之间的overlap关系,将成对reads拼接(merge)成一条序列。结合序列首尾两端的barcode和引物区分样品,调整序列的方向,得到有效序列。使用Uchime软件对拼接好的序列剔除嵌合体,采用Uparse软件按照97%的相似度对有效序列聚成不同的操作分类单元(operational taxonomic units,OTU)。通过Mothur和Qiime软件计算不同地区魔芋根际土壤中细菌基于OTU分类水平的生物多样性指数,主要包括Ace指数、Chao1指数、Shannon指数以及Simpson指数,用以探索不同地区魔芋根际土壤样本间细菌群落组成的相似性和差异性。
1.3.4 魔芋根际土壤理化性质的测定
魔芋根际土壤pH、有机质、速效磷、速效钾、碱解氮:参照《土壤农化分析》中的方法测定[23]。
1.3.5 数据处理
实验数据的处理采用Excel 2016,显著性差异分析采用SPSS 20.0。
2 结果与分析
2.1 不同地区魔芋根际土壤理化性质分析
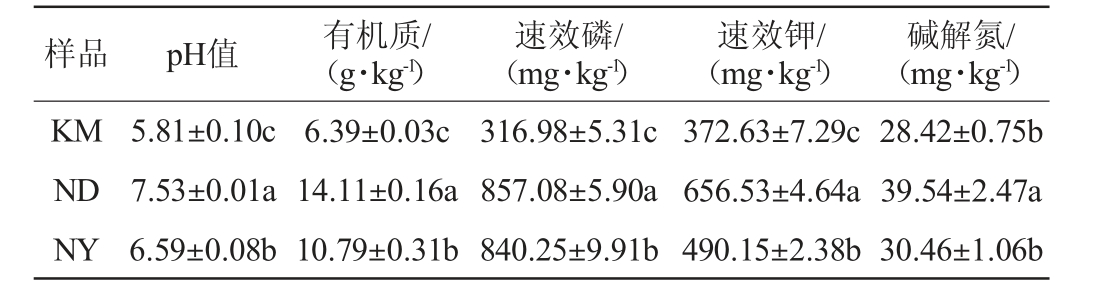
昆明(KM)、宁德(ND)和南阳(NY)魔芋根际土壤样品的pH、有机质、速效磷、速效钾和碱解氮含量测定结果见表1。
表1 不同地区魔芋根际土壤理化性质
Table 1 Physicochemical properties of konjac rhizosphere soil samples from different regions
注:同列不同小写字母代表差异显著(P<0.05)。
样品 pH值 有机质/(g·kg-1)速效磷/(mg·kg-1)速效钾/(mg·kg-1)碱解氮/(mg·kg-1)KM ND NY 5.81±0.10c 7.53±0.01a 6.59±0.08b 6.39±0.03c 14.11±0.16a 10.79±0.31b 316.98±5.31c 857.08±5.90a 840.25±9.91b 372.63±7.29c 656.53±4.64a 490.15±2.38b 28.42±0.75b 39.54±2.47a 30.46±1.06b
由表1可知,宁德(ND)魔芋根际土壤的pH、有机质、速效磷和速效钾显著高于昆明(KM)和南阳(NY)(P<0.05);碱解氮指标宁德(ND)与昆明(KM)、南阳(NY)差异显著(P<0.05),南阳(NY)与昆明(KM)差异不显著(P>0.05)。表明不同地区魔芋根际土壤的理化性质存在明显的差异。
2.2 不同地区魔芋根际土壤细菌群落结构和多样性分析
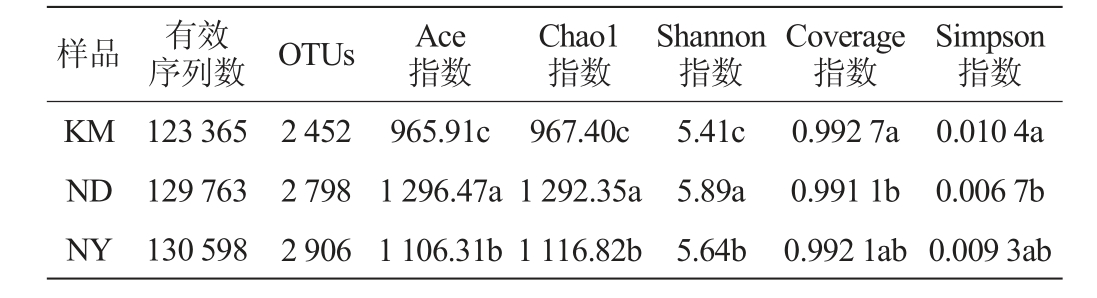
基于OTU分类水平,对3处魔芋根际土壤样品进行多样性指数分析。通过对KM、ND、NY 3处魔芋根际土壤样品中细菌进行高通量测序,结果见表2。
表2 不同地区魔芋根际土壤样品中细菌菌群多样性分析结果
Table 2 Analysis results of bacterial community diversity of konjac rhizosphere soil samples from different regions
注:同列不同小写字母代表差异显著(P<0.05)。
样品 有效序列数 OTUs Ace指数Chao1指数Shannon指数Coverage指数Simpson指数KM ND NY 0.010 4a 0.006 7b 0.009 3ab 123 365 129 763 130 598 2 452 2 798 2 906 965.91c 1 296.47a 1 106.31b 967.40c 1 292.35a 1 116.82b 5.41c 5.89a 5.64b 0.992 7a 0.991 1b 0.992 1ab
由表2可知,KM、ND、NY样品得到有效序列数分别为123 365、129 763、130 598。在97%相似度水平下划分OTU,3处样品共得到8 156个OTU。Chao1指数和Ace指数常用来表述群落丰富度,其数值越大表明群落物种丰富度越高。Shannon指数和Simpson指数用来表述群落多样性,Shannon指数越大,Simpson指数越小,表明群落物种的多样性越高。由表2可知,ND样品的Chao1指数(1292.35)、Ace指数(1296.47)均显著高于KM和NY(P<0.05),说明其土壤中细菌群落物种丰富度最高;ND样品的Shannon指数(5.89)显著高于KM和NY(P<0.05),Simpson指数(0.006 7)显著低于KM(P<0.05),表明其土壤中细菌群落多样性最高。
选取OTU序列,绘制Venn图见图1。
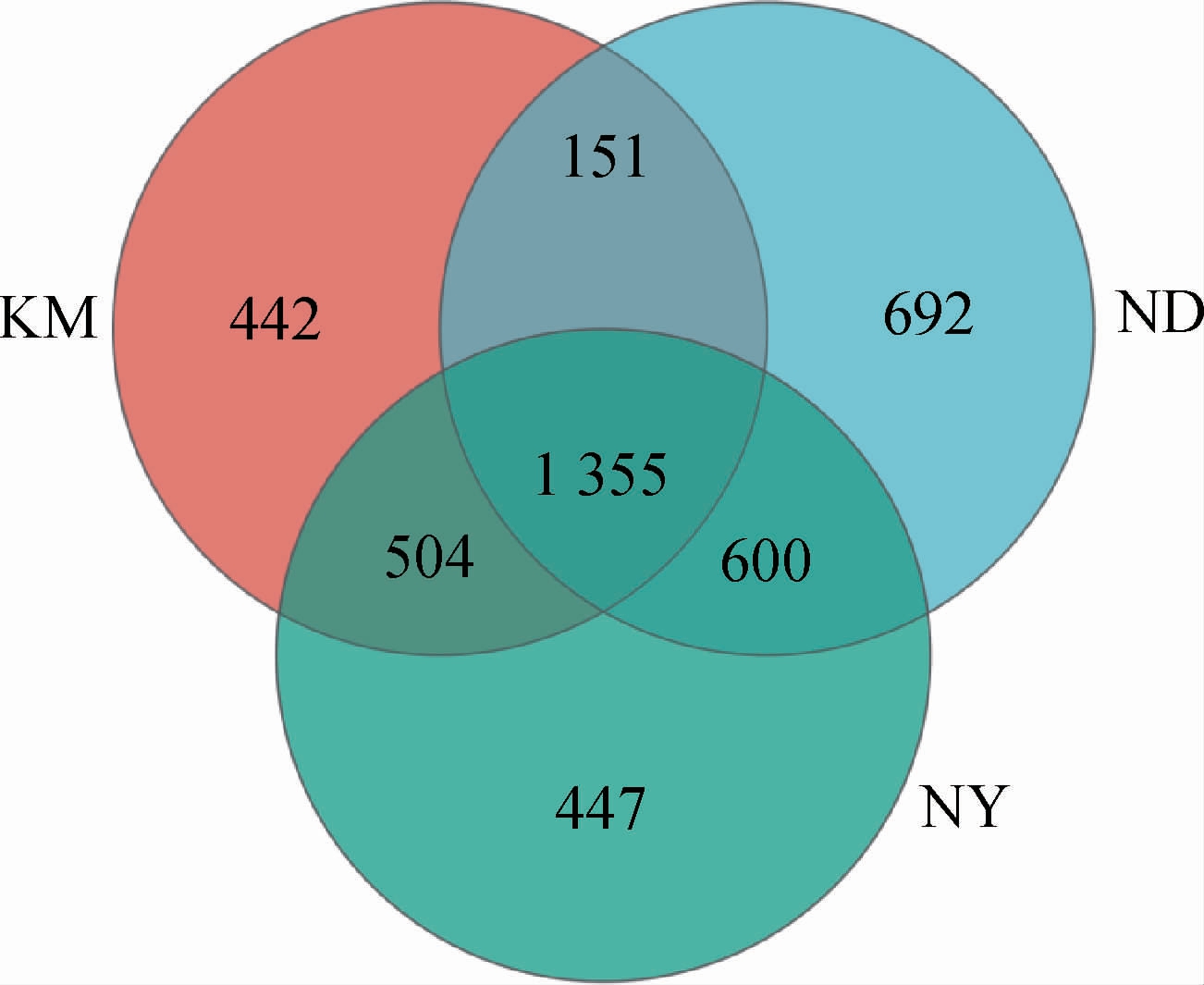
图1 不同地区魔芋根际土壤样品中细菌OTU分布Venn图
Fig.1 Venn diagram of bacterial OTU of konjac rhizosphere soil samples from different regions
由图1可知,KM、ND、NY 3处地点的土壤样品共有的OTU数量为1355个,特有的OTU数量分别为442、692、447个,其余OTU为不同地区间共有。3处地点的土壤样品测序的覆盖率均>99%,表明试验的取样合理,测序数据较好,能真实反映样本的实际情况,可进行后续数据分析。
基于土壤样品中细菌的OTU得到Sobs稀释曲线见图2。由图2可知,随着抽样数的增加,3处地点的土壤样品9个土壤样品的Sobs指数没有趋于平缓,表明继续加大测序量可能会发现更多OTU。

图2 不同地区魔芋根际土壤样品Sobs稀释曲线
Fig.2 Sobs rarefaction curves of konjac rhizosphere soil samples from different regions
Shannon-Wiener曲线是反映样品中微生物多样性的指数,利用各样品的测序量在不同深度时的微生物多样性指数构建曲线,以此反映各样品在不同测序数量时的微生物多样性。当曲线趋向平缓时,说明测序数据量足够大,可以准确反映样品中绝大多数的微生物信息。以KM、ND和NY 3处地点的9个土壤样品的香农指数和序列数绘制Shannon-Wiener曲线见图3。由图3可知,随着测序序列数的增加,3处地点的9个土壤样品的香农指数趋于平缓,表明测序数据量足够大,可以准确反映样品中微生物群落的多样性。
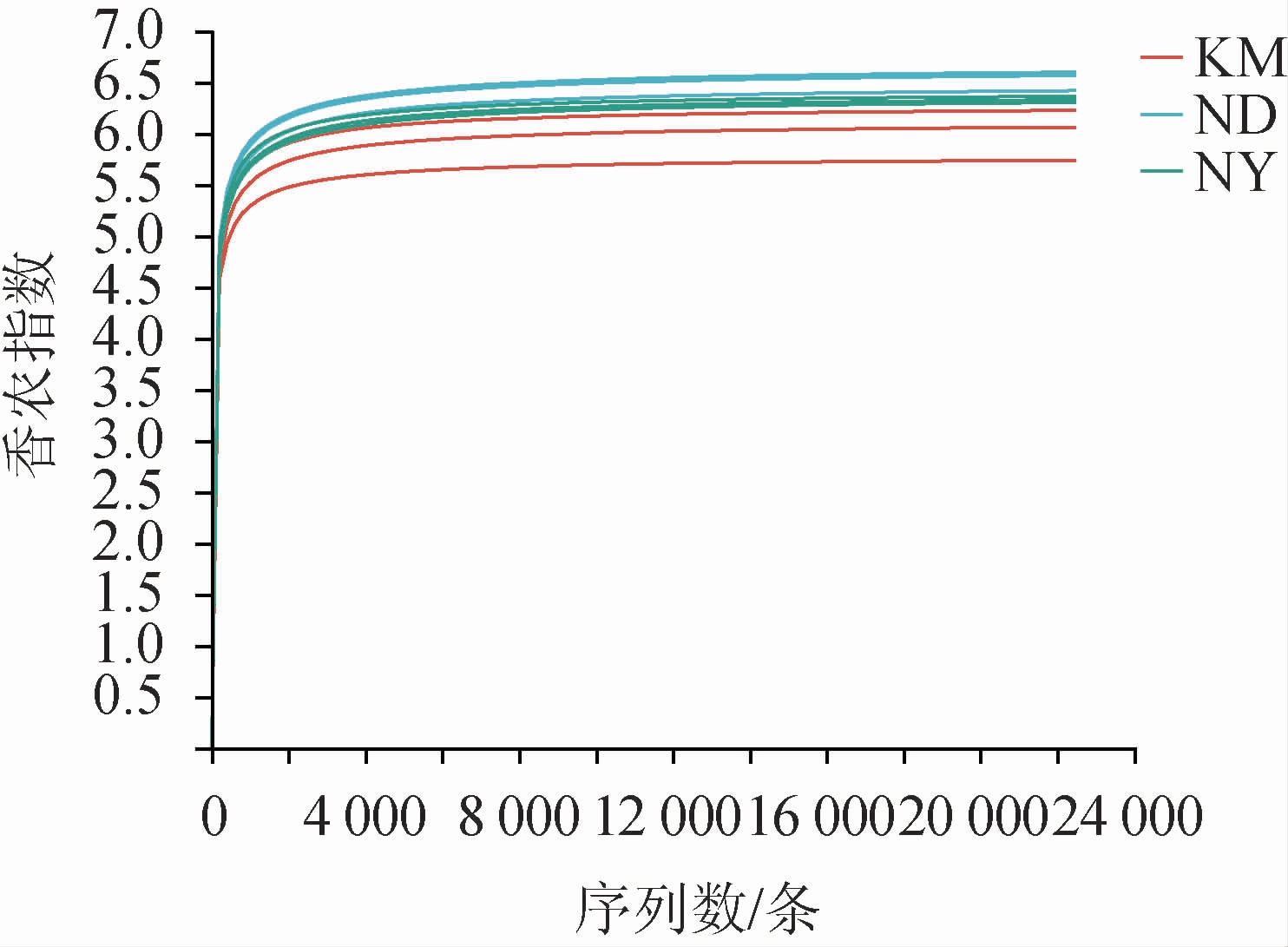
图3 不同地区魔芋根际土壤样品Shannon-Wiener曲线
Fig.3 Shannon-Wiener curves of konjac rhizosphere soil samples from different regions
2.3 属水平上不同地区魔芋根际土壤细菌的种类和相对丰度分析
在属水平上,基于高通量测序技术对不同地区的魔芋根际土壤中细菌的种类和相对丰度进行分析。根据相对丰度将样本中的菌属分为优势菌属(相对丰度≥1.0%)和次要菌属(相对丰度<1.0%),且将次要菌属归类于其他(others),结果见图4。
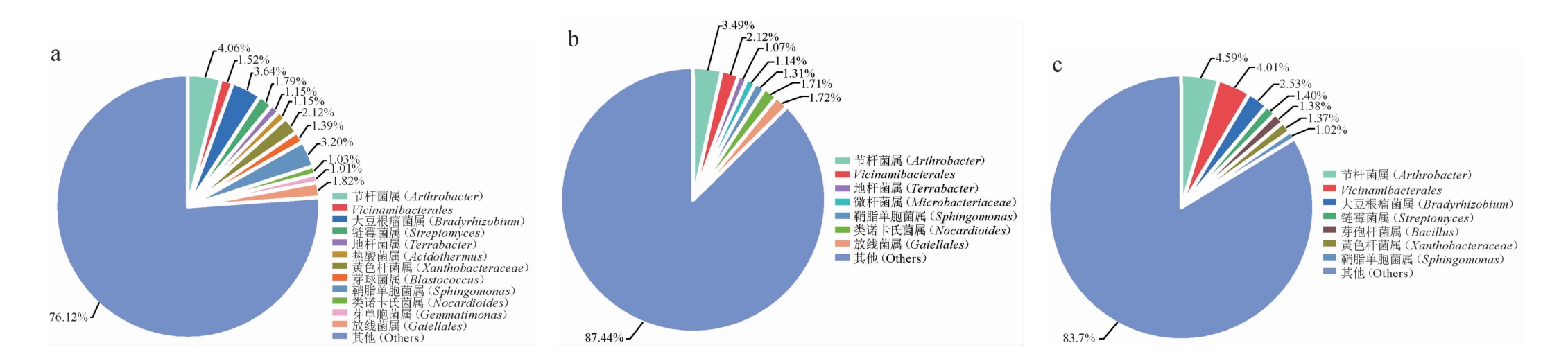
图4 基于属水平KM(a)、ND(b)及NY(c)魔芋根际土壤样品中细菌种类及相对丰度
Fig.4 Species and relative abundance of bacteria in KM (a), ND (b) and NY(c)konjac rhizosphere soil samples based on genus level
由图4a可知,KM样本中优势菌属有12个,其中节杆菌属(Arthrobacter)相对丰度最高,为4.06%;其次为大豆根瘤菌属(Bradyrhizobium),相对丰度为3.64%;其余依次为鞘脂单胞菌属(Sphingomonas)(3.20%)、黄色杆菌属(Xanthobacter)(2.12%)、放线菌属(Gaiellales)(1.82%)、链霉菌属(Streptomyces)(1.79%)、Vicinamibacterales(1.52%)、芽球菌属(Blastococcus)(1.39%)、热酸菌属(Acidothermus)(1.15%)、地杆菌属(Terrabacter)(1.15%)、类诺卡氏菌属(Nocardioides)(1.03%)、芽单胞菌属(Gemmatimonas)(1.01%)。
由图4b可知,ND样本中优势菌属有7个,节杆菌属(Arthrobacter)相对丰度最高,为3.49%;其余依次为Vicinamibacterales(2.12%)、放线菌属(Gaiellales)(1.72%)、类诺卡氏菌属(Nocardioides)(1.71%)、鞘脂单胞菌属(Sphingomonas)(1.31%)、微杆菌属(Microbacterium)(1.14%)、地杆菌属(Terrabacter)(1.07%)。
由图4c可知,NY样本中优势菌属有7个,其中节杆菌属(Arthrobacter)相对丰度最高,为4.59%;其余依次为Vicinamibacterales(4.01%)、大豆根瘤菌属(Bradyrhizobium)(2.53%)、链霉菌属(Streptomyces)(1.40%)、芽孢杆菌属(Bacillus)(1.38%)、黄色杆菌属(Xanthobacter)(1.37%)、鞘脂单胞菌属(Sphingomonas)(1.02%)。
对比昆明(KM)、宁德(ND)、南阳(NY)3个地区魔芋根际土壤细菌优势菌属的种类,发现不同地区之间存在种类上的差异。节杆菌属(Arthrobacter)、Vicinamibacterales、鞘脂单胞菌属(Sphingomonas)为3个地区所共有,热酸菌属(Acidothermus)和芽单胞菌属(Gemmatimonas)为昆明样品独有;微杆菌属(Microbacterium)为宁德样品特有;芽孢杆菌属(Bacillus)则只在南阳样品中发现。表明不同地区魔芋根际土壤细菌优势菌属种类存在差异,这与彭玉娇等[24]的研究结果一致。
3 结论
本研究运用Illumina MiSeq高通量测序技术对昆明、宁德和南阳魔芋根际土壤中细菌的群落结构和多样性进分析比较,结果表明,昆明、宁德和南阳地区魔芋根际土壤的细菌丰富度和物种多样性存在显著性差异(P<0.05),其中昆明样品中优势菌属有12个,热酸菌属(Acidothermus)和芽单胞菌属(Gemmatimonas)为独有;宁德样品中优势菌属有7个,微杆菌属(Microbacterium)为其特有;南阳样品中优势菌属有8个,芽孢杆菌属(Bacillus)为其独有。
[1]邓慧君.白魔芋实生种子休眠特性研究[D].重庆:西南大学,2018.
[2]DEVARAJ R D,REDDY C K,XU B.Health-promoting effects of konjac glucomannan and its practical applications: A critical review[J].Int J Biozogical Macromol,2019,126:273-281.
[3]何斐,冯明月,王箐轩,等.花魔芋响应软腐病菌侵染初期的转录组分析[J].西北农业学报,2022,31(6):777-786.
[4]周中凯,赵亚丽,杨星月.魔芋低聚糖对高脂饮食小鼠脂代谢的影响[J].食品科学,2019,40(1):149-154.
[5]郭金玲,张丹阳,樊雪莲,等.β-甘露聚糖酶产生菌的筛选及魔芋低聚糖制备工艺的研究[J].中国酿造,2018,37(8):123-127.
[6]吴拥军,孟望霓,蔡金藤,等.魔芋葡甘露低聚糖的提取及其发酵产物对耐氧双歧杆菌的促生长作用[J].食品工业科技,2002,23(9):41-43.
[7]李勇超,刘瑞雪,李亚琦,等.添加魔芋低聚糖对益生菌增殖和酸奶品质的影响[J].中国乳品工业,2016,44(5):62-64.
[8]杨艳燕,李小明,李顺意,等.魔芋低聚糖对小鼠血糖含量和抗氧化能力的影响[J].中草药,2001,32(2):142-145.
[9]陈黎,杨艳燕,闫达中.魔芋低聚糖降脂作用的初步研究[J].中国生化药物杂志,2002,23(4):181-182.
[10]焦丹,李浩霞,吴丹丹,等.魔芋低聚糖缓解高糖水平诱发的大鼠代谢综合征及相关机制[J].食品科学,2019,40(13):137-142.
[11]王苗,蒋敏,李恒,等.乳酸菌对魔芋甘露聚糖的降解与利用[J].食品与发酵工业,2016,42(11):20-24.
[12]冯莉,刘瑞雪,张波.魔芋低聚糖作为新食品原料的应用研究[J].中国食品添加剂,2014(7):153-157.
[13]李剑芳,邬敏辰,程科,等.β-甘露聚糖酶制备魔芋葡甘露低聚糖的研究[J].食品与发酵工业,2007,33(1):21-24.
[14]季海蕊,张鑫,姜静,等.乳酸菌β-甘露聚糖酶研究进展[J].食品工业科技,2021,42(4):325-329.
[15]甄红敏,华晓晗,马俊文,等.产黄青霉β-甘露聚糖酶的高效表达、性质及应用[J].食品科学,2021,42(8):98-105.
[16]杨苗.β-甘露聚糖酶产生菌的筛选鉴定、产酶条件优化及酶学特性研究[D].武汉:湖北工业大学,2016.
[17]MEENAKSHI,SING G,BHALLA A,et al.Solid state fermentation and characterization of partially purified thermostable from Bacillus sp.MG-33[J].Bioresources,2010,5(3):1689-1701.
[18] TITAPOKA S, KEAWSOMPONG S, HALTRICH D, et al.Selection and characterization of mannanase-producing bacteria useful for the formation of prebiotic manno-oligosaccharides from copra meal[J].World J Microbiol Biotechn,2008,24(8):1425-1433.
[19]雷高,李珊珊,王佰涛,等.产β-甘露聚糖酶菌株的筛选鉴定及产酶条件优化[J].中国酿造,2022,41(4):99-104.
[20]郭尚旭,王瑶,那金,等.细菌β-甘露聚糖酶研究进展[J].中国农学通报,2017,33(27):61-65.
[21]李珊珊,刘德海,刁文涛,等.黑曲霉β-甘露聚糖酶ManA耐热突变体的筛选[J].中国酿造,2022,41(3):62-68.
[22]田庚,高伟强,陈晓波,等.热稳定性高β-甘露聚糖酶产生菌的筛选、鉴定及酶学性质研究[J].食品工业科技,2020,41(19):127-131.
[23]鲍士旦.土壤农化分析[M].北京:中国农业出版社,2000:24-114.
[24]彭玉娇,崔学宇,覃礼蒙,等.不同地区沙田柚果园土壤细菌群落结构和多样性分析[J].四川农业大学学报,2020,38(6):715-722.
[25]罗长财,缪静,李国莹,等.一株产耐高温β-甘露聚糖酶菌株的筛选鉴定[J].生物技术通讯,2018,29(2):233-237.