我国地域广阔,涵盖了多种气候类型,因此能够种植许多蔬菜。但是绝大多数蔬菜如辣椒等不耐储存。而经过微生物发酵或者腌制加工成发酵蔬菜(如鲊广椒、泡菜、酸菜等),就能存放较长时间,且微生物发酵能改善蔬菜的滋味、气味和质构等特征[1]。鲊广椒就是这类传统发酵食品的代表,其在我国多个省区,如四川、湖北、重庆、贵州和湖南等地均有制作,且是日常的调味佐料,能用来制作鲊广椒排骨、鲊广椒土豆片、鲊广椒炒腊肉等常见菜肴。
鲊广椒制作技艺已经成为恩施土家族苗族自治州的州级非遗代表项目,其制作原料一般使用鲜红辣椒和玉米粉或者大米粉,再添加大蒜、食盐、白酒等密封发酵数月即可食用。不同地区的工艺或者原料,略有差异,但是差别不大。研究显示,由于鲊广椒采用自然发酵方式,未添加任何发酵剂,且制作过程中的原料和容器均未消毒处理,因此发酵过程中的菌群主要来自原料和制作环境,且由于不同地区原料和制作环境中微生物的不同而使鲊广椒成品品质具有差异。既往研究显示,鲊广椒中含有复杂的微生物菌群,且主要是细菌中的乳酸菌。按照最新的乳酸菌科分类单元,鲊广椒中常见优势菌属包括:伴生乳杆菌属(Companilactobacillus),乳植物杆菌属(Lactiplantibacillus),乳杆菌属(Lactobacillus),魏斯氏菌属(Weissella),明串珠菌属(Leuconostoc),片球菌属(Pediococcus)和促生乳杆菌属(Levilactobacillus)[2-4],一些鲊广椒中还含有醋酸杆菌属(Acetobacter),棒状杆菌属(Corynebacterium)和假单胞菌属(Pseudomonas)[2,4]。
高通量测序技术具有准确率高,成本较低,且测序周期短的优点,因此在食品、医学、环境等许多研究领域有了广泛的应用。尽管通过高通量测序技术无法得到微生物菌株,但其不依赖于培养基和培养条件,因此对微生物菌群的解析结果不受培养基、培养条件等因素限制,得到的结果更加客观[5]。恩施土家族苗族自治州,是湖北喀斯特地貌的主要分布区域,地理环境较为特别,因此该地区的鲊广椒中可能含有一些特别的微生物类群。前期基于传统培养方法得出,恩施鲊广椒富含乳汁乳杆菌属和粘液乳杆菌属(Limosilactobacillus)[6]。鉴于传统培养方法的局限性以及样品收集数量较少,因此,为进一步明确恩施州的鲊广椒微生物组成,本研究通过第二代高通量测序对鲊广椒中的微生物多样性进行测序与生物信息学分析,并进行功能预测,以期明确恩施鲊广椒样品中特有及核心微生物菌群,为鲊广椒产业提供基础数据支撑。
1 材料与方法
1.1 材料与试剂
鲊广椒样品:分别采集自湖北省恩施土家族苗族自治州利川市,共采集14个样品,分别编号为LC01~LC14,样品采集地的经纬度分别为:108.35°~109.30°E,29.70°~30.65°N;另外,还在恩施州恩施市采集了26个样品,编号为ES01~ES26,样品采集的区域为:109.08°~109.98°E,29.84°~30.66°N。另外,为方便进行组间分析,分别将来自利川市与恩施市的样品编号为LC和ES。
用于聚合酶链式反应(polymerase chain reaction,PCR)扩增的引物对:338F(5'-ACT CCT ACG GGA GGC AGC A-3')和806R(5'-GGA CTA CHV GGG TWT CTAAT-3'),由上海桑尼生物科技有限公司合成;脱氧核糖核苷三磷酸(deoxy-ribonucleoside triphosphates,dNTPs)Mix、Ex PremierTM脱氧核糖核酸(deoxyribonucleic acid,DNA)聚合酶:宝生物工程大连有限公司;E.Z.N.A.Rfood DNA试剂盒:美国Omega Bio-Tek公司。
1.2 仪器与设备
EDC-810型黑金刚基因扩增仪:北京东胜创新生物科技有限公司;WD-9413B型凝胶成像分析系统、DYY-6C型电泳仪电源:北京六一生物科技有限公司;K5800H自动检测超微量分光光度计:北京凯奥科技发展有限公司。
1.3 方法
1.3.1 样品采集
鲊广椒样品采集自湖北省恩施土家族苗族自治州。样品采集标准:无不良气味与滋味,且不同编号的鲊广椒均通过自然发酵方式制作,来自不同的家庭。样品采集后,迅速装入无菌瓶,然后装入保温箱,带回微生物实验室,放入-70 ℃冰箱备用。
1.3.2 PCR扩增与高通量测序
在进行样品总DNA提取前,将样品从冰箱中取出,使用E.Z.N.A.Rfood DNA试剂盒,按照附带的说明提取样品宏基因组DNA。提取完DNA后,使用琼脂糖凝胶电泳和微量紫外分光光度计检测提取DNA的纯度和质量。将检测合格的样品总DNA作为模板,用于细菌16S rRNA的V3-V4区域的扩增,具体的PCR扩增体系及扩增程序参照GUO Z等[7]的方法。同样的,将PCR产物使用琼脂糖凝胶电泳和微量紫外分光光度计检测后,装入干冰,送至上海美吉生物医药科技公司基于Illumina HiSeq第二代测序平台测序。
1.3.3 生物信息学分析
首先使用TrimGalore v0.6.6(https://github.com/Felix Krueger/TrimGalore)对从HiSeq测序平台得到的双端原始测序结果进行过滤,去掉低质量序列和接头。然后将产生的高质量序列提交到QIIME v1.9.1平台进行后续的生物信息学分析[8]:首先对双端序列进行合并,对于合并后的序列,先去掉嵌合体序列[9],然后使用UCLUST划分操作分类单元(operational taxonomic unit,OTU),阈值为97%[10]。基于核糖体数据库项目(ribosomal database project,RDP),使用最新版的RDP分类器对随机抽取的代表性OTU序列进行注释[11]。为比较两个地区的微生物多样性,使用QIIME平台自带脚本分别对鲊广椒样品的α与β多样性进行分析及组间比较。基于OTU表和对应的序列,通过PICRUSt2对菌群功能进行预测[12],并使用HUMAnN3对菌群代谢通路的丰度进行标准化处理。
1.3.4 统计分析
对收集到的恩施州利川市与恩施市鲊广椒样品的聚类分析使用R包Factoextra v4.2.2(https://cran.r-project.org/web/packages/factoextra)进行。组间菌群差异比较使用置换多元方差分析(permutational multivariate analysis of variance,PerMANOVA)多元方差分析法,具体使用R包pairwiseAdonis(https://github.com/pmartinezarbizu/pairwiseAdonis)完成。通过PICRUSt2的菌群功能分析,使用STAMP v2.1.3进行统计检验与作图[13]。组间门和属相对含量差异分析使用威尔科克森符号秩检验(Wilcoxon test),使用R包ggpubr完成(https://cran.r-project.org/web/packages/ggpubr/)。组间生物标志物的确定使用线性判别分析效应大小(linear discriminant analysis effect size,LEfSe),线性判别分析值(linear discriminant analysis,LDA)值设为3.0[14]。使用R软件包ggplot(https://github.com/tidyverse/ggplot2)或ggpubr进行作图。
2 结果与分析
2.1 微生物多样性分析
2.1.1 鲊广椒菌群α多样性分析
使用高通量测序技术,对恩施州不同地区来源的鲊广椒进行了分析,经过去除低质量序列,去除序列标签(barcode)和嵌合体序列后,共得到1 199 308条高质量序列。在此基础上,对恩施鲊广椒的微生物多样性进行了分析。使用97%的阈值进行OTU聚类,结果共得到12 212个OTU。接下来使用RDP分类器对每个代表性OUT序列进行了注释,共注释到22个菌门,37个菌纲,73个菌目,146个菌科以及343个菌属。分别对恩施州恩施市和利川市来源的鲊广椒α多样性进行了计算与统计分析,结果见图1。
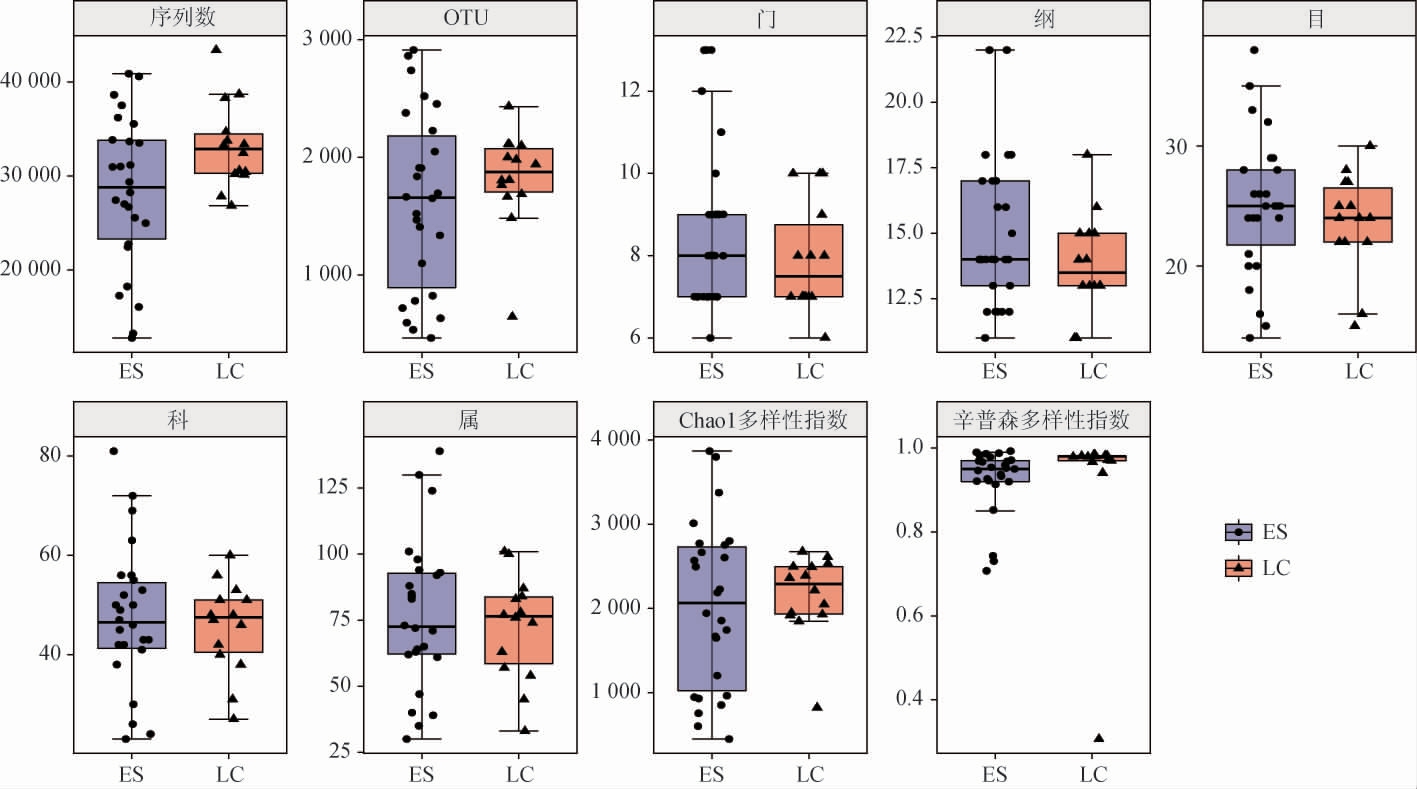
图1 恩施州不同地区鲊广椒样品序列与微生物多样性
Fig.1 Sequence number and microbial diversity of Zha-chili samples from different regions in Enshi prefecture
由图1可知,基于非参数的威尔科克森符号秩检验显示,利川市来源的鲊广椒辛普森多样性指数显著高于恩施市鲊广椒样品,而Chao1指数无显著差异(P>0.05),表明尽管利川市的各分类单元数量低于恩施市,但是利川市的微生物多样性高于恩施市,而丰富度无显著差异(P>0.05)。总体上,来自恩施市的鲊广椒各个分类单元数量多与利川市,但是无显著差异(P>0.05)。
2.1.2 鲊广椒菌群主坐标分析
比较了采集自恩施州两个地区的鲊广椒样品的β多样性,结果见图2。
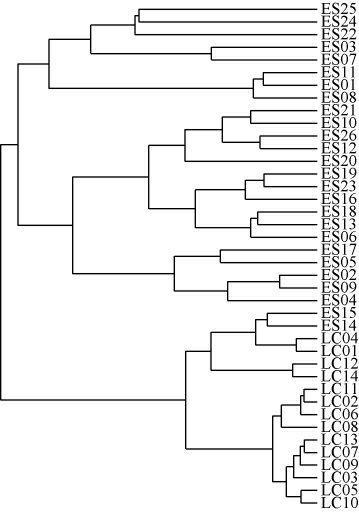
图2 恩施州不同地区鲊广椒样品聚类分析
Fig.2 Cluster analysis of Zha-chili samples from different regions in Enshi prefecture
由图2聚类分析结果表明,多数采集自恩施市的鲊广椒能聚集在一起,另外两个采集自恩施市的鲊广椒样品与利川市的鲊广椒样品聚集在一起,形成两个大的分支。另外,还通过vegan包对鲊广椒的样品进行了主坐标分析(principal co-ordinates analysis,PCoA)分析,结果见图3。
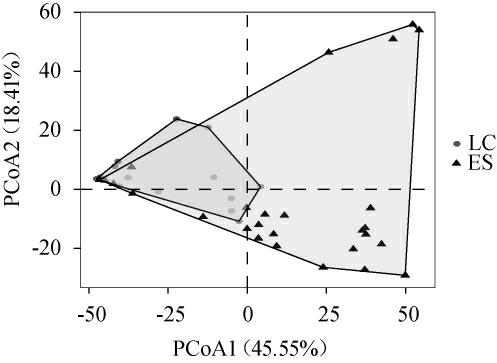
图3 恩施州不同地区鲊广椒样品β多样性分析
Fig.3 β diversity analysis Zha-chili samples from different regions in Enshi prefecture
由图3可知,基于欧式距离的PCoA1和PCoA2总体上能解释大约64%的菌群总体变量。实际上,采集自两个地区的样品,总体在PCoA1上差别较大。图3PerMANOVA分析表明,两个不同地区的鲊广椒样品菌群差异极显著(P=0.001)。另外,采集自利川市来源的鲊广椒样品之间组内差异较小,而恩施市样品组内差异较大,在PCoA1与PCoA2两个方向上分布均较为分散。
2.2 鲊广椒菌群物种组成分析
为进一步分析采集自恩施州两个地区鲊广椒的差异,从物种组成中提取出门和属的信息进行统计分析,结果见图4。本研究中,将相对含量>1%的门或属定义为优势菌门或者优势菌属。
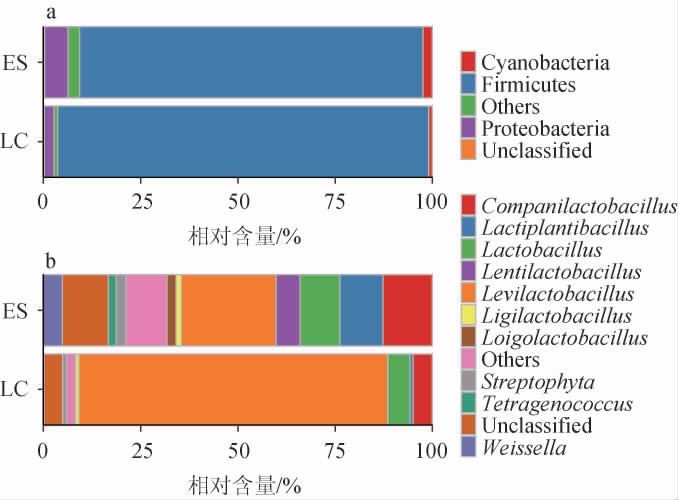
图4 基于门(a)和属(b)水平恩施州不同地区鲊广椒样品优势微生物平均相对含量
Fig.4 Average relative abundance of dominant microbe of Zha-chili samples from different regions in Enshi prefecture based on phylum(a) and genus(b) levels
由图4a可知,在门的分类水平上,恩施州鲊广椒共包含3个优势菌门,它们分别是厚壁菌门(Firmicutes),变形菌门(Proteobacteria)和蓝细菌门(Cyanobacteria)。值得注意的是,厚壁菌门在两个地区的鲊广椒中均占据绝对地位,相对含量均超过了85%。统计分析表明,恩施州两个地区的鲊广椒中含的3个优势菌门在相对含量上均无显著差异(P>0.05)。
由图4b可知,在属的分类水平上,共检测到10个优势属,分别是促生乳杆菌属(Levilactobacillus)(43.20%),伴生乳杆菌属(Companilactobacillus)(9.88%),乳杆菌属(Lactobacillus)(8.54%),乳植物杆菌属(Lactiplantibacillus)(7.38%),迟缓乳杆菌属(Lentilactobacillus)(3.97%),魏斯氏菌属(Weissella)(3.16%),链霉菌属(Streptomyces)(1.97%),肇源腐败乳杆菌属(Loigolactobacillus)(1.51%),四联球菌属(Tetragenococcus)(1.30%)和联合乳杆菌属(Ligilactobacillus)(1.20%)。总体上,乳酸菌分别在利川市和恩施市来源的鲊广椒中分别占到总的91.36%和74.07%。经过统计分析发现,有6个优势属在恩施州两个地区的鲊广椒中存在差异:采集自利川市的鲊广椒中促生乳杆菌属的相对含量显著较高(P<0.01),在利川市鲊广椒的相对含量为78.88%,在恩施市的鲊广椒中相对含量为23.99%;另外5个属,即乳植物杆菌属、魏斯氏菌属、乳杆菌属、肇源腐败乳杆菌和链球菌属在恩施市的鲊广椒中相对含量较高(P<0.05),在恩施市的鲊广椒中含量分别为:10.96%,4.79%,10.10%,2.29%和2.51%,而在利川鲊广椒中的相对含量为:0.72%,0.14%,5.63%,0.05%和0.96%。
恩施州鲊广椒的多数优势细菌属,也出现在湖南怀化和贵州松桃鲊广椒中,然而链霉菌属是恩施州鲊广椒中特有[4,15-17]。值得注意的是,链霉菌是多种生物活性代谢产物的主要来源[18-19],可能对醡广椒中有害菌具有抑制能力;同时,链霉菌能产生淀粉酶和脂肪酶,也可能对鲊广椒的风味起到重要作用。另外,鲊广椒在制作过程中,添加了食盐,而据报道,具有较强盐耐受能力的四联球菌能增强食品的风味物质含量[20-21],因此推测,四联球菌与鲊广椒的风味品质有关,而四联球菌和链霉菌与鲊广椒风味的关系,有待于进一步的实验证实。
2.3 不同地区鲊广椒菌群差异分析
为进一步分析两个地区来源鲊广椒中的生物标志物,使用LEfSe进一步基于OTU数据,对所有分类单元的差异情况进行分析,结果见图5。
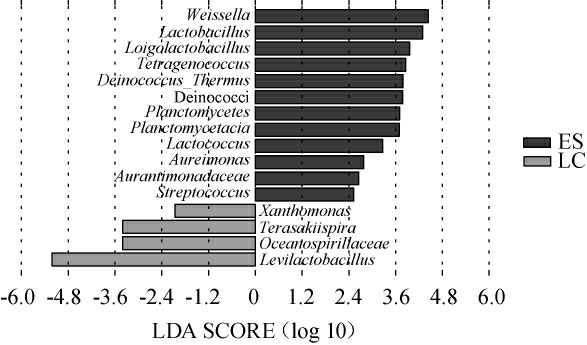
图5 基于LEfSe分析的恩施州不同地区鲊广椒菌群样品生物标志物
Fig.5 Biomarkers of Zha-chili samples flora from different regions in Enshi prefecture based on LEfSe analysis
由图5可知,除了优势属促生乳杆菌属(Levilactobacillus)之外,在利川市鲊广椒中还富集有非优势属—黄单胞菌属(Xanthomonas)和Terasakiispira。同样的,在恩施市鲊广椒中优势属魏斯氏菌属(Weissella)、乳杆菌属(Lactobacillus)、肇源腐败乳杆菌属(Loigolactobacillus)等以及3个非优势属:Aureimona,乳球菌属(Lactococcus)和链球菌属(Streptococcus)显著富集。根据这些菌属在LEfSe分析中得到的LDA值大小,可以将促生乳杆菌属(Levilactobacil lus)和魏斯氏菌属(Weissella)分别作为利川市和恩施市鲊广椒的生物标志物。另一项研究显示,促生乳杆菌与鲊广椒的特征风味酸味呈正相关,而与苦味的回味(异味)呈负相关,说明了该菌在鲊广椒的滋味中的作用。
2.4 恩施州鲊广椒优势属相关性分析
自然发酵环境中微生物菌落结构较为复杂,有些不同微生物间存在相互促进作用,另一些可能存在拮抗作用。为分析鲊广椒中微生物菌群间的相互作用关系,基于优势属相对含量,分析了它们的相关性,结果见图6。
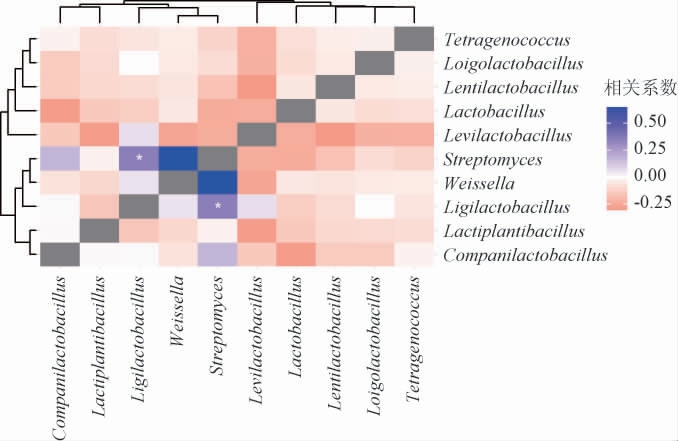
图6 恩施州不同地区鲊广椒样品基于优势菌属相对丰度的菌群相关性分析
Fig.6 Correlation analysis of bacterial community of Zha-chili samples from different regions in Enshi prefecture based on relative abundance of dominant genera
蓝色越深表明正相关性越强,红色越深表明负相关性越强,“*”表示显著相关(P<0.05)。
由图6可知,在利川市与恩施市鲊广椒的10个优势属中,仅联合乳杆菌属和链霉菌属呈显著正相关关系(P<0.05),其他菌属间相关性均不显著(P>0.05)。可能这两个属之间的微生物能合成对彼此生长有益的代谢物。研究显示,链霉菌能产生一系列的生物活性物质(如guadinomine B)以及铁载体、抗生素来抑制一些有害微生物;同时链霉素还能产生酯酶、淀粉酶和蛋白酶,将生物大分子降解成小分子化合物[22],这些小分子化合物可能对联合乳杆菌的生长有益。另外,乳酸菌可合成短链脂肪酸、氨基酸、维生素等[23],推测这些物质可能促进了链霉菌的生长,因此导致这两类微生物存在正相关关系。
2.5 菌群功能预测
使用PICRUSt2对恩施鲊广椒样品的菌群的功能进行了分析,获得了各样品基于京都基因与基因组百科全书(kyoto encyclopedia of genes and genomes,KEGG)的代谢通路丰度。然后,提取菌群代谢途径中丰度最高的15个代谢途径,进行统计分析,结果见图7。
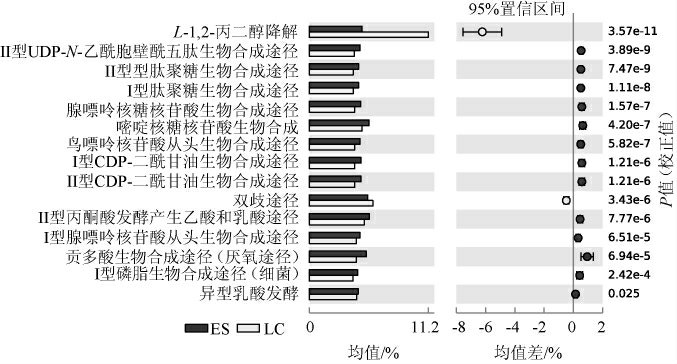
图7 恩施州不同地区鲊广椒样品菌群功能分析
Fig.7 Functional analysis of Zha-chili samples flora from different regions in Enshi prefecture
由图7可知,菌群丰度最高的15个代谢途径分别为:L-1,2-丙二醇降解途径,II型UDP-N-乙酰胞壁酰五肽生物合成途径,II型肽聚糖生物合成途径等。在这些代谢途径中,有4条代谢途径与核酸代谢有关,5条代谢途径与碳水化合物有关,还有一些与细菌细胞壁有关。微生物多样性分析显示,恩施地区鲊广椒中乳酸菌相对含量占绝对优势;同样的,菌群功能分析检测到异型乳酸发酵代谢途径,且相对丰度较高,这可能是鲊广椒中富含乳酸的原因[3]。除了异型乳酸发酵途径外,还检测到了双歧途径,该途径产物中也有乙酸和乳酸[24]。另外,在这些代谢途径中,两个地区间鲊广椒差异最大的是L-1,2-丙二醇降解途径,该途径能为微生物的生命活动提供三磷酸腺苷(adenosine triphosphate,ATP)和还原力,而一些乳酸菌也能代谢L-1,2-丙二醇[25]。在这些丰度最高的代谢途径中,有多个代谢途径与鲊广椒滋味物质合成有关,如乳酸、核苷酸和丙二醇分别与食品的酸味,鲜味和甜味有关。另外,恩施市与利川市鲊广椒的菌群具有差异,但是预测的代谢途径多数无显著差异(P>0.05),而这可以从微生物菌群中的功能冗余性来解释[26]。
3 结论
从恩施州利川市与恩施市采集的鲊广椒样品中共有优势属有促生乳杆菌属、伴生乳杆菌属、乳杆菌属等,其中的乳酸菌占据绝对优势,乳酸菌中的促生乳杆菌属的平均相对含量超过总体的40%。统计分析表明,利川市和恩施市的鲊广椒在菌群组成上具有显著差异,优势属中有6个优势属的相对含量具有显著差异(P<0.05)。基于LEfSe分析发现,伴生乳杆菌属和魏斯氏菌属在恩施市与利川市的鲊广椒中相对含量差异最大,能分别将它们作为利川市和恩施市的生物标志物,来区分这两个地区的鲊广椒。菌群功能分析发现,与核苷酸和碳水化合物代谢与脂肪代谢的途径丰度较高,而地区间多数丰度较高的代谢途径丰度无显著差异。
[1]De SOUZA E L,DE OLIVEIRA K Á R,DE OLIVEIRA M E G.Influence of lactic acid bacteria metabolites on physical and chemical food properties[J].Curr Opin Food Sci,2023,49:100981.
[2] CAI W, WANG Y, HOU Q, et al.PacBio sequencing combined with metagenomic shotgun sequencing provides insight into the microbial diversity of zha-chili[J].Food Biosci,2021,40:100884.
[3]CAI W,WANG Y,HOU Q,et al.Rice varieties affect bacterial diversity,flavor,and metabolites of zha-chili[J].Food Res Int,2021:147,110556.
[4] GUO Z,WANG Y,XIANG F,et al.Evaluating the flavor and divergent bacterial communities in corn-based zha-chili[J].Food Biosci,2022,46(3):101563.
[5]吴成,王春晓,王晓丹,等.高通量测序技术在酿酒微生物多样性研究中的应用[J].食品科学,2019,40(3):348-355.
[6]雷炎,马佳佳,雷敏,等.恩施鲊广椒乳酸菌的分离鉴定及其对挥发性风味物质的影响[J].中国酿造,2019,38(7):126-130.
[7]GUO Z,WANG Y,XIANG F,et al.Bacterial diversity in pickled Cowpea(Vigna unguiculata [Linn.] Walp) as determined by Illumina MiSeq sequencing and culture-dependent methods[J].Curr Microbiol, 2021, 78(4):1286-1297.
[8]CAPORASO J G,KUCZYNSKI J,STOMBAUGH J,et al.QIIME allows analysis of high-throughput community sequencing data[J].Nat Methods,2010,7(5):335-336.
[9]HAAS B J,GEVERS D,EARL A M,et al.Chimeric 16S rRNA sequence formation and detection in Sanger and 454-pyrosequenced PCR amplicons[J].Genome Res,2011,21(3):494-504.
[10] EDGAR R C.Search and clustering orders of magnitude faster than BLAST[J].Bioinformatics,2010,26(19):2460-2461.
[11]COLE J R,WANG Q,FISH J A,et al.Ribosomal Database Project:data and tools for high throughput rRNA analysis[J].Nucleic Acids Res,2014,42:D633-D642.
[12] DOUGLAS G M, MAFFEI V J, ZANEVELD J R, et al.PICRUSt2 for prediction of metagenome functions[J].Nat Biotechnol, 2020, 38(6):685-688.
[13]PARKS D H,TYSON G W,HUGENHOLTZ P,et al.STAMP:statistical analysis of taxonomic and functional profiles[J].Bioinformatics,2014,30(21):3123-3124.
[14] SEGATA N, IZARD J, WALDRON L, et al.Metagenomic biomarker discovery and explanation[J].Genome Biol,2011,12(6):R60.
[15]李娜,崔梦君,马佳佳,等.基于Illumina MiSeq测序和传统可培养方法的洪湖鲊广椒乳酸菌多样性研究[J].食品与发酵工业,2021,47(4):110-115.
[16]张逸舒,王玉荣,陈芸曼,等.保康鲊广椒中乳酸菌的分离鉴定及其发酵特性的评价[J].食品研究与开发,2019,40(16):159-165.
[17]向凡舒,王玉荣,葛东颖,等.湖北夷陵地区鲊广椒中乳酸菌的分离鉴定及其对产品品质的影响[J].食品科技,2019,44(4):23-29.
[18]BERNAL M G,MARRERO R M,CAMPA-CÓRDOVA Á I,et al.Probiotic effect of Streptomyces strains alone or in combination with Bacillus and Lactobacillus in juveniles of the white shrimp Litopenaeus vannamei[J].Aquacult Int,2017,25(2):927-939.
[19]ZHANG L,GUO B,YANG H,et al.A novel strain of moderately thermophilic Streptomyces from the Fenjiu-flavor Daqu[J].Open Access Libr J,2022,9(2):1-9.
[20] SANG X, MA X, ZHANG Y, et al.Assessment of the distribution and safety of Tetragenococcus muriaticus for potential application in the preparation of Chinese grasshopper sub shrimp paste[J].Front Microbial,2021,12:628838.
[21]LI C,LI W,LI L,et al.Microbial community changes induced by a newly isolated salt-tolerant Tetragenococcus muriaticus improve the volatile flavor formation in low-salt fish sauce[J].Food Res Int,2022,156:111153.
[22] BUTT U D, KHAN S, LIU X, et al.Present status, limitations, and prospects of using Streptomyces bacteria as a potential probiotic agent in aquaculture[J].Probiotics Antimicrob Proteins,2023,doi:10.1007/s12602-023-10053-x.
[23]SZUTOWSKA J.Functional properties of lactic acid bacteria in fermented fruit and vegetable juices:A systematic literature review[J].Eur Food Res Technol,2020,246(3):357-372.
[24] KILLINGER B J, WHIDBEY C, SADLER N C, et al.Activity-based protein profiling identifies alternating activation of enzymes involved in the bifidobacterium shunt pathway or mucin degradation in the gut microbiome response to soluble dietary fiber[J].NPJ Biofilms Microbiomes,2022,8:60.
[25] KROONEMAN J, FABER F, ALDERKAMP A C, et al. Lactobacillus diolivorans sp.nov., a 1,2-propanediol-degrading bacterium isolated from aerobically stable maize silage[J].Int J Syst Evol Microbiol,2002,52(2):639-646.
[26]LOUCA S,POLZ M F,MAZEL F,et al.Function and functional redundancy in microbial systems[J].Nat Ecol Evol,2018,2(6):936-943.