鲊广椒又称酢辣椒、包谷酸辣子或鲊辣椒等,其主要以玉米面或大米粉和辣椒为原料,加辅料剁碎拌匀经密封发酵15~30 d而成,具有酸、辣和鲜的特点[1]。王玉荣等[2]通过研究发现湖北省当阳地区鲊广椒中主要细菌类群以乳酸杆菌(Lactobacillus)为主,其所在团队亦对湖北省秭归[3]、荆州[4]和洪湖[5]地区以及贵州省遵义地区[6]鲊广椒细菌菌群多样性进行了解析,亦发现Lactobacillus为其优势细菌属。尹小庆等[7]对鲊广椒中微生物和风味之间的关联性进行了揭示,结果发现Lactobacillus与47种挥发性化合物显著相关(P<0.05),且鲊广椒特征风味的形成与乳酸菌类群构成息息相关[8]。由此可见,鲊广椒中蕴含了丰富的乳酸菌资源且其对产品风味品质的形成具有积极作用。
CAI W等[9]对湘西地区鲊广椒和糯米酸细菌菌群多样性进行了解析,结果发现虽然两类淀粉质发酵食品均富含乳酸菌,但由于其所使用的原料分别为大米和糯米,因而两者细菌类群存在显著的差异。CAI W等[10]亦发现,重庆和湖北地区的鲊广椒细菌类群均以乳酸菌为主,但不同地区的鲊广椒其乳酸菌类群构成存在一定差异。湖南省位于长江中游,大部分区域在洞庭湖以南,其中湖南西北部主要包括张家界市和湘西土家苗族自治州,多山地峰林,生态环境较好[11]。湘西土家苗族自治州地处低纬度带,而张家界地处北中纬度,且两地相差100多km,气候和环境均存在一定差异。由此可见,对采集自张家界和湘西地区的鲊广椒细菌菌群多样性进行比较分析,其研究数据不仅可以进一步加深研究人员对鲊广椒这一特色发酵食品的了解,亦可以论证地域因素是否会对鲊广椒的细菌群落结构产生影响。
本研究从湖南省张家界市采集了18份鲊广椒样品进行了MiSeq测序,同时从MGRAST数据库下载了湖南省湘西土族苗族自治州12份鲊广椒样品的细菌MiSeq测序数据,并在此基础上对2份数据进行了合并分析,对2个地区鲊广椒中关键细菌类群进行了揭示,探讨了地域因素对鲊广椒细菌类群的影响,通过本研究的开展以期为鲊广椒细菌多样性和功能的研究提供数据支撑。
1 材料与方法
1.1 材料与试剂
DNeasy mericon Food Kit脱氧核糖核酸(deoxyribonucleic acid,DNA)基因组提取试剂盒:德国QIAGEN公司;引物338F/806R:上海桑尼生物科技有限公司;10×聚合酶链式反应(polymerase chain reaction,PCR)缓冲液、脱氧核糖核苷三磷酸、DNA聚合酶:北京全式金生物技术有限公司;Axygen清洁试剂盒:康宁生命科学吴江有限公司;高通量测序配套试剂:美国Illumina公司。
1.2 仪器与设备
Veriti FAST梯度PCR仪:美国ABI公司;ND-2000C微量紫外分光光度计:美国Nano Drop公司;DYY-12电泳仪:北京六一仪器厂;Fluor Chem FC3化学发光凝胶成像系统:美国Protein Simple公司;PE300高通量测序平台:美国Illumina公司;R920机架式服务器:美国DELL公司。
1.3 实验方法
1.3.1 样品采集
于2019年10月下旬从湖南省张家界市(E109°40′~111°20′,N28°52′~29°48′)采集鲊广椒样品18份,样品均满足以下标准:1)发酵时间均为30 d左右;2)无异味及发霉现象;3)色泽微红;4)所有原材料均在本地种植或生产。将样品按照ZZJ1~ZZJ18进行编号后,分别装入采样袋中密封保存并在常温下带回实验室进行后续研究。
1.3.2 宏基因组DNA提取、PCR扩增和高通量测序
每份样品各取2 g,使用试剂盒进行DNA提取,使用加入核苷酸标签(Barcord)的引物338F/806R对细菌16S rRNA V3-V4区进行PCR扩增,扩增体系和程序参照向凡舒等[12]的方法。将扩增合格的产物置于干冰中寄至上海美吉生物医药科技公司进行测序。
1.3.3 生物信息学分析
参照WANG Y等[13]的方法对张家界地区鲊广椒细菌类群下机序列进行质控,质控合格的序列合并为一个fna文件。从MGRAST数据库(https://www.mg-rast.org/)下载CAI W等[9]上传的湖南省湘西土族苗族自治州12份鲊广椒MiSeq测序数据,数据编号为mgp103301,并将其与张家界地区鲊广椒测序数据进行合并,合并后的fna文件上传至QIIME(V1.9.1)平台进行生物信息学分析[14],具体参照HOU Q等[15]的分析流程。首先按照97%相似性对序列集进行划分获得操作分类单元(operational taxonomic units,OTU)矩阵,继而从每个OTU中选取一条代表性序列使用RDP(ribosomal database project 11.5)和Greengenes 13.5数据库对序列进行比对,明确其分类学地位,最后构建系统发育树,并在此基础上进行α多样性和β多样性分析。
1.3.4 鲊广椒样品细菌类群代谢通路的预测
基于Greengenes数据库注释后的代表性序列,使用PICRUSt(phylogeneticinvestigationofcommunitiesbyreconstruction of unobserved states)软件[16]和蛋白质直系同源簇(clusters of orthologous groups of proteins,COG)数据库[17]对2个地区鲊广椒细菌类群有差异的主要代谢途径进行预测。
1.3.5 数据分析
使用Past3软件进行数据处理,使用Mann-Whitney检验对2个地区鲊广椒细菌类群的α多样性指标、优势种属以及基因预测指标进行差异性分析。使用R软件、Origin2017、GraphPad9.0软件、STAMP软件、AI软件和Excel2016进行绘图及修改。使用Huttenhower实验室Galaxy Server进行线性判别分析效应大小(linear discriminant analysis effect size,LEfSe)分析。
2 结果与分析
2.1 两个地区鲊广椒细菌类群α和β多样性分析
纳入本研究的18份张家界鲊广椒样品通过MiSeq测序共获得703 576条原始序列,经质控后得到702 540条有效序列,平均每个样品包含39 030条序列。通过将其与从数据库中下载的湖南省湘西土族苗族自治州12份鲊广椒样品数据进行合并分析,本研究计算了2个地区鲊广椒细菌类群的α多样性指标,结果见图1。
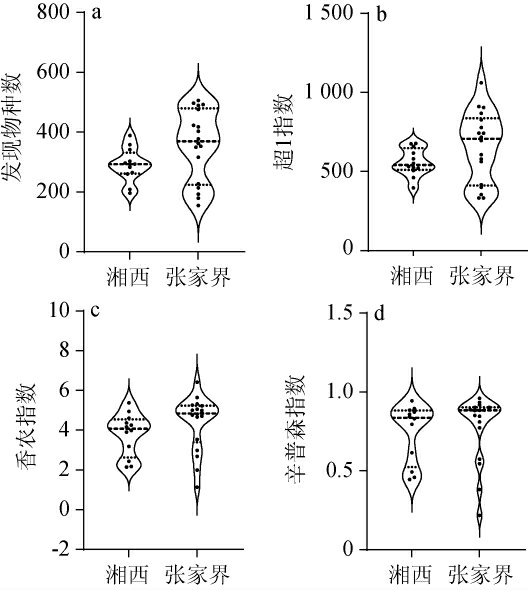
图1 两个地区鲊广椒样品细菌类群α多样性指标的比较分析
Fig.1 Comparative analysis of α-diversity indexes among zha-chili samples collected from 2 regions
由图1可知,经Mann-Whitney检验发现,2个地区鲊广椒细菌类群发现物种数、超1指数、香农指数和辛普森指数差异均不显著(P>0.05),由此可见,2个地区鲊广椒细菌类群的丰度和多样性差异均无明显差异(P>0.05)。本研究进一步基于加权和非加权UniFrac距离对2个地区鲊广椒细菌类群的群落结构进行解析,结果见图2。
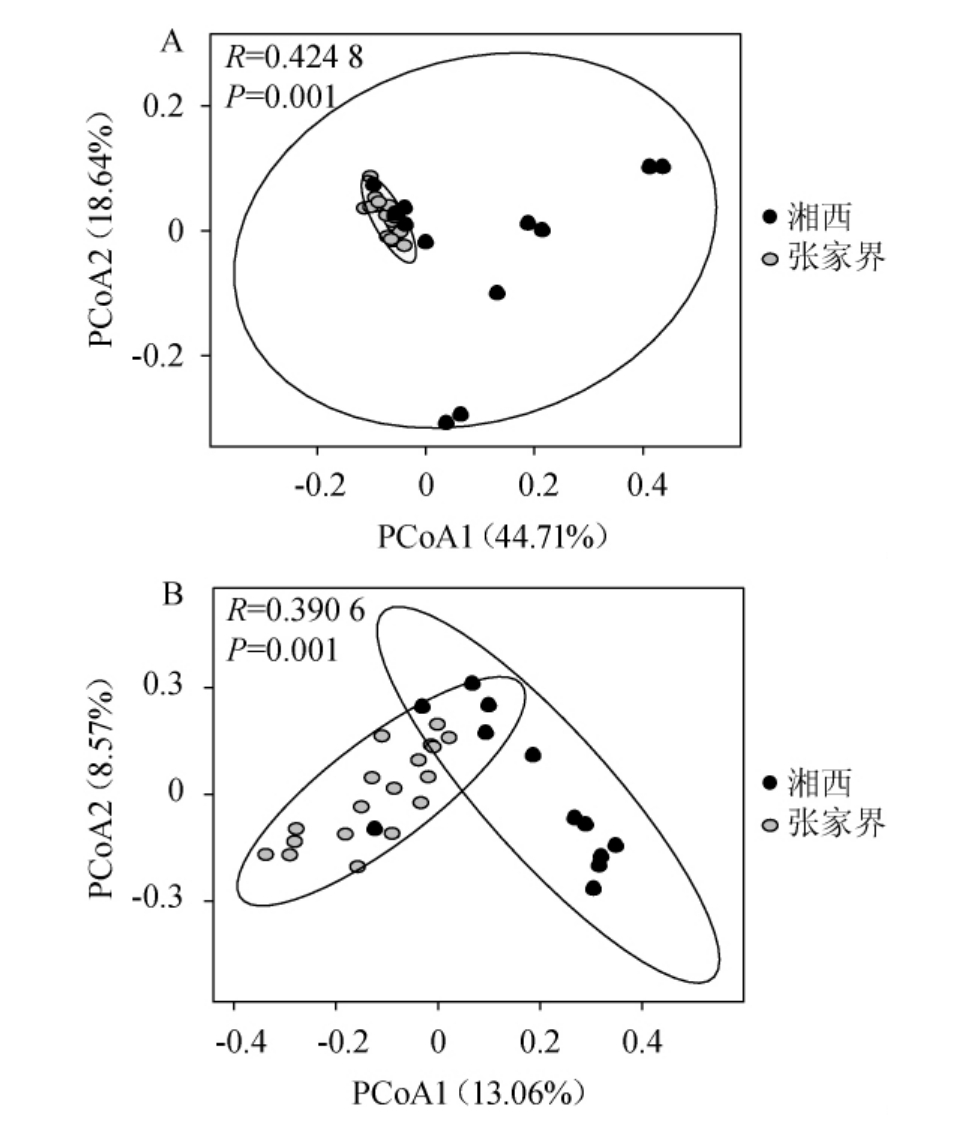
图2 基于加权(A)和非加权距离(B)的2个地区鲊广椒样品细菌类群主成分分析
Fig.2 Principal component analysis of bacterial groups in zha-chili samples collected from 2 regions based on weighted (A) and unweighted (B) UniFrac distance
由图2可知,无论是基于加权还是非加权UniFrac距离,在主成分分析得分图中2个地区的鲊广椒样品均呈现出明显的分离趋势,这说明两者细菌类群群落结构存在明显差异。经多元方差分析(multivariate analysis of variance,MANOVA)发现,两者差异显著(P<0.05)。由此可见,虽然2个地区鲊广椒细菌的丰度和多样性无明显差异,但其细菌类群群落结构存在明显的差异。
2.2 两个地区鲊广椒细菌类群比较分析
经序列比对发现,张家界和湘西2个地区共30份鲊广椒样品中的细菌类群划分为24个门和397个属。基于门和属水平,2个地区平均相对含量大于1.0%的细菌类群比较分析见图3。
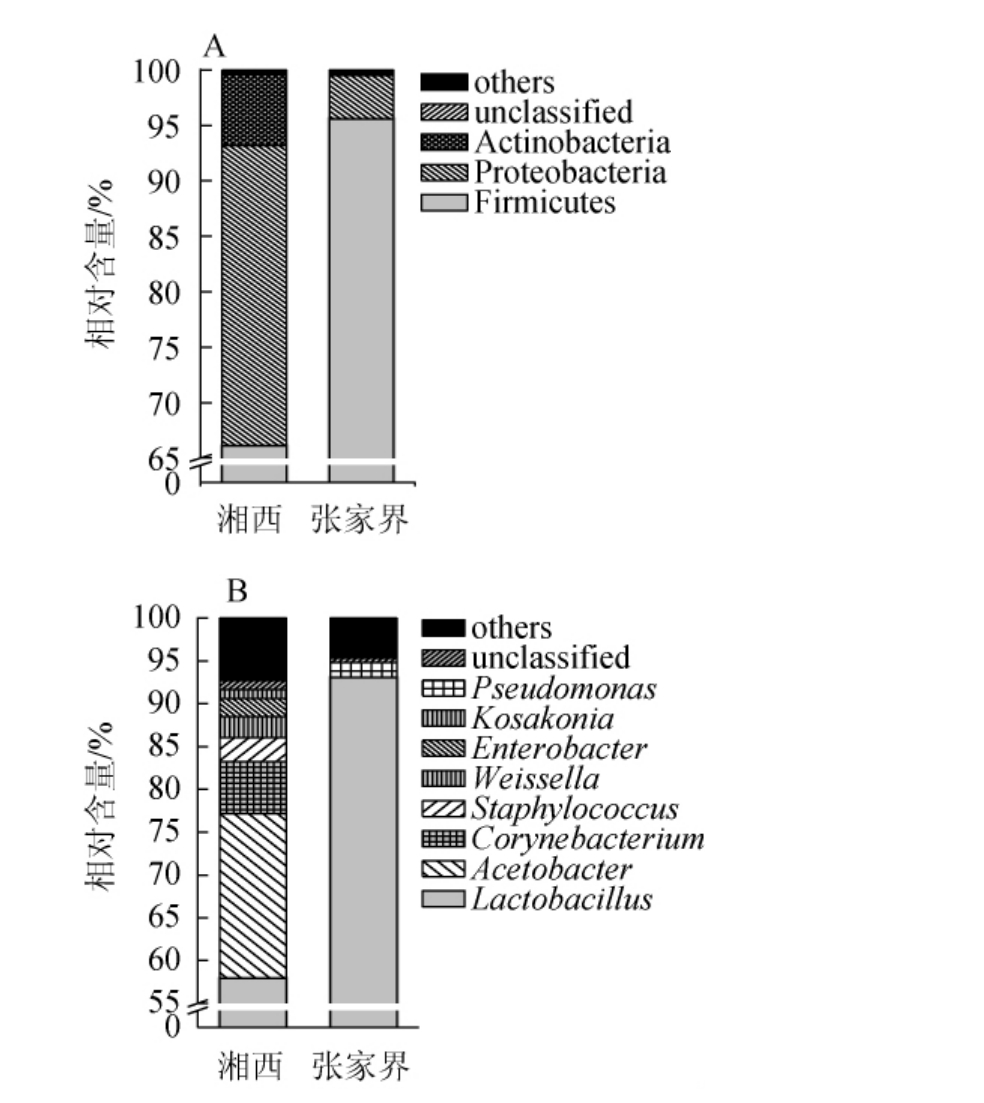
图3 基于门(A)和属(B)水平2个地区鲊广椒样品细菌类群比较分析
Fig.3 Comparative analysis of bacterial groups in zha-chili samples collected from 2 regions at the phylum (A) and genus level(B)
由图3A可知,张家界地区鲊广椒中平均相对含量大于1.0%的细菌门主要为硬壁菌门(Firmicutes)和变形菌门(Proteobacteria),平均相对含量分别为95.59%和3.94%,而湘西地区鲊广椒中平均相对含量大于1.0%的细菌门主要为硬壁菌门(Firmicutes)、变形菌门(Proteobacteria)和放线菌门(Actinobacteria),平均相对含量分别为66.08%、27.12%和6.45%。经Mann-Whitney检验发现,3个细菌门在2个地区鲊广椒中差异均非常显著(P<0.01)。由图3B可知,张家界地区鲊广椒中平均相对含量大于1.0%的细菌属主要为乳酸杆菌(Lactobacillus)和假单胞菌属(Pseudomonas),平均相对含量分别为93.04%和1.78%,而湘西地区鲊广椒中平均相对含量大于1.0%的细菌属为乳酸杆菌(Lactobacillus)、醋酸杆菌属(Acetobacter)、棒状杆菌属(Corynebacterium)、葡萄球菌属(Staphylococcus)、魏斯氏菌属(Weissella)、肠杆菌属(Enterobacter)和科萨克氏菌属(Kosakonia),平均相对含量分别为57.89%、19.26%、6.09%、2.82%、2.45%、2.08%和1.10%。张家界地区鲊广椒中Lactobacillus和Pseudomonas相对含量显著偏高(P<0.05),而Acetobacter、Corynebacterium、Staphylococcus、Weissella、Enterobacter和Kosakonia等含量显著偏低(P<0.05)。
研究表明,乳酸杆菌是产自中国[18]、土耳其[19]和韩国[20]地区泡菜的优势细菌类群,鲊广椒发酵原料中亦含有辣椒等蔬菜制品,且发酵方式亦为厌氧发酵。由此可见,由于发酵原料和发酵方式的相似性使得鲊广椒和泡菜均蕴含着丰富的乳酸菌资源。虽然张家界和湘西地区相距仅100多公里,但本研究发现2个地区鲊广椒细菌菌群多样性存在显著的差异,这可能与其生产工艺、使用原料和生态环境不同有关[21]。本研究团队从鲊广椒中发现并命名了“恩施乳杆菌(L.enshiensis)”[22]、“鲊广椒乳杆菌(L.zhachilii)”[23]、“安琪乳杆菌(Secundilactobacillus angelii sp.nov.)”[24]、“土家乳杆菌(Levilactobacillus tujiae sp.nov.)”[24]和“宜都促生乳杆菌(L.yiduensis sp.nov.)”[25]共5个乳酸杆菌新物种,这表明鲊广椒中乳酸菌类群的构成具有一定的独特性,本研究亦揭示了不同地区鲊广椒的细菌多样性存在差异,因而在后续研究中对华中乃至西南地区鲊广椒中蕴含的乳酸菌资源进行挖掘保护是极为必要的。
2.3 两个地区鲊广椒共有细菌类群分析
本研究进一步对在2个地区30份鲊广椒样品中均存在的OTU进行了统计,结果见图4。
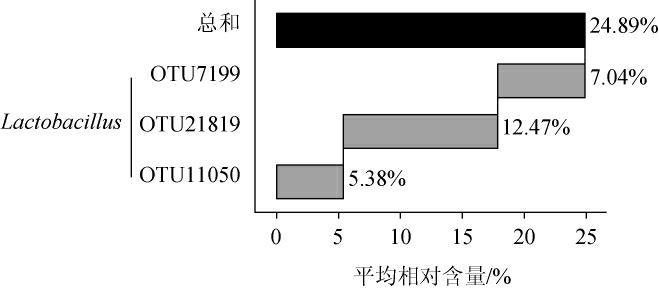
图4 两个地区鲊广椒样品共有细菌类群分析
Fig.4 Core bacterial groups in zha-chili samples collected from 2 regions
由图4可知,在张家界和湘西地区鲊广椒中均存在的OTU有3个,分别为OTU11050、OTU21819和OTU7199,相对含量分别为5.38%、12.47%和7.04%,三者累计相对含量为24.89%。本研究在每个OTU中选取一条代表性序列在数据库中进行比对,结果发现其均隶属于乳酸杆菌(Lactobacillus)。由此可见,虽然2个地区鲊广椒细菌类群存在显著的差异,但亦共有部分乳酸杆菌类群,因而乳酸杆菌对鲊广椒品质的形成可能具有积极的作用。
2.4 两个地区鲊广椒关键细菌类群的甄别
基于LEfSe分析两个地区鲊广椒关键细菌类群的水平条形图见图5。由图5可知,LEfSe的线性判别得分为3.0时,可筛选出22个丰度差异明显的类群,其中9个类群在张家界地区鲊广椒中明显偏高,在属水平上主要为乳酸杆菌(Lactobacillus)和假单胞菌(Pseudomonas),13个类群在湘西地区鲊广椒中明显偏高,在属水平上主要为异杆菌属(Allobaculum)、寡养单胞菌(Stenotrophomonas)、产丙酸单孢菌属(Propionicimonas)、藤黄单孢菌属(Luteimonas)和醋酸杆菌属(Acetobacter)。值得一提的是,异杆菌属(Allobaculum)、寡养单胞菌(Stenotrophomonas)、产丙酸单孢菌属(Propionicimonas)和藤黄单孢菌属(Luteimonas)在2个地区30份鲊广椒中的含量均偏低,平均相对含量分别为0.000 3%、0.005 2%、0.000 6%和0.001 4%。由此可见,Lactobacillus和Pseudomonas可作为张家界地区鲊广椒中的生物标志物,而醋酸杆菌属(Acetobacter)可作为湘西地区鲊广椒中的生物标志物。
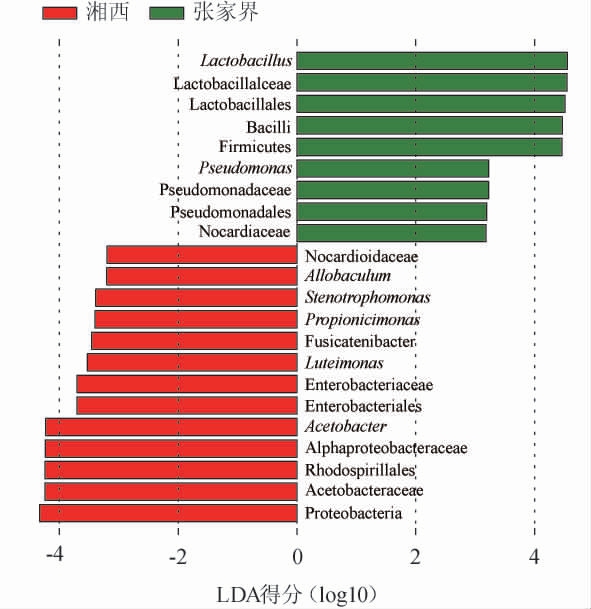
图5 基于LEfSe分析两个地区鲊广椒样品关键细菌类群的水平条形图
Fig.5 Horizontal bar chart of key bacterial groups in zha-chili samples collected from 2 regions based on LEfSe analysis
2.5 两个地区鲊广椒细菌类群差异性代谢通路的预测
本研究使用PICRUSt分析了2个地区鲊广椒细菌类群的代谢途径,共注释到4 793个同源蛋白簇,获得了8个存在显著差异的代谢途径,结果见图6。
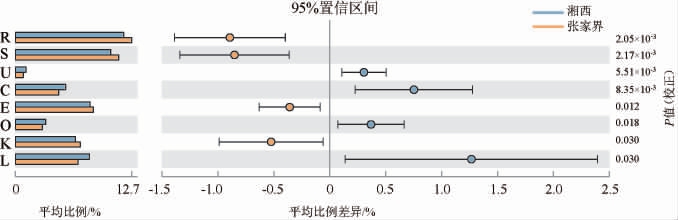
图6 两个地区鲊广椒样品细菌类群差异性代谢通路的预测
Fig.6 Prediction of differential metabolic pathways in zha-chili samples collected from 2 regions
C为能量生产和转换;E为氨基酸转运与代谢;K为转录;L为复制、重组和修复;O为翻译后修饰、蛋白质周转、伴随蛋白;R为一般功能预测;
S为功能未知;U为细胞内运输、分泌和囊泡运输。
由图6可知,代谢途径R(一般功能预测)、S(功能未知)、E(氨基酸转运与代谢)和K(转录)在张家界地区鲊广椒细菌类群中显著高表达(P<0.05),而代谢途径U(细胞内运输、分泌和囊泡运输)、C(能量生产和转换)、O(翻译后修饰、蛋白质周转、伴随蛋白)和L(复制、重组和修复)显著低表达(P<0.05),这说明张家界地区鲊广椒中细菌类群对原料中的蛋白质潜在利用率更高。
3 结论
张家界地区鲊广椒细菌类群以乳酸杆菌(Lactobacillus)和假单胞菌(Pseudomonas)为主。较之湘西地区鲊广椒,虽2个地区鲊广椒细菌的丰度和多样性无明显差异,但其细菌类群群落结构存在明显的差异。Lactobacillus和Pseudomonas可作为张家界地区鲊广椒中的生物标志物,而醋酸杆菌属(Acetobacter)可作为湘西地区鲊广椒中的生物标志物。此外,代谢途径R(一般功能预测)、S(功能未知)、E(氨基酸转运与代谢)和K(转录)在张家界地区鲊广椒细菌类群中显著高表达。由此可见,2个地区鲊广椒细菌类群和功能存在明显差异。
[1]张曼,汤艳燕,魏雪,等.不同产地鲊辣椒风味成分比较分析[J].食品与发酵工业,2021,47(14):264-270.
[2]王玉荣,沈馨,董蕴,等.鲊广椒细菌多样性评价及其对风味的影响[J].食品与机械,2018,34(4):25-30.
[3]席啦,向凡舒,张彦,等.秭归地区鲊广椒中乳酸菌及细菌多样性研究[J].中国酿造,2021,40(11):122-126.
[4]葛东颖,王玉荣,向凡舒,等.荆州地区鲊广椒乳酸菌多样性解析及其分离株发酵特性的评价[J].中国食品添加剂,2019,30(4):72-79.
[5]李娜,崔梦君,马佳佳,等.基于Illumina MiSeq测序和传统可培养方法的洪湖鲊广椒乳酸菌多样性研究[J].食品与发酵工业,2021,47(4):110-115.
[6]崔梦君,王玉荣,葛东颖,等.遵义地区莽椒细菌多样性及PICRUSt基因功能预测分析[J].食品与发酵工业,2020,46(4):106-112.
[7]尹小庆,汤艳燕,阚建全,等.小米辣鲊辣椒发酵过程中风味物质及微生物多样性的变化[J].食品与发酵工业,2020,46(5):74-82.
[8]尹小庆,汤艳燕,杜木英,等.两种鲊辣椒发酵过程中香气特征及其差异分析[J].食品与发酵工业,2019,45(16):266-274.
[9]CAI W,WANG Y,HOU Q,et al.Rice varieties affect bacterial diversity,flavor,and metabolites of zha-chili[J].Food Res Int,2021,147(1):110556.
[10]CAI W,TANG F,WANG Y,et al.Bacterial diversity and flavor profile of Zha-Chili,a traditional fermented food in China[J].Food Res Int,2021,141(3):110112.
[11]袁东海.湘西地区森林碳汇发展存在的主要问题及其对策[J].资源信息与工程,2022,37(2):156-158.
[12]向凡舒,张婷,董蕴,等.糯米酸细菌多样性解析及其表型和基因功能预测[J].中国粮油学报,2021,36(1):14-20.
[13]WANG Y,CAI W,WANG W,et al.Analysis of microbial diversity and functional differences in different types of high-temperature Daqu[J].Food Sci Nutr,2021,9(2):1003-1016.
[14] PALMER R J, COTTON S L, KOKARAS A S, et al.Analysis of oral bacterial communities: comparison of HOMI NGS with a tree-based approach implemented in QIIME[J].J Oral Microbiol,2019,11(1):1586413.
[15] HOU Q, WANG Y, NI H, et al.Deep sequencing reveals changes in prokaryotic taxonomy and functional diversity of pit muds in different distilleries of China[J].Ann Microbiol,2022,72(1):1-14.
[16]WILKINSON T J,HUWS S A,EDWARDS J E,et al.CowPI: a rumen microbiome focussed version of the PICRUSt functional inference software[J].Front Microbiol,2018,9(5):1095.
[17] GALPERIN M Y, KRISTENSEN D M, MAKAROVA K S, et al.Microbial genome analysis:the COG approach[J].Brief Bioinform,2019,20(4):1063-1070.
[18] HE Z, CHEN H, WANG X, et al.Effects of different temperatures on bacterial diversity and volatile flavor compounds during the fermentation of suancai, a traditional fermented vegetable food from northeastern China[J].LWT-Food Sci Technol,2020,118(1):108773.
[19] BADER ELMAC S, TOKATL M, DURSUN D, et al.Phenotypic and genotypic identification of lactic acid bacteria isolated from traditional pickles of the ubuk region in Turkey[J].Folia Microbiologica, 2015,60(3):241-251.
[20]JUNG J Y,LEE S H,KIM J M,et al.Metagenomic analysis of kimchi,a traditional Korean fermented food[J].Appl Environ Microbiol,2011,77(7):2264-2274.
[21] CAI W, WANG Y, HOU Q, et al.PacBio sequencing combined with metagenomic shotgun sequencing provides insight into the microbial diversity of zha-chili[J].Food Biosci,2021,40(4):100884.
[22]ZHANG Z,WANG Y,HOU Q,et al.Lactobacillus enshiensis sp.nov.,a novel arsenic-resistant bacterium[J].Int J Syst Evolut Microbiol,2020,70(4):2580-2587.
[23]ZHANG Z,HOU Q,WANG Y,et al.Lactobacillus zhachilii sp.nov.,a lactic acid bacterium isolated from Zha-Chili[J].Int J Syst Evolut Microbiol,2019,69(8):2196-2201.
[24] ZHANG Z, WANG Y, DONG Y, et al.Characterization of two novel pentose-fermenting and GABA-producing species:Levilactobacillus tujiorum sp.nov.and Secundilactobacillus angelensis sp.nov.isolated from a solid-state fermented zha-chili[J].System Appl Microbiol,2022,45(5):126344.
[25] DONG Y, WANG Y, ZHAO H, et al. Levilactobacillus yiduensis sp.nov.,isolated from Zha-chili in Yidu county,Hubei province,China[J].Curr Microbiol,2023,80(4):112.