清香型白酒是中国白酒三大基本香型之一,独特的工艺结合独特的微生物菌群结构造就了清香型白酒清香纯正的风格特征[1]。汾酒大曲是一种以生料大麦、豌豆为原料,自然接种环境中的微生物,经过微生物的生长代谢和能量代谢形成的微生物生态制品,具有糖化、发酵、增香的作用[2-4]。
大麦属禾本科植物,富含碳水化合物、蛋白质等大分子物质,此外,含有钙、磷等矿质元素,适合多种微生物生长,是清香型白酒的主要制曲原料[5-7]。不同品种的大麦由于其生长地区、气候、储藏条件的不同,其内生微生物、表皮微生物的差异,会对制曲造成一定的影响。近年来,已经有较多采用高通量测序技术解析植物表皮微生物群落结构的研究[8-9]。张二豪等[10]利用高通量测序技术对苹果表皮及根际土壤微生物的群落结构与多样性进行研究,并对优势微生物菌属进行关联性分析;张世伟等[11]采用高通量测序技术研究了不同品种葡萄表面的微生物群落结构差异,表明不同品种是最关键的影响因素。何萍等[12]采用高通量测序技术对不同品种葡萄果实表皮微生物群落组成进行研究,结果表明,不同品种间微生物群落结构组成及相对丰度有较大的差异。目前,有关大麦表皮微生物多样性的研究尚非常少,且有关不同大麦品种原料对于大曲发酵影响的相关研究也知之甚少。
本研究采用Illumina Miseq高通量测序技术对以上三个不同品种的大麦表皮细菌和真菌群落结构进行解析,分析不同品种大麦表皮细菌和真菌的多样性,旨在为清香型白酒制曲原料大麦的质量把控提供理论依据。
1 材料与方法
1.1 材料与试剂
三个不同品种的大麦:DM1(甘啤6号,产地甘肃,清香型白酒现用大麦品种),DM2(汾麦30,产地山西)、DM3(大麦3018,产地山西)。DM2、DM3为本项目团队优选大麦父本、母本,采用系谱法选育得到的新大麦品种,具有迟播早收、生育期短、抗逆性强、亩产高、容重高的优良种植特性,适宜山西南部临汾市、运城市及黄淮冬麦区种植。
使用经体积分数75%的酒精消毒后的镊子按照“五点取样法”取大麦种子样品,置于无菌自封袋中,-20 ℃冷冻备用。每个样品取3个样本作为平行样。
MoBio PowerSoil 脱氧核糖核酸(deoxyribonucleic acid,DNA)Isolation Kit(100)试剂盒:德国QIAGEN公司;2×Taq Plus Master Mix试剂盒:南京诺唯赞生物科技股份有限公司;AgencourtR AMPureR XP(核酸纯化试剂盒):美国贝克曼库尔特有限公司。
1.2 仪器与设备
SORVALL RC6 Plus高速冷冻离心机:赛默飞世尔科技(中国)有限公司;GelDoc-It凝胶成像系统:美国UVP公司;NanoDrop2000超微量分光光度计:美国赛默飞公司;Agilent 2100生物分析仪:安捷伦科技有限公司;Labchip GX生物大分子分析仪:珀金埃尔默仪器有限公司;ABI 9700基因扩增聚合酶链式反应(polymerase chain reaction,PCR)仪,ABI 7500实时荧光定量PCR(real-time fluorescent quantitive PCR,RT-fqPCR)仪:美国ABI公司;Illumina Miseq PE300高通量二代测序仪:美国Illumina公司。
1.3 方法
1.3.1 DNA提取
在50 mL无菌离心管中加入10 g大麦样品和30 mL 0.01 mol/L磷酸盐缓冲液(phosphate buffered saline,PBS),旋涡振荡,使大麦表皮的微生物富集于PBS中,直接提取缓冲液中的微生物。使用DNA Isolation Kit试剂盒提取基因组DNA,DNA浓度和纯度利用Nano Drop 2000进行检测。
1.3.2 PCR扩增
以总基因组DNA为模版进行PCR扩增,细菌16S rRNA基因的V3-V4区相关引物对为338F(5'-ACTCCTACGGGAGGCAGCAG-3')和806R(5'-GGACTACHVGGGTWTCTAAT-3')。真核ITS2区相关引物对为:ITS3(5'-GCATCGATGAAGAACGCAGC-3')和ITS4(5'-TCCTCCGCTTATTGATATGC-3')。
PCR扩增体系和扩增条件参照相关文献方法[13]。PCR扩增产物委托北京奥维森基因科技有限公司使用Illumina Miseq PE300高通量测序平台进行测序。
1.3.3 高通量测序结果分析
利用QIIME1(v1.8.0)软件对测序所得的原始序列进行去噪、拼接和质控后,使用UPARSE软件(version 7.1)根据97%的相似度对优质序列进行操作分类单元(operational taxonomic unit,OTU)聚类,并进行物种注释。使用QIIME1(v1.8.0)软件进行Alpha多样指数分析。使用R(v3.6.0)软件进行物种组成柱状图分析和主成分分析(principal component analysis,PCA)。
2 结果与分析
2.1 样品Venn图分析
对9个大麦样品表皮微生物细菌16S rRNA基因的V3-V4区以及真菌ITS2区进行PCR扩增和测序,对各样本优化后共得到677 155条有效细菌序列和896 479条有效真菌序列,在97%相似度下划分OTU。
Venn图可直观分析各个样本的OTU数目和各样本间的重叠情况[14-15]。由图1a可知,9个大麦表皮样本共915个细菌OTUs,DM1、DM2、DM3样品所产生的OTU数分别为566个、668个、517个;共有细菌OTU数为279个,特有细菌OTU数分别为119个、152个、87个。由图1b可知,9个大麦表皮样本共产生了397个真菌OTUs,DM1、DM2、DM3样品所产生的OTU数分别为334个、228个、199个;共有真菌OTU数为131个,特有真菌OTU数分别为125个、19个、20个。以上结果表明,不同品种大麦表皮微生物群落结构组成差异较大。
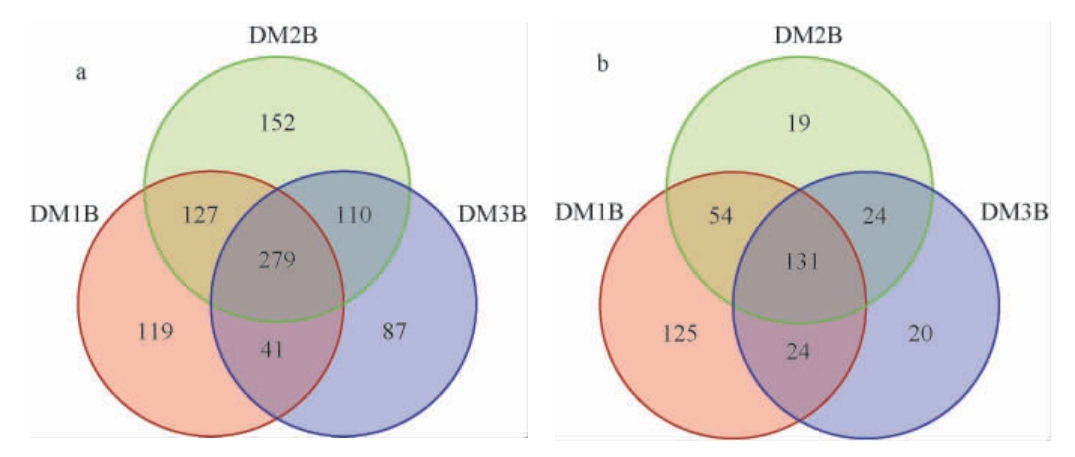
图1 不同品种大麦样品基于细菌(a)、真菌(b)操作分类单元的Venn图
Fig.1 Venn diagram of operational taxonomic unit of different varieties of barley samples based on bacteria (a) and fungi (b)
2.2 稀释性曲线
稀释性曲线可反映样品的测序深度,可用来说明测序的数据量是否合理。由图2可知,细菌与真菌的测序深度合理,能反映样本中微生物的实际情况。
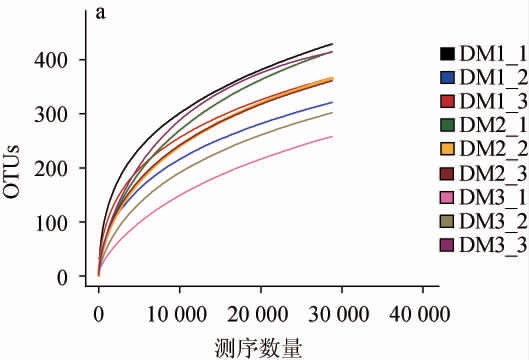
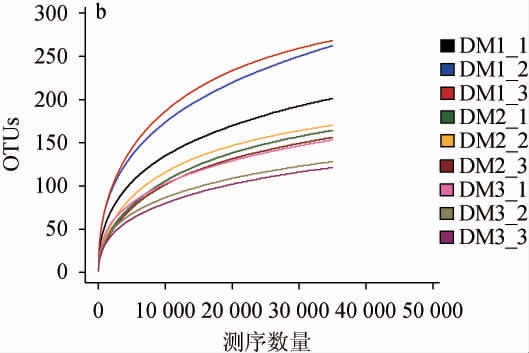
图2 不同品种大麦样品细菌(a)、真菌(b)稀释性曲线
Fig.2 Dilution curves of bacteria (a) and fungi (b) of different varieties of barley samples
2.3 Alpha多样性分析
Chao1指数表示菌种丰富度,Shannon指数用来表征菌种多样性[16]。由图3可知,DM1样品的细菌和真菌丰富度和多样性最高,DM3样品的细菌和真菌丰富度和多样性最低。
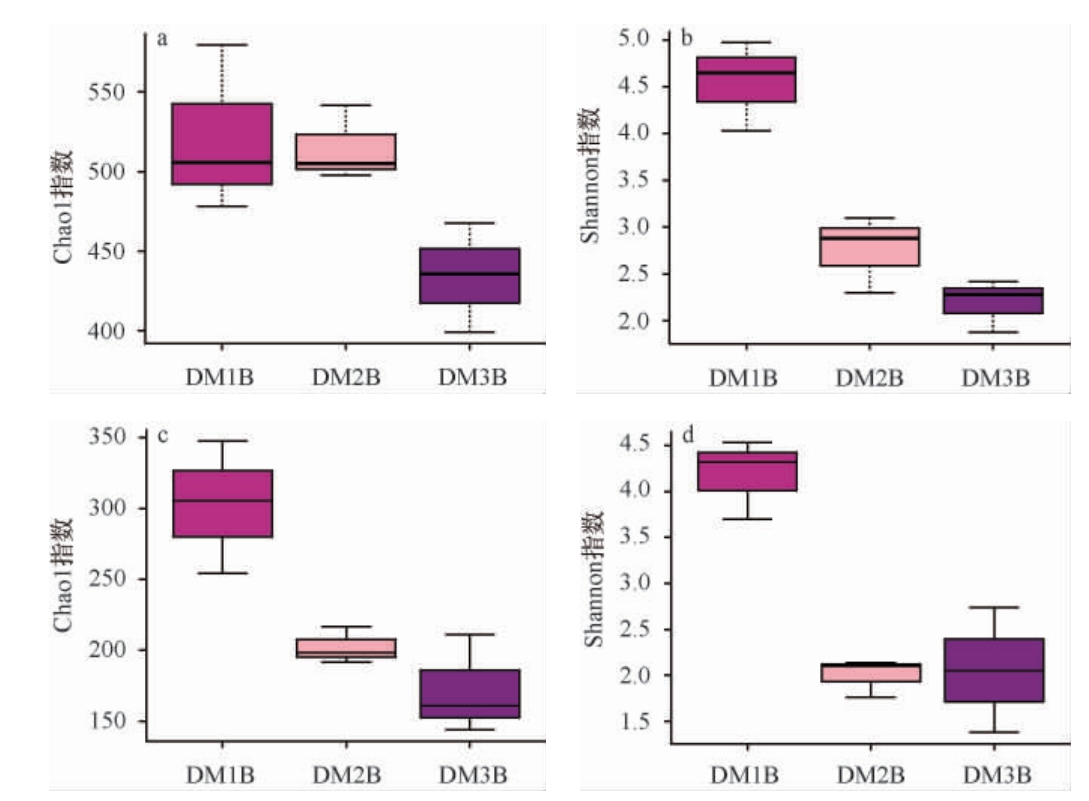
图3 不同品种大麦样品细菌(a、b)及真菌(c、d)Alpha多样性箱图
Fig.3 Box diagram of bacterial (a,b) and fungal (c,d) alpha diversity of different varieties of barley samples
2.4 细菌群落多样性分析
由图4(a)可知,基于门水平,从三个大麦样本中共得到5个优势细菌门(平均相对含量>1%),分别为变形菌门(Proteobacteria)、厚壁菌门(Firmicutes),拟杆菌门(Bacteroidota)、放线菌门(Actinobacteriota)和蓝细菌门(Cyanobacteria)。DM1样品中的优势细菌门为变形菌门、厚壁菌门、拟杆菌门、放线菌门和蓝细菌门,平均相对含量分别是70.8%、12.5%、12.3%、2.6%、1.4%。DM2样品中的优势细菌门为变形菌门、厚壁菌门、蓝细菌门、拟杆菌门和放线菌门,平均相对含量分别是82.5%、5.3%、8.6%、2.1%和1.0%。DM3样品中的优势细菌门为变形菌门、厚壁菌门和蓝细菌门,平均相对含量分别是92.9%、2.3%和2.9%。以上结果表明,三个大麦表皮样本菌群结构差异较大,且变形菌门在三个品种大麦样本中均占绝对优势。

图4 基于门(a)和属(b)水平不同品种大麦样品细菌群落结构
Fig.4 Bacterial community structure of different varieties of barley samples based on phylum (a) and genus (b) levels
由图4(b)可知,基于属水平,DM1的表皮细菌多样性显著高于DM2和DM3。DM1样品共有7个优势细菌属(平均相对丰度>1%),分别为假单胞菌属(Pseudomonas)、马赛菌属(Massilia)、泛菌属(Pantoea)、鞘氨醇单胞菌属(Sphingomonas)、类芽孢菌属(Paenibacillus)、Pedobacter、薄层菌属(Hymenobacter),平均相对丰度分别为20.8%、17.8%、12.3%、11.7%、9.06%、5.05%、4.20%。DM2、DM3样品中均有2个优势菌属(平均相对丰度>1%),分别是泛菌属(Pantoea)和假单胞菌属(Pseudomonas),其平均相对丰度为60.9%、62.3%和11%、26.3%。泛菌属是一类广泛存在于自然界且功能多样的微生物[17]。假单孢菌属普遍分布于植物中,对植物生长、生物防治和养分利用等方面具有有益作用[18-21]。研究发现,泛菌属存在于多种植物的种子中,且多为优势类[22]。在连续3代的萝卜种子中,发现泛菌属和假单胞菌属也是主要类群[23];LEVEAU J H J等[24]研究发现,霞多丽葡萄上的优势菌属为假单胞菌属。RAHMAN M等[25]研究表明,Pantoea和Pseudomonas具有促进植物生长和抵抗真菌性病害等功能。综上所述,DM2和DM3样品中富含与植物生长和生物防治相关的微生物。
2.5 真菌群落多样性分析
由图5(a)可知,基于门水平,从三个大麦样本中,共得到3个优势真菌门(平均相对丰度>1%),分别为担子菌门(Basidiomycota)、子囊菌门(Ascomycota)和毛霉门(Mucoromycota)。DM1样品中的优势真菌门为担子菌门、子囊菌门和毛霉门,平均相对丰度分别是62.0%、27.1%、10.5%。DM2样品中的优势真菌门为担子菌门和子囊菌门,平均相对丰度分别是85.9%、13.8%。DM3样品中的优势真菌门为子囊菌门和担子菌门,平均相对丰度分别是80.7%、18.9%。以上结果表明,大麦表皮样本中,主要存在担子菌门、子囊菌门这两类,三个大麦样本菌群结构差异较大。
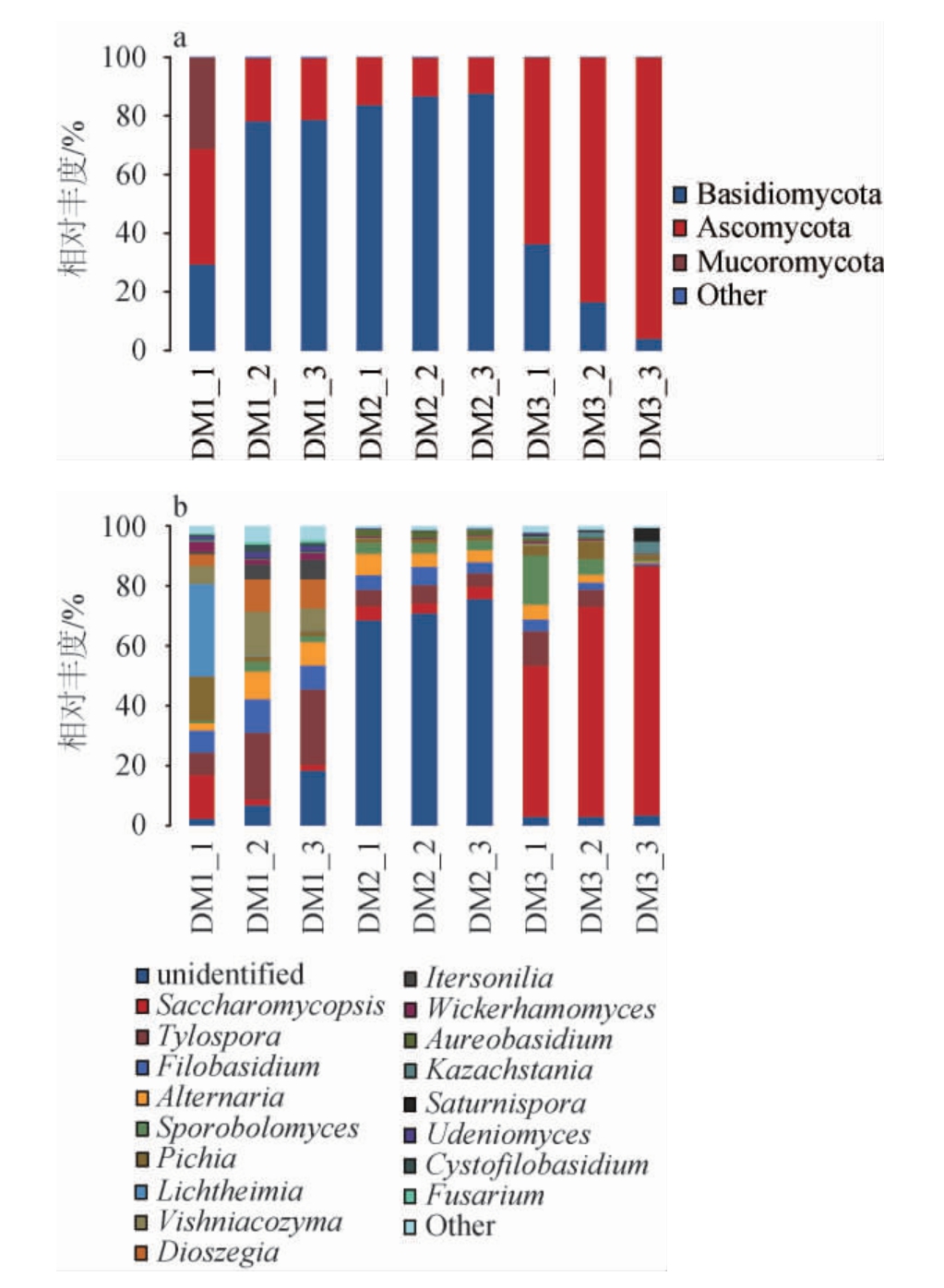
图5 基于门(a)和属(b)水平不同品种大麦样品真菌群落结构
Fig.5 Fungal community structure of different varieties of barley samples based on phylum (a) and genus (b) levels
由图5(b)可知,基于属水平,DM1的表皮真菌多样性显著高于DM2和DM3。DM1样品中共有9个能准确鉴定的优势菌属(平均相对丰度>1%),分别是类型孢子菌属(Tylospora)、横梗霉属(Lichtheimia)、维希尼克氏酵母菌属(Vishniacozyma)、线黑粉酵母属(Filobasidium)、Dioszegia、链格孢属(Alternaria)、复膜孢酵母属(Saccharomycopsis)、毕赤酵母属(Pichia)和锁霉属(Itersonilia),其平均相对丰度分别是18.2%、10.4%、9.3%、8.8%、8.2%、6.5%、6.2%、6.2%和4.0%。DM2样品中共有6个优势细菌属(平均相对丰度>1%),分别是担子菌门一类未鉴定的属、类型孢子菌属(Tylospora)、链格孢属(Alternaria)、线黑粉酵母属(Filobasidium)、复膜孢酵母属(Saccharomycopsis)和掷孢酵母属(Sporobolomyces),平均相对丰度分别是71.7%、5.2%、5.2%、4.8%、4.1%和3.6%。DM3样品中共有5个优势细菌属(平均相对丰度>1%)分别是复膜孢酵母属(Saccharomycopsis)、掷孢酵母属(Sporobolomyces)、类型孢子菌属(Tylospora)、毕赤酵母属(Pichia)和链格孢属(Alternaria),平均相对丰度分别是67.8%、7.3%、5.8%、3.6%和2.6%。三个大麦品种优势菌属差异较大,可能与产地、气候和品种等因素有关。复膜孢酵母属是清香型白酒大曲上霉的主要微生物,可分泌淀粉酶、蛋白酶、β-葡萄糖苷酶等水解酶类,降解大分子底物,为酿酒酵母等其他白酒发酵微生物的生长提供营养[26]。DM3样品中复膜孢酵母属为绝对优势真菌属。链格孢属是一类世界范围广泛分布的真菌,存在于自然界的不同基质上,可作为腐生菌、寄生菌和内生菌,是重要植物病原菌,可造成多种作物的严重病害[27]。DM3样品中链格孢属相对丰度最低。
2.6 基于细菌、真菌OTU水平的不同品种大麦样品主成分分析
主成分分析(PCA)可直观地将样品间、样品组间差异程度体现在二维坐标图上,样品间距离越近表示其物种组成越相似。
由图6a可知,第一主成分PC1方差贡献率为61.09%,第二主成分PC2方差贡献率为12.9%。DM3中两个样本与DM2距离较近,DM1与DM2、DM3距离较远,表明三个大麦品种细菌群落结构差异较大。由图6b可知,第一主成分PC1方差贡献率为49.92%,第二主成分PC2方差贡献率为34.23%。三个大麦品种组分布距离较远,表明三个大麦品种真菌群落结构差异较大。
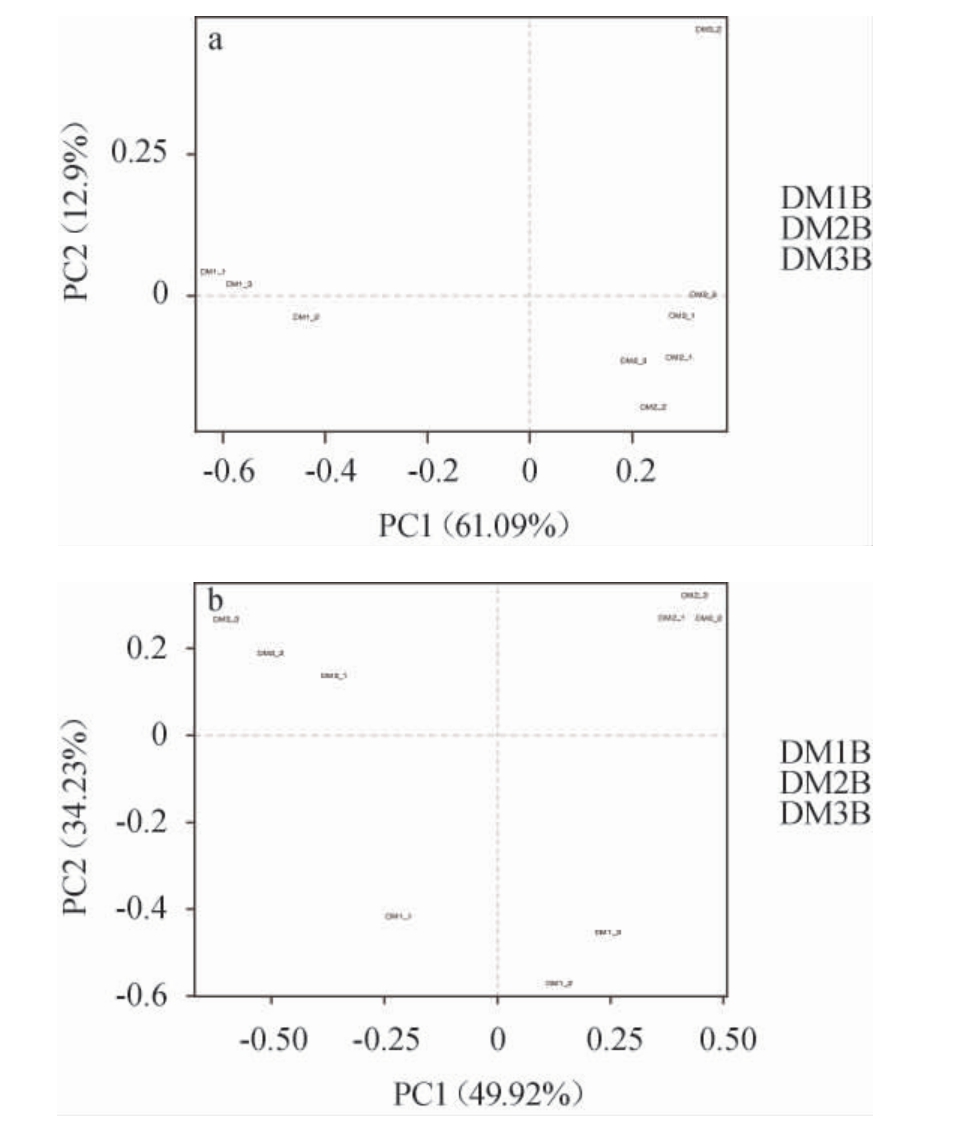
图6 基于细菌(a)、真菌(b)操作分类单元水平的不同品种大麦样品主成分分析
Fig.6 Principal component analysis of different varieties of barley samples based on operational taxonomic unit level of bacteria (a) and fungi (b)
3 结论
采用Illumina Miseq高通量测序技术对不同大麦品种表皮微生物群落结构研究发现,甘啤6号的细菌和真菌丰富度和多样性均最高,大麦3018的细菌和真菌丰富度和多样性均最低。甘啤6号与汾麦30、大麦3018细菌群落结构差异较大,三个大麦品种真菌群落结构差异较大,甘啤6号产地为甘肃,汾麦30、大麦3018产地为山西,由此表明大麦的生长环境对大麦表皮细菌群落结构的影响较大,大麦品种对其表皮真菌群落结构的影响较大。从细菌群落结构分析来看,甘啤6号表皮样本的优势细菌属相对丰度较高的依次是假单胞菌属(Pseudomonas)、马赛菌属(Massilia)、泛菌属(Pantoea)、鞘氨醇单胞菌属(Sphingomonas)、类芽孢菌属(Paenibacillus)等。汾麦30、大麦3018样本中,相对丰度较高的依次是泛菌属(Pantoea)、假单胞菌属(Pseudomonas)等。从真菌群落结构分析来看,甘啤6号中相对丰度较高的依次是类型孢子菌属(Tylospora)、横梗霉属(Lichtheimia)、维希尼克氏酵母菌属(Vishniacozyma)、线黑粉酵母属(Filobasidium)、Dioszegia等。汾麦30样本中担子菌门一类未鉴定的属为优势菌属;大麦3018样本中复膜孢酵母属(Saccharomycopsis)为优势菌属。本研究揭示了不同品种大麦表皮微生物群落结构的差异,为清香型白酒制曲原料大麦的选择和质量把控提供了理论依据,并为进一步完善制曲大麦质量评价指标提供了科学依据。
[1]沈怡方.白酒生产技术全书[M].北京:中国轻工业出版社,2009:332.
[2]孟娇娇,崔海灏.大曲贮存过程中理化指标及微生物变化研究[J].酿酒,2015,42(9):43-45.
[3]张春林.泸州老窖大曲的质量、微生物与香气成分关系[D].无锡:江南大学,2012.
[4]张志刚,吴生文,陈飞.大曲酶系在白酒生产中的研究现状及发展方向[J].中国酿造,2011,30(1):13-16.
[5]朱睦元,张京.大麦(青稞)营养分析及其食品加工[M].杭州:浙江大学出版社,2015:47-59.
[6]ABDEL-AAL E,WOOD P.Specialty grains for food and feed[M].Canada:American Association of Cereal Chemists,2005:167-202.
[7]董海洲.大麦理化特性及其挤压膨化加工机理与应用研究[D].北京:中国农业大学,2002.
[8]李俊锋.基于16S rRNA和宏基因组高通量测序的微生物多样性研究[D].北京:清华大学,2015.
[9]杜贞娜,程斐,单之初,等.高通量测序技术及其在黄酒微生物多样性研究中的应用[J].中国酿造,2021,40(8):14-19.
[10]张二豪,杨雪,张鑫,等.藏东南产区苹果表皮及根际土壤微生物多样性研究[J].中国酿造,2022,41(5):118-123.
[11]张世伟,陈曦,钟其顶,等.不同品种酿酒葡萄表皮微生物群落多样性分析[J].生物技术通报,2017,33(3):128-137.
[12]何萍,王健强,高潭,等.西藏林芝地区不同品种葡萄果实表皮微生物群落结构组成分析[J].中国酿造,2022,41(12):144-149.
[13]任婷月,韩英,甄攀,等.不同品种大麦内生菌的研究分析[J].酿酒科技,2022(4):22-29.
[14]FOUTS D E,SZPAKOWSKI S,PURUSHE J,et al.Next generation sequencing to define prokaryotic and fungal diversity in the bovine rumen[J].PLoS One,2012,7(11):e48289.
[15]MCCALL M N,KENT O A,YU J S,et al.MicroRNA profiling of diverse endothelial cell types[J].BMC Med Genomics,2011,4(1):78.
[16] LIU W J,XI X X,SUDU Q,et al.High-throughput sequencing reveals microbial community diversity of Tibetan naturally fermented yak milk[J].Ann Microbiol,2015,65(3):1741-1751.
[17]郭鹤宝,何山文,王星,等.水稻种子内生泛菌(Pantoea spp.)系统发育多样性及其促生功能[J].微生物学报,2019,59(2):2285-2295.
[18]赵疆,梁世军,杨涛,等.促生细菌的分离及复配菌剂对甘肃贝母产量的影响[J].中国实验方剂学杂志,2021,27(24):163-170.
[19]LEE S A,KIM Y,KIM J M,et al.A preliminary examination of bacterial,archaeal, and fungal communities inhabiting different rhizocompartments of tomato plants under real-world environments[J].Sci Rep,2019,9:9300.
[20] DE JESUS SOUSA J A, OLIVARES F L.Plant growth promotion by Streptomycetes:ecophysiology,mechanisms and applications[J].Chem Biol Technol Agr,2016,3(1):1-12.
[21] LU Y Z, ZHANG E H, HONG M S, et al.Analysis of endophytic and rhizosphere bacterial diversity and function in the endangered plant Paeonia ludlowii[J].Arch Microbiol,2020,202(7):1717-1728.
[22]ZHANG J,ZHANG C W,YANG J,et al.Insights into endophytic bacterial community structures of seeds among various Oryza sativa L.rice genotypes[J].J Plant Growth Regul,2019,38(1):93-102.
[23]REZKI S,CAMPION C,SIMONEAU P,et al.Assembly of seed-associated microbial communities within and across successive plant generations[J].Plant Soil,2018,422:67-79.
[24]LEVEAU J H J,TECH J J.Grapevine microbiomics:bacterial diversity on grape leaves and berries revealed by high-throughput sequence analysis of 16S rRNA amplicons[C].International Symposium on Biological Control of Postharvest Diseases:Challenges and Opportunities 905,2010:31-42.
[25] RAHMAN M, FLORY E, KOYRO H W, et al.Consistent associations with beneficial bacteria in the seed endosphere of barley(Hordeum vulgare L.)[J].Syst Appl Microbiol,2018,41(4):386-398.
[26]王居伟,韩培杰,王雪薇,等.白酒发酵过程中常见酵母菌扣囊复膜酵母的研究进展[J].广西科学,2020,27(1):1-9.
[27]冯中红,孙广宇.链格孢属及相关属分类研究新进展[J].菌物研究,2020,18(4):294-303.