发酵蔬菜主要是以新鲜蔬菜为主要原料,经盐、糖、醋、酱、虾油和其他调味品腌制,借助蔬菜表面自然粘附的有益微生物或接种纯菌在厌氧环境下进行发酵而成的蔬菜制品[1]。目前国内自然发酵蔬菜代表性的产品有四川涪陵榨菜[2]、四川泡菜[3]、东北酸菜[4]、贵州辣椒酱以及以甘肃兰州和陕西陕南为代表的浆水菜[5]等。发酵蔬菜在我国历史悠久,至今已有三千多年的历史,因其独特的滋味和香气及营养价值而深受欢迎[6-8]。
沙芥(Pugionium cornutum)为十字花科沙芥属二年生草本植物,为我国特有种[9-11]。沙芥是一种药食兼用的沙生野生蔬菜,具有“沙漠人参”的美称[12-13]。沙芥富含粗蛋白、氨基酸、糖、纤维素和矿物质等多种营养成分及甙类、酚类、鞣质、有机酸、皂甙、黄酮、内酯、香豆素、生物碱、甾醇等化学成分[14-16]。据《中药大辞典》记载,沙芥可入中药,具有行气、止痛、消食、解毒,治疗消化不良、胸胁胀满、食物中毒的作用[17-18]。沙芥作为沙漠地区人们喜食的特色野生蔬菜之一,其根、茎、叶均可食用[19]。在鄂尔多斯高原地区有食用沙芥的传统,当地蒙古族在夏、秋季采集沙芥的幼苗及嫩茎叶,做沙芥汤、凉拌或加酸乳清进行腌渍后自然发酵制成发酵沙芥[20]。内蒙古地区沙芥的传统腌制方法根据是否添加乳清情况可分为添加乳清或不添加乳清工艺。杨帆等[21]研究了乳清液对发酵沙芥理化指标及微生物变化的影响,结果表明,添加乳清液较自然发酵具有明显优势,2种发酵方式微生物变化、亚硝酸盐变化趋势一致,但乳清添加组亚硝酸盐含量的峰值早且低于自然发酵组,更具食用安全性;添加乳清发酵液可使乳酸菌快速成为优势菌群,抑制其他杂菌生长,有助于稳定产品质量。马程[22]对沙芥茎叶青汁的乳酸菌发酵工艺进行优化,发现通过沙芥与益生菌的结合,使其活菌数增多,产酸能力增强,从而有利于调节人的消化系统,维持菌群平衡;挥发性风味物质明显增加,沙芥原有辛辣的风味得到改善,更适合广大消费者。目前,腌制过程中发酵沙芥微生物菌群多样性的研究鲜见报道。
本研究以内蒙古农户自制的传统发酵沙芥(添加乳清和未添加乳清)为研究对象,采用Illumina MiSeq高通量测序技术对发酵沙芥样品中的细菌群落结构进行解析,为今后沙芥产品研发以及产业化、工业化生产提供理论和技术支持。
1 材料与方法
1.1 材料与试剂
1.1.1 样品
4份发酵沙芥样品(YZ1、YZ2、RQ1、RQ2):采集于内蒙古鄂尔多斯市乌审旗农户,-80 ℃保存,其中样品RQ1、RQ2中有添加乳清。样品YZ1和RQ1采集自同一家农户,样品YZ2和RQ2采集自同一家农户。
1.1.2 试剂
E.Z.N.A.R Soil DNA Kit脱氧核糖核酸(deoxyribonucleic acid,DNA)抽提试剂盒:美国Omega公司;琼脂糖:西班牙Biowest公司;Fast Pfu Polymerase:中国TransGen公司;AxyPrep DNA Extraction Kit:美国Axygen公司;NEXTFLEX Raoid DNA-Seq Kit建库试剂盒:美国Bioo Scientific公司;MiSeq Reagent Kit v3/NovaSeq Reagent Kits测序试剂盒:美国Illumina公司。
1.2 仪器与设备
JY202电子天平:上海浦春计量仪器有限公司;JL-CJH1B超净工作台:上海靳澜仪器制造有限公司;FastPfu Poly merase:北京全式金生物技术有限公司;ABI GeneAmpR 9700型聚合酶链式反应(polymerase chain reaction,PCR)仪:美国ABI公司;DYY-6C电泳仪:北京六一生物科技有限公司;QuantusTM Fluorometer微型荧光剂:美国Promega公司。
1.3 方法
1.3.1 DNA抽提、PCR扩增与高通量测序
采用E.Z.N.A.R Soil DNA kit试剂盒提取发酵沙芥样品的基因组DNA,以其为模板,使用携带Barcode序列的引物799F(5'-AACMGGATTAGATACCCKG-3')和引物1193R(5'-ACGTCATCCCCACCTTCC-3')对细菌菌群的16SrRNA V3-V4可变区基因序列进行PCR扩增。PCR扩增体系:5×TransStart FastPfu缓冲液4 μL,2.5 mmol/L脱氧核糖核苷三磷酸(deoxy-ribonucleoside triphosphate,dNTPs)2 μL,上游引物(5 μmol/L)0.8 μL,下游引物(5 μmol/L)0.8 μL,TransStart FastPfu DNA聚合酶0.4 μL,模板DNA 10 ng,双蒸水(ddH2O)补足至20 μL。PCR扩增程序:95 ℃预变性3 min,27个循环(95 ℃变性30 s,55 ℃退火30 s,72 ℃延伸30 s),然后72 ℃再延伸10 min,最后在4 ℃进行保存。每个样本3个重复。将同一样本的PCR产物混合后使用2%琼脂糖凝胶检测PCR产物,利用AxyPrepDNA GelExtractionKit回收纯化PCR产物,2%琼脂糖凝胶电泳检测,并用QuantusTMFluorometer对回收产物进行检测定量。PCR扩增产物委托至上海美吉生物医药科技有限公司Miseq PE300平台进行高通量测序。
1.3.2 高通量数据统计分析
所有的数据分析均在美吉生物云平台(https://cloud.majorbio.com)上进行。使用fastp(https://github.com/Open Gene/fastp,version 0.19.6)软件对双端原始测序序列进行质控,使用FLASH(http://www.cbcb.umd.edu/software/flash,version 1.2.11)软件进行拼接;使用UPARSE软件,根据97%的相似度对质控拼接后的序列进行操作分类单元(operational taxonomic unit,OTU)聚类并剔除嵌合体。采用mothur软件计算Alpha多样性指数(超1(Chao 1)、香农(Shannon)指数等);使用基于bray-curtis距离算法的主坐标分析(principal coordinate analysis,PCoA)检验样本间微生物群落结构的相似性等;采用线性判别分析效应大小(linear discriminant analysis effect size,LEfSe)确定不同发酵沙芥样品的差异性物种。
2 结果与分析
2.1 不同发酵沙芥样本高通量测序结果统计
从供试样品中共获得有效序列693 078条,每条序列碱基平均长度为377.52 bp,去除叶绿体和线粒体的序列数量之后,得到658 626条序列,序列碱基平均长度为377 bp。基于97%相似度的分类水平,共注释到93个OTUs,隶属于6个门、8个纲、21个目、37个科和52个属,68个种。
2.2 不同发酵沙芥样品细菌菌群Alpha多样性分析
Alpha多样性指数包括Sobs指数、Chaol指数、Ace指数、Shannon指数和辛普森(Simpson)指数,其中Sobs指数、Chao1指数和Ace指数越大,说明物种丰富度越高,即群落中物种数量越多;Simpson指数越小,Shannon指数越大,说明群落多样性越高,即个体分配越均匀[23]。样本Coverage值表明测序的数据能够覆盖目前状态下样品中细菌的种类,其值越高说明测序深度越高,样本中序列均能被测出,能反映样品真实的菌群情况[24]。由表1可知,4个发酵沙芥样品的Coverage值均>99%,说明测序结果可以真实反映出4种发酵沙芥样品中的细菌群落丰富度及群落多样性。样品YZ2的Sobs指数、Chaol指数和Ace指数均最低,说明其细菌群落丰富度最低,而样品YZ1、RQ1和RQ2的群落丰富度指数差异不大。对于Shannon指数,样品RQ2>RQ1>YZ2>YZ1;对于Simpson指数,样品YZ1>YZ2>RQ1>RQ2,说明添加乳清发酵沙芥样品的细菌群落多样性高于未添加乳清发酵沙芥样品。
表1 发酵沙芥样本中细菌菌群的Alpha多样性分析结果
Table 1 Alpha diversity analysis results of bacteria community in fermented Pugionium cornutum samples
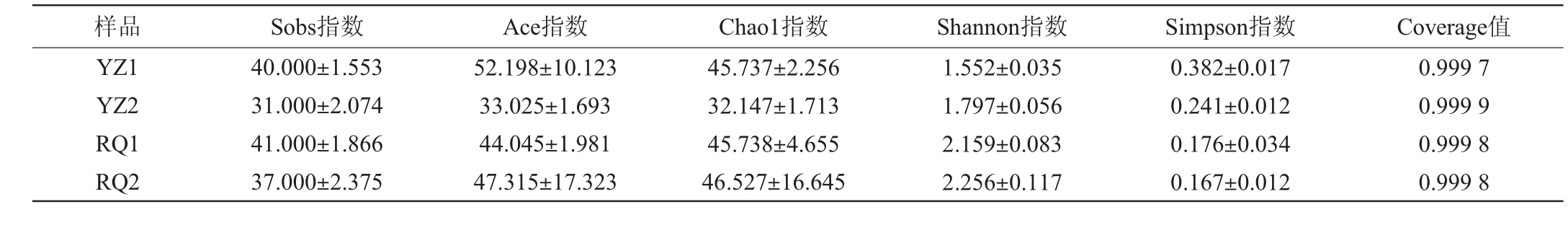
样品 Sobs指数 Ace指数 Chao1指数 Shannon指数 Simpson指数 Coverage值YZ1 YZ2 RQ1 RQ2 40.000±1.553 31.000±2.074 41.000±1.866 37.000±2.375 52.198±10.123 33.025±1.693 44.045±1.981 47.315±17.323 45.737±2.256 32.147±1.713 45.738±4.655 46.527±16.645 0.999 7 0.999 9 0.999 8 0.999 8 1.552±0.035 1.797±0.056 2.159±0.083 2.256±0.117 0.382±0.017 0.241±0.012 0.176±0.034 0.167±0.012
为了进一步证明样品数量的合理性,基于OTU水平绘制Sobs指数及Shannon指数稀释曲线,结果见图1。
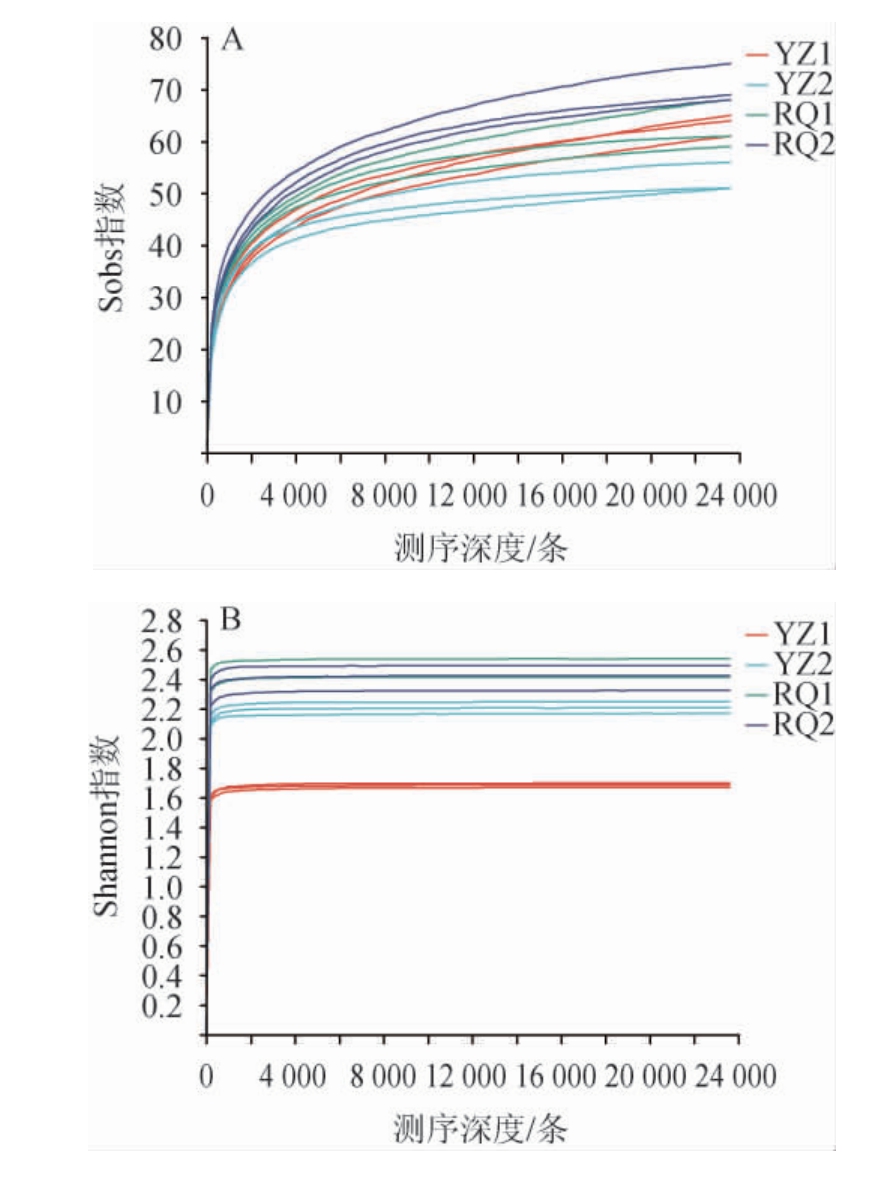
图1 基于OTU水平不同发酵沙芥样品细菌群落的Sobs指数(A)及Shannon指数(B)稀释曲线
Fig.1 Dilution curves of Sobs (A) and Shannon index (B) of bacteria community in different fermented Pugionium cornutum samples based on OTU level
由图1可知,随着测序深度的增加,Sobs指数及Shannon指数逐渐平缓,说明4种样本测序数量足够、合理,能够真实反映4种发酵沙芥的细菌群落组成结构。为了表明样品物种的多样性绘制Rank Abundance图,结果见图2。由图2可知,Rank Abundance能直观地反映样品中物种的丰富度和均匀度,水平方向的曲线跨度体现了物种丰富度,曲线在横轴上的范围较大,表明物种丰度较高;垂直方向的曲线渐进平缓,表明物种分布均匀[25]。结果表明,曲线在横轴上的范围较大且渐进平缓,表明4种发酵沙芥样品的物种丰富度较高以及物种分布均匀。
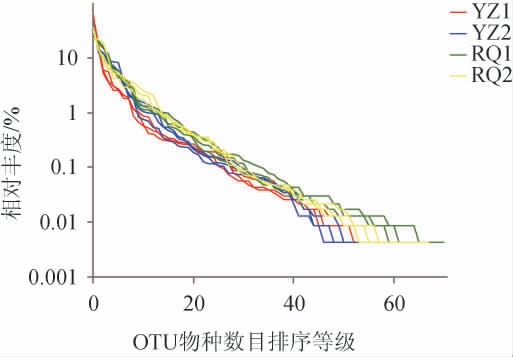
图2 基于OTU水平不同发酵沙芥样品细菌菌落Rank abundance曲线图
Fig.2 Rank abundance curves of bacteria community in different fermented Pugionium cornutum samples based on OTU level
2.3 不同发酵沙芥样品细菌菌群结构分析
2.3.1 韦恩图分析
由图3可知,在97%的序列相似度下,4种发酵沙芥样品的细菌菌群共划分为93个OTUs,其中,样品YZ1、YZ2、RQ1和RQ2的细菌OTU数分别为76个、64个、79个和70个。4种发酵沙芥样品共有的OTU数为47个。YZ1样本中没有特有的OTU,而YZ2、RQ1和RQ2样品中特有的OTU数分别为3个、4个、4个。除共有物种和特有物种外,不同样品间均有相同或相似的物种。综上,各样品共有物种较多,特有物种较少,且添加乳清发酵沙芥中的特有物种数量大于未添加乳清发酵沙芥。
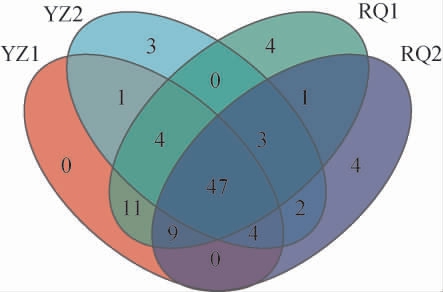
图3 不同发酵沙芥样品细菌群落OTU韦恩图
Fig.3 OTU venn diagram of bacterial community in different fermented Pugionium cornutum samples
2.3.2 菌群结构分析
由图4可知,从4个发酵沙芥样品中共注释到6个细菌门,平均相对丰度较高的细菌门分别为厚壁菌门(Firmicutes)(73.13%)、变形菌门(Proteobacteria)(16.02%)、放线菌门(Actinobacteriota)(10.62%)。4个发酵沙芥样品中厚壁菌门(Firmicutes)的相对丰度均最高,在样品YZ1、YZ2、RQ1、RQ2中的相对丰度分别为90.89%、83.61%、65.30%、52.71%,而变形菌门(Proteobacteria)的相对丰度均较低,分别为8.50%、8.26%、33.52%、13.87%。添加乳清的发酵沙芥较未添加乳清的发酵沙芥厚壁菌门的相对丰度较小,但变形菌门的相对丰度较高。
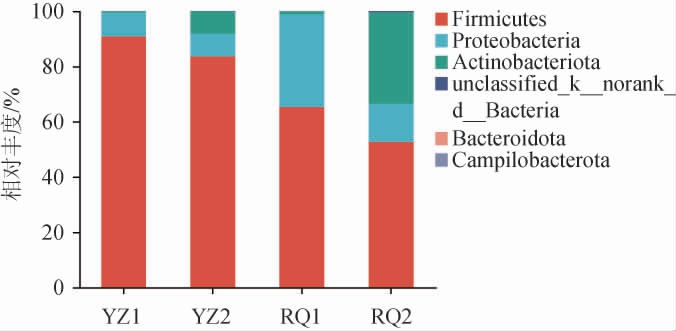
图4 基于门水平不同发酵沙芥样品细菌菌群结构
Fig.4 Bacterial community structure of different fermented Pugionium cornutum samples based on phylum level
others:平均相对丰度<1%的细菌门。
由图5可知,从4个发酵沙芥样品中共注释到52个细菌属,平均相对丰度较高的细菌属有葡萄球菌属(Staphylococcus)(19.56%)、乳酸杆菌属(Lactobacillus)(17.08%)、海洋芽孢杆菌属(Oceanobacillus)(12.13%)、魏斯氏菌属(Weissella)(10.53%)、棒状杆菌属(Corynebacterium)(9.25%)。不同发酵沙芥样本中细菌属相对丰度大小存在差异。YZ1样品中乳酸杆菌属(Lactobacillus)相对丰度较高(58.87%),YZ2和RQ1样品中葡萄球菌属(Staphylococcus)相对丰度较高,分别为40.50%、29.50%。RQ2样品中海洋芽孢杆菌属(Oceanobacillus)相对丰度较高(29.01%)。
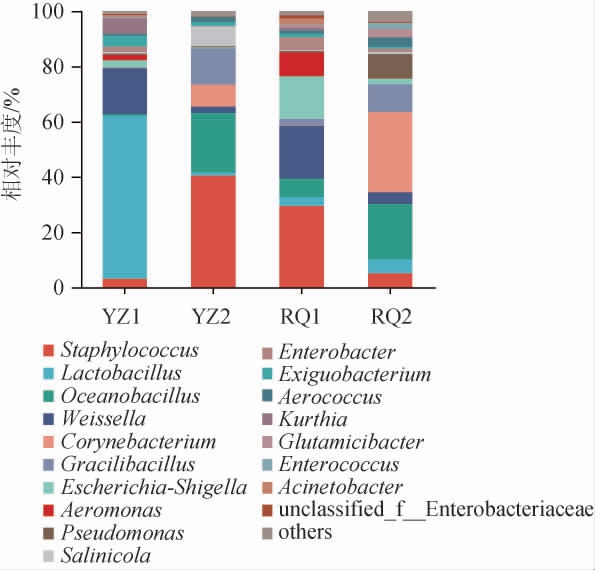
图5 基于属水平不同发酵沙芥样品细菌菌群结构
Fig.5 Bacterial community structure of different fermented Pugionium cornutum samples based on genus level
others:平均相对丰度排名18之后的细菌属。
2.4 不同发酵沙芥样品细菌菌群聚类分析
基于OTU水平对4个发酵沙芥样品中细菌群落的差异进行PCoA,结果见图6。
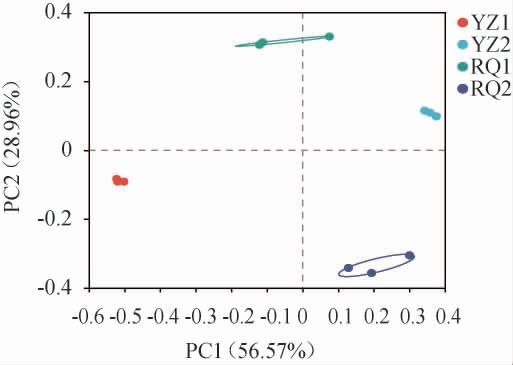
图6 基于OTU水平不同发酵沙芥样品主坐标分析结果
Fig.6 Principal coordinate analysis results of different fermented Pugionium cornutum samples based on OTU level
由图6可知,PCo1的方差贡献率为56.57%,PCo2的方差贡献率为28.96%,累积方差贡献率为85.53%,说明两个主要成分能够解释85.53%的细菌OTU差异,能很好的区分4种发酵沙芥细菌群落分布差异情况。由图6亦可知,4种发酵沙芥样品分布于不同象限,均各自聚类,表明4种发酵沙芥细菌群落组成具有明显差异。样品YZ1和RQ1之间距离较近、样品YZ2和RQ2之间距离较近,表明样品YZ1和RQ1的细菌菌落组成较为相似,以及样品YZ2和RQ2细菌菌落组成较为相似。可能由于样品YZ1和RQ1、YZ2和RQ2分别取自同一家农户自制,造成其细菌菌落组成的相似。
2.5 不同发酵沙芥样品细菌菌群的LEfSe分析结果
图7中显示线性判别分析分值大于设定值(4.0)的物种,不同颜色表示不同样品的物种,柱状图的长度代表差异物种的影响大小。由图7可知,4种发酵沙芥样品中含有16种差异性细菌属,其中,YZ1样品中含有乳杆菌属(Lactobacillus)、微小杆菌属(Exiguobacterium),YZ2样品中含有葡萄球菌属(Staphylococcus)、海洋芽孢杆菌属(Oceanobacillus)、纤细芽孢杆菌属(Gracilibacillus)、短芽孢杆菌属(Brevibacterium)、Marinilactibacillus,样品RQ1中含有棒状杆菌属(Corynebacterium)、谷氨酸杆菌属(Glutamicibacter)、气球菌属(Aerococcus)、肠球菌属(Enterococcus),RQ2样品中含有魏斯氏菌属(Weissella)、志贺氏菌属(Escherichia-Shigella)、气单胞菌属(Aeromonas)、肠杆菌属(Enterobacter)、不动杆菌属(Acinetobacter)。
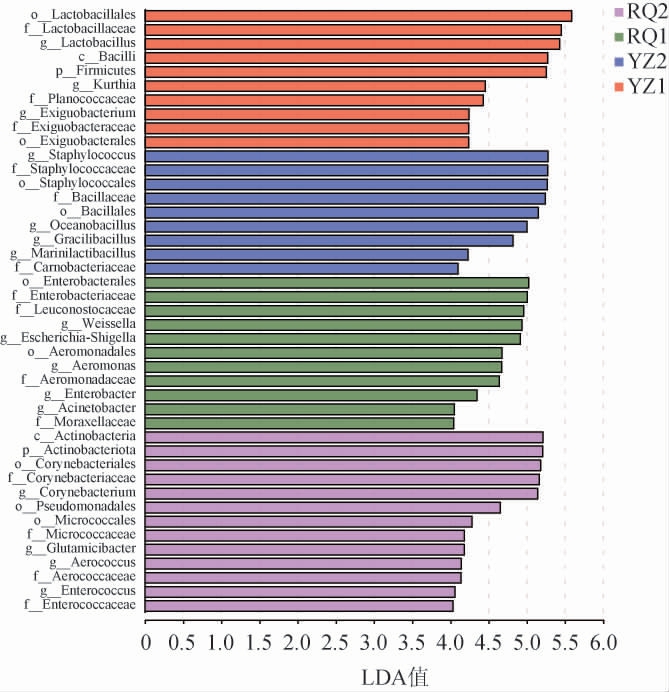
图7 不同发酵沙芥样品细菌菌群的LEfSe分析结果
Fig.7 LEfSe analysis results of bacterial community in different fermented Pugionium cornutum samples
3 结论
本研究从4个发酵沙芥样品中细菌菌群共获得693 078条有效序列,注释到93个操作分类单元(OTUs),隶属于6个门、8个纲、21个目、37个科、52个属、68个种。4个样品中相对丰度最高的细菌门均为厚壁菌门(Firmicutes),而各样品中细菌属相对丰度各有差异,YZ1、YZ2、RQ1、RQ2样品中相对丰度最高的细菌属分别为Lactobacillus、Staphylococcus、葡萄球菌属、Oceanobacillus。PCoA发现4种发酵沙芥样品细菌群落组成具有明显差异。LEfSe分析结果显示,4种发酵沙芥中含有16种差异性细菌属。与未添加乳清发酵沙芥样品(YZ1、YZ2)相比,添加乳清发酵沙芥样品(RQ1、RQ2)的细菌菌群多样性及特有物种数较高,且变形菌门(Proteobacteria)相对丰度较高,但厚壁菌门(Firmicutes)相对丰度较小。综上,添加乳清可使发酵沙芥细菌群落结构发生改变。本研究结果将加强对发酵沙芥中复杂细菌组成和微生物群变异的理解,这些资源对沙芥发酵产品的开发具有潜在的应用价值。
[1]AN F Y,SUN H J,WU J R,et al.Investigating the core microbiota and its influencing factors in traditional Chinese pickles[J].Food Res Int,2021,147(6):110543-110550.
[2]郑恒光,陈君琛,汤葆莎,等.中外蔬菜发酵加工技术的对比研究[J].安徽农业科学,2018,46(5):171-175.
[3]李雪,和兴萍,陈丹,等.乳酸菌发酵混合蔬菜的初步研究[J].中国酿造,2016,35(9):176-179.
[4]汪铃,王亮.传统发酵蔬菜制作工艺、品质特征及影响因素研究概况[J].中国酿造,2022,41(7):21-25.
[5]向书娅,翟茹,张海燕,等.不同地区发酵浆水中微生物群落结构比较及优势菌群的鉴定[J].现代食品科技,2023,39(3):121-128.
[6]李子怡,邓淼,王燕,等.发酵蔬菜中的挥发性成分及其影响因素探讨[J].中国酿造,2022,41(12):26-30.
[7]侯强川,郭壮,张家超,等.俄罗斯卡尔梅克共和国发酵蔬菜中细菌多样性研究[J].食品与发酵工业,2014,40(7):16-22.
[8]侯小艺,王建辉,邓娜,等.乳酸菌对发酵蔬菜风味影响研究进展[J].食品与机械,2023,39(4):232-240.
[9]高子舒,徐康宁,乌仁格格.沙芥的研究现状分析[J].广东化工,2018,45(8):138-157.
[10]秦鑫愿,常仲林,黄修梅,等.沙芥属植物花粉和果实形态特征研究[J].北方农业学报,2022,50(4):109-115.
[11]于晓婧,王萍,武兆昕,等.沙芥幼苗叶片小热激蛋白基因的克隆与表达[J].湖南农业大学学报(自然科学版),2021,47(6):669-676.
[12]张东,郑清岭,郝丽珍,等.自然脱水对沙芥种子抗氧化系统的影响[J].种子,2018,37(11):64-68.
[13]鲍红春,郝丽珍,张凤兰,等.沙芥果皮浸提液对白菜种子化感作用及其化感物质分析[J].北方农业学报,2019,47(4):8-14.
[14]张凤兰,杨忠仁,郝丽珍,等.5种野生蔬菜叶片营养成分分析[J].华北农学报,2009,24(2):164-169.
[15] HAOYU L, LI C, ZHAN C, et al.Compositional and gastrointestinal prokinetic studies of Pugionium(L.)[J].Food Chem,2015,186:285-291.
[16]胡和珠拉,财吉拉胡.药食同源之沙芥的研究进展[J].内蒙古中医药,2022,41(10):160-161.
[17]江苏新医学院.中药大辞典[M].上海:上海人民出版社,1977:1162.
[18]袁学松.肉桂酸处理对沙芥属幼苗生理生化基础的影响[D].呼和浩特:内蒙古农业大学,2019.
[19]许灵媛,张丹阳,吕博威,等.沙生蒙药沙芥生物活性及应用研究进展[J].农产品加工,2020(18):79-83.
[20]额尔德木图.鄂尔多斯高原地区蒙古族民间野生食用植物的调查研究[D].呼和浩特:内蒙古师范大学,2007.
[21]杨帆,刘学勤.乳清液对发酵沙芥理化指标及微生物变化的影响[J].安徽农业科学,2018,46(35):159-161.
[22]马程.乳酸菌发酵沙芥茎叶青汁的工艺条件优化及其挥发性风味物质的变化研究[D].呼和浩特:内蒙古农业大学,2022.
[23]许艺,李红,巩雪峰,等.基于高通量测序分析辣椒/玉米套作对辣椒根际土壤细菌多样性的影响[J].西南农业学报,2022,35(4):772-779.
[24]张守梅,郭玉秋,张娜娜,等.基于高通量测序分析玉米浸泡副产物中细菌群落结构和多样性[J].山东农业科学,2021,53(12):143-148.
[25]马静,张琳琳,柴沙驼,等.基于高通量测序技术分析青藏高原牦牛和犏牛乳中微生物多样性的研究[J].食品工业科技,2021,42(9):122-128.