菌草最早是1986年由福建农林大学林占熺研究员提出的[1]。目前,根据三级系统筛选法已经获得菌草品种45种,主要为巨菌草、柠檬香茅、紫象草、五节芒、绿洲1号等[2]。巨菌草(Cenchrus fungigraminus Z.X.Lin&D.M.Lin&S.R.Lan sp.Nov.)具有产量高、纤维、粗蛋白和糖分含量高的特点[3]。柠檬香茅(Cymbopogon citratus Stapf)含有丰富的多糖、多酚、黄酮类及香茅精油[4]。师静等[5]通过对巨菌草纤维素的酶解条件进行研究,发现巨菌草酶解后含有丰富的还原糖,可制备乙醇。童金华[6]以巨菌草、柠檬香茅、紫花苜蓿等为主要原料,成功研制了菌草复合饮料。这些研究为巨菌草和柠檬香茅作为菌草酒的发酵原料提供了理论参考。范锦琳等[7]采用巨菌草和柠檬香茅为酿造原料,以安琪白酒曲为糖化发酵剂,采用固态发酵法发酵,经过多次蒸馏后得到具有独特风味的菌草酒。
我国白酒的发酵主要通过自然富集和混菌发酵,大量研究表明,自然发酵过程中复杂的自然菌群对成品酒的酿造具有积极作用[8-11]。目前,针对发酵过程中菌草酒细菌菌群组成及变化鲜有研究。杨旭等[12]采用高通量测序技术,探究大曲细菌菌群结构,发现细菌组成的差异性是导致白酒品质差异的主要原因之一。王春艳等[13]运用高通量测序技术,研究宋河浓香型白酒不同窖龄窖泥细菌菌群结构,发现窖龄越久细菌菌落结构越复杂。可见微生物在白酒的酿造中发挥着极其重要的作用,不同菌群微生物在酿酒过程中的活动会影响白酒的品质[14]。因此,研究菌草酒发酵过程中的自然菌群,阐明其群落结构及组成,是后续深入研究菌草酒酒体特征形成机理的基础,对提高菌草酒的出酒率、改善菌草酒的品质至关重要[15]。
高通量测序技术能够同时对几十万甚至几百万条脱氧核糖核酸(deoxyribonucleic acid,DNA)序列进行测定,具有精确性高、重复性好,无需建库、无克隆误差,速度快、测速时间短等优点,被广泛应用于酿酒微生物的研究[16-20]。本研究首次利用高通量测序技术探究菌草酒酿造过程中细菌菌群的变化。通过探究菌草酒酿造过程中酒醅的细菌菌群结构变化,为建立菌草酒微生物信息数据库、探究后续菌草酒发酵机理、提升菌草酒品质提供科学依据。
1 材料与方法
1.1 材料与试剂
巨菌草、柠檬香茅:由福建农林大学旗山校区菌草中心基地提供;白酒曲:安琪酵母股份有限公司;白砂糖:市售;Ezup柱式土壤基因组DNA抽提试剂盒:上海生工生物工程有限公司;无水乙醇:上海阿拉丁试剂有限公司;琼脂糖:西班牙Biowest Agarose公司;DNA Marker:大连Takara公司;Axy Prep DNA凝胶回收试剂盒:美国Axygen公司;Gengreen染料:上海赛百盛有限公司;引物:北京奥维森基因科技有限公司。本研究所用试剂均为分析纯或生化试剂。
1.2 仪器与设备
IlluminaMiseqPE250高通量测序仪:美国Illumina公司;T100 Thermal Cycler聚合酶链式反应(polymerase chain reaction,PCR)仪:美国BIO-RAD公司;InvitrogenQubit3.0荧光计:美国Life公司;ZF-288凝胶成像系统:美国UVP公司。
1.3 方法
1.3.1 酒醅样品制备及预处理
新鲜巨菌草、柠檬香茅经过清洗和切分,在发酵温度为29 ℃,初始糖度为24°Bx,酒曲接种量为0.45 g/L的条件下自然发酵30 d[7]。分别采集0、0.5 d、1 d、2 d、3 d、5 d、7 d、10 d、15 d、21 d和30 d的酒醅样品,标记为D0、D0.5、D1、D2、D3、D5、D7、D10、D15、D21和D30。在超净工作台上,从发酵罐的上、中、下层三个不同的位置分别取50 g酒醅,进行等量混匀为一个样品,并且置于无菌采样袋密封。样品采集后迅速置于盛有干冰的泡沫箱中快速冷冻,于-80 ℃的低温环境中保存[21]。
1.3.2 酒醅样品细菌菌群基因组DNA的提取、PCR扩增及高通量测序
参照DNA Kit基因组DNA抽提试剂盒说明书提取酒醅样品细菌菌群的基因组DNA,以其为模板,采用引物338F(5'-GTACTCCTACGGGAGGCAGCA-3')和引物806R(5'-GTGGACTACHVGGGTWTCTAAT-3')PCR扩增细菌菌群的16S rDNA V3~V4区基因序列。PCR扩增体系(50 μL):10×PCR buffer 5 μL,脱氧核糖核苷三磷酸(deoxy-ribonucleoside triphosphate,dNTP)(10 mmol/L)0.5 μL,DNA 10 ng,上下游引物(50 μmol/L)各0.5 μL,Plantium Taq(5 U/μL)0.5 μL,加双蒸水(ddH2O)到50 μL。PCR扩增参数:95 ℃预变性5 min;95 ℃变性45 s,55 ℃退火50 s,72 ℃延伸45 s,28个循环;72 ℃再延伸10 min。PCR扩增产物用2%的琼脂糖凝胶电泳检测,使用Axy Prep DNA凝胶回收试剂盒回收PCR扩增产物,Tris-HCl洗脱,2%琼脂糖电泳检测,凝胶成像观察。对所有DNA样本等量混合后,依托北京奥维森基因科技有限公司进行Illumina Miseq高通量测序。
1.3.3 数据分析
使用FLASH(V1.2.7)和UCHIME Algorithm软件(http://www.drive5.com/usearch/manual/uchime_algo.html)对测序序列进行质控,得到有效序列[22-23]。通过Uparse软件(http://drive5.com/uparse/)将序列按照相似性>97%阈值进行操作分类单元(operational taxonomic unit,OTU)聚类,利用RDP Classifier与GreenGene数据库对OTUs代表序列进行物种注释(阈值为0.8~1.0)[24-25]。利用PyNAST软件(V1.2)与GreenGene数据库中“Core Set”数据信息进行多序列比对,得到所有OTUs代表序列的系统发生关系[26]。基于每个样品的Alpha多样性指数分析,用稀释曲线、超1(Chao1)指数、香农(Shannon)指数等来评估样品中微生物群落的物种丰度和多样性[27-28]。通过Beta多样性分析样品之间群落组成的相异性[29]。
2 结果与分析
2.1 高通量测序结果及Alpha多样性分析
稀释曲线的变化趋势可以直观反映出测序数据量与鉴定得到物种数量的相关性,在一定程度上可以反映实验测序量是否能充分覆盖样品中的实际物种数量[30-31]。稀释曲线后期逐渐平稳,表明测序数据量已充分覆盖样品中的物种[32]。各酒醅样品的稀释曲线见图1。
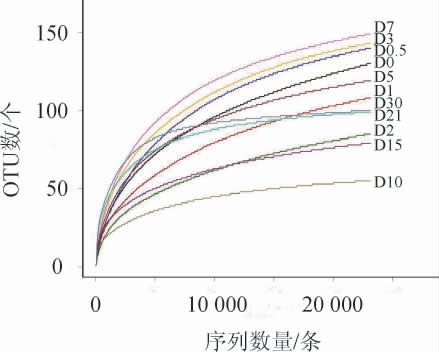
图1 不同发酵时期酒醅样品的稀释曲线
Fig.1 Rarefaction curves of fermented grains samples at different fermentation periods
由图1可知,11条稀释曲线在测序量达到20 000条以后均趋向平坦,说明不同发酵时期酒醅样品测序数据量合理,包含了大多数菌群,能够比较真实地反映样本的微生物群落多样性。从11个酒醅样品中共注释到14个门、32个纲、62个目、89个科、117个属。序列统计和Alpha多样性评估见表1。由表1可知,通过对测序所得的序列进行预处理,去除低质量序列和模糊序列,从不同发酵时期的11个酒醅样品中共得到406 080条原始序列,过滤后得到382 048条有效序列。11个酒醅样品文库的覆盖率均>99.70%,说明测序结果可以真实反映样本内的细菌实际存在情况。通过归类共得到1 343个OTU。由表1亦可知,菌草酒发酵过程中细菌菌群微生物的Alpha多样性指数均呈波动式变化。OTUs和Chao1指数整体均呈发酵前期(0~7 d)>发酵末期(15~30 d)>发酵中期(7~15 d)的规律,且均在发酵10 d时最低。可能是由于发酵10 d时酒精度达到最高,许多微生物耐受力较弱,引起这些指标急剧下降[33]。发酵10 d后,这些指标呈缓慢上升趋势,可能与发酵环境中的氧气浓度、酒精度和有机酸含量的变化有关,导致菌群数量发生变化[34]。而细菌菌群的Shannon指数总体表现出发酵末期>发酵中期>发酵前期的趋势。发酵2 d时Shannon指数最小,表明菌群多样性在发酵2 d时最小。菌草酒发酵过程中细菌菌群丰富度和多样性在发酵的前2 d呈下降趋势,发酵的第2天到第7天逐渐上升,第10天又急剧下降,之后开始缓慢上升至趋于稳定。推测可能是由于发酵初期酒精度增加较快,抑制了部分微生物的生长繁殖,到发酵第2天时大部分微生物逐渐适应同时开始缓慢生长[35]。结果表明,菌草酒发酵过程中细菌菌群在不断调整和平衡。
表1 各样本的测序数据、操作分类单元及多样性指数统计
Table 1 Sequencing data, operational taxonomic units and diversity index statistics of each sample
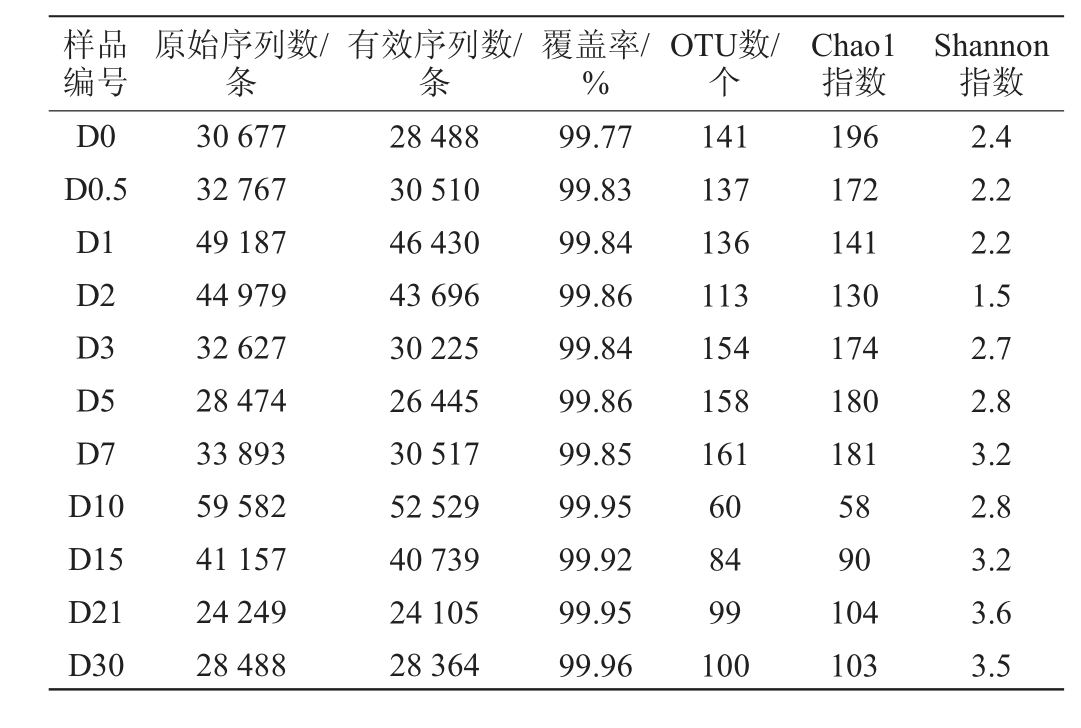
样品编号原始序列数/条有效序列数/条覆盖率/%OTU数/个Chao1指数Shannon指数D0 D0.5 D1 D2 D3 D5 D7 D10 D15 D21 D30 30 677 32 767 49 187 44 979 32 627 28 474 33 893 59 582 41 157 24 249 28 488 28 488 30 510 46 430 43 696 30 225 26 445 30 517 52 529 40 739 24 105 28 364 99.77 99.83 99.84 99.86 99.84 99.86 99.85 99.95 99.92 99.95 99.96 141 137 136 113 154 158 161 60 84 99 100 196 172 141 130 174 180 181 58 90 104 103 2.4 2.2 2.2 1.5 2.7 2.8 3.2 2.8 3.2 3.6 3.5
2.2 菌草酒发酵过程酒醅细菌菌群结构分析
2.2.1 基于门水平菌草酒不同发酵过程酒醅细菌菌群结构分析
基于门水平菌草酒不同发酵过程酒醅细菌菌群结构结果见图2。由图2可知,从酒醅样品中共注释到14个细菌门,包括3个优势细菌门(平均相对丰度>1.00%),分别为厚壁菌门(Firmicutes)(39.48%)、蓝藻菌门(Cyanobacteria)(38.96%)、变形菌门(Proteobacteria)(20.79%)。各酒醅样品中这3类细菌门的相对丰度之和均>98%,充分说明在门水平上,菌草酒不同发酵过程酒醅的细菌菌群结构具有稳定性。发酵前期主要以蓝藻菌门(Cyanobacteria)为绝对优势菌群,发酵中后期主要以厚壁菌门(Firmicutes)为绝对优势菌群。厚壁菌门(Firmicutes)随着发酵时间逐渐增加相对丰度整体呈增加趋势,其中发酵第0天时相对丰度最低,为8.43%;发酵第10天时相对丰度最高,为77.14%。蓝藻菌门(Cyanobacteria)相对丰度整体呈减少趋势,其中发酵第0天时相对丰度最高,为72.10%,发酵第30天时相对丰度最低,为2.77%。变形菌门(Proteobacteria)相对丰度变化幅度相对较小,相对丰度在10.59%~31.04%之间浮动,表明发酵环境抑制其生长繁殖。
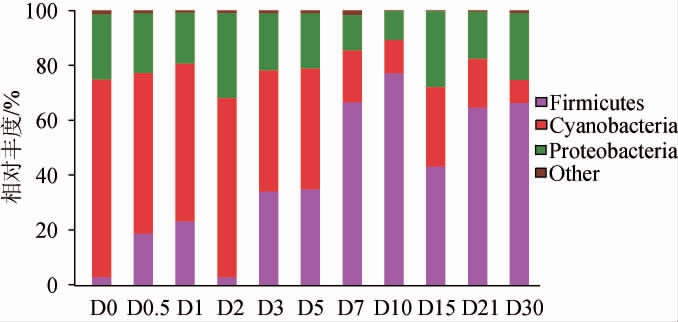
图2 基于门水平菌草酒发酵过程中酒醅样品细菌菌群结构
Fig.2 Bacterial community structure of fermented grains samples during the fermentation process of Juncao wine based on phylum level
Other表示平均相对丰度<1.00%的细菌门。
2.2.2 基于属水平菌草酒不同发酵过程酒醅细菌菌群结构分析
基于属水平菌草酒不同发酵过程酒醅细菌菌群结构结果见图3。由图3可知,从酒醅样品中共注释到117个细菌属,包括7个优势细菌属(平均相对丰度>1.00%),分别为未知菌属(unidentified)(52.37%)、片球菌属(Pediococcus)(13.85%)、魏斯氏菌属(Weissella)(12.73%)、乳酸杆菌属(Lactobacillus)(9.66%)、乳球菌属(Lactococcus)(4.81%)、泛生菌属(Pantoea)(1.78%)、芽孢杆菌属(Bacillus)(1.53%),不同酒醅样品中优势细菌属的相对丰度之和为95.29%~99.74%,而其他细菌属的相对丰度之和<5%。
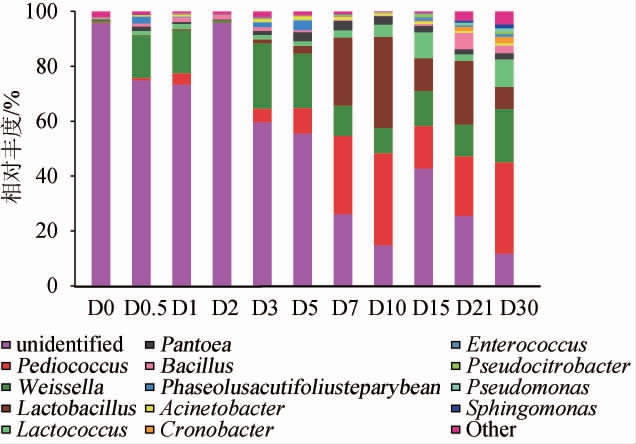
图3 基于属水平菌草酒发酵过程中酒醅样品细菌菌群结构
Fig.3 Bacterial community structure of fermented grains samples during the fermentation process of Juncao wine based on genus level
Other表示相对丰度<1.00%的细菌属。
由图3亦可知,在整个发酵期均含有未知菌属(unidentified),其中在发酵前期相对丰度较高,可能是由于采用生料发酵酿造,而菌草原料自身含有丰富的细菌菌群。随着发酵时间的延长,未知菌属(unidentified)相对丰度逐渐下降,分析原因可能与相关微生物不耐酸,对酒精的耐受力也比较低有关[36]。片球菌属(Pediococcus)在发酵前期相对丰度较低,发酵第5天到发酵第7天,相对丰度大幅提高,从9.33%迅速上升到28.48%,发酵第10天相对丰度达到最高值,为33.51%。分析其变化的原因可能是因为片球菌属(Pediococcus)为兼性厌氧菌,因此在发酵中后期通常相对丰度较高[37]。魏斯氏菌属(Weissella)在整个发酵过程中相对丰度较高,整体呈先升高后降低趋势,发酵第3天时相对丰度达到最高,为23.72%。菌草酒酒醅中的乳酸杆菌属(Lactobacillus)在发酵初期相对丰度极低,发酵中后期相对丰度大幅增长,发酵第10天时相对丰度达到最大值,为33.10%。王海燕[38]研究发现,乳酸菌对不同环境的适应能力较强,因此通常在发酵过程中占据优势地位。王群[39]研究发现,乳酸杆菌属(Lactobacillus)多数为兼性厌氧菌,所以在发酵中后期通常相对丰度较高,与本研究结果一致,因此推测菌草酒的乳酸等有机酸可能主要是在发酵中后期生成的。综上,菌草酒发酵过程中酒醅的微生物菌群变化是动态的。
2.3 基于属水平菌草酒发酵过程中酒醅样品的主成分分析
基于属水平,通过R软件对菌草酒发酵过程中酒醅样品进行主成分分析(principal component analysis,PCA),结果见图4。由图4可知,第1主成分和第2主成分的累计方差贡献率为83.88%。说明两主成分可以解释发酵过程中细菌菌群变化的83.88%。由图4亦可知,发酵0 d和2 d的样本距离较为接近;发酵0.5 d、1 d、3 d和5 d的样本距离较为接近;发酵7 d、10 d、15 d和21 d的样本遗传距离较为接近;发酵30 d的样本与其他样品距离最远。说明不同发酵时期菌草酒酒醅样品的细菌菌群相似程度具有阶段性,且与发酵时间有关。
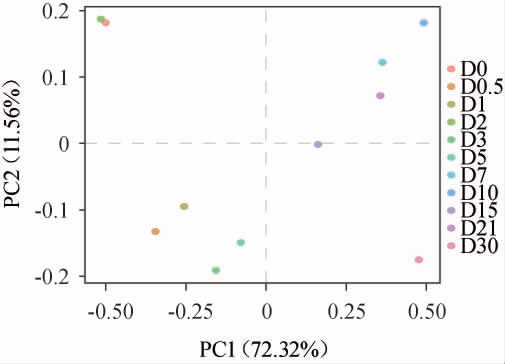
图4 基于属水平菌草酒发酵过程中酒醅样品的主成分分析结果
Fig.4 Results of principal component analysis of fermented grains samples during the fermentation process of Juncao wine based on genus level
3 讨论
本研究通过高通量测序技术,对菌草酒发酵过程中11个不同发酵时期酒醅样本的细菌群落进行测序,初步解析了菌草酒自然发酵过程中细菌菌群结构及多样性。结果表明,发酵过程中菌草酒酒醅的微生物菌群结构会随发酵时间发生动态变化,细菌菌群组成及丰度均存在较大差异。
在门水平上,发酵前期菌群组成较丰富,且厚壁菌门(Firmicutes)、变形菌门(Proteobacteria)和蓝藻菌门(Cyanobacteria)是菌草酒酒醅中所有分离样本的优势细菌门类。这与姜丽[40]对糯米酒发酵过程中的细菌菌群结构研究结果一致。邓杰等[41]利用高通量测序技术分析浓香型白酒窖池细菌微生物菌群结构,发现厚壁菌门(Firmicutes)为绝对优势菌群,相对丰度为79.00%,放线菌门(Actinobacteria)相对丰度为4.90%,拟杆菌门(Bacteroidetes)相对丰度为4.10%,黏胶球形菌门(Lentisphaerae)相对丰度为3.50%,与本研究的结果基本一致。
在属水平上,发现除了大部分可培养的微生物外,菌草酒酒醅中还存在着大量的未知微生物,尤其在发酵初期大量的未知菌群占绝对优势。张源等[42]通过高通量测序技术分析草莓酒自然发酵过程中的细菌群落的变化,发现未知菌属(unidentified)与酒体特征和风味物质的形成有关。其他相对丰度较高的细菌属包括片球菌属(Pediococcus)、魏斯氏菌属(Weissella)和乳酸杆菌属(Lactobacillus)。该结果与刘延波等[43]对不同储藏期浓香型白酒大曲的微生物多样性分析结果基本一致。菌草酒酒醅中的乳酸杆菌属(Lactobacillus)在发酵过程中的相对丰度有所不同,呈先上升后下降的趋势。乳酸杆菌属(Lactobacillus)在发酵过程中,主要参与糖类的代谢,进一步生成乳酸,还能以乳酸为底物产生乙酸、琥珀酸、丙酸和丁酸等其他有机酸,而这些有机酸通过酯化反应生成相应的乙酯类化合物[44]。李莹莹等[45]通过研究葡萄酒乳酸菌发酵剂,发现乳酸杆菌在连续的胁迫环境下也能很快地适应,通过丙酮酸代谢途径生成大量乳酸等有机酸,对葡萄酒的风味具有积极的促进作用。张煜晨等[46]通过研究苹果酸-乳酸发酵中乳酸菌的分离及其对葡萄酒香气成分的影响,发现乳酸菌能显著增加香气物质的种类和含量,赋予葡萄酒果香味、玫瑰味、脂肪味、咖啡香味和茴香味。HONG X T等[47]研究发现,在黄酒发酵初期,黄酒腐败主要是由短乳杆菌(Lactobacillus brevis)的大规模生长引起的。综上可见乳酸杆菌属对酒的发酵风味至关重要。BOKULICH N A等[48]通过对绍兴黄酒发酵过程中微生物菌群变化规律进行研究,发现发酵初期主要是以葡萄球菌属(Staphylococcus)和芽孢杆菌属(Bacillus)为主,随着发酵过程的进行,逐渐转变乳酸杆菌属(Lactobacillus)为优势菌群,与本研究结果一致。黄和强等[49]运用高通量测序技术研究夏季青稞酒发酵过程中的微生物菌群结构和多样性,发现酒醅中的魏斯氏菌属(Weissella)和泛生菌属(Pantoea)在整个发酵过程占主导地位,乳酸菌属(Lactobacillus)从发酵第5天开始相对丰度逐渐增加,在发酵末期成为绝对优势菌属,与本研究结果基本一致。刘选明等[50]从香茅草内分离鉴定出泛生菌属的菠萝泛菌(Pantoea ananatis)可产生以柠檬醛为主要成分的萜类化合物,推测本研究结果中泛生菌属(Pantoea)为优势菌属之一,可能与采用柠檬香草为原料有关,且该类微生物代谢的香类物质可能与菌草酒的独特风味形成有关。
4 结论
本研究通过高通量测序技术对菌草酒不同发酵过程酒醅样品中的细菌群落结构进行多样性分析。Alpha多样性分析结果表明,菌草酒发酵过程中细菌菌群在不断调整和平衡。菌群结构分析表明,从酒醅样品中共注释到14个门、32个纲、62个目、89个科、117个属。在门水平上,酒醅样品的优势细菌门(平均相对丰度>1%)为厚壁菌门(Firmicutes)、蓝藻菌门(Cyanobacteria)和变形菌门(Proteobacteria),其在各样品中的对丰度总和均>98%。发酵前期主要以蓝藻菌门(Cyanobacteria)为绝对优势菌群,发酵中后期主要以厚壁菌门(Firmicutes)为绝对优势菌群。在属水平上,酒醅样品的优势细菌属(平均相对丰度>1%)为未知菌属(unidentified)(52.37%)、片球菌属(Pediococcus)(13.85%)、魏斯氏菌属(Weissella)(12.73%)、乳酸菌属(Lactobacillus)(9.66%)、乳球菌属(Lactococcus)(4.81%)、泛生菌属(Pantoea)(1.78%)、芽孢杆菌(Bacillus)(1.53%)。其中,未知菌属(unidentified)、片球菌属(Pediococcus)和乳酸杆菌属(Lactobacillus)的相对丰度在发酵过程中存在较大差异,说明细菌群落在发酵过程中具有不稳定性。片球菌属(Pediococcus)的相对丰度在发酵过程中整体呈先上升后下降的趋势。乳酸菌属(Lactobacillus)和乳球菌属(Lactococcus)在发酵中后期丰度较高,乳酸菌属(Lactobacillus)在发酵第10天和第21天相对丰度大幅升高,魏斯氏菌属(Weissella)与之相反。芽孢杆菌(Bacillus)丰度变化主要发生在发酵末期。PCA结果表明,发酵0 d和2 d的样品较为接近,发酵0.5 d、1 d、3 d和5 d的样品较为接近,发酵7~21 d的样品较为接近,发酵30 d的样品与其他样本较远,说明菌草酒细菌菌群在发酵过程中样本遗传距离具有明显的阶段性,且与发酵时间有关。本研究首次解析菌草酒发酵过程中细菌的菌群结构及其变化,为后续探究菌草酒的风味物质形成与微生物群落特征之间的关系提供一定的科学依据,为将来筛选菌草酒酿造的优势微生物菌株奠定基础,为菌草酒的现代化、规模化、标准化生产提供新思路和理论依据。
[1]林占熺.菌草学概论[M].第3版.北京:中国农业出版社,2019:1-3.
[2]梅嘉沼,葛红柳,刘翠翠,等.菌草种质资源AFLP 多样性分析[J].热带农业科学,2017,37(11):32-38.
[3]丁铭,白璐,王龙清.巨菌草引进试验及栽培种植技术[J].农村科技,2013(12):60-61.
[4]谷瑶,朱永杰,周丽珠,等.柠檬香茅草总黄酮和多糖含量的测定[J].应用化工,2018,47(4):846-848.
[5]师静,林占熺,林冬梅,等.巨菌草纤维素的酶解条件[J].草业科学,2014,31(4):760-765.
[6]童金华.菌草复合饮料配方的研究[J].农产品加工,2021,13:26-29,33.
[7]范锦琳,张丽丽,张煜隆,等.菌草酒酿造工艺优化及其品质分析[J].福建农林大学学报(自然科学版),2023,52(1):98:105.
[8]陈慧颖,李慧星,许彬,等.高通量测序研究浓香型酒醅的细菌菌群多样性及共变性[J].工业微生物,2021,51(4):35-43.
[9]李欣,王彦华,林静怡,等.高通量测序技术分析酱香型白酒酒醅的微生物多样性[J].福建师范大学学报,2017,33(1):51-59.
[10]刘雪婷,王子媛,刘继明,等.清香型白酒酒醅发酵菌株分离鉴定及细菌群落结构分析[J].中国酿造,2021,40(1):39-43.
[11]李凯敏,付桂明,吴酬飞,等.特香型白酒酿造过程中真核微生物菌群演替[J].食品科学,2017,38(22):131-136.
[12]杨旭,马歌丽,王光路,等.高通量测序分析原香型白酒高温大曲的细菌群落[J].酿酒科技,2020,3:14-17.
[13]王春艳,宋建阳,吕慧鑫,等.基于高通量测序的宋河浓香型白酒不同窖龄窖泥细菌群落结构分析[J].中国酿造,2019,38(9):163-166.
[14]沈才萍,李德林,敖宗华,等.酒醅微生物的分子生物学研究[J].技术与市场,2013,20(6):40-42.
[15]邱并生.混菌发酵对白酒液态发酵效率和风味物质的影响[J].微生物学通报,2014,41(7):1477-1478.
[16]陈一钒,吴余宁,徐春燕,等.基于高通量测序技术分析黄酒微生物多样性的研究进展[J].现代食品,2020,23(11):38-43.
[17]陈申习,张磊,宿智新,等.基于高通量测序技术对清香型和酱香型酒曲细菌群落特征研究[J].酿酒,2022,49(1):42-46.
[18]左乾程,黄永光,郭敏,等.酱香型白酒机械化制曲发酵细菌群落的演替[J].食品科学,2021,42(18):150-156.
[19]刘延波,赵志军,陈黄曌,等.高通量测序技术分析浓香型白酒中温曲和高温曲的细菌群落结构[J].现代食品科技,2018,34(5):229-235.
[20]唐贤华.高通量测序技术初步解析浓香型白酒窖泥细菌群落结构[J].中国酿造,2020,39(1):160-164.
[21] LI X R,MA E B,YAN L Z,et al.Bacterial and fungal diversity in the traditional Chinese liquor fermentation process[J].Int J Food Microbiol,2011,146(1):31-37.
[22] EDGAR R C, HAAS B J, CLEMENTE J C, et al.UCHIME improves sensitivity and speed of chimera detection[J].Bioinformatics,2011,27(16):2194-2200.
[23]HAAS B J,GEVERS D,EARL A M,et al.Chimeric 16S rRNA sequence formation and detection in Sanger and 454-pyrosequenced PCR amplicons[J].Genome Res,2011,21(3):494-504.
[24]WANG Q,GARRITY G M,TIEDJE J M,et al.Naive Bayesian classifier for rapid assignment of rRNA sequences into the new bacterial taxonomy[J].Appl Environ Microbiol,2007,73(16):5261-5267.
[25]DESANTIS T Z,HUGENHHOLTZ P,LARSEN N,et al.Greengenes,a chimera-checked 16S rRNA gene database and workbench compatible with ARB[J].Appl Environ Microbiol,2006,72(7):5069-5072.
[26] CAPORASO J G, BITTINGER K, BUSHMAN F D, et al.PyNAST: a flexible tool for aligning sequences to a template alignment[J].Bioinformatics,2010,26(2):266-267.
[27]张海燕,康三江,张霁红,等.苹果酵素发酵过程中微生物群落与风味物质的相关性分析[J].中国酿造,2022,41(12):110-119.
[28] SHANNON C E.A mathematical theory of communication[J].ACM Sigcomm Comp Com,2001,5(1):3-55.
[29]XU C L,SUN R,QIAO X J,et al.Protective effect of glutamine on intestinal injury and bacterial community in rats exposed to hypobaric hypoxia environment[J].World J Gastroenterol,2014,20(16):4662-4674.
[30]彭宇科.太湖富营养化本体中细菌群落结构特征和功能与蓝藻水华相关性的研究[D].南京:南京大学,2017.
[31]杨婧,李娜娜,刘子恒,等.高粱单宁对清香型大曲白酒发酵过程中真菌群落结构的影响[J].食品科技,2022,47(7):1-9.
[32]陈圣宾,欧阳志云,徐卫华,等.Beta多样性研究进展[J].生物多样性,2010,18(4):323-335.
[33]周志立.绍兴黄酒生麦曲的微生物群落演替驱动力研究与制曲工艺优化[D].无锡:江南大学,2021.
[34]周袁璐,师苑,陆跃乐,等.传统发酵过程微生物互作研究进展[J].中国食品学报,2021,21(11):349-358.
[35]汪江波,周敏,沈永祥,等.白酒机械化酿造过程中主要风味物质的生成规律[J].湖北农业科学,2014,53(22):5499-5503.
[36]彭丽娟,李孟伟,杨承剑,等.不同乳酸菌和酵母菌添加量及发酵天数对甘蔗尾青贮发酵品质及营养价值的影响[J].畜牧与兽医,2022,54(3):21-27.
[37]邢敏钰,杜海,徐岩.芝麻香型白酒发酵过程中乳酸菌多样性及其演替规律[J].微生物学通报,2018,45(1):19-28.
[38]王海燕.PCR-DGGE技术对清香型汾酒微生物群落结构演变规律的研究[D].无锡:江南大学,2014.
[39]王群.芝麻香型白酒用枯草芽孢杆菌脱水菌剂研究[D].济南:齐鲁工业大学,2016.
[40]姜丽.黑糯米酒发酵过程中微生物多样性及风味品质研究[D].贵阳:贵州大学,2020.
[41]邓杰,黄治国,卫春会,等.基于高通量测序的浓香型白酒窖池细菌群落结构分析[J].现代食品科技,2015,31(7):50-55.
[42]张源,兰伟,王明跃,等.基于高通量测序的草莓酒发酵过程中的细菌群落分析[J].宿州学院学报,2018,33(8):98-102,106.
[43]刘延波,王琳琳,金尚萍,等.不同储藏期浓香型白酒大曲的微生物多样性分析[J].中国酿造,2022,41(4):105-110.
[44] GODÁLOVÁ Z, KRAKOVÁ L, PUSKÁROVÁ A, et al.Bacterial consortia at different wine fermentation phases of two typical central european grape varieties:Blaufrankisch and Gruner Veltliner[J].Int J Food Microbiol,2016,217:110-116.
[45]李莹莹,刘叶.葡萄酒乳酸菌发酵剂研究进展[J].酿酒科技,2016(11):100-103.
[46]张煜晨,张丹丹,林可,等.苹果酸-乳酸发酵中乳酸菌的分离及其对葡萄酒香气成分的影响[J].食品与发酵工业,2020,46(15):84-92.
[47]HONG X T,CHEN J,LIU L,et al.Metagenomic sequencing reveals the relationship between microbiota composition and quality of Chinese rice wine[J].Sci Rep,2016,6:26621.
[48]BOKULICH N A,OHTA M,LEE M,et al.Indigenous bacteria and fungi drive traditional kimoto sake fermentations[J].Appl Environ Microbiol,2014,80(17):5522-5529.
[49]黄和强,张芬军,李善文,等.夏冬两季青稞酒发酵过程中酒醅微生物菌群多样性分析[J].酿酒,2021,48(2):71-76.
[50]刘选明,张鑫,徐婷,等.一株香茅草内生细菌Pantoea ananatis CcSh-1的分离、鉴定及产香条件优化研究[J].湖南大学学报(自然科学版),2017,44(6):112-118.