大曲是中国白酒的糖化发酵剂,是在开放条件下自然接种形成的微生物菌群的载体,与白酒香型、品质及风格的形成等密切相关[1-4]。黄永光等[5]研究发现,从酱香大曲中分离得到的芽孢杆菌(Bacillus)对酱味贡献较大的菌种依次为地衣芽孢杆菌(Bacillus licheniformis)、解淀粉芽孢杆菌(Bacillus amyloliquefaciens)与枯草芽孢杆菌(Bacillus subtilis)。郭成栓等[6]从酱香大曲中分离到枯草芽孢杆菌E20,其对苯基乙醛、4-甲基-2,6-二叔丁基-苯酚、吲哚、α-呋喃甲醇、2,3,5-三甲基吡嗪、苯乙醇等酱香白酒风味骨架成分的形成具有重要作用。杨春霞等[7]研究发现,芽孢杆菌产生酯类化合物,对于牛栏山二锅头酒体风味形成具有一定帮助。因此,从大曲中筛选功能微生物,尤其是枯草芽孢杆菌与地衣芽孢杆菌,是认识与了解白酒香型、风格及品质形成的前提与基础。
大曲中目标菌种的筛选与鉴定目前还依赖于传统的研究思路:形态观察、生理生化试验与分子生物学相结合的方法[8-9]。但繁琐的操作、较大的工作量、较长的周期导致这项工作的效率较低。基质辅助激光解吸电离-飞行时间-质谱(matrix assisted laser desorption ionization-time of flightmass spectrometry,MALDI-TOF-MS)是一种简单和高效的新型软电离生物质谱,是基质辅助激光解吸电离(MALDI)技术与飞行时间质谱(TOF-MS)的结合,用激光照射样品与基质形成的共结晶薄膜,基质从激光中吸收能量传递给生物分子,电离过程中将质子转移到生物分子或从生物分子得到质子,而使生物分子电离;电离的离子在电场作用下加速飞过飞行管道,根据到达检测器的飞行时间不同而被检测[10]。该技术通过测定微生物蛋白指纹图谱实现了对菌种的快速鉴定[11-13]。近些年,该技术已推广至沙门氏菌(Salmonella)、霍乱弧菌(Vibrio cholera)、单核细胞增生李斯特氏菌(Listeria monocytogenes)等微生物的检测[14]。但是,非标准化的体系仍限制其广泛应用。
本研究试图对MALDI-TOF-MS的检测流程与体系进行改进与优化,并引入至浓香型白酒大曲中芽孢杆菌的初筛,以进一步缩短目标菌种的筛选与鉴定周期,提高工作效率,为后续芽孢杆菌对浓香型白酒风味的研究提供基础条件。同时,也为复杂样品中目标菌株的筛选提供参考方案。
1 材料与方法
1.1 材料与试剂
1.1.1 菌株与材料
枯草芽孢杆菌(Bacillus subtilis)168、地衣芽孢杆菌(Bacillus licheniformis)ATCC 14580、大肠杆菌(Escherichia coli)DH5α等模式菌株:实验室保存;地衣芽孢杆菌(Bacillus licheniformis)CICC 10037、地衣芽孢杆菌(Bacillus licheniformis)CICC 20007:中国工业微生物菌种保藏管理中心,分别为分离自北京、四川绵竹某酒企的菌种;浓香型大曲样品:成都某浓香型酒企。
1.1.2 试剂
基质α-氰基-4-羟基肉桂酸(alpha-cyano-4-hydroxycinnamic acid,CHCA)(色谱纯):美国Fisher Scientific公司;T-Vector pMD19、MiniBEST Bacteria Genomic脱氧核糖核酸(deoxyribonucleic acid,DNA)Extraction Kit Ver.3.0、Ex TaqR Hot Start Version:日本Takara公司;其他试剂均为国产分析纯。
1.1.3 培养基
牛肉膏蛋白胨培养基[15]:蛋白胨10 g,牛肉膏5 g,氯化钠10 g,琼脂粉20 g,去离子水1 000 mL,pH 7.0~7.4。121 ℃高压蒸汽灭菌15 min。
1.2 仪器与设备
Autof ms 1000基质辅助激光解吸电离-飞行时间-质谱仪及配套96孔靶板:中国安图生物公司;MyCycler聚合酶链式反应(polymerase chain reaction,PCR)扩增仪、Sub-Cell GT水平电泳槽:美国Bio-Rad公司;DHP-9402电热恒温培养箱:上海齐欣科学仪器有限公司。
1.3 方法
1.3.1 MALDI-TOF-MS体系的优化
(1)MALDI-TOF-MS体系优化
利用4株具有完整信息的B.subtilis 168、B.licheniformis 14580、B. licheniformis 10037、B. licheniformis 20007为测试菌株对现有文献报道[16-17]的方法进行改进与优化。
直接涂抹法[16]:利用接种环或移液器枪头取待检单菌落直接涂抹于靶板进行基质覆盖与质谱检测。
甲酸-乙腈裂解法[17]:取待检单菌落于无菌水中水洗,体积分数70%乙醇重悬,离心沉淀,随后依次加入甲酸与乙腈进行裂解、点样、干燥、基质覆盖、干燥与质谱检测。基于此,对甲酸-乙腈法进行改进优化。首先,利用灭菌牙签或移液器枪头将不同菌株单菌落划短线转接至牛肉膏蛋白胨培养基平板培养,挑取肉眼可见菌体量直接采用甲酸-乙腈裂解,省去水洗、乙醇沉淀等操作,提高测试菌株检出率至75%。其次,对甲酸-乙腈裂解时间、上样体积、干燥温度等参数进行调整,使测试菌株检出率稳定至100%。优化后流程与具体参数为:挑取转接平板上肉眼可见量的菌落于10 μL裂解液(甲酸∶乙腈=4∶3,V/V),混匀,室温裂解3~10 min,点样1.0~1.5 μL,待30 ℃干燥后覆盖基质CHCA 1.0~5 μL,干燥后质谱检测。检测过程为:打开仪器配备软件,校准仪器,将含样品靶板放入质谱仪,选择数据采集方法,调整仪器为氮基光源、线性阳离子检测模式,每个样本设激光随机射击100个点,每次射击5次,采集范围为2 000~20 000 Da,每个样品采集20张图谱,采用质谱仪Autof Analyzer软件比对菌株蛋白指纹图谱并根据可信度分值进行鉴定结果判定。
(2)MALDI-TOF-MS结果判定
可信度分值标准为:得分为[9.5-10.0],种水平可信;得分为[9.0-9.5),属水平可信,种水平参考;得分为[6.0-9.0),属水平参考;得分为[0.0-6.0),不可信。
1.3.2 大曲样品处理
样品处理:为避免样本污染,于超净工作台内取一定量大曲断裂面中心位置处样品、无菌研钵内研磨、称质量,将1 g大曲溶解于装有9 mL无菌水离心管中,振荡混匀、静置分层、梯度稀释,每梯度分别取200 μL涂布于牛肉膏蛋白胨培养基平板,37 ℃静置培养12~16 h。每个试验重复两次。
样品热处理[18]:操作同上,振荡混匀后置于80 ℃水浴10 min,冷却至室温,梯度稀释,每梯度分别取200 μL涂布于牛肉膏蛋白胨培养基平板,37 ℃静置培养12~16 h。每个试验重复两次。
1.3.3 大曲样品中菌株的分离与纯化
挑选菌落分布均匀的平板,利用灭菌牙签或移液器枪头将单菌落划短线转接至新牛肉膏蛋白胨培养基平板,并对菌株编号,37 ℃培养,培养时间根据需求设定。
1.3.4 基于MALDI-TOF-MS法芽孢杆菌的筛选及鉴定
将转接平板上不同编号的菌株按照改进后的流程进行MALDI-TOF-MS检测与鉴定,完成大曲样品中菌株的规模化筛选,并初步确定目标菌株枯草芽孢杆菌与地衣芽孢杆菌。
1.3.5 MALDI-TOF-MS法的验证
利用分子生物学再次鉴定目标菌株信息,以验证MALDI-TOF-MS法的可行性与准确性。采用Takara MiniBEST Bacteria Genomic DNA Extraction Kit提取基因组DNA,以此为模板,利用通用引物27F和1492R PCR扩增目标菌株的16S rDNA,PCR扩增体系和条件按照文献[19]进行。PCR扩增产物进行T-A(载体为T-Vector pMD19)克隆,阳性克隆子送至四川擎科生物公司测序。测序结果经美国国家生物技术信息中心(national center of biotechnology information,NCBI)的GenBank数据库进行基本局部比对搜索工具(basic local alignment search tool,BLAST)比对分析。选取同源性较高的菌株的16S rDNA基因序列,采用MEGA-X软件中邻接(neighbor-joining,NJ)法构建系统发育树[20]。
2 结果与分析
2.1 MALDI-TOF-MS体系的改进与优化
目前,MALDI-TOF-MS技术无标准化流程,报道较多的为直接涂抹[16]和甲酸-乙腈法[17]。本研究以4株已知芽孢杆菌菌株作为测试菌株,采用这两种方法进行检测发现,直接涂抹法虽流程上操作简单,但是存在菌落涂抹不易掌握、信号弱或无信号响应等问题。甲酸-乙腈法在信号响应、菌株检出率等方面虽有一定改善,但单菌落状态的样品因水洗、乙醇沉淀等过程造成损失,致使检出率仅为50%。因此,对甲酸-乙腈法中取样方式、裂解时间、上样体积、干燥温度等过程与参数进行改进与优化,使测试菌株检出率稳定至100%。在此优化条件下,4株测试菌株的信号响应及蛋白指纹图谱见图1,鉴定结果判定见表1。
表1 测试菌株的MALDI-TOF-MS鉴定结果
Table 1 Identification results of test strains by MALDI-TOF-MS

测试菌株 MALDI-TOF-MS匹配菌株 可信度分值/分B.subtilis 168 B.licheniformis 14580 B.licheniformis 10037 B.licheniformis 20007 Bacillus subtilis subsp.subtilis Bacillus licheniformis Bacillus licheniformis Bacillus licheniformis 9.577 9.298 9.466 9.380
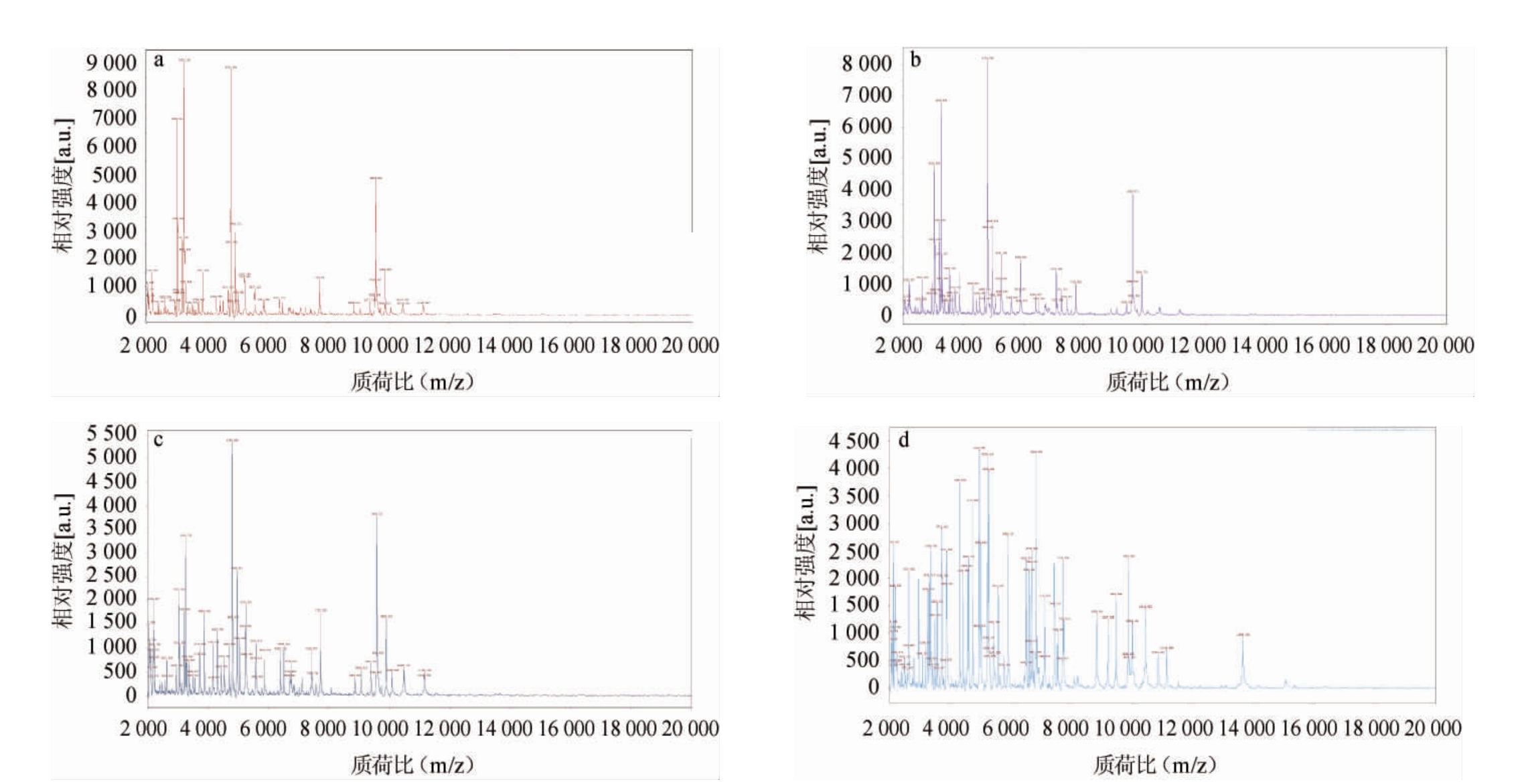
图1 测试菌株的蛋白指纹图谱
Fig.1 Protein fingerprints spectrum of test strains
a~d分别为B.licheniformis 14580、B.licheniformis 10037、B.licheniformis 20007、B.subtilis 168。
由图1可知,在改进优化后的流程下4株测试菌株蛋白指纹图谱清晰,信号响应均较好;种水平一致的菌株有相似的蛋白指纹图谱;地衣芽孢杆菌主要在3 020 Da、3039Da、3 193 Da、3 250 Da、4 791 Da、4 946 Da、7 719 Da、9 588 Da、9 895 Da和10 455 Da出现m/z信号,其中3 020 Da、3 250 Da、4 791 Da和9 588 Da 4个m/z信号强度较高。检测过程中m/z信号在±4 Da波动。而枯草芽孢杆菌的m/z信号构成比较复杂,说明其蛋白构成与地衣芽孢杆菌蛋白构成具有较大差异。所以,通过蛋白指纹图谱能够识别菌株。
由表1可知,4株测试菌株的可信度分值均在9.0以上,其中B. subtilis 168得分为9.577,为种水平可信;另外3株B.licheniformis分值均处于[9.0-9.5),为属水平可信、种水平参考。但是,4株测试菌株均为分子生物学手段鉴定种水平已知的菌株。因此,优化后的MALDI-TOF-MS体系的判定结果比预期更理想,在保证信号响应与菌株检出率的情况下,可信度分值在[9.0-10.0]之间的菌株均高度可信,均可视为种水平可信。
2.2 菌株的分离及基于MALDI-TOF-MS技术芽孢杆菌的筛选鉴定
采用传统微生物培养分离法首次从浓香型大曲中分离得到65株菌株,利用优化后的MALDI-TOF-MS技术对分离得到的菌株进行检测与鉴定,结果见表2。由表2可知,64株菌株判定结果的可信度分值均>9.0,1株菌株判定结果的可信度分值在[0.0-6.0),不可信占比<3%,可忽略,不影响大曲样品菌株总体鉴定结果。65株菌株的检测可于2 h内完成,即菌株筛选可于微生物培养结束当天完成,这比传统的形态观察、生理生化试验及分子生物学相结合的方法至少节约1~2周时间。这一结果不仅表明了MALDITOF-MS方法的高效,也说明了本研究中改进体系的稳定。根据菌株判定结果可知,阴沟肠杆菌(Enterobacter cloacae)与考氏科萨克氏菌(Enterobacter cowanii)分别占筛选菌株的19%与6%,而蜡样芽孢杆菌(B.cereus)与B.subtilis则分别占62%与13%,进一步说明了芽孢杆菌属构成了本研究大曲样品菌株的优势菌群。
表2 基于MALDI-TOF-MS技术大曲样品分离菌株的鉴定结果
Table 2 Identification results of strains isolated from Daqu sample based on MALDI-TOF-MS
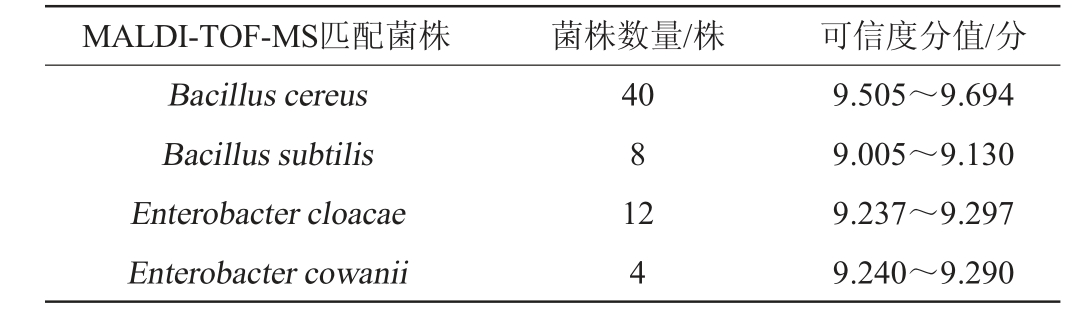
MALDI-TOF-MS匹配菌株 菌株数量/株 可信度分值/分Bacillus cereus Bacillus subtilis Enterobacter cloacae Enterobacter cowanii 40 8 12 4 9.505~9.694 9.005~9.130 9.237~9.297 9.240~9.290
有研究报道称,B.subtilis与B.licheniformis对白酒酒体风味影响较大[21-25]。从本研究大曲菌株鉴定结果可知,B.subtilis在成都酒企大曲样品中占据较高的优势,而非优势菌株B. licheniformis则未检出。为了提高非优势菌株B.licheniformis的检出率,进一步对大曲样品进行了热处理,再次采用传统微生物培养分离法从浓香型大曲中分离得到61株菌株。采用MALDI-TOF-MS技术对这61株菌株进行检测与筛选,结果见表3。由表3可知,被检61株菌株可信度分值均>9.0,为属水平可信;除首次检测发现的优势菌株Enterobacter cloacae、B.cereus、B.subtilis外,此次还发现了极少量的非优势菌株B.licheniformis、索诺拉沙漠芽孢(B.sonorensis)与阿氏芽孢杆菌(B.aryabhattai),说明热处理提高了样品中非优势菌株的检出率。
表3 基于MALDI-TOF-MS技术热处理大曲样品分离菌株的鉴定结果
Table 3 Identification results of strains isolated from heat-treated Daqu sample based on MALDI-TOF-MS
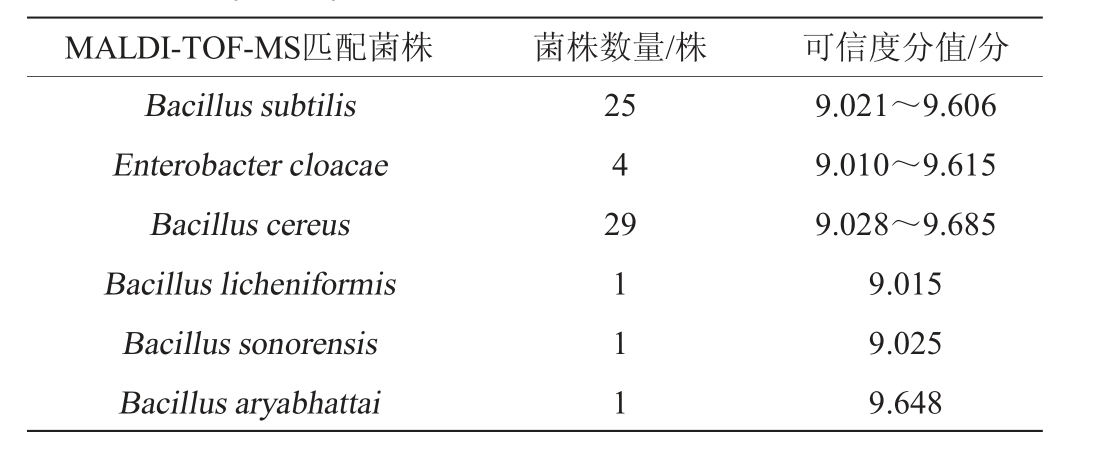
MALDI-TOF-MS匹配菌株 菌株数量/株 可信度分值/分Bacillus subtilis Enterobacter cloacae Bacillus cereus Bacillus licheniformis Bacillus sonorensis Bacillus aryabhattai 25 4 29 1 1 1 9.021~9.606 9.010~9.615 9.028~9.685 9.015 9.025 9.648
基于以上结果可知,利用MALDI-TOF-MS技术可于较短时间内从126株菌株中筛选到目标菌株地衣芽孢杆菌与枯草芽孢杆菌,大幅提高了工作效率,节约了人力成本。为进一步验证MALDI-TOF-MS技术的可靠性与菌株鉴定信息的准确性,将可信度分值为9.015、编号为CD-11的B.licheniformis设定为1#目标菌株,且其蛋白指纹图谱与3株B.licheniformis测试菌株具有较高一致性。B.subtilis在样品中属优势菌株,丰度较高,将编号为CD-1、得分为9.451的B.subtilis设定为2#目标菌株,采用分子生物学技术对目标菌株进行鉴定验证。
2.3 目标菌株的分子生物学鉴定
基于16S rDNA基因序列构建目标菌株CD-1和CD-11的系统发育树,结果见图2。由图2可知,两目标菌株分别处于亲缘关系较远的两分支;菌株CD-1与B.subtilis ZIM3/168等聚于同一分支,亲缘关系最近,故而鉴定菌株CD-1为枯草芽孢杆菌(Bacillus subtilis);而菌株CD-11与B. licheniformis 14ADL4聚于同一分支,因此菌株CD-11被鉴定为地衣芽孢杆菌(Bacillus licheniformis)。
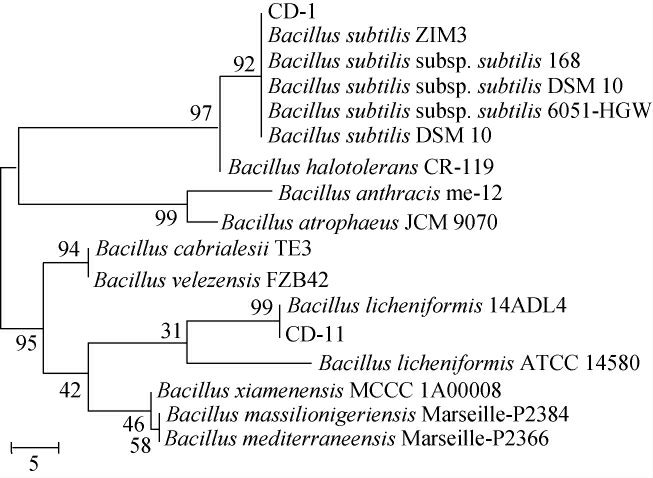
图2 基于16S rDNA基因序列菌株CD-1与CD-11的系统发育树
Fig.2 Phylogenic tree of strains CD-1 and CD-11 based on 16S rDNA gene sequences
目标菌株的分子生物学鉴定结果与MALDI-TOF-MS技术的鉴定结果一致,这表明,MALDI-TOF-MS技术参与的大曲样品中目标菌株的筛选与鉴定是可行的,且准确性极高。枯草芽孢杆菌与地衣芽孢杆菌的高效筛选也将为下一步浓香白酒风味研究奠定了基础。同时,MALDI-TOF-MS技术参与的大曲样品目标菌株的筛选鉴定也为其他菌株的获得、菌群结构与丰度的解析提供了另一种可能。
3 结论
该研究利用已知菌株作为测试菌株对MALDI-TOFMS技术进行了流程与体系优化,通过平板转接、裂解液体积、点样量、基质覆盖量等确保了检测过程中的信号响应与结果判定的准确性,使被检菌株可信度分值均在9.0以上、达到属水平可信。随后,将优化后的体系引入至浓香大曲样品中芽孢杆菌菌株的筛选。从浓香型大曲中分离得到的126株菌株中,检测到了可信度分值在9.0分以上的菌株125株。以编号为CD-1的B.subtilis和编号为CD-11的B.licheniformis为目标菌株,利用分子生物学手段进行进一步验证,结果显示,与MALDI-TOF-MS技术筛选鉴定结果一致。因此,借助MALDI-TOF-MS技术,可于短时间内从大曲样品上百株菌株中完成枯草芽孢杆菌与地衣芽孢杆菌菌株的筛选、鉴定,为复杂样品中目标菌株的筛选与鉴定提供了一条简洁与高效的路径。
[1]ZHENG X W,TABRIZI M R,NOUT M,et al.Daqu-a traditional Chinese liquor fermentation starter[J].J I Brewing,2011,117(1):82-90.
[2] WEI Y, ZOU W, SHEN C H, et al.Basic flavor types and component characteristics of Chinese traditional liquors[J].J Food Sci,2020,85(12):4096-4107.
[3]张治刚,张彪,赵书民,等.中国白酒香型演变及发展趋势[J].中国酿造,2018,37(2):15-18.
[4]范广璞,张安宁,王传荣,等.论浓香型白酒的流派[J].酿酒科技,2004(1):81-83.
[5]黄永光,杨国华,张肖克,等.产酱香风味芽孢杆菌类菌株发酵代谢产物及其酶分析研究[J].酿酒科技,2013(1):41-45.
[6]郭成栓,欧阳蒲月,谢和.枯草芽孢杆菌E20发酵产生挥发性风味成分的GC/MS分析[J].中国酿造,2010,29(9):153-155.
[7]杨春霞,廖永红,刘峻雄,等.牛栏山二锅头酒醅中芽孢杆菌分离鉴定及发酵风味分析[J].食品工业科技,2012,33(9):69-74.
[8]WOESE C R,GUTELL R,GUPTA R,et al.Detailed analysis of the higherorder structure of 16S-like ribosomal ribonucleic acids[J].Microbiol Rev,1983,47(3):621-669.
[9]张崇军,唐贤华,周文.五粮浓香包包曲中高产蛋白酶功能细菌的筛选及鉴定[J].酿酒,2018,45(5):56-58.
[10]DARIE-ION L,WHITHAM D,JAYATHIRTHA M,et al.Applications of MALDI-MS/MS-based proteomics in biomedical research[J].Molecules,2022,27(19):6196.
[11]STEUER A E,POETZSCH M,KRAEMER T.MALDI-MS drug analysis in biological samples: opportunities and challenges[J].Bioanalysis,2016,8(17):1859-1878.
[12]JANG K S,KIM Y H.Rapid and robust MALDI-TOF MS techniques for microbial identification: a brief overview of their diverse applications[J].J Microbiol,2018,56(4):209-216.
[13] PAN C, XU S, ZHOU H, et al.Recent developments in methods and technology for analysis of biological samples by MALDI-TOF-MS[J].Anal Bioanal Chem,2007,387(1):193-204.
[14]顾春华.MALDI-TOF MS技术及其在食品微生物检测方面的应用[J].中国酿造,2019,38(9):24-27.
[15]商曰玲,范莹,余岚,等.啤酒糟中细菌的分离鉴定及其在啤酒糟发酵中的作用[J].中国酿造,2022,41(12):68-73.
[16]BARBUDDHE S B,MAIER T,SCHWARZ G,et al.Rapid identification and typing of Listeria species by matrix-assisted laser desorption ionization-time of flight mass spectrometry[J].Appl Environ Microbiol,2008,74(17):5402-5407.
[17]GIEBEL R,WORDEB C,RUST S M,et al.Microbial fingerprinting using matrix-assisted laser desorption ionization time-of-flight mass spectrometry(MALDI-TOF MS) applications and challenges[J].Adv Appl Microbiol,2010,71:149-184.
[18]周平,罗惠波,黄丹,等.中高温大曲中一株耐热细菌的分离鉴定及其风味代谢产物分析[J].食品工业科技,2016,37(24):215-220.
[19]YAKOUBOU S,COTE J C.Assessment of a short phylogenetic marker based on comparisons of 3'end 16S rDNA and 5'end 16S-23S ITS nucleotide sequences of the Bacillus cereus group[J].Nat Sci,2010,2(12):1113-1118.
[20]徐麓凯,刘一平,韩袁彤,等.抗副溶血弧菌海洋微生物的筛选、鉴定及抑菌谱研究[J].中国酿造,2022,41(11):61-65.
[21]吴树坤,杨磊,杨玲麟,等.沉香型酒醅中产香芽孢杆菌的分离鉴定及代谢产物分析[J].中国酿造,2018,37(1):35-40.
[22]吴文睿,李安军,汤有宏,等.古井贡酒窖泥中芽孢杆菌发酵风味的研究[J].食品科技,2016,41(3):27-30.
[23]钟姝霞,邓杰,汪文鹏,等.酱香型酒醅产香芽孢杆菌的分离鉴定及其代谢产物分析[J].现代食品科技,2017,33(4):89-95,88.
[24]王霜,缪礼鸿,张明春,等.浓酱兼香型酒醅中产酱香芽孢杆菌的筛选及发酵风味成分分析[J].中国酿造,2017,36(10):61-65.
[25]明红梅,董瑞丽,许德富,等.浓香型大曲中优势菌的分离及初步鉴定[J].酿酒科技,2013(12):57-60.