生姜(Zingiber officinale Roscoe),又名姜根、因地辛等,为多年生草本植物的新鲜根茎,其口感辛辣,还带有独特的刺激性香味,是世界上使用最广泛的香辛调味料之一[1]。作为一种良好的中药材,食之两三片生姜可对人体发挥其温肺止咳、发散风寒和温中止呕等功效[2]。生姜中含有丰富的功能性成分和营养物质,据研究表明,它所含的姜油酮和姜酚等具有显著的抗氧化、抗炎、抗糖尿病和抗癌活性等多种效用,此外,生姜中主要刺激性成分二芳基七萜和姜辣素相关化合物对多种癌细胞系具有化学预防和化疗作用[3-5]。
作为南宁酸嘢传统小吃之一的酸姜片,其因咸酸可口、清爽开胃、营养丰富以及既可用作佐料又可用作零食的特点,而备受食客青睐。其工艺流程一般为:选择新鲜、块状饱满、水分充足和无虫害的生姜进行清洗、去皮、晾干和切片,将姜片置于干净无油的容器中,加入少许盐并倒入米醋,米醋的量以没过姜片为宜,常温放置一晚上,待其入味后,次日再放入冰箱进行腌制。关洪全等[6]研究结果显示,当生姜与较低浓度的醋酸混合一段时间后,会表现出一定的协同抗菌活性;田程飘等[7]通过比较生姜与酸姜片的抗氧化、抗肿瘤和抑菌活性研究发现,酸姜片的抗氧化活性、抑菌功能在一定程度上优于生姜。目前,对于酸姜片的研究主要集中于该食品的保健功能上,而有关自然发酵酸姜片微生物多样性和菌株发掘的研究尚少。
Illumina MiSeq高通量测序技术能检测到发酵食品中非培养和纯培养技术不能发现的稀有物种及一些未知的物种[8],具有省时、高效和灵敏等特点[9]。本研究从广西南宁地区共采集酸姜片样品7 份,采用高通量测序技术解析酸姜片样品中细菌群落结构组成的同时使用PICRUSt软件对该样品中微生物基因进行功能预测,并结合传统的微生物培养方法对酸姜片样品中的乳酸菌进行分离鉴定与保存,以期为后续酸姜片工艺条件的优化和相关菌株的发掘提供一定的理论依据和数据支撑。
1 材料与方法
1.1 材料与试剂
1.1.1 原料
酸姜片:采集自广西壮族自治区南宁市(E104°26'~112°04',N20°54'~26°24')淡村菜市场和三元菜市场,一共7个样品,分别编号为SJ1~SJ7。
1.1.2 化学试剂
感受态大肠杆菌(Escherichia coli)top10:本团队制备;脱氧核糖核苷三磷酸(deoxy-ribonucleoside triphosphates,dNTPs)Mix、FastPfu Fly脱氧核糖核酸(deoxyribonucleic acid,DNA)Polymerase、5×TransStartTM FastPfu Buffer:北京全式金生物技术有限公司;QIAGEN DNeasy mericon Food Kit DNA基因组提取试剂盒:德国QIAGEN公司;琼脂糖:索来宝生物技术有限公司。
1.1.3 培养基
MRS培养基、LB培养基:国药集团化学试剂有限公司。
1.2 仪器与设备
DG250型厌氧工作站:英国Don Whitley公司;LRH-250生化恒温培养箱:上海力辰仪器科技有限公司;UVPCDS8000凝胶成像分析系统:美国ProteinSimple公司;Veriti FAST梯度聚合酶链式反应(polymerase chain reaction,PCR)仪:美国ABI公司;Illumina MiSeq高通量测序平台:美国Illumina公司;R920机架式服务器:美国DELL公司。
1.3 方法
1.3.1 宏基因组DNA提取、PCR扩增和测序
按照规定步骤使用基因组提取试剂盒提取酸姜片样品DNA。参照刘晓柱等[10]的方法扩增细菌16S rRNA基因序列,并将检测合格的PCR产物通过IlluminaMiSeq平台进行测序。
1.3.2 生物信息学分析
参照杨放晴等[11]的方法对下机数据进行拼接和序列的剔除。应用QIIME(v1.70)平台对质控合格的序列进行生物学信息分析,步骤如下:PyNAST软件排齐[12],两步UCLUST法聚类并构建操作分类单元(operational taxonomic unit,OTU)[13],ChimeraSlayer去除嵌合体,最后在核糖体数据库项目(Ribosomal Database Project,RDP)、SILVA和Greengenes数据库中同源性比对,明确其分类学水平,并计算超1指数和香农指数等α多样性指数。
1.3.3 基因功能预测
酸姜片样品中细菌序列功能采用PICRUSt软件分析[14],并根据直系同源蛋白(clusters of orthologous groups,COG)数据库注释其功能[15]。
1.3.4 乳酸菌的分离与鉴定
使用倍比稀释涂布法参照刘逸群等[16]的操作流程对酸姜片样品中乳酸菌进行分离,并对各菌株进行2~3次划线纯化,使用30%的甘油对分离到的菌种进行保存。使用十六烷基三甲基溴化铵(cetyl trimethyl ammonium bromide,CTAB)法对样品中分离的菌株进行DNA提取,并以其为模板,按照葛东颖等[17]的PCR扩增体系和扩增条件进行PCR扩增,然后对扩增成功的PCR产物进行清洁、产物连接、转化和鉴定,选择上海桑尼生物科技有限公司对挑选的阳性克隆子进行测序。将纳入研究的菌株核酸序列数据提交至美国国家生物技术信息中心(National Center of Biotechnology Information,NCBI)网站(https://www.ncbi.nlm.nih.gov)进行GenBank登录号的申请,获得的登录号为MZ749626~MZ749634。菌株系统发育树采用MEGA 7.0软件中的邻接(neighbor joining,NJ)法构建。
1.3.5 数据处理
使用R软件对各酸姜片样品中OTU的分布情况及相对含量和优势细菌门、属的相对含量进行了可视化分析,然后进行基因功能预测热图的绘制;利用相关性网络图展示菌株间的关系。
2 结果与分析
2.1 细菌序列丰富度和多样性分析
采用MiSeq高通量测序技术对纳入本研究的7份酸姜片样品进行了测序,结果共产生413 172 条高质量16S rRNA V3-V4序列,每份样品平均序列数59 025 条,各样品的测定分析结果见表1。
表1 样品测序结果及各分类地位数量统计分析
Table 1 Sequencing results of samples and quantitative statistical analysis of each classification status
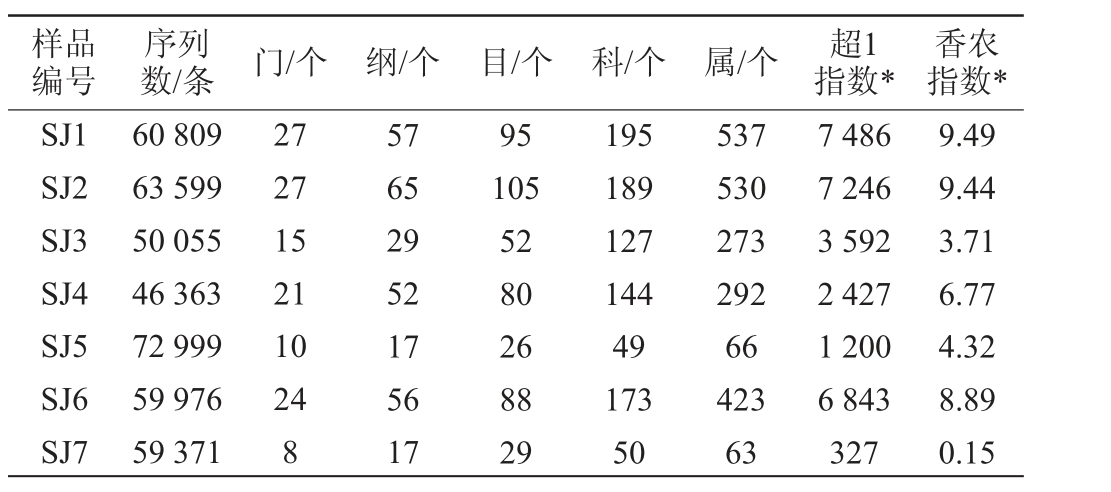
注:“*”表示该数据是在测序深度为45 010条时计算所得。
样品编号数/条 门/个 纲/个 目/个 科/个 属/个 超1指数*序列 香农指数*SJ1 SJ2 SJ3 SJ4 SJ5 SJ6 SJ7 9.49 9.44 3.71 6.77 4.32 8.89 0.15 60 809 63 599 50 055 46 363 72 999 59 976 59 371 27 27 15 21 10 24 8 57 65 29 52 17 56 17 95 105 52 80 26 88 29 195 189 127 144 49 173 50 537 530 273 292 66 423 63 7 486 7 246 3 592 2 427 1 200 6 843 327
由表1可知,样品SJ1、SJ2、SJ3、SJ4、SJ5、SJ6和SJ7的序列数分别为60 809、63 599、50 055、46 363、72 999、59 976和59 371 条,基于UCLUST两步法对序列进行了划分后并比对,发现共鉴定到34个门、84个纲、139个目、241个科和698个属,其中无法鉴定到门和属水平的序列分别仅有0.28%和6.29%。经α多样性计算,7份样品中SJ1的超1指数和香农指数均最高,分别为7 486和9.49,由此说明样SJ1相较于其他样品,其细菌物种丰富度与群落多样性均为最高。
2.2 基于OTU水平的统计分析
在97%的相似度下全部有效序列划分为22 940个OTU,每份样品平均3 277个OTU。一般情况下,将不同样品中均存在的OTU定义为核心OTU,平均相对含量>1.00%的OTU为优势OTU,7份酸姜片样品中OTU的分布情况见图1。
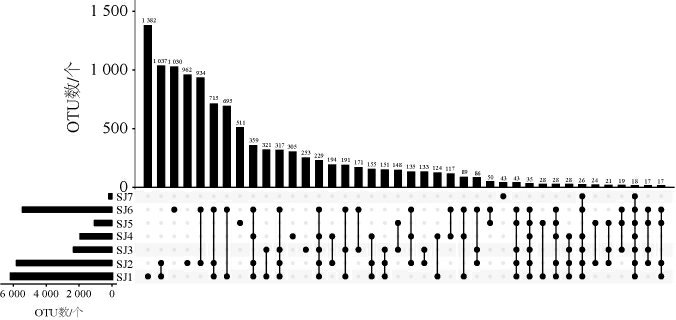
图1 酸姜片样品中OTUs的分布情况
Fig.1 Distribution of OTUs in acid ginger slices samples
由图1可知,在酸姜片样品SJ1~SJ7中,其特有OTU分别有1 382、962、253、305、511、1 030和43个,而核心OTU仅存在18个,占到OTU总数的0.08%,由此可见,不同样品之间细菌菌落结构差异比较大。值得一提的是,SJ7样品中特有OTU数量为43个,仅占特有OTU总数的9.59%,且综合表1中该样品的超1指数为327,香农指数为0.15,从整体上看,相较于其他6 份样品而言,SJ7样品的微生物多样性最小。
本研究进一步分析了该样品中核心OTU的平均相对含量,结果发现核心优势OTU仅有4个,占总核心OTU总数的22.22%。其平均相对含量分布见图2。
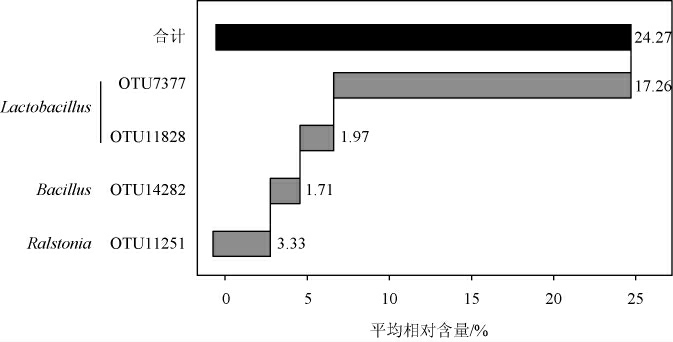
图2 酸姜片样品中平均相对含量>1.0%的核心OTU分析
Fig.2 Analysis of core OTU with average relative content greater than 1.0% in acid ginger slices samples
由图2可知,OTU7377和OTU11828被鉴定隶属于乳杆菌属(Lactobacillus),累计平均相对含量为19.23%;隶属于劳尔氏菌属(Ralstonia)和芽孢杆菌属(Bacillus)的核心优势OTU各有1个,分别为OTU11251和OTU14282,其平均相对含量分别为3.33%和1.71%。由此可见,Lactobacillus在酸姜片的发酵过程中可能有着重要的影响,并有研究表明,Lactobacillus具有嗜酸的特点,可以发酵糖类产生乳酸,当发酵环境中pH下降到一定值时有利于抑制有害菌群和酶的活性,能缩短发酵时间,从而提高产品的风味和营养价值[18]。因此,Lactobacillus在发酵蔬菜的腌制过程中发挥着不可或缺的作用。
2.3 基于门和属水平的酸姜片细菌群落结构分析
进一步在门和属水平上对酸姜片样品菌群结构进行分析,结果见图3。
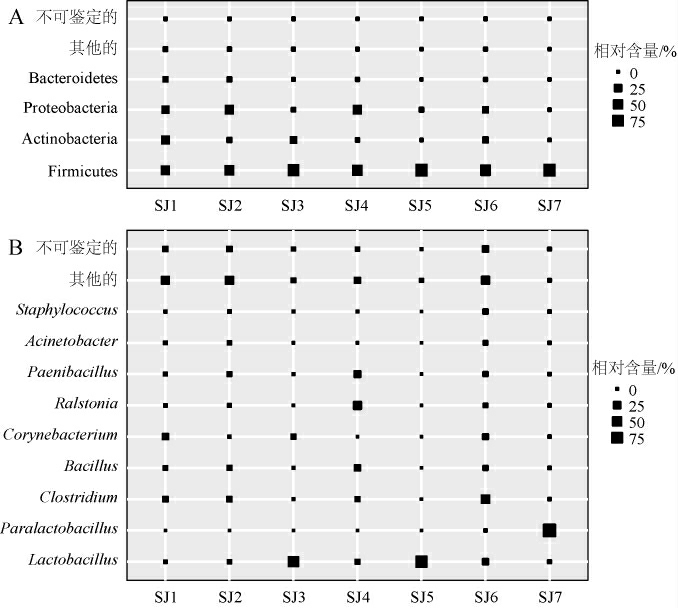
图3 酸姜片样品中细菌门(A)和属(B)的相对含量分析
Fig.3 Analysis of relative contents of bacteria phylum (A) and genus (B) in acid ginger slices samples
由图3可知,优势细菌门(平均相对含量>1.00%)分别为硬壁菌门(Firmicutes)、放线菌门(Actinobacteria)、变形菌门(Proteobacteria)和拟杆菌门(Bacteroidetes),平均相对含量分别为71.62%、16.08%、9.17%和1.75%;优势细菌属(平均相对含量>1.00%)分别为乳酸杆菌属(Lactobacillus)、副乳杆菌属(Paralactobacillus)、梭菌属(Clostridium)、芽孢杆菌属(Bacillus)、棒状杆菌属(Corynebacterium)、劳尔氏菌属(Ralstonia)、类芽孢杆菌属(Paenibacillus)、不动杆菌属(Acinetobacter)和葡萄球菌属(Staphylococcus),平均相对含量依次为28.11%、14.18%、9.14%、5.53%、4.68%、4.20%、1.43%和1.18%。值得注意的是,7 份样品在门水平上就已展现出了一定的差异性,如Firmicutes虽为样品中的优势细菌门,但在SJ1中相对含量仅为38.97%,而在SJ7中其相对含量高达99.77%,由此可见,不同样品在细菌群落结构上存在一定的差异。
本研究中酸姜片样品优势细菌属有9个,然而隶属于其中的部分菌种大多为致病腐败菌,如梭菌属中部分菌种在发酵食品中会增加亚硝酸的含量,在胃肠道的酸性环境中亚硝酸盐会转化为具有强烈致癌作用的亚硝胺[19];在产品贮藏过程中芽孢杆菌属中的部分菌种在适宜条件下会激活,开始生长繁殖进而产酸产气,导致食品变质,甚至有些菌种可以产生一种或者多种肠毒素而引起食物中毒,如蜡样芽孢杆菌[20];葡萄球菌属中的金黄色葡萄球菌为一种食源性致病菌,可产生DNA酶、脂肪酶过氧化氢酶和溶血素类等酶和毒素,引起人类的很多疾病[21];劳尔氏菌属中部分菌种会使人体中毒,有的则是植物病原菌,其病原菌致病力强,寄主范围广,能够侵染多种植物,导致烟草青枯病[22];不动杆菌属中部分菌种为条件致病菌,例如鲍曼不动杆菌为人体肺炎、脑膜炎和败血症等感染的病原体[23];类芽孢杆菌属中的类芽孢杆菌亦为一种腐败菌[24]。究其原因,可能是与农户制作酸姜片的开放环境、工艺流程和贮藏条件有关,由于开放的制作环境,容易导致原材料在前期加工过程中被污染,且在后期不恰当的贮藏条件影响下,会引起腐败菌的滋生。所以在后期酸姜片的研究中,改善工艺与贮藏环境、优化工艺条件和对发酵菌株的纯化筛选,从而提高产品的安全性显得尤为重要[25]。
2.4 酸姜片中细菌属的相关性分析
为揭示样品酸姜片中细菌属彼此间的相关关系,本研究进一步对其中优势细菌属的相关性进行了分析,相关性网络图见图4。
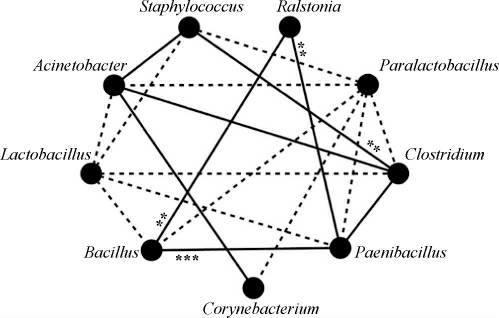
图4 酸姜片样品中优势细菌属相关性网络图
Fig.4 Correlation network diagram of dominant bacteria genera in acid ginger slices samples
实线表示正相关,虚线表示负相关;“***”表示极显著相关(P<0.001),“**”表示显著相关(P<0.01)。
由图4可知,芽孢杆菌属(Bacillus)与劳尔氏菌属(Ralstonia)呈显著正相关(R=0.90,P<0.01),与类芽孢杆菌属(Paenibacillus)呈极显著正相关(R=0.99,P<0.001);类芽孢杆菌属(Paenibacillus)与劳尔氏菌属(Ralstonia)呈显著正相关(R=0.91,P<0.01);葡萄球菌属(Staphylococcus)与梭菌属(Clostridium)呈显著正相关(R=0.92,P<0.01)。综合分析表明,7份酸姜片样品中潜在的致病细菌类群,在很大程度上存在着一定的共生关系。
2.5 基因功能预测
基于对酸姜片样品中细菌类群的解析,本研究进一步预测了该样品中细菌菌群潜在基因功能。结果见图5。
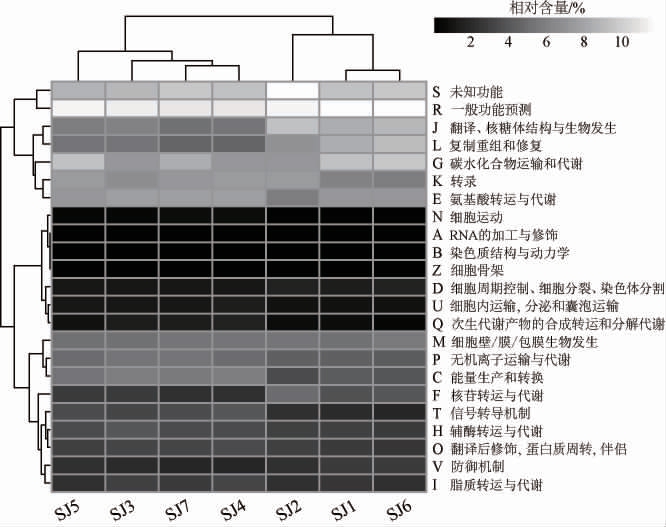
图5 酸姜片样品中细菌序列基因功能预测热图
Fig.5 Heat map of gene function prediction of bacterial sequences in acid ginger slices samples
本研究从酸姜片样品所有细菌序列中注释到的COG隶属于23个功能类别,共有4 544个。由图5可知,在样品细菌序列中占比相对较高的功能类别为“碳水化合物运输和代谢”与“氨基酸转运与代谢”,而“细胞骨架”、“染色质结构与动力学”和“RNA的加工与修饰”这3 个功能类别在样品中序列比例较低。有研究表明,碳水化合物是植物生长的基本营养物质,与果实品质的形成密切相关;氨基酸是蛋白质合成的基础,在发酵过程中,它的存在会促进食品的风味特征与品质[26];此外,Lactobacillus作为酸姜片中的优势细菌属,在发酵过程中经各种代谢活动会产生大量氨基酸和有机酸等代谢物质[27],由此推测,南宁酸姜片中微生物具有“碳水化合物运输和代谢”与“氨基酸转运与代谢”等潜在功能更有利于原料发酵、营养物质及风味的形成。
2.6 乳酸菌的分离鉴定
通过以上分析发现Lactobacillus为南宁酸姜片样品中的优势细菌属。为进一步明确样品中的乳酸菌菌种,采用传统微生物学方法进行了菌株的分离纯化,共分离出疑似乳酸菌菌株8株,编号为HBUAS6001、HBUAS6003~HBUAS6009,其系统发育树见图6。
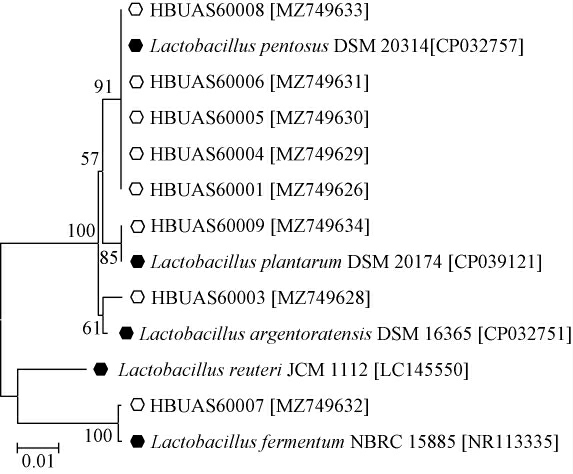
图6 基于16S rRNA基因序列筛选乳酸菌的系统发育树
Fig.6 Phylogenetic tree of screened lactic acid bacteria based on 16S rRNA gene sequences
由图6可知,菌株HBUAS6001、HBUAS6004、HBUAS 6005、HBUAS6006和HBUAS6008被鉴定为戊糖乳杆菌(Lactobacillus pentosus);菌株HBUAS6009被鉴定为植物乳杆菌(Lactobacillus plantarum);菌株HBUAS6003被鉴定为Lactobacillus argentoratensis;菌株HBUAS6007被鉴定为发酵乳杆菌(Lactobacillus fermentum)。综上,本研究分离的8株乳酸菌被鉴定为4个种,其中L.pentosus有5株,占总分离菌株的62.50%。由于受培养条件的限制,通常会有一定比例的菌株不可培养,因而传统纯培养的分析结果不能完全反映样本中微生物群落情况[28],但在酸姜片样品中分离鉴定的菌株均隶属于Lactobacillus,可以进一步说明该菌属在酸姜片自然发酵过程中的重要性。
3 结论
本研究采用Illumina MiSeq高通量测序技术和传统微生物学手法对广西南宁地区7 份酸姜片进行细菌多样性解析发现,Firmicutes、Proteobacteria、Actinobacteria和Bacteroidetes为优势细菌门;Lactobacillus、Paralactobacillus、Clostridium、Bacillus、Corynebacterium、Ralstonia、Paenibacillus、Acinetobacter和Staphylococcus为优势细菌属;经纯培养技术获得的8株分离株均隶属于Lactobacillus。同时通过对细菌菌群的基因功能进行预测发现,酸姜片中细菌具有较强的碳水化合物和氨基酸转运与代谢功能。但潜在致病菌的存在显示,为提高食品的安全性,该产品在工艺条件的优化和贮藏条件的改善方面有待开展。
[1]任昌娟,朱云龙.生姜多糖提取技术工艺优化及效益分析[J].中国调味品,2021,46(9):117-120.
[2]雷丹,洪滔,伍庆华,等.基于网络药理学对生姜药用活性成分的发现和作用机制分析[J].江西中医药大学学报,2021,33(4):83-89.
[3] DUGASANI S, PICHIKA M R, NADARAJAH V D, et al.Comparative antioxidant and anti-inflammatory effects of 6-gingerol, 8-gingerol, 10-gingerol and 6-shogaol[J].J Ethnopharmacol,2010,127(2):515-520.
[4]CHAND B.Antibacterial effect of garlic(Allium sativum)and ginger(Zingiber officinale) against Staphylococcus aureus, Salmonella typhi, Escherichia coli and Bacillus cereus[J].J Microbiol Biotechnol Food Sci,2013,2(4):2481-2491.
[5] SEMWAL R B, SEMWAL D K, COMBRINCK S, et al.Gingerols and shogaols:Important nutraceutical principles from ginger[J].Phytochemistry,2015,117:554-568.
[6]关洪全,栗田启幸,大荒田素子,等.生姜与醋酸协同抗菌作用的实验研究[J].中国中医药信息杂志,2000,46(8):23-24.
[7]田程飘,朱伟伟,宋雅玲,等.生姜与醋泡姜抗氧化、抑菌和抗肿瘤活性比较研究[J].食品工业科技,2019,40(14):18-23.
[8] MASOUD W M, TAKAMIYAN F K, VOGENSEN S, et al.Characterization of bacterial populations in Danish raw milk cheeses made with different starter cultures by denaturating gradient gel electrophoresis and pyrosequencing[J].Int Dairy J,2011,21(3):142-148.
[9]WEN C Q,WU L Y,QIN Y J,et al.Evaluation of the reproducibility of amplicon sequencing with Illumina MiSeq platform[J].Plos One,2017,12(4):e0176716.
[10]刘晓柱,张远林,李银凤,等.高通量测序技术分析刺梨自然发酵过程中细菌多样性[J].中国酿造,2020,39(8):82-85.
[11]杨放晴,何丽英,杨丹,等.基于高通量测序技术分析不同地区自然陈化广陈皮表面微生物群落多样性[J].中国酿造,2021,40(9):31-36.
[12] CAPORASO J G, BITTINGER K, BUSHMAN F D, et al.PyNAST: a flexible tool for aligning sequences to a template alignment[J].Bioinformatics,2010,26(2):266-267.
[13] EDGAR R C.Search and clustering orders of magnitude faster than BLAST[J].Bioinformatics,2010,26(19):2460-2461.
[14] CHEN Y,TIAN W,SHAO Y, et al.Miscanthus cultivation shapes rhizosphere microbial community structure and function as assessed by Illumina MiSeq sequencing combined with PICRUSt and FUNGUIld analyses[J].Arch Microbiol,2020,202(5):1157-1171.
[15] HUERTA-CEPAS J, SZKLARCZYK D, HELLER D, et al.EggNOG 5.0:a hierarchical,functionally and phylogenetically annotated orthology resource based on 5090 organisms and 2502 viruses[J].Nucleic Acids Res,2018,47(D1):D309-D314.
[16]刘逸群,党娜,武岳,等.内蒙古赤峰地区传统发酵乳制品中乳酸菌的分离与鉴定[J].中国乳品工业,2021,49(6):23-26,32.
[17]葛东颖,王玉荣,向凡舒,等.荆州地区鲊广椒乳酸菌多样性解析及其分离株发酵特性的评价[J].中国食品添加剂,2019,30(4):72-79.
[18]胡晓清,潘露,王汝毅.发酵蔬菜中乳酸菌的功能性与安全性研究进展[J].现代食品科技,2012,28(11):1606-1611.
[19]AKHTAR S,PAREDES-SABJA D,TORRES J A,et al.Strategy to inactivate Clostridium perfringens spores in meat products[J].Food Microbiol,2009,26(3):272-277.
[20]DRIEHUIS F.Silage and the safety and quality of dairy foods:a review[J].Agr Food Sci,2013,22(1):16-34.
[21]SANDEL M K,MCKILLIP J L.Virulence and recovery of Staphylococcus aureus relevant to the food industry using improvements on traditional approaches[J].Food Control,2004,15(1):5-10.
[22]向立刚,郭华,周浩,等.健康与感染青枯病烟株根际土壤与茎秆真菌群落结构与多样性[J].植物保护,2020,46(1):189-196,228.
[23]SMITH M G,GIANOULIS T A,PUKATZKI S,et al.New insights into Acinetobacter baumannii pathogenesis revealed by high-density pyrosequencing and transposon mutagenesis[J].Genes Dev,2007,21(5):601-614.
[24] RANIERI M L, BOOR K J.Tracking and eliminating sporeformers in dairy systems[J].Aust J Dairy Technol,2010,65(2):74-80.
[25] ZHANG X D.Study on microbial detection technology in food safety[J].E3S Web of Conferences,2021,233(10):02029.
[26]黄子粤,刘文君,覃仁柳,等.不同品种南瓜内生细菌多样性及PICRUSt基因功能预测分析[J].中国农业科学,2021,54(18):4018-4032.
[27]XIONG T,CHEN J K,HUANG T,et al.Fast evaluation by quantitative PCR of microbial diversity and safety of Chinese Paocai inoculated with Lactobacillus plantarum NCU116 as the culture starter[J].LWT-Food Sci Technol,2019,101:201-206.
[28]曹荣,张井,孟辉辉,等.高通量测序与传统纯培养方法在牡蛎微生物群落分析中的应用对比[J].食品科学,2016,37(24):137-141.