苹果酸-乳酸发酵(malolactic fermentation,MLF)是继酿酒酵母主导的酒精发酵(alcoholic fermentation,AF)后,由乳酸菌将L-苹果酸脱羧成L-乳酸的二次发酵过程[1-3]。MLF是酿造优质红葡萄酒必不可少的工艺环节,其顺利进行会降低葡萄酒的酸度,增加微生物稳定性,增强风味和香气复杂性,提升葡萄酒品质[4-6]。
目前,已确定能够进行MLF的乳酸菌包括酒球菌属(Oenococcus)、明串珠菌属(Leuconostoc)、乳杆菌属(Lactobacillus)和片球菌属(Pediococcus)[7-8]。在应用推广方面,由于酒酒球菌快速启动MLF的能力[9]以及对环境的高耐受性[10-11]等优越表现,酿酒业中广泛采用酒酒球菌(Oenococcus oeni)进行MLF[12]。研究表明,酒酒球菌抵御葡萄酒多因素胁迫的能力具有累积效应,包括低pH、高乙醇和二氧化硫含量、低温和生长抑制化合物等[13-14]。
细胞膜是由蛋白质、脂质和糖类构成的一层覆盖性薄膜,这是细菌应对外界胁迫的第一层防护。MAULUCCI G等[15]研究表明,通过改变细胞膜的脂肪酸组成,细菌可以有效提高在恶劣环境下的存活率。BASTARD A等[2]将细胞膜和浮游细胞接种在发酵的葡萄酒中以研究细胞膜的作用,结果表明细胞膜能够增加酒酒球菌对葡萄酒环境压力的抗性以及MLF活性。当酒酒球菌处于低温、酸和酒精胁迫时,细菌的膜流动性会发生变化。有研究证明,酸胁迫会使细胞膜硬化,但是并不影响细胞的活性[16]。小分子热休克蛋白Lo18与经受热激、暴露于乙醇或苯甲醇(已知对细胞膜有影响的试剂)的酒酒球菌细胞膜关联[17]。
在低温、酸和酒精的压力下,酒酒球菌膜脂肪酸组分会受到影响[18]。低pH和乙醇胁迫下,环丙烷脂肪酸(cyclopropane fatty acid,CFA)含量增加、油酸含量的减少[19]。此外,研究还发现,cfa基因显著上调,该基因可以编码一种环丙烷脂肪酸合酶,而环丙烷脂肪酸合酶能够催化不饱和脂肪酸磷脂生物合成CFA。研究发现,与初始pH 7.4组相比,初始pH 6.8组植物乳杆菌LIP-1为适应酸胁迫环境,上调了脂肪酸合成相关基因,提高了CFA和不饱和脂肪酸的相对含量[20]。研究结果表明,高温和低pH会改变处理细胞的脂肪酸组成,特别是不饱和脂肪酸和饱和脂肪酸的比例,与对照细胞相比,应激菌株的不饱和脂肪酸和饱和脂肪酸比值显著增加[21]。研究结果表明,在铜绿假单胞菌对数生长阶段,观察到亚致死剂量的长波紫外线照射诱导去饱和酶编码基因desA和desB的表达,并增加了不饱和脂肪酸的比例,此外,膜的流动性也可能增加[22]。
本研究通过比较不同生长时期适应期(3 h)、对数期(36 h)和稳定期(108 h)的酒酒球菌耐酸突变株b1和酸敏突变株b2在酸胁迫条件(pH 3.0)以及无胁迫条件(pH 4.8)下膜脂肪酸的动态变化和差异,初步验证不饱和脂肪酸合成关键基因fabF在酒酒球菌酸压力响应中的功能,为酒酒球菌响应酸胁迫机制研究及酸耐受菌种选育提供理论依据。
1 材料与方法
1.1 材料与试剂
1.1.1 菌株
耐酸突变菌株b1、酸敏突变株b2[23]:本实验室前期离子注入诱变获得;植物乳杆菌(Lactobacillus plantarum)XJ25:西北农林科技大学葡萄酒学院保藏;大肠杆菌(Escherichia coli)DH5α:生工生物工程(上海)股份有限公司;感受态植物乳杆菌XJ25、过表达质粒pNZ-POL2(增加了氯霉素抗性):本课题组改造获得[24]。
1.1.2 化学试剂
蛋白胨、胰蛋白胨、酵母浸粉、牛肉膏、葡萄糖、磷酸氢二钾、柠檬酸二铵、无水乙酸钠、硫酸锰、硫酸镁、吐温80、琼脂粉、L-盐酸半胱氨酸、HCl、NaOH、氯化钠、氯霉素粉末、氯化镁、磷酸二氢钾、氢氧化钾、甘油、蔗糖、Tris、乙二胺四乙酸二钠、冰醋酸、苹果酸:杨凌索莱宝生物化学玻璃仪器供应站;番茄汁、双氧水(ddH2O):本实验室自制;Lysis buffer:宝日医生物技术(北京)有限公司;2×Taq Master Mix:苏州近岸蛋白质科技股份有限公司;PrimeSTAR Max Premix:南京诺唯赞生物科技有限公司;上样缓冲液10×Loading Buffer、DL5 000 DNA Marker、Gel Red核酸染液、胶回收试剂盒:北京天漠科技开发有限公司;质粒脱氧核糖核酸(deoxyribonucleic acid,DNA)小提试剂盒:广州飞扬生物工程有限公司。
1.1.3 培养基
MRS液体培养基:蛋白胨10 g/L、酵母浸粉5 g/L、牛肉膏10 g/L、葡萄糖20 g/L、磷酸氢二钾2 g/L、柠檬酸二铵2 g/L、无水乙酸钠5 g/L、硫酸锰0.25 g/L、硫酸镁0.58 g/L、吐温-80 1 mL/L,pH 6.5。
MRS固体培养基:MRS液体培养基中加入15 g/L琼脂粉。
植物乳杆菌电转化复苏培养基:5 mL灭菌MRS液体培养基(试管装)中加入1.25 mL灭菌的1.5 mol/L蔗糖溶液,涡旋混匀。
MRS酸性液体培养基:蛋白胨10 g/L、酵母浸粉5 g/L、牛肉膏10 g/L、葡萄糖20 g/L、磷酸氢二钾2 g/L、柠檬酸二铵2 g/L、无水乙酸钠5 g/L、硫酸锰0.25 g/L、硫酸镁0.58 g/L、吐温-80 1.0 mL/L,pH 3.8。
ATB液体培养基:蛋白胨10 g/L、酵母浸粉5 g/L、番茄汁250 mL/L、葡萄糖10 g/L、硫酸镁0.2 g/L、硫酸锰0.02 g/L、L-盐酸半胱氨酸0.5 g/L。
LB培养基:胰蛋白胨10 g/L、酵母浸粉5 g/L、氯化钠10 g/L,pH 7.0。
上述培养基均在115 ℃条件下灭菌20 min。除MRS酸性液体培养基用苹果酸调pH外,其他培养基灭菌前用HCl和NaOH溶液调节培养基pH值。
1.1.4 引物
本实验所用引物见表1。
表1 本实验所用引物序列
Table 1 Primer sequences used in this experiment
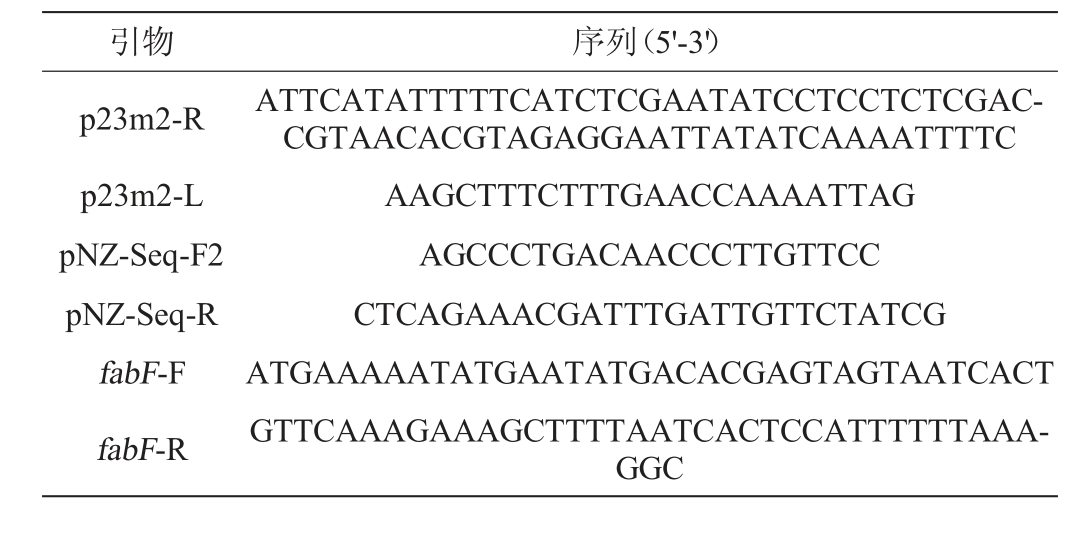
引物 序列(5'-3')p23m2-R p23m2-L pNZ-Seq-F2 pNZ-Seq-R ATTCATATTTTTCATCTCGAATATCCTCCTCTCGACCGTAACACGTAGAGGAATTATATCAAAATTTTC AAGCTTTCTTTGAACCAAAATTAG AGCCCTGACAACCCTTGTTCC CTCAGAAACGATTTGATTGTTCTATCG fabF-F fabF-R ATGAAAAATATGAATATGACACGAGTAGTAATCACT GTTCAAAGAAAGCTTTTAATCACTCCATTTTTTAAAGGC
1.2 仪器与设备
Nanodrop1000聚合酶链式反应(polymerase chain reaction,PCR)仪、MicroPulser电穿孔仪、电击杯(2 mm):美国Bio-Rad公司;MJ-250B恒温培养箱:上海跃进医用光学器械厂;TGL-16B高速台式离心机:天津市泰斯特仪器有限公司;SS325高压蒸汽灭菌锅:日本TOMY公司;PHS-3C pH计:上海雷磁仪器厂;SW-CJ-1超净工作台:苏州安泰空气技术有限公司;UV-680紫外分光光度计:岛津仪器(中国)有限公司;ZQTY-70S磁力搅拌器:济南欧莱博科学仪器有限公司;5804R高速冷冻离心机:德国Eppendorf公司;TRACE1310-ISQLT气相色谱-质谱联用仪:赛默飞世尔科技(中国)有限公司;WM-WAXMS强极性毛细管色谱柱(60 m×0.25 mm×0.25 μm):月旭科技(上海)股份有限公司;JY92-IIDN超声波破碎仪:宁波新芝生物科技有限公司;JY300HC 通用电泳仪、ChampGel 5000plus全自动凝胶成像仪:北京君意电泳设备有限公司。
1.3 试验方法
1.3.1 浓缩菌液制备
参考吕茜等[25]获取浓缩菌液方法并改进,将冷冻保存的酒酒球菌b1、b2菌株转接至pH 4.8 ATB液体培养基中,26 ℃恒温培养箱内活化3 d,稳定一代后再在26 ℃恒温培养箱内扩大培养至对数期(OD600 nm值约为1.0,约36 h),收集菌体,按4%的接种量接种到pH 3.0(酸胁迫)与pH 4.8的ATB液体培养基中,振荡摇匀,于26 ℃恒温培养3 h、36 h和108 h后再将培养基中的菌液在4 ℃高速离心(10 000 r/min,10 min),弃去上清,获得菌体,然后加入1 mL灭菌的生理盐水重新悬浮。
1.3.2 样品制备方法
取1 mL浓缩菌液直接加入2 mL 1 mol/L固体甲醇钠(CH3NaO),4 ℃振摇10 min,用1 mL正己烷萃取脂肪酸甲酯。
1.3.3 细胞膜脂肪酸检测
采用气相色谱质谱联用技术对样品进行细胞膜脂肪酸检测,试验重复3次。
气相色谱条件:WM-WAXMS强极性毛细管色谱柱(60 m×0.25 mm×0.25 μm),高纯氦气(He)为载气,流速0.800 mL/min;进样口温度为230 ℃。柱温升温规则是以80 ℃为初始温度,保持1 min后,以50 ℃/min的速率迅速增至175 ℃,再保持1 min,然后以5 ℃/min的速率增至200 ℃并维持1 min。该实验设置的不分流进样方式,进样量为1 μL。柱温升温是以2 ℃/min的速率迅速增至210 ℃并维持1 min,然后以5 ℃/min的速率增至230 ℃并保持35 min。质谱条件:电子电离(electronic ionization,EI)源,离子源温度为280 ℃,电子能量70 eV,四级杆温度150 ℃,扫描质量范围为40~400 m/z。
定性、定量方法:总离子流色谱图经美国国家标准技术研究所(national institute of standards and technology,NIST)2014S标准谱库检索定性鉴定出挥发性成分,定量采用面积归一法,每种脂肪酸的相对含量可通过计算各脂肪酸峰面积和总体脂肪酸峰面积和的比值表示。
1.3.4 线性载体与目的基因胶回收
将酒酒球菌b1和b2在ATB试管中26 ℃培养36 h,离心收集1.5 mL菌体,加入20 μL Lysis buffer,80 ℃裂解15 min,离心后取1 μL上清作为PCR扩增模板。
将过夜活化的乳酸乳球菌pNZ-Pol2划线于含有相应抗生素的M17平皿,放置于37 ℃培养箱培养,待平皿上长出单菌落后(约48 h),使用灭菌牙签轻轻蘸取少量菌体,利用细菌细胞裂解液处理后,获得用于基因扩增的质粒模板pNZ-Pol2。
参考孟强[26]的PCR扩增体系与PCR扩增程序,改进并采用简石生物的TD407-50琼脂糖凝胶DNA回收试剂盒回收DNA。
1.3.5 重组植物乳杆菌鉴定
于冰盒上将线性化载体与目的片段以一定的摩尔比加入到离心管中进行重组反应;转化改进并采用普因特生物工程有限公司大肠杆菌DH5α感受态细胞说明书;挑取单菌落进行菌落PCR验证。使用omega质粒小提试剂盒来提取重组质粒。参考孟强[26]的植物乳杆菌XJ25感受态细胞制备与电转化方法。
1.3.6 菌株酸胁迫处理
菌株活化:挑取构建好的重组植物乳杆菌L1(过表达菌株b1的目的基因)、L2(过表达菌株b2的目的基因)和插入空质粒的重组植物乳杆菌L0单菌落接种于pH 6.5的MRS液体培养基中,37 ℃过夜培养。
生长曲线的测定:吸取适量菌液转接于新的50 mL pH 3.8 MRS培养基中,每1 h取一次样测OD600 nm值,使用软件Graphpad Prism 8.0.2绘制其生长曲线。
2 结果与分析
2.1 不同生长时期酸胁迫下细胞膜脂肪酸的变化与差异分析
不同生长时期酸胁迫下细胞膜脂肪酸的变化分别见表2~表4。由表2~表4可知,酸胁迫下的突变酒酒球菌b1、b2细胞膜脂肪酸主要成分为肉豆蔻酸(C14:0)、棕榈酸(C16:0)、9-十六碳烯酸(C16:1)、2-己基-环丙烷辛酸(C17:1)、硬脂酸(C18:0)、油酸(C18:1)以及亚油酸(C18:2)、2-己基环丙烷-1-癸酸(C19:1),这几种脂肪酸的含量超过了总脂肪酸含量的95%。
表2 适应期3 h细胞膜脂肪酸相对含量
Table 2 Relative contents of cell membrane fatty acids at adaptation stage 3 h
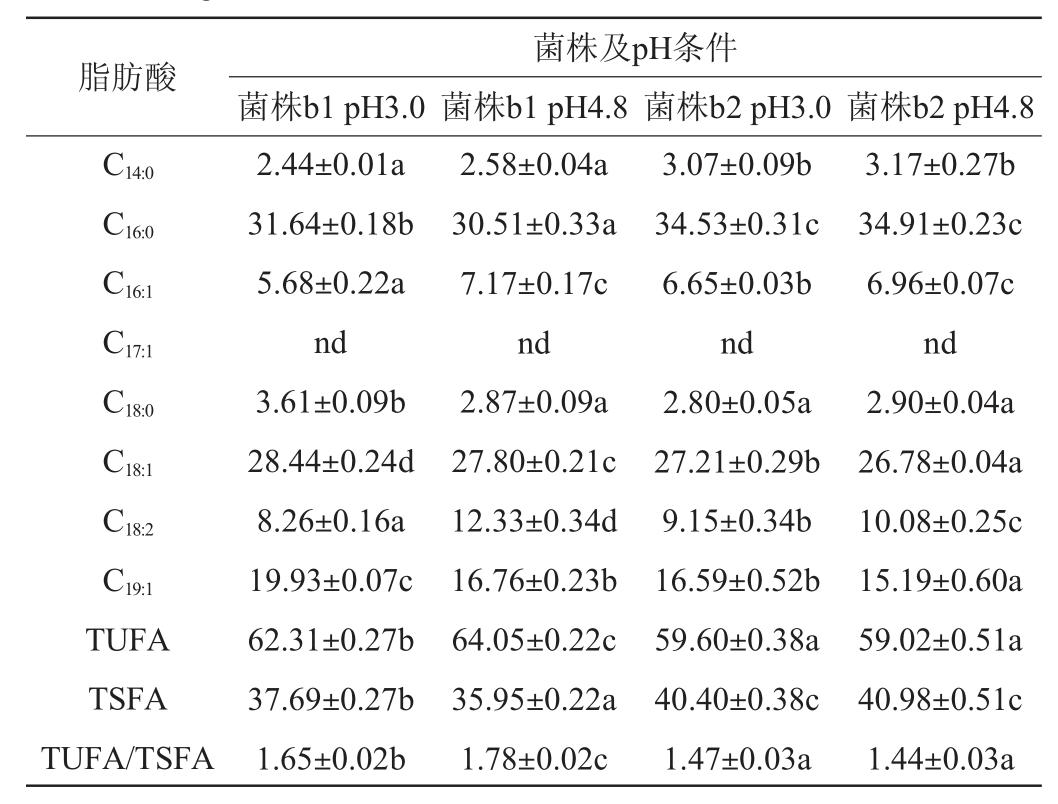
注:“nd”表示未检出;同一行数据肩字母不同表示显著性差异(P<0.05)。下同。
脂肪酸菌株及pH条件菌株b1 pH3.0 菌株b1 pH4.8 菌株b2 pH3.0 菌株b2 pH4.8 C14:0 C16:0 C16:1 C17:1 C18:0 C18:1 C18:2 C19:1 TUFA TSFA TUFA/TSFA 2.44±0.01a 31.64±0.18b 5.68±0.22a nd 3.61±0.09b 28.44±0.24d 8.26±0.16a 19.93±0.07c 62.31±0.27b 37.69±0.27b 1.65±0.02b 2.58±0.04a 30.51±0.33a 7.17±0.17c nd 2.87±0.09a 27.80±0.21c 12.33±0.34d 16.76±0.23b 64.05±0.22c 35.95±0.22a 1.78±0.02c 3.07±0.09b 34.53±0.31c 6.65±0.03b nd 2.80±0.05a 27.21±0.29b 9.15±0.34b 16.59±0.52b 59.60±0.38a 40.40±0.38c 1.47±0.03a 3.17±0.27b 34.91±0.23c 6.96±0.07c nd 2.90±0.04a 26.78±0.04a 10.08±0.25c 15.19±0.60a 59.02±0.51a 40.98±0.51c 1.44±0.03a
2.1.1 适应期3 h时菌株b1和b2酸胁迫下细胞膜脂肪酸的变化与差异分析
一般在胁迫环境下,细胞膜总饱和脂肪酸(total saturated fatty acid,TSFA)含量减少,总不饱和脂肪酸(total un saturated fatty acid,TUFA)增加,从而提高脂肪酸的不饱和度,增强细胞膜的流动性,更利于细胞生存。适应期3 h时菌株b1和b2酸胁迫下细胞膜脂肪酸含量测定结果见表2。
由表2可知,适应期3 h、pH 3.0时,菌株b1的总不饱和脂肪酸含量显著高于菌株b2(P<0.05),菌株b1的总饱和脂肪酸显著低于菌株b2(P<0.05),可见适应期3 h时,菌株b1较b2更容易适应pH 3.0的酸性环境。另一方面,已有报道指出,环丙烷脂肪酸能够增强乳酸菌对胁迫环境的耐受力,从而增加这些脂肪酸的灵活性和流动性,以维持细胞膜的柔性并避免细胞死亡。适应期3 h、pH 3.0时,菌株b1的2-己基环丙烷-1-癸酸(C19:1)含量显著高于菌株b2(P<0.05),这也与菌株的耐酸性相关。与此前的研究发现[19-20]基本一致,即低pH胁迫下,植物乳杆菌会增加环丙烷脂肪酸和不饱和脂肪酸的相对含量、减少油酸(C18:1)含量。
2.1.2 对数期36 h时菌株b1和b2酸胁迫下细胞膜脂肪酸的变化与差异分析
对数期36 h时菌株b1和b2酸胁迫下细胞膜脂肪酸含量测定结果见表3。
表3 对数期36 h细胞膜脂肪酸相对含量
Table 3 Relative contents of cell membrane fatty acids at logarithmic stage 36 h
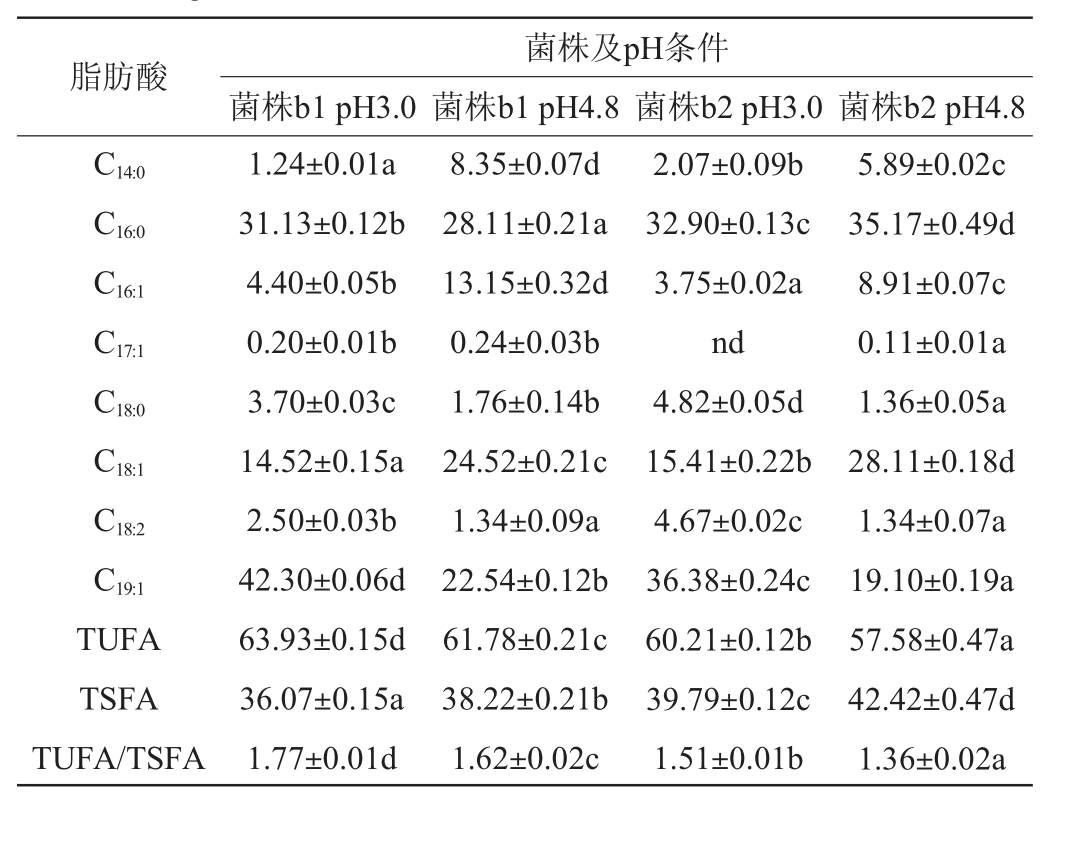
脂肪酸菌株及pH条件菌株b1 pH3.0 菌株b1 pH4.8 菌株b2 pH3.0 菌株b2 pH4.8 C14:0 C16:0 C16:1 C17:1 C18:0 C18:1 C18:2 C19:1 TUFA TSFA TUFA/TSFA 1.24±0.01a 31.13±0.12b 4.40±0.05b 0.20±0.01b 3.70±0.03c 14.52±0.15a 2.50±0.03b 42.30±0.06d 63.93±0.15d 36.07±0.15a 1.77±0.01d 8.35±0.07d 28.11±0.21a 13.15±0.32d 0.24±0.03b 1.76±0.14b 24.52±0.21c 1.34±0.09a 22.54±0.12b 61.78±0.21c 38.22±0.21b 1.62±0.02c 2.07±0.09b 32.90±0.13c 3.75±0.02a nd 4.82±0.05d 15.41±0.22b 4.67±0.02c 36.38±0.24c 60.21±0.12b 39.79±0.12c 1.51±0.01b 5.89±0.02c 35.17±0.49d 8.91±0.07c 0.11±0.01a 1.36±0.05a 28.11±0.18d 1.34±0.07a 19.10±0.19a 57.58±0.47a 42.42±0.47d 1.36±0.02a
由表3可知,对数期36 h时,菌株b1和b2 pH 3.0的总不饱和脂肪酸和2-己基环丙烷-1-癸酸(C19:1)较b1和b2 pH 4.8都是显著升高(P<0.05),b1和b2 pH 3.0的总饱和脂肪酸含量较b1和b2 pH 4.8显著降低(P<0.05),这说明在酸胁迫环境下,细胞膜通过提高不饱和脂肪酸和饱和脂肪酸的比例,增加细胞膜流动性,从而生存下去,实验结果与此前研究结果一致[19-20]。
对数期36 h时,菌株b1和b2 pH 3.0的总不饱和脂肪酸和2-己基环丙烷-1-癸酸(C19:1)含量较对数期3 h都是显著升高的(P<0.05),b1和b2的总饱和脂肪酸较对数期3 h都是显著降低(P<0.05),可见随着时间的进行,菌株b1和b2都在通过调节膜脂肪酸的比例,从而让自己更加适应胁迫环境。对数期36 h、pH 3.0时,菌株b1的总不饱和脂肪酸和2-己基环丙烷-1-癸酸(C19:1)含量较b2都是显著升高(P<0.05),菌株b1的总饱和脂肪酸较b2都是显著降低(P<0.05),而且菌株b1中检测出了2-己基-环丙烷辛酸(C17:1)含量,而菌株b2则未检出。这说明菌株b1比菌株b2更容易通过增加细胞膜不饱和膜脂肪酸和环丙烷脂肪酸的含量。结果表明,随着酸胁迫时间的延长,2-己基-环丙烷辛酸(C17:1)开始被检测出来,而且含量不高,可能在酸胁迫下对细胞的生存至关重要。
2.1.3 稳定期108 h时b1和b2酸胁迫下膜脂肪酸的变化与差异分析
稳定期108 h时菌株b1和b2酸胁迫下细胞膜脂肪酸含量测定结果见表4。
表4 稳定期108 h细胞膜脂肪酸相对含量
Table 4 Relative contents of cell membrane fatty acids in stable stage 108 h
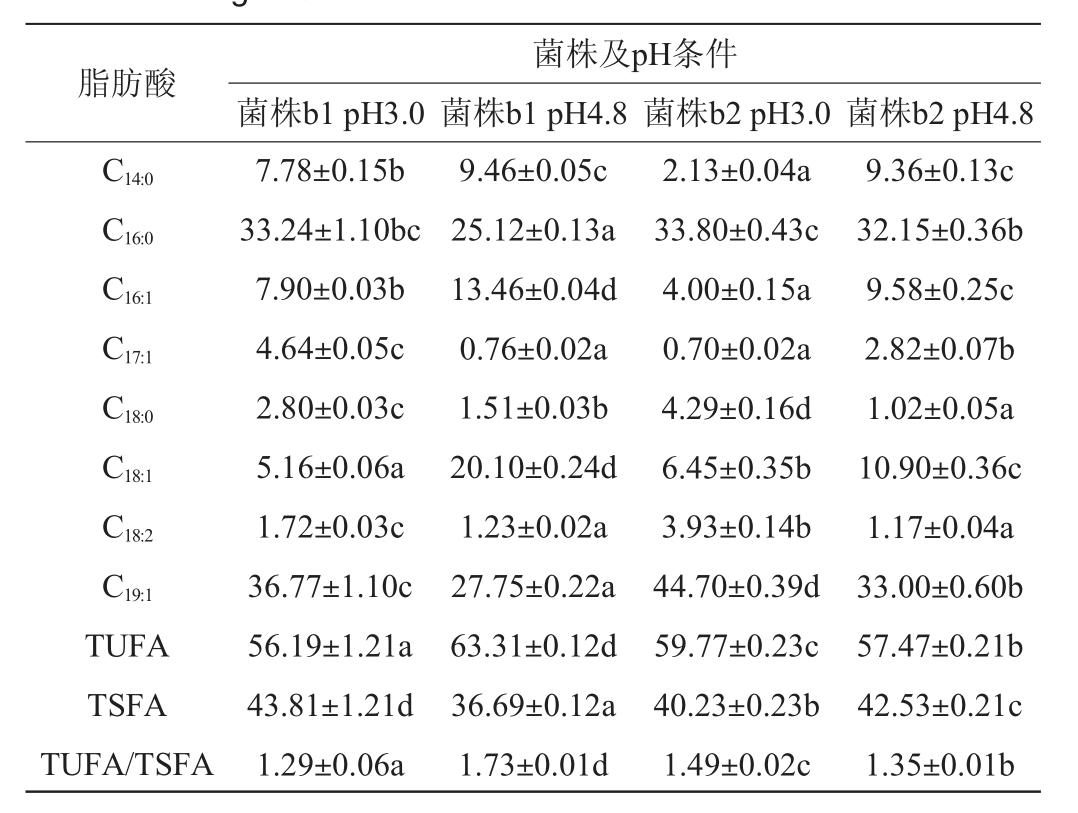
脂肪酸菌株及pH条件菌株b1 pH3.0 菌株b1 pH4.8 菌株b2 pH3.0 菌株b2 pH4.8 C14:0 C16:0 C16:1 C17:1 C18:0 C18:1 C18:2 C19:1 TUFA TSFA TUFA/TSFA 7.78±0.15b 33.24±1.10bc 7.90±0.03b 4.64±0.05c 2.80±0.03c 5.16±0.06a 1.72±0.03c 36.77±1.10c 56.19±1.21a 43.81±1.21d 1.29±0.06a 9.46±0.05c 25.12±0.13a 13.46±0.04d 0.76±0.02a 1.51±0.03b 20.10±0.24d 1.23±0.02a 27.75±0.22a 63.31±0.12d 36.69±0.12a 1.73±0.01d 2.13±0.04a 33.80±0.43c 4.00±0.15a 0.70±0.02a 4.29±0.16d 6.45±0.35b 3.93±0.14b 44.70±0.39d 59.77±0.23c 40.23±0.23b 1.49±0.02c 9.36±0.13c 32.15±0.36b 9.58±0.25c 2.82±0.07b 1.02±0.05a 10.90±0.36c 1.17±0.04a 33.00±0.60b 57.47±0.21b 42.53±0.21c 1.35±0.01b
由表4可知,稳定期108 h pH 3.0时,菌株b1的总不饱和脂肪酸和2-己基环丙烷-1-癸酸(C19:1)含量较菌株b2都是显著降低的(P<0.05),菌株b1的总饱和脂肪酸较菌株b2显著升高(P<0.05),这说明在菌株b1和菌株b2的生长过程中,菌株b1比b2在酸性pH 3.0环境下长得更好,并不总是通过调节不饱和膜脂肪酸的比例,从而让自己更加适应胁迫环境的,比如说在108 h、pH 3.0时,菌株b1的2-己基-环丙烷辛酸(C17:1)含量约为菌株b2的7倍以上,可见除了已经研究[19-20]的增加细胞膜不饱和脂肪酸比例,增加流动性更适于生存外,也有可能是这些对酸胁迫起着关键作用的环丙烷脂肪酸含量,比如说其详细功能特性未知的2-己基-环丙烷辛酸(C17:1),具体结论有待进一步研究。随着酸胁迫时间的延长,突变株b1、b2的油酸(C18:1)、亚油酸(C18:2)含量均显著降低(P<0.05),而2-己基-环丙烷辛酸(C17:1)、2-己基环丙烷-1-癸酸(C19:1)含量均显著升高(P<0.05),可能这两种脂肪酸存在相互转化的关系以适应酸胁迫。
2.2 不饱和脂肪酸合成关键基因fabF在酒酒球菌酸压力响应中的功能
酸胁迫过程中,大量饱和脂肪酸在向不饱和脂肪酸转化,由此可见,提高不饱和脂肪酸含量,增加细胞膜流动性,可能也是酒酒球菌抵御酸胁迫的一个应答机制。基因fabF是与主要不饱和脂肪酸合成相关的beta-ketoacyl-ACP synthase II的基因,因此推测该基因与酒酒球菌酸胁迫响应密切相关。此外,通过基因序列比对,发现菌株b1与b2的该基因存在有一个非同义突变,推测该基因突变可能一定程度增加了酸耐受菌b1的酸耐受能力。因此,分别构建了菌株b1与b2 fabF基因的过表达的重组植物乳杆菌L1和L2,对比了其在酸胁迫条件下生长情况,验证这两个fabF在酸胁迫响应中的作用及差异。
2.2.1 线性载体与目的基因胶回收
胶回收来源于菌株b1与b2 fabF基因以及乳酸乳球菌pNZ-Pol2的线性载体基因,结果见图1。
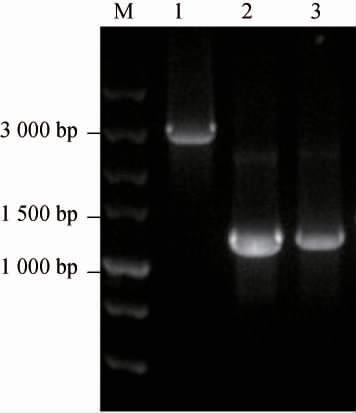
图1 线性载体及目的基因胶回收结果
Fig.1 Results of gel recovery of linear vector and target gene
M:DL5 000 DNA marker;1:线性载体PCR扩增产物;2:b1-fabF PCR扩增产物;3:b2-fabF PCR扩增产物。
由图1可知,线性载体PCR扩增产物碱基数为3 000 bp处出现单一、明亮的条带,并且无杂带;纯化后的b1-fabF基因及纯化后的b2-fabF基因,均在1 200 bp出现单一、明亮的条带,并且无杂带;表明扩增并纯化成功,可进行后续连接实验。将扩增产物交由北京奥科进行测序,并将测序所获得的序列与预期图谱进行比对,结果表明线性载体与目的基因胶回收成功。
2.2.2 重组植物乳杆菌鉴定
在将重组质粒引入植物乳杆菌XJ25感受态细胞后,挑选单个菌落进行菌落PCR,并借助电泳技术进行检测,结果见图2。
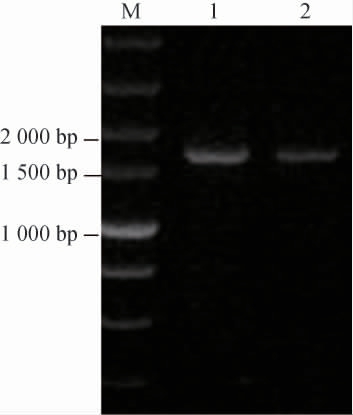
图2 重组植物乳杆菌的鉴定结果
Fig.2 Identification results of recombinant Lactobacillus plantarum
M:DL5 000 DNA marker;1:重组植物乳杆菌b1-fabF;2:重组植物乳杆菌b2-fabF。
由图2可知,b1-fabF和b2-fabF重组载体单菌落PCR产物在1 600 bp处出现明亮的条带,出现条带表明目的基因已经连接载体,质粒构建成功。将扩增产物交由北京奥科进行测序,并将测序所获得的序列与预期图谱进行比对,结果表明重组菌构建成功。
2.2.3 酸胁迫下重组植物乳杆菌生长曲线
测定酸胁迫下重组植物乳杆菌生长曲线见图3。由图3可知,在酸胁迫条件下,重组植物乳杆菌L1(过表达菌株b1的fabF基因)、L2(过表达菌株b2的fabF基因)稳定期OD600 nm值较插入空质粒的重组植物乳杆菌L0分别增加了约12.8%和11.5%,菌体生长的涨幅L1>L2的长势更好。与E J J等[20]研究发现在胁迫环境下,一般细胞膜饱和脂肪酸含量减少,不饱和脂肪酸增加,从而提高脂肪酸的不饱和度,增强细胞膜的流动性,更利于细胞生存研究结果一致。综合两株重组菌和对照菌生长情况的测定,结果表明,酒酒球菌fabF基因的外源插入对于植物乳杆菌在酸性环境中的表现有明显提升,但是也说明酒酒球菌fabF基因存在的碱基突变并不是引起耐酸菌株b1和酸敏菌株b2在耐酸能力方面存在差异的关键基因。
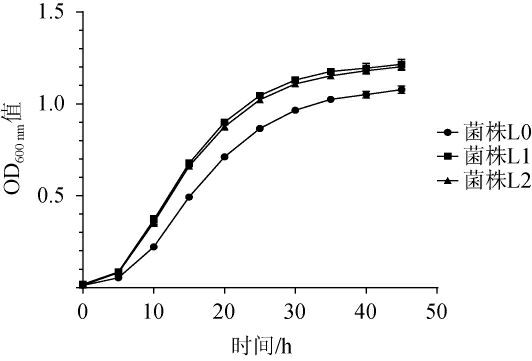
图3 酸胁迫下重组植物乳杆菌生长曲线
Fig.3 Growth curves of recombinant Lactobacillus plantarum under acid stress
3 结论
为研究引起耐酸菌株b1和酸敏菌株b2不同耐酸表型的关键突变基因,利用气质联用(GC)仪测定了两株不同酸耐受表型酒酒球菌(Oenococcus oeni)突变株在酸胁迫及无酸胁迫条件下不同生长时期的细胞膜脂肪酸种类与含量以及分别构建了菌株b1与b2有一个非同义突变的fabF基因过表达的重组植物乳杆菌L1和L2,对比了其在酸胁迫条件下生长情况,验证这两个fabF在酸胁迫响应中的作用及差异。结果发现随着培养时间的延长,有2-己基-环丙烷辛酸(C17:1)被检测出来;随着酸胁迫时间的延长,大量的油酸(C18:1)、亚油酸(C18:2)可能向2-己基环丙烷-1-癸酸(C19:1)转化,使得2-己基环丙烷-1-癸酸(C19:1)含量几乎占到了所有脂肪酸含量的一半;此外,培养3 h和36 h的酸耐受突变株b1的2-己基-环丙烷辛酸(C17:1)、2-己基环丙烷-1-癸酸(C19:1)含量及总不饱和脂肪酸与总饱和脂肪酸比值均显著高于酸敏感突变株b2,培养108 h的菌株b1的2-己基-环丙烷辛酸(C17:1)含量约为菌株b2的7倍。在酸胁迫条件下,重组植物乳杆菌L1、L2稳定期OD600 nm值较菌株L0分别增加了约12.8%和11.5%,菌体生长的涨幅为菌株L1>L2。结果表明,含量不多的2-己基-环丙烷辛酸(C17:1)、2-己基环丙烷-1-癸酸(C19:1)以及不饱和脂肪酸在酒酒球菌抵御酸胁迫时有重要作用,不饱和脂肪酸合成相关的fabF基因在酒酒球菌酸压力响应过程中具有重要作用,但是也说明酒酒球菌fabF基因存在的碱基突变并不是引起耐酸菌株b1和酸敏菌株b2在耐酸能力方面存在差异的关键基因。
[1] RUIZ P, IZQUIERDO P M, SESEÑA S, et al.Malolactic fermentation and secondary metabolite production by Oenoccocus oeni,strains in low pH wines[J].J Food Sci,2012,77(10):579-585.
[2] BASTARD A, COELHO C, BRIANDET R, et al.Effect of biofilm formation by Oenococcus oeni on malolactic fermentation and the release of aromatic compounds in wine[J].Front Microbiol,2016,7:613.
[3] CAPOZZI V, TUFARIELLO M, DE SIMONE N, et al.Biodiversity of oenological lactic acid bacteria: species and strain-dependent plus/minus effects on wine quality and safety[J].Ferment,2021,7(1):1-17.
[4]MALHERBE S,TREDOUX A G J,NIEUWOUDT H H,et al.Comparative metabolic profiling to investigate the contribution of Oenococcus oeni,MLF starter cultures to red wine composition[J].J Ind Microbiol Biotechnol,2012,39(3):477-494.
[5] LASIK M.The application of malolactic fermentation process to create good-quality grape wine produced in cool-climate countries:A review[J].Eur Food Res Technol,2013,237:843-850.
[6]BARTOWSKY E J.Oenococcus oeni and the genomicera[J].FEMS Microbiol Rev,2017,41:S84-S94.
[7]SICO M A,BONOMO M G,SALZSNO G.Isolation and characterization of Oenococcus oeni from Aglianico wines[J].World J Microb Biot,2008,24(9):1829-1835.
[8]CRUZ-PIO L E,POVEDA M,ALBERTO M R,et al.Exploring the biodiversity of two groups of Oenococcus oeni isolated from grape musts and wines:Are they equally diverse[J].Syst Appl Microbiol,2017,40(1):1-10.
[9]CAMPBELL-SILLS H,LORENTZEN M,LUCAS P M.Genomic evolution and adaptation to wine of Oenococcus oeni BT-biology of microorganisms on grapes,in must and in wine[M].Berlin:Springer,2017:457-468.
[10] RODR GUEZ-NOGALES J M, VILA-CRESPO J, FERNNDEZ-FERN NDEZ E.Immobilization of Oenococcus oeni in lentikats to develop malolactic fermentation in wines[J].Biotechnol Prog,2013,29(1):60-65.
[11]BORDAS M,ARAQUE I,ALEGRET J O,et al.Isolation,selection,and characterization of highly ethanol-tolerant strains of Oenococcus oeni from south Catalonia[J].Int Microbiol,2013,16(2):113-123.
[12] ROMERO J,ILABACA C,RUIZ M, et al. Oenococcus oeni in chilean red wines:Technological and genomic characterization[J].Front Microbiol,2018,9:1-12.
[13]BONOMO M G,TOMASO K,CALABRONE L,et al.Ethanol stress in Oenococcus oeni: transcriptional response and complex physiological mechanisms[J].Appl Microbiol Biot,2018,125(1):2-15.
[14] LORENTZEN M P G, LUCAS P M.Distribution of Oenococcus oeni populations in natural habitats[J].Appl Microbiol Biot,2019,103(7):2937-2945.
[15]MAULUCCI G,COHEN O,DANIEL B,et al.Fatty acid-related modulations of membrane fluidity in cells:detection and implications[J].Free Radic Res Commun,2016,50(1):S40-S50.
[16]SON C K,TOURDOT-MARECHAL R,Marechal P A,et al.Combined,cold,acid,ethanol shocks in Oenococcus oeni:effects on membrane fluidity and cell viability[J].BBA-Biomembranes,2005,1717(2):118-124.
[17] COUCHENEY F, GAL L, BENEY L, et al.A small HSP, Lo18, interacts with the cell membrane and modulates lipid physical state under heat shock conditions in a lactic acid bacterium[J].BBA-Biomembranes,2005,1720(1-2):92-98.
[18]ZHANG G,FAN M T,LV Q,et al.The effect of cold,acid and ethanol shocks on synthesis of membrane fatty acid, freeze-drying survival and malolactic activity of Oenococcus oeni[J].Ann Microbiol,2013,63(2):477-485.
[19]GRANDVALET C,ASSAD-GARCÍA J S,CHU-KY S.Changes in membrane lipid composition in ethanol- and acid-adapted Oenococcus oeni cells: characterization of the CFA gene by heterologous complementation[J].Microbiology,2008,154:2611-2619.
[20]E J J,CHEN J,CHEN Z C,et al.Effects of different initial pH values on freeze-drying resistance of Lactiplantibacillus plantarum LIP-1 based on transcriptomics and proteomics[J].Food Res Int,2021,149:110694.
[21]HADDAJI N,MAHDHI A K,BEN ISMAIIL M B,et al.Effect of environmental stress on cell surface and membrane fatty acids of Lactobacillus plantarum[J].Arch Microbiol,2017,199(9):1243-1250.
[22]PEZZONI M,DE TROCH M,PIZARRO R A,et al.Homeophasic adaptation in response to UVA radiation in pseudomonas aeruginosa:Changes of membrane fatty acid composition and induction of desA and desB expression[J].Photochem Photobiol,2022,98(4):886-893.
[23]陈其玲,任晓宁,王玲.酒酒球菌β-葡萄糖苷酶活性与耐酸胁迫能力的相关性分析[J].食品科学,2017,38(1):115-120.
[24]孟强.外源添加甲硫氨酸在植物乳杆菌酸胁迫应答中的作用机制研究[D].杨凌:西北农林科技大学,2022.
[25]吕茜,张国强,樊明涛.酒酒球菌膜脂肪酸成分分析的样品处理方法比较[J].西北农业学报,2012,21(1):192-197.
[26]孟强.葡萄酒植物乳杆菌SA-680耐酸标记基因的功能研究[D].杨凌:西北农林科技大学,2018.