酒精饮品是一种以蔬菜、水果、谷物(小麦、大米、玉米、高粱)为原料[1],在酵母(酵母属或非酵母属)和细菌等微生物的共同作用下酿造加工而成的发酵产品[2]。传统意义上的酒精饮品包括啤酒[3]、白酒[4]、红酒[5]、黄酒[6]等产品,研究发现,多种微生物共同参与了这些酒精饮品的发酵过程,并代谢产生了酯、醇、酮、醛、酸、萜烯、硫化物等风味物质[7-9],它们决定了酒精饮品的口味和品质。除风味物质外,在微生物的作用下,酒精饮品的发酵过程中还会产生新的营养物质和功能因子等对人体健康有益的活性成分。例如红酒中含有白藜芦醇、花青素、原花青素、槲皮素、儿茶素等[10]多种营养物质。然而在酒精饮品的发酵过程中也难以避免地会产生如氨基甲酸乙酯、组胺[11]等有害物质,当人体摄入这些物质的含量超过一定阈值后,就会严重危害身体健康[12],因而需要严格控制有害物质在酒精饮品中的含量。因此,为了稳定酒精饮品的风味与品质,需要对酿酒菌株进行定向的驯化与筛选。从自然环境中分离得到的野生菌株的生理和生产性能通常不能准确满足工业生产的需要,因此需要改进菌株筛选的方法与技术,以便快速、准确地获得性能优良的工业菌株。随着高通量筛选(highthroughput screening,HTS)技术的日趋成熟,这种高效的方法已经成为优良菌株选育的新兴方法之一[13]。和传统的工业微生物育种相比(图1),高通量菌株筛选技术可大幅度提升菌株筛选的速率和准确率。目前,高通量筛选技术不仅已经应用于酒精饮品菌株进化的研究中,而且还应用于其他基因工程改造菌株的筛选过程中[14]。本文从风味的提升(减少硫化物等物质)、生产性能的提升(提高产量及耐受性等)、降低有害物质的生成三个方面,对基于颜色或荧光以及生物量等检测参数的高通量筛选技术在酒精饮品生产菌株筛选中的应用进展进行综述。
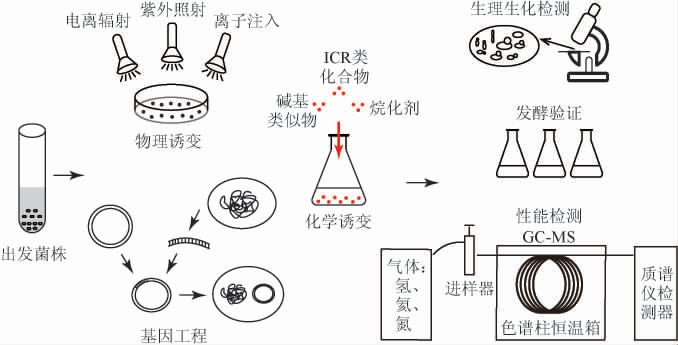
图1 工业微生物育种
Fig.1 Breeding of industrial microorganism
1 酒精饮品生产菌株高通量筛选技术
传统的菌株筛选技术存在耗时、准确率低等问题,使用高通量筛选技术可以快速筛选到理想菌株,减少菌株筛选周期,降低时间成本与经济成本,基于该技术优势,可以加快菌种进化进程,筛选获得环境耐受性、底物利用能力以及代谢物生产能力等更强的工业微生物[15]。高通量筛选是利用自动化的设备,对模式生物、细胞、途径或分子水平上进行快速检测的一项技术[16]。随着该技术的不断发展,该技术可利用更低的检测量完成筛选[17],这大幅度提升了筛选工作的效率。高通量筛选技术的发展推动了不同领域的研究进展,其可被应用于药物筛选[18]、蛋白质定向进化[19]、菌株选育[20]、发酵优化[21]等方面。在菌株选育中,通过建立高通量筛选平台,可以获得高稳定性、高活性或高产某代谢物的目的菌株[22],例如LIU Y F等[23]通过建立高通量筛选平台(该平台由基于色氨酸特异性适配体的生物传感器和荧光激活液滴分选技术组成),从大量菌株中筛选出高产色氨酸的菌株,与原始菌株相比,目的菌株的色氨酸产量提高了165.9%。
目前,高通量菌株筛选法的主要步骤(图2)可概括为:①对含有目的菌株的样品进行采样以及收集;②采用特定的培养基对目的菌株进行富集;③纯化菌株后进行分离;④孔板发酵以确定该菌株的生产性能[24]。
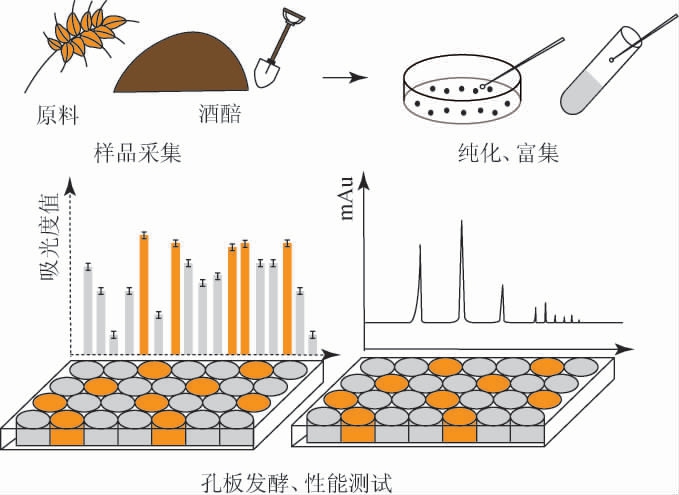
图2 高通量筛选技术的主要步聚
Fig.2 Main procedure of high-throughput screening technology
高通量筛选技术需要使用相应的仪器和自动化系统来处理和分析样品(图3),例如流式细胞仪[25]、酶标仪[26]、QPix 420微生物克隆筛选系统[27]、Biomek FXP自动工作站[28]等。

图3 高通量筛选技术常用仪器
Fig.3 Common instruments of high-throughput screening technology
流式细胞仪能够同时进行定量和定性分析[29],可以在单细胞水平对细胞进行筛选从而达到高纯度,实现高通量。酶标仪是高通量筛选中最常用的设备之一,可对吸光度、荧光强度等参数进行检测,该方法通常用于检测菌体的生长情况、风味物质产量等。酶标仪还可以和孔板发酵相结合,建立高通量菌株筛选平台,以筛选出目的产物高产菌株[30]。Qpix 420微生物克隆筛选系统可以识别微生物单菌落,特定的荧光通道会对产生荧光的菌落进行筛选并自动接至孔板中培养[31]。Biomek FXP自动工作站不仅可以自动化处理液体,还可以实现分析与处理一体化,形成一个自动化系统,并且可将以上设备进行联用,构建相应的高通量筛选平台[32]。传统高通量筛选过程的纯富集技术在揭示微生物菌群的多样性方面具有局限性,随着第二代测序技术的建立与发展,高通量测序技术进入人们视野,通过边合成边测序,准确挖掘未知微生物,因此在酒精饮品生产菌株性能改良方面具有重要地位[33]。目前,主要的高通量测序平台包括罗氏的454测序技术平台、Illumina公司的Solexa测序平台和ABI公司的SOLiD测序平台[34]。
杨祖明等[35]从四个方面对菌种进化过程中的高通量筛选技术进行了综述,将高通量菌株筛选技术分为基于颜色或荧光、细胞生长、生物传感器和液滴微流体的四种技术。目前,应用在酒精饮品生产菌株的高通量菌株筛选技术[36],主要有以下两种,基于颜色或荧光检测的高通量筛选技术和基于生物量检测的高通量筛选技术。
1.1 基于颜色或荧光检测的高通量筛选技术
基于颜色或荧光检测的高通量筛选技术应用到菌株的筛选中,能够直观且高效的对菌株进行筛选,主要有比色法、微量滴定板筛选法、荧光激活细胞分选(fluorescenceactivatedcellsorting,FACS)技术、荧光探针分析检测技术[35]。通过比色法可直观地筛选出代谢物所具有的特征性颜色或荧光,并且通过颜色的类别和深浅判断出产物种类和合成情况,例如硝基酚类[37]和烟酰胺腺嘌呤二核苷酸磷酸(nicotinamide adenine dinucleotide phosphate,NADPH)等可以作为酶的发光底物,β-胡萝卜素[38]和番茄红素[39]是带有颜色的代谢产物。微量滴定板筛选法可关联菌株代谢产物与吸光度,确定菌株的合成能力(或产品评估),应用该方法可在短时间内仅用150 μL的反应试剂完成辣根过氧化物酶突变体的筛选[40]。荧光激活细胞分选技术可对胞内带荧光的代谢物或可被荧光染色的物质进行筛选,通过设置特定波长的激发光激活细胞内荧光信号,对相应物质进行检测[41]。DE VRIES A R G等[42]提出了一种高通量筛选方法,即双染料染色后将不同菌株进行杂交育种,然后采用荧光激活细胞分选技术富集相应杂合细胞,从而分离出不同的杂合酿酒酵母(Saccharomyces cerevisiae)。荧光探针在分析检测过程中可与目标物质特异性结合并释放荧光信号,实现目标物质的高通量检测筛选[43]。例如为高产聚苹果酸酵母设计的荧光探针的荧光信号与聚苹果酸含量间具有良好的定量关系,可对高产聚苹果酸酵母进行高通量筛选[44]。
1.2 基于生物量检测的高通量筛选技术
基于生物量检测的高通量筛选技术使用微量滴定板缩小发酵规模,该技术可关联细胞生长与代谢物浓度以达到筛选目的,目前已被广泛应用于高产特定代谢物与酶的菌株的筛选中,如甲羟戊酸[45]和脂肪酶A[46]等,其中筛选高产甲羟戊酸菌株是通过细胞的荧光强度来监测甲羟戊酸的产量,最终获得20倍高产菌株。该技术可将生产菌株生长状况作为筛选依据,初筛获得生长能力更强的菌株后,再对该菌株的内源性代谢物种类和含量进行检测,例如乙醇代谢能力、乳酸生产能力,从而获得风味物质生产能力选择性显著提高的菌株,以达到提升酒精饮料发酵过程中风味物质含量的目的[47]。
2 高通量筛选技术在性能改良菌株筛选中的应用
2.1 风味的改良
2.1.1 高通量表型筛选
低温发酵可以通过影响风味物质的种类和含量提升葡萄酒的风味和品质。为了筛选获得低温生长缺陷的酿酒酵母菌株,在12 ℃的温度下使用96孔板液体培养突变体菌株合集,并结合积分光密度(integral optical density,IOD)测量方法进行监测,最终确定能够适应低温发酵的菌株[48]。高通量菌株筛选技术还可以和其他进化策略进行联用,如常压室温等离子体(atmospheric room temperature plasma,ARTP)诱变、高通量筛选(HTS)和适应性实验室进化(adaptive laboratory evolution,ALE)等组合策略,筛选出耐酸性的酿酒酵母菌株。将诱变后的菌落用Qpix 420自动采集仪接种至pH为3.0的96孔板上培养,然后使用微孔板阅读器测量OD600nm值,而未接种的培养基作为对照。通过这种方法,获得了一株具有较好的发酵性能的高耐酸菌株ET008-c54,在酸性环境下,该菌株的存活率可提高10倍,发酵时间缩短,香味化合物的含量显著增加52%[49]。窖泥微生物是浓香型白酒发酵的核心,但是目前对窖泥中功能菌株的研究还较少,宋连宝等[50]利用微包埋培养法模拟窖泥生境培养,采用流式细胞仪将包有菌的经活细胞染色的微球分选到含加富培养基的96孔细胞培养板中,分离出传统培养分离方法所得不到的一株产香味物质较为丰富的细菌,该菌经证实能够增加白酒香气物质,提高白酒酿造品质。
2.1.2 高通量测序筛选
采用新一代测序(next generation sequencing,NGS)和高通量筛选技术联用的方法,可筛选出酚类异味(phenolic off-flavor,POF)合成阴性的酒香酵母菌株CRL-90。首先,孔板菌株采用Biomek FXP自动化工作站进行一般液体处理和体外分析表型,然后分离基因组脱氧核糖核酸(deoxyribonucleic acid,DNA)至Illumina HiSeq4000全基因组测序,最终确定了DaPAD1基因缺陷型菌株,由于该基因的缺失使菌株无法将阿魏酸转化为4-乙基愈创木酚(4-ethyl guaiacol,4-EG)和4-乙基苯酚(4-ethylphenol,4-EP),从而在发酵过程中不会合成酚类异味物质,改良酒精饮品的风味和品质[51]。糖化是传统白酒酿造的基础,糖化率越高,产酒率越高。通过利用高通量测序技术分析白酒发酵过程的菌落演替,并与理化指标和香气成分变化相结合,从特香型白酒大曲中筛选得到了高糖化力的黑曲霉(Aspergillus niger)NCUF413.1,该菌株可显著提高发酵出酒率,增加产香风味物质种类[52]。
2.2 生产性能的提升
2.2.1 高通量表型筛选
96微孔板筛选法是将突变体96微孔板的培养和基于酶标仪的酶活测定结合对突变库进行筛选,其优势在于筛选结果灵敏可靠、减少测定所需样品的体积,是常用的菌株筛选方法之一。β-1,3-1,4-葡聚糖酶是啤酒生产中糖化环节的关键酶,其主要来源于芽孢杆菌(Bacillus),增加该酶含量可提高啤酒的过滤速度,因此,为获得高产β-1,3-1,4-葡聚糖酶的菌株从而提升啤酒生产品质,目前针对主要的高地芽孢杆菌(Bacillus alitudinis)进行了一系列的诱变育种,将用于检测β-1,3-1,4-葡聚糖酶酶活的传统3,5-二硝基水杨酸(3,5-dinitrosalicylic acid,DNS)法和96孔板筛选法进行结合,筛选获得了β-1,3-1,4-葡聚糖酶产量显著提高的目的菌株[53]。郭立芸[54]从燕京科研中心提供的12°P酿酒酵母菌株出发,经过梯度浓度麦汁固体培养基培养、抗性平板分离等定向选育方法,得到了抗葡萄糖阻遏效应表型的突变株;将突变株接种至48孔板,并用酶标仪测量其吸光度值,分离获得抗葡萄糖阻遏效应的啤酒酵母;获得的啤酒酵母经过发酵验证,发现其酒精耐受能力提高,发酵速率提高,发酵时间缩短,酒精度有所提高,并且驯化菌株生产的啤酒中含有较低浓度的乙酸乙酯和适宜浓度的高级醇,使啤酒口感显著改善。96深孔板也可用于测定菌株的沉降速率,通过与高通量技术相结合,可筛选获得在葡萄酒发酵后期具有自絮凝沉降性状的酵母菌株,在葡萄酒生产中不仅可以降低过滤成本,还可以提高葡萄酒的产量和质量[55]。除此之外,流式细胞仪也可用于对菌株絮凝性的筛选[56]。
2.2.2 高通量测序筛选
乳酸与乙醇发生酯化反应后会形成乳酸乙酯,是白酒中的一种重要的风味物质[57],适量的乳酸可以改善白酒的风味[58]。但对于酱香型白酒的生产而言,过量的乳酸会严重影响基酒的质量和档次[59]。因此,需要对白酒中的乳酸含量进行相应的控制,从而保证酱香型白酒的质量。杨帆[60]采用ARTP诱变结合高通量筛选方法,筛选出不同产乳酸能力的优势乳酸菌,即首先利用高通量测序技术解析酒醅中细菌群落结构和乳酸变化规律,确定主要的产乳酸微生物并分离培养,最后通过结合菌株数量及产酸能力筛选获得对乳酸贡献度不同的多种乳酸菌。乙醇是酿酒酵母的重要产物,乙醇过量积累会降低黄酒发酵效率,影响黄酒产品品质。杨昳津[61]对黄酒传统发酵样品中的酵母菌株进行紫外(ultraviolet,UV)诱变、定向驯化和原生质融合后,采用高通量测序技术对选育得到的优良菌株进行全基因组测序,并结合基因差异,对该选育株的乙醇耐受机制进行解析,最终获得起酵能力与发酵性能均增强的乙醇耐受性酵母菌株,该菌株能够有效提高黄酒的发酵效率和风味品质。
2.3 有害物质含量的控制
2.3.1 高通量表型筛选
在啤酒的酿造过程中,含硫化合物会导致啤酒产生腐败的硫化味[62]。硫化氢是其中一种含硫化合物,它的存在会影响啤酒的风味。WINTER G等[63]开发了一种在发酵过程中高通量检测硫化氢的方法。二价硫离子是一种还原剂,可将亚甲基蓝进行还原并使其脱色。因此,将亚甲基蓝加到培养基中可对发酵过程中菌株产生的硫化氢进行实时检测,在此原理上可以结合具有液体处理和多孔分光光度读数能力的高通量筛选系统得到低硫化氢的酿造微生物。除此之外,氨基甲酸乙酯是酒精饮品中普遍存在的一种潜在致癌物质[12]。2007年世界卫生组织(world health organization,WHO)国际癌症研究机构(international agency for research on cancer,IARC)将其列为2A类致癌物。刘晓慧[27]运用高通量筛选技术从高温大曲中分离得到3株可产氨基甲酸乙酯水解酶活性的细菌,这些细菌具有降解氨基甲酸乙酯的能力。尿素是氨基甲酸乙酯的前体物质之一[12],因此控制酿造生产过程中的尿素含量也至关重要。程艳等[64]从黄酒生产工业酿酒酵母XZ-11出发,采用恒温常压等离子体(ARTP)诱变及高通量筛选策略,筛选获得代谢过程尿素积累量更低的菌株,具体筛选策略是采用超高速流式分选系统分选被碘化丙啶(propidiumiodide,PI)染色的活细胞,转移至含氮源的孔板中培养并检测发酵液中尿素量。丁霞[65]采用Qpix 420高通量筛选技术从浓香型白酒的酒醅中分离得到一株能够同时降低氨基甲酸乙酯及其前体尿素的解淀粉芽孢杆菌(Bacillus amyloliquefaciens)JP21,以及一株能够降低瓜氨酸、尿素和氨基甲酸乙酯的地衣芽孢杆菌(Bacillus licheniformis)DX530。
2.3.2 高通量测序筛选
由于精氨酸可被利用转化为氨基甲酸乙酯的另一前体物质瓜氨酸,仇钰莹等[66]运用Qpix 420高通量筛选系统将单菌落接种至96深孔板的精氨酸利用培养基中,从浓香型白酒的酒醅中分离具有高精氨酸利用率的菌株,并通过基因型和表型验证以及窖内发酵模拟实验确定了菌株对控制氨基甲酸乙酯含量的效果,为阐明白酒发酵过程中氨基甲酸乙酯形成机制提供依据。张鹏等[67-68]在黄酒的研究中应用了适应性实验室进化(ALE)策略与高通量筛选技术,首先对酿酒酵母N85单倍体菌株尿素代谢能力进行进化,然后对突变体菌株使用微孔板分光光度计初筛和流式细胞仪分选单个活细胞,最后基因组测序和数据分析,从而筛选获得一株低产尿素的适应性进化突变菌株。
3 结论与展望
菌株的生理和生产性能是决定发酵酒精饮品品质的基础。在基因工程菌株细胞工厂的构建中,可通过一系列诱变或突变技术实现生产菌株发酵性状的改变,可在更短的时间内获得大量的突变菌株。使用高效的高通量筛选技术,基于环境耐受性、底物消耗率、代谢物生成率等检测参数,可快速获得所需的表型突变菌株。因此,高通量筛选技术在菌种进化方面起到了不可替代的作用。目前,国内外高通量筛选技术已经发展到了一个比较成熟的阶段,已经建立了推动工业微生物菌种进化的各种高通量筛选平台[69],并通过与高效液相色谱、质谱、气质联用、液质联用等分析仪器进行组合,可快速得到分析结果。
在酒精饮品行业,将一系列传统育种方法(如物理、化学诱变技术及基因组重排)与高通量筛选技术进行结合之后,能够快速从大量突变菌株中获得工业生产所需菌株。基于不同的筛选原理和不断发展的新筛选技术,可更高通量、高精度、低成本地筛选理想的菌株,使目的菌株在发酵酒精饮品生产中产生更多风味物质、具备更高生产性能、合成更少的有害物质。虽然高通量筛选技术在实际菌种进化应用中具有明显的优势和广泛的应用前景,但是酒精饮品生产菌株高通量筛选技术目前的应用范围还不够广泛,仅适用于部分发酵性能、代谢物和酶含量及合成路径的筛选。因此,随着未来生物信息学以及人工智能领域的发展,可进一步提高高通量筛选技术的精度和适用范围,建立更多的高通量筛选联合策略,扩展基于生物传感器和液滴微流体技术的高通量筛选方式,进而推动符合工业4.0时代酒精饮品发酵所需生产菌株的改良进程。
[1]YLDRM H K.Insights into the role of yeasts in alcoholic beverages[M].Salt Lake City:Academic Press,2021.
[2] NIKOLAOU E,SOUFLEROS E H,BOULOUMPASI E, et al.Selection of indigenous Saccharomyces cerevisiae strains according to their oenological characteristics and vinification results[J].Food Microbiol,2006,23(2):205-211.
[3] HABSCHIED K, VELIC' N, TIŠMA M, et al.The effect of barley and wheat microflora on malt and beer quality[J].Food Technol Biotechnol,2011,6:100-111.
[4]赵亮,王莉,汪地强,等.白酒微生物群落研究技术现状与二代测序数据分析方略[J].酿酒科技,2016(7):88-96.
[5]GSCHAEDLER A.Contribution of non-conventional yeasts in alcoholic beverages[J].Curr Opin Food Sci,2017,13:73-77.
[6]张中华.绍兴黄酒麦曲中微生物群落结构的研究[D].无锡:江南大学,2012.
[7]孙优兰,钟方达,韦露露,等.中国白酒风味组学研究进展[J].酿酒科技,2021(5):50-55.
[8]杨珍,贺立东.反吹-气相色谱法快速检测啤酒中的主要风味物质[J].中国酿造,2019,38(10):171-174.
[9]刘春艳,张静,李栋梅,等.葡萄酒风味物质研究进展[J].食品工业科技,2017,38(14):310-313.
[10]SUN B S,SPRANGER M I.Changes in phenolic composition of Tinta Miúda red wines after 2 years of ageing in bottle:effect of winemaking technologies[J].Eur Food Res Technol,2005,221(3):305-312.
[11]RODRÍGUEZ-SAAVEDRA M,GONZÁLEZ DE LLANO D,MORENOARRIBAS M V.Beer spoilage lactic acid bacteria from craft brewery microbiota: microbiological quality and food safety[J].Food Res Int,2020,138:109762.
[12]DI Y H,LI J H,CHEN J,et al.Simulation and control of the formation of ethyl carbamate during the fermentation and distillation processes of Chinese Baijiu[J].Foods,2023,12(4):821.
[13] GAO H, LIU M, ZHOU X L, et al.Identification of avermectin-highproducing strains by high-throughput screening methods[J].Appl Microbiol Biotechnol,2010,85(4):1219-1225.
[14] LEAVELL M D, SINGH A H, KAUFMANN-MALAGA B B.Highthroughput screening for improved microbial cell factories, perspective and promise[J].Curr Opin Biotechnol,2020,62:22-28.
[15] TONG Q Q, LI Y L, WANG S C, et al.High-throughput screening of Streptomyces virginiae strains using microtiter plates for the high-titer production of virginiamycin[J].Anal Lett,2020,53(8):1233-1246.
[16] ATTENE-RAMOS M S, AUSTIN C P, XIA M.Encyclopedia of toxicology[M].Amsterdam:Elsevier,2014:916-917.
[17]TAN J,CHU J,HAO Y Y,et al.High-throughput system for screening of Cephalosporin C high-yield strain by 48-deep-well microtiter plates[J].Appl Biochem Biotechnol,2013,169:1683-1695.
[18] VERNOOIJ L, BATE-EYA L T, ALLES L K, et al.High-throughput screening identifies idasanutlin as a resensitizing drug for venetoclax-resistant neuroblastoma cells[J].Mol Cancer Ther, 2021, 20(6): 1161-1172.
[19]YONG K J,SCOTT D J.Rapid directed evolution of stabilized proteins with cellular high-throughput encapsulation solubilization and screening(chess)[J].Biotechnol Bioeng,2015,112(3):438-446.
[20] WEIS R.High-throughput screening and selection of Pichia pastoris strains[J].Methods Mol Biol,2019,1923:169-185.
[21]廖建国,洪铭,储炬.运用高通量筛选技术优化红霉素A发酵的合成培养基[J].中国抗生素杂志,2018,43(1):55-62.
[22]KOVACˇEVIC' G,OSTAFE R,BALAŽ A M,et al.Development of GFPbased high-throughput screening system for directed evolution of glucose oxidase[J].J Biosci Bioeng,2019,127(1):30-37.
[23] LIU Y F, YUAN H L, DING D Q, et al.Establishment of a biosensorbased high-throughput screening platform for tryptophan overproduction[J].ACS Synth Biol,2021,10(6):1373-1383.
[24] PANDEY B R, GHIMIRE S, BHATTARAI S, et al.Isolation, growth,enzyme assay and identification via 16S rRNA full sequencing of cellulolytic microbes from Nepal for biofuel production[J].Renew Energy,2019,132:515-526.
[25]KWON K K,LEE D H,KIM S J,et al.Evolution of enzymes with new specificity by high-throughput screening using DmpR-based genetic circuits and multiple flow cytometry rounds[J].Sci Rep,2018,8:2659.
[26]CHOUDHARI V G,ODANETH A A,LALI A M.Application of highthroughput screening for evaluating hydrolytic potential of cellulases[J].Biomass Convers Biorefin,2019,9(4):659-667.
[27]刘晓慧.氨基甲酸乙酯水解酶的筛选及分子改造[D].无锡:江南大学,2016.
[28]龙燕,刘然,梁恒宇,等.Nisin高产菌株的高通量筛选[J].微生物学报,2018,58(7):1298-1308.
[29]STAVRAKIS S,HOLZNER G,CHOO J,et al.High-throughput microfluidic imaging flow cytometry[J].Curr Opin Biotech,2019,55:36-43.
[30]常富城,任尧,加宇星,等.γ-聚谷氨酸微孔板微缩发酵与快速测定[J].食品科技,2021,46(7):278-284.
[31]李慧真.从蓝细菌光敏色素和藻胆蛋白筛选近红外荧光蛋白生物探针的研究[D].武汉:华中农业大学,2017.
[32]曹晓梅.阿维菌素高产菌株的定向选育[D].无锡:江南大学,2018.
[33] PAN C T, LI G, BANDYOPADHYAY A, et al.Guide RNA librarybased CRISPR screens in plants:opportunities and challenges[J].Curr Opin Biotech,2023,79:102883.
[34]DI BELLA J M,BAO Y,GLOOR G B,et al.High throughput sequencing methods and analysis for microbiome research[J].J Microbiol Meth,2013,95(3):401-414.
[35]杨祖明,王颖,姚明东,等.高通量筛选技术在菌种进化中的研究进展[J].化工进展,2019,38(5):343-353.
[36] JONG C D, HAZELWOOD L A, DIJKSTRA A, et al.Flavour science[M].Salt Lake City:Academic Press,2014:355-359.
[37]REETZ M T.Laboratory evolution of stereoselective enzymes:a prolific source of catalysts for asymmetric reactions[J].Angew Chem Int Ed Engl,2011,50(1):138-174.
[38]LI J,SHEN J,SUN Z Q,et al.Discovery of several novel targets that enhance β-carotene production in Saccharomyces cerevisiae[J].Front Microbiol,2017,8:1116.
[39]ALPER H,MIYAOKU K,STEPHANOPOULOS G.Construction of lycopene-overproducing E.coli strains by combining systematic and combinatorial gene knockout targets[J].Nat Biotechnol,2005,23(5): 612-616.
[40] AGRESTI J J, ANTIPOV E, ABATE A R, et al.Ultrahigh-throughput screening in drop-basec microfluidics for directed evolution[J].Proc Natl Acad Sci,2010,107(9):4004-4009.
[41]LÜLSDORF N,PITZLER C,BIGGEL M et al.A flow cytometer-based whole cell screening toolbox for directed hydrolase evolution through fluorescent hydrogels[J].Chem Commun,2015,51(41):8679-8682.
[42]DE VRIES A R G,KOSTER C C,WEENING S M,et al.Phenotype-independent isolation of interspecies Saccharomyces hybrids by dual-dye fluorescent staining and fluorescence-activated cell sorting[J].Front Microbiol,2019,10:871.
[43]刘宜嵩,王壬,钮成拓,等.荧光探针在高通量筛选技术上的应用进展[J].食品与发酵工业,2021,47(11):266-271.
[44]LI H Q,LI T F,ZUO H,et al.A novel rhodamine-based fluorescent pH probe for high-throughput screening of high-yield polymalic acid strains from random mutant libraries[J].Rsc Adv,2016,6(97):94756-94762.
[45] HENNING H, LEGGEWIE C, POHL M, et al.Identification of novel benzoylformate decarboxylases by growth selection[J].Appl Environ Microbiol,2006,72(12):7510-7517.
[46]BOLES E,OREB M.A growth-based screening system for hexose transporters in yeast[J].Methods Mol Biol,2018,1713:123-135.
[47] ZHAO X D, PROCOPIO S, BECKER T.Flavor impacts of glycerol in the processing of yeast fermented beverages: a review[J].J Food Sci Technol,2015,52(12):7588-7598.
[48] SALVADÓ Z, RAMOS-ALONSO L, TRONCHONI J, et al.Genomewide identification of genes involved in growth and fermentation activity at low temperature in Saccharomyces cerevisiae[J].Int J Food Microbiol,2016,236:38-46.
[49]TIAN T T,WU D H,NG C T,et al.A multiple-step strategy for screening Saccharomyces cerevisiae strains with improved acid tolerance and aroma profiles[J].Appl Microbiol Biotechnol,2020,104:3097-3107.
[50]宋连宝,赵辉.包埋培养法筛选白酒窖泥中一株功能细菌[J].食品科学,2017,38(6):159-165.
[51] COLOMER M S, CHAILYAN A, FENNESSY R T, et al.Assessing population diversity of Brettanomyces yeast species and identification of strains for brewing applications[J].Front Microbiol,2020,11:637.
[52]潘菲.高糖化力霉菌的筛选及其对特香型白酒酿造出酒率和风味的影响[D].南昌:南昌大学,2018.
[53]陈计.β-1,3-1,4-葡聚糖酶高产菌诱变选育及酶的定向进化研究[D].南京:南京农业大学,2014.
[54]郭立芸.18°P高浓酿造抗葡萄糖阻遏效应啤酒酵母的选育[J].食品与发酵工业,2015,41(4):91-96.
[55] VARELA C, BARTEL C, NANDORFY D E, et al.Identification of flocculant wine yeast strains with improved filtration-related phenotypes through application of high-throughput sedimentation rate assays[J].Sci Rep,2020,10:2738.
[56] VAN DEN BROEK M, BOLAT I, NIJKAMP J F, et al.Chromosomal copy number variation in Saccharomyces pastorianus is evidence for extensive genome dynamics in industrial Lager brewing strains[J].Appl Environ Microbiol,2015,81(18):6253-6267.
[57]李伟,李佳,王宇鹏,等.清香型白酒乳酸利用菌的筛选及鉴定[J].中国酿造,2017,36(9):87-91.
[58]李维青.浓香型白酒与乳酸菌、乳酸、乳酸乙酯[J].酿酒,2010,37(3):90-93.
[59]YANG F,CHEN L Q,LIU Y F,et al.Identification of microorganisms producing lactic acid during solid-state fermentation of Maotai flavour liquor[J].J I Brewing,2019,125(1):171-177.
[60]杨帆.酱香型白酒中乳酸代谢机理及调控策略的研究[D].无锡:江南大学,2020.
[61]杨昳津.优良黄酒酵母选育及其乙醇耐受机制研究[D].上海:上海理工大学,2018.
[62]徐春凤,常昕.酵母添加对降低啤酒硫化味影响的试验[J].啤酒科技,2007(11):47,51.
[63]WINTER G,CURTIN C.In situ high throughput method for H2S detection during micro-scale wine fermentation[J].J Microbiol Meth,2012,91(1):165-170.
[64]程艳,堵国成,周景文,等.高通量筛选诱变菌株降低黄酒发酵氨基甲酸乙酯前体积累[J].微生物学报,2017,57(10):103-112.
[65]丁霞.酒醅中氨基甲酸乙酯及其前体的控制与消除[D].无锡:江南大学,2018.
[66]仇钰莹,方芳,周新虎,等.酒醅中精氨酸利用菌株的分离筛选及其对浓香型白酒中瓜氨酸积累的影响[J].微生物学报,2016,56(10):1638-1646.
[67]张鹏.酿酒酵母氮代谢阻遏机制解析和低产尿素黄酒发酵菌株选育[D].无锡:江南大学,2017.
[68]ZHANG W P,CHENG Y,LI Y D,et al.Adaptive evolution relieves nitrogen catabolite repression and decreases urea accumulation in cultures of the Chinese rice wine yeast strain Saccharomyces cerevisiae XZ-11[J].J Agr Food Chem,2018,66(34):9061-9069.
[69]ZENG W Z,GUO L K,XU S,et al.High-throughput screening technology in industrial biotechnology[J].Trends Biotechnol,2020,38(8):888-906.