中国白酒按香型划分可分为浓香、酱香、清香、米香四大基本香型,在此基础上不断演变、完善到至今的十二大香型[1-2]。“香型融合”是白酒生产技术发展的趋势,在继承传统工艺的基础上持续创新,在借鉴中寻求突破,以提高白酒质量、丰富产品风格,适应市场的多元化需求及消费者口味变化[3-4]。在传统浓香型白酒生产中借鉴酱香型堆积工艺,将高温大曲、中温大曲等按照一定比例应用于浓香型酒醅中,在高温条件下利用功能微生物的生长代谢产生丰富的香味成分及其前体物质,形成具有酱香、芝麻香和浓香复合香味的特殊风味调味酒[5-7]。大曲作为糖化发酵剂富含多种微生物和酶系,麸皮为微生物生长提供营养物质,两者影响着微生物的种类与数量,在堆积和入窖发酵中有着举足轻重的作用,与白酒品质有着密切的联系。因此,阐明酒醅堆积阶段微生物群落的演替规律对复合香调味酒酿造工艺控制要点的把握及产品风格的影响意义重大。
近年来,高通量测序技术被广泛运用到浓香型、清香型、酱香型等白酒酿造的微生物群落结构分析中[8-10],包括白酒酒醅堆积过程中微生物群落演替研究。姜明慧等[11]分析酱香型白酒下沙轮次堆积发酵过程中微生物,结果表明,其优势细菌属为乳酸菌,占比高达85%以上,优势真菌属均为生香酵母;张春林等[12]研究发现,酱香型白酒酒二轮次堆积发酵酒醅中曲霉属(Aspergillus)、假丝酵母属(Candida)、裸胞壳属(Emericella)、红曲霉属(Monascus)等8种真菌属平均相对丰度较高,芽孢杆菌属(Bacillus)、海洋芽孢杆菌属(Oceanobacillus)、乳杆菌属(Lactobacillus)等7种细菌属平均相对丰度较高;张瀚之等[13]分析酱香型白酒堆积发酵酒醅的真菌菌群结构发现,嗜热真菌属(Thermomyces)、嗜热子囊菌属(Thermoascus)、Aspergillus是堆积过程中重要的真菌属。然而,堆积过程中关键工艺控制要点对微生物群落变化的影响机制仍不够明确。
为明确糖化发酵剂及麸皮对复合香调味酒堆积过程中功能微生物的影响,本研究利用可培养技术实时监测堆积过程中细菌、酵母和霉菌动态变化规律,结合高通量测序技术解析酒醅堆积工序的细菌和真菌群落结构,并对酒醅堆积过程中微生物与理化因子进行相关性分析,以期剖析堆积发酵过程中理化指标对微生物群落的影响机制,为更好地稳定和改善复合香调味酒生产工艺提供一定的指导及理论依据。
1 材料与方法
1.1 材料与试剂
麸皮、高温大曲:本地市售;酒醅、中温大曲Ⅰ、中温大曲Ⅱ:由生产试验车间提供。
NaOH、酚酞、次甲基蓝、HCl、葡萄糖、CuSO4·5H2O、酒石酸钾钠(均为分析纯):成都市科龙化工试剂厂;FastDNA SPIN Kit脱氧核糖核酸(deoxyribonucleic acid,DNA)试剂盒:美国MP Biomedicals公司;NEXTFLEX TM Rapid DNA-Seq Kit建库试剂盒:美国Bioo Scientific公司。
SPIN Kit脱氧核糖核酸(deoxyribonucleic acid,DNA)试剂盒:美国MP Biomedicals公司;NEXTFLEX TM Rapid DNA-Seq Kit建库试剂盒:美国Bioo Scientific公司。
1.2 仪器与设备
ABIGeneAmp 9700 聚合酶链式反应(polymerase chain reaction,PCR)仪、ABI7300型荧光定量PCR仪:美国ABI公司;NanoDrop2 000核酸微量测定仪:美国Thermo Fisher Scientific公司;QuantusTM Fluorometer微型荧光计:美国Promega公司;Illumina Miseq PE300高通量测序仪:美国Illumina公司。
9700 聚合酶链式反应(polymerase chain reaction,PCR)仪、ABI7300型荧光定量PCR仪:美国ABI公司;NanoDrop2 000核酸微量测定仪:美国Thermo Fisher Scientific公司;QuantusTM Fluorometer微型荧光计:美国Promega公司;Illumina Miseq PE300高通量测序仪:美国Illumina公司。
1.3 方法
1.3.1 样品采集与保存
3种糖化发酵剂和麸皮的组合方式见表1。根据实际生产中酒醅堆积60 h分别在0(开始堆积)、12 h、24 h、36 h、48 h及60 h(堆积结束)取样,每个取样时间点取样,分别在距离表面0~5 cm和20 cm处的酒醅处取样,并置于无菌取样袋中立即密封混合均匀,样品根据取样时间进行编号,样品1#(ZP1-1、ZP1-2、ZP1-3、ZP1-4、ZP1-5、ZP1-6),样品2#(ZP2-1、ZP2-2、ZP2-3、ZP2-4、ZP2-5、ZP2-6),样品3#(ZP3-1、ZP3-2、ZP3-3、ZP3-4、ZP3-5、ZP3-6),共计36个酒醅样品。再将其分为2份,一份用于理化实验分析(4 ℃保存),一份用于高通量测序(-20 ℃保存)。从堆积开始时用窖池温度计记录温度数值,以跟踪记录不同取样时间的堆积温度。
表1 不同糖化发酵剂及麸皮的组合方式
Table 1 Combination of different saccharifying starter cultures and bran

1.3.2 分析检测
理化分析:参照文献[14]的方法。淀粉含量的测定采用酸水解法;酸度的测定采用氢氧化钠滴定法;水分含量的测定采用干燥法。菌落总数的测定:参照GB 4789.2—2022《食品安全国家标准食品微生物学检验菌落总数测定》;霉菌和酵母菌数的测定:参照GB 4789.15—2016《食品安全国家标准食品微生物学检验霉菌和酵母计数》。
1.3.3 酒醅样品总DNA的提取及PCR扩增
酒醅样品微生物总DNA的提取:采用FastDNA SPINKit试剂盒提取,使用正向引物338F(5'-ACTCCTACGGGAGGCAGCAG-3')和反向引物806R(5'-GGACTACHVGGGTWTCTAAT-3')扩增细菌16S rDNA的V3-V4区域,使用正向引物ITS1F(5'-CTTGGTCATTTAGAG GAAGTAA-3')和反向引物ITS2R(5'-TGCGTTCTTCATCGATGC-3')扩增真菌ITS1区域。
SPINKit试剂盒提取,使用正向引物338F(5'-ACTCCTACGGGAGGCAGCAG-3')和反向引物806R(5'-GGACTACHVGGGTWTCTAAT-3')扩增细菌16S rDNA的V3-V4区域,使用正向引物ITS1F(5'-CTTGGTCATTTAGAG GAAGTAA-3')和反向引物ITS2R(5'-TGCGTTCTTCATCGATGC-3')扩增真菌ITS1区域。
PCR扩增:PCR扩增体系(20 μL):模板DNA10 ng,5×TransStartFastPfu缓冲液4 μL,2.5mmol/L 脱氧核糖核苷三磷酸(deoxy-ribonucleoside triphosphate,dNTPs)2 μL,引物(5 μmol/L)各0.8 μL,TransStartFastPfu DNA聚合酶0.4 μL,双蒸水(ddH2O)补足。PCR扩增条件:95 ℃预变性3 min;95 ℃变性30 s,55 ℃退火30 s,72 ℃延伸30 s,共27个循环;72 ℃再延伸10 min。将PCR产物使用2%琼脂糖凝胶电泳检测和切胶回收,并利用DNA QuantusTM Fluorometer对纯化后的回收产物进行检测定量。
1.3.4 实时荧光定量PCR
针对酒醅中芽孢杆菌设计特异性引物(B1:5'-TTGACATCCTCTGACAACCCT-3';B2:5'-GAATGCTGGCAACTAAGATCA-3')用于实时荧光定量PCR(real-time fluorescent quantitative PCR,RT-fqPCR)扩增[15]。采用绝对定量法跟踪酒醅堆积过程中芽孢杆菌的生物量[16],其中标准品为重组质粒(pMD18-T载体),根据重组质粒的拷贝数(y)与扩增循环数(x)之间的线性关系构建芽孢杆菌标准曲线[16],样品的质量浓度与模板拷贝数换算公式如下:
式中:C为模板拷贝数,copies/μL;M为质量浓度,ng/μL;N为阿佛加德罗常数,6.02×1023;L为分子质量(总长度为靶片段加载体之和),bp;D为转换因子,6.6×102g/(mol·bp)。
qPCR体系(20 μL):2×ChamQSYBR Color qPCR Master Mix 10 μL,上、下游引物(5 μmol/L)各0.8 μL,模板2 μL,ddH2O补足。将质粒标准品以10倍稀释梯度进行稀释,共制备10-3~10-8共6个稀释梯度,浓度范围为4.39×102~4.39×107(copies/μL),每个梯度取2 μL为模板建立标准曲线。qPCR的扩增程序为:变性阶段95 ℃、3min;循环阶段95 ℃、5 s,58 ℃、30 s,72 ℃1 min,40个循环。qPCR扩增结束后通过熔解曲线分析,验证扩增的特异性。
以白酒酒醅样品DNA为模板进行RT-fqPCR特异性扩增,并根据标准曲线回归方程计算酒醅中芽孢杆菌的拷贝数。每份样本先稀释10倍,然后取稀释液DNA 2 μL作为反应量,每个样本进行3次重复检测。获得的芽孢杆菌标准曲线回归方程为y=-3.267 8x+39.797(相关系数为R2=0.998 1),R2>0.99表明标准曲线可以用于荧光定量分析,从而根据标准曲线回归方程计算酒醅中芽孢杆菌的拷贝数。
1.3.5 高通量测序
利用Illumina MiseqPE300平台进行测序,委托上海美吉生物医药科技有限公司。
使用fastp(version 0.20.0)软件对原始测序序列进行质控,过滤Read尾部质量值20以下的碱基,设置50 bp的窗口,如果窗口内的平均质量值<20,从窗口开始截去后端碱基,过滤质控后50 bp以下的Reads;使用FLASH(version 1.2.7)软件进行拼接,根据PEreads之间的Overlap关系将成对的序列拼接成一条序列[17];使用Qiime2流程中的DADA2插件对质控拼接之后的优化序列进行降噪处理[18]。DADA2降噪处理之后的序列被称为扩增子序列变体(amplicon sequence variants,ASVs)。基于Sliva138 16S rRNA数据库和Unite8.0 ITS数据库,使用Qiime2中的Naivebayes分类器对ASVs进行物种分类学分析,并按照70%置信度阈值进行物种注释。
1.3.6 数据分析
为了降低测序深度对后续Alpha多样性和Beta多样性等分析结果的影响,按最小样本序列数抽平后进行后续分析。通过上海美吉生物医药科技有限公司的多样性云分析平台进行统计分析及数据可视化。使用冗余分析(redundancy analysis,RDA)及典型相关分析(canonical correlation analysis,CCA)用来反映酒醅理化指标对微生物群落结构的影响。首先采用Species-sample做数据收集分析(data collection analysis,DCA),若Lengths of gradient第一轴的大小≥3.5,选择CCA;若第一轴的大小<3.5,则选用RDA[15]。基于Spearman相关性|R|>0.6且P<0.05,挑选丰度前10的优势细菌属及真菌属进行相关性网络图分析[19]。
2 结果与分析
2.1 酒醅堆积过程中的理化指标检测结果
酒醅堆积过程中理化指标检测结果见图1。
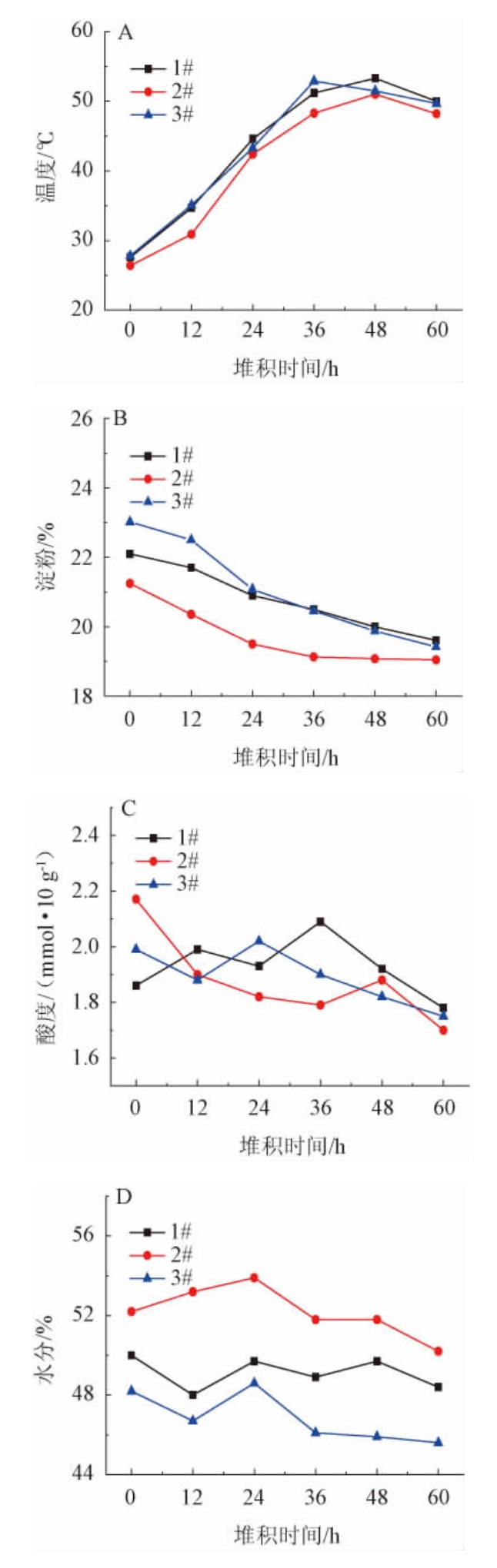
图1 酒醅堆积过程中理化指标检测结果
Fig.1 Determination results of physicochemical indexes of fermented grains during the stacking process
堆积过程中酒醅温度的变化是微生物生长代谢产生生物热与散失热达到相对平衡的结果[20]。由图1A可知,3组样品的温度随堆积时间的变化趋势一致,均呈先升高后下降的趋势。酒醅样品1#、2#、3#的温度分别在48 h、48 h、36 h达到最大值,分别为53.3 ℃、51.0 ℃、52.9 ℃。堆积开始阶段,营养丰富的酒醅具备细菌、酵母菌及霉菌等微生物快速繁殖的良好条件,微生物产生的生物热大于散失的热能,表现为堆积温度逐渐升高[21]。堆积时间为0~36 h时,与酒醅样品2#相比,同时添加了麸皮的酒醅样品1#与3#在堆积过程中,整体升温较快,推测麸皮的添加提高了酒醅疏松度和含氧量,有利于堆积过程中微生物的生长繁殖[22]。当堆积温度升至46 ℃左右时,酒醅表面出现部分白色粉末状或结块状,此时酒醅有浓郁的酒香味,说明酵母菌等微生物代谢活动旺盛,这为入窖发酵富集酵母奠定了坚实的基础[23]。因此,3组样品温度变化达到堆积发酵筛选和驯化微生物的要求。
堆积工序是一个微生物有序地利用、分解酒醅淀粉等基质产生丰富的酿酒酶系和代谢产物的工艺过程[24]。由图1B可知,3组样品的淀粉含量随堆积时间的变化趋势一致,均呈下降趋势。堆积时间为0~24 h时,酒醅堆积过程中的淀粉含量均呈较快下降趋势,其中酒醅样品3#淀粉含量下降速率最快,其原因可能是微生物代谢旺盛,产生较多的酶作用于淀粉类物质产生还原糖[15];当堆积时间>24 h,淀粉含量下降趋势趋于平缓。此外,酒醅样品3#堆积前后淀粉消耗量高于酒醅样品1#和2#。其原因可能是,此时作用于淀粉分解的酶主要源于酒醅中耐热微生物[20]。适当提高堆积温度有助于淀粉的分解,这与酒醅中糖化酶最适温度一般在40~60 ℃之间有关[25]。堆积阶段,淀粉降解生成系列可以被微生物快速吸收的物质并形成香味成分与前提物质,且堆积结束后淀粉含量为19%~20%,为后续入窖提供了良好的原材料。
由图1C可知,堆积时间为0~36 h时,酒醅样品2#的酸度呈下降趋势;堆积时间为36~48 h时,酸度有所上升;当堆积时间>48 h时,酸度下降。堆积时间为0~36 h时,酒醅样品1#的酸度呈先上升后下降再上升的趋势;堆积时间>36 h时,酒醅样品1#的酸度呈下降趋势。堆积时间为0~24 h时,酒醅样品3#的酸度呈先下降再上升的趋势;堆积时间>24 h时,酒醅样品3#的酸度呈下降趋势。总的来说,酒醅堆积过程中酸度变化曲线呈起伏状,但整体呈现下降趋势,堆积结束后酒醅酸度低于起始堆积酸度,且未加麸皮的酒醅样品2#堆积前后酸度降幅高于酒醅样品1#和3#。酒醅堆积起始酸度主要来源于粮食本身及上一排蒸馏后残留酸度,堆积过程中酒醅酸度降低的原因在于其可挥发酸性物质在开放式环境中挥发,加之酸性物质在相关菌系或酶系作用下生成相应酯类物质[20]。3组样品堆积结束的酸度范围为1.7~1.8 mmol/10 g,该范围内的酸度适宜入窖发酵时有益微生物的生长需要[10]。
由图1D可知,堆积时间为0~12 h内,酒醅样品1#、3#的水分含量呈逐渐下降的趋势;当堆积时间为12~24 h内,水分含量逐渐增加;当堆积时间>36 h时,酒醅样品1#的水分含量变化整体呈平稳趋势,酒醅样品3#的水分含量变化整体呈下降趋势。当堆积时间为0~24 h内,酒醅堆积过程中样品2#的水分含量逐渐增加;当堆积时间为24 h时,水分含量达最大值,为54.10%;堆积时间>24 h时,酒醅样品2#的水分含量变化整体呈下降趋势。原因可能是,堆积前期微生物大量繁殖产生水分,而堆积后期随着温度的升高与不耐热微生物的死亡,酒醅水分因自然挥发而逐渐降低,且酒醅温度的增加将加速水分的挥发,故堆积过程水分含量将因温度等多因素条件而变化,堆积结束后水分含量为45%~50%,该范围内的水分符合后续发酵时所需水分[20]。
综上所述,3组酒醅样品的温度、淀粉、酸度和水分理化指标在堆积过程中均处于适宜的范围内,为微生物生长提供了有利条件。
2.2 酒醅堆积过程中的微生物指标检测结果
酒醅堆积过程中微生物指标检测结果见图2。
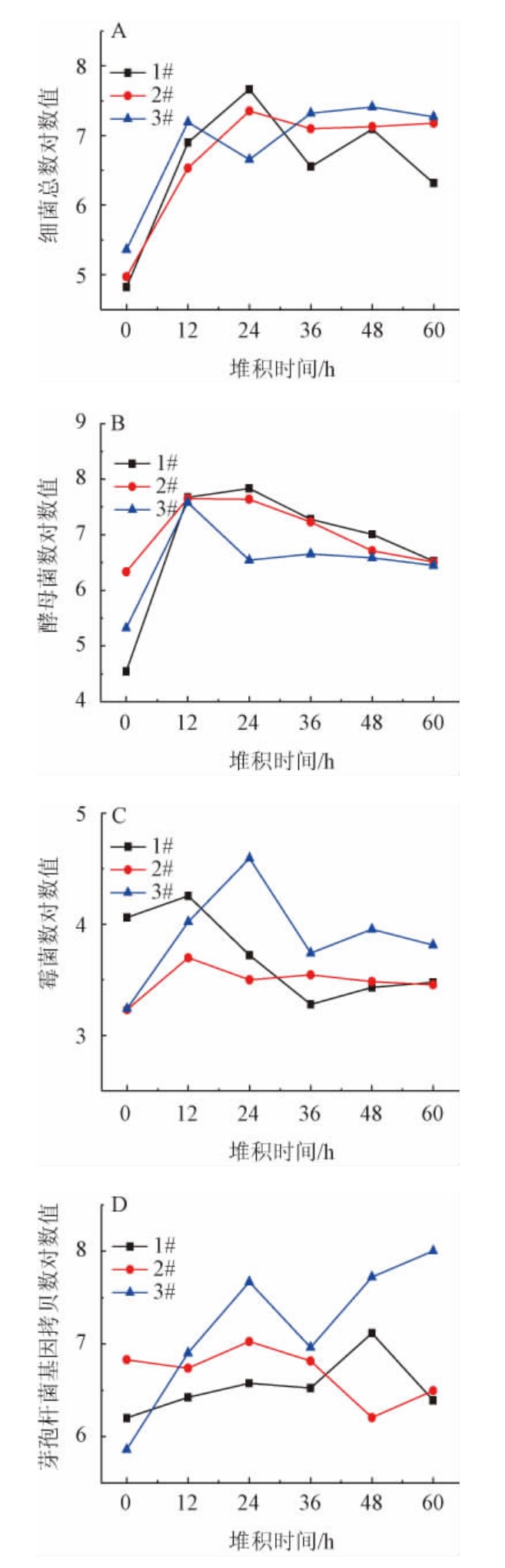
图2 酒醅堆积过程中微生物指标检测结果
Fig.2 Determination results of microbial indexes during the stacking process
由图2A可知,堆积时间为0~24 h,酒醅样品1#、2#中的细菌总数对数值逐渐增加;堆积时间>24 h,酒醅样品2#中的细菌总数对数值呈平稳趋势,而酒醅样品1#的细菌总数对数值呈先下降后上升再下降的趋势。堆积时间为0~12 h,酒醅样品3#的细菌总数对数值逐渐增加;堆积时间为12~24 h,细菌总数对数值呈下降趋势;堆积时间为24~36 h,细菌总数对数值呈上升趋势,随后趋于平稳。到堆积结束时,酒醅样品1#、2#、3#的细菌总数对数值分别为6.32、7.18、7.27,其中酒醅样品3#的细菌总数对数值最高。
由图2B可知,当堆积时间为0~60 h时,酒醅样品的酵母菌数对数值变化趋势一致,均呈先上升后下降的趋势。当堆积时间分别为24 h、24 h、12 h时,酒醅样品1#、2#、3#的酵母菌数对数值分别达到最大值,为7.58、7.83、7.65;堆积结束时,酵母数对数值均稳定至6.5。酵母菌在酒醅堆积前12 h内快速增长,当堆积温度>35 ℃,不耐热酵母菌的生长繁殖受到限制,酵母菌数量下降[26]。
由图2C可知,当堆积时间为0~60 h时,酒醅样品的霉菌数对数值变化趋势一致,均呈先上升后下降的趋势。当堆积时间分别为12 h、12 h、24 h时,酒醅样品1#、2#、3#的霉菌数对数值达到最大值,分别为4.26、3.70、4.59;到堆积结束时,酒醅样品1#、2#、3#的霉菌对数值分别为3.48、3.45、3.81。霉菌代谢产生大量的淀粉酶、糖化酶,促进生成大量的还原糖[27]。刚开始堆积时,酒醅样品1#的霉菌数对数值高于酒醅样品2#与3#。随着堆积时间的增加,细菌和酵母菌大量繁殖,释放生物热使温度升高,不耐热的霉菌数量下降[28]。
实时荧光定量分析结果包括样品中的死菌和活菌[29]。由图2D可知,当堆积时间为0~60 h,酒醅样品1#中芽孢杆菌基因拷贝数对数值呈先上升后下降趋势。酒醅样品2#和3#中芽孢杆菌的基因拷贝数对数值呈先增加后降低再增加的趋势。当堆积时间分别为48 h、24 h、60 h达到最大值,分别为7.12、7.03、8.00。堆积结束后,酒醅样品3#中芽孢杆菌基因拷贝数对数值明显高于酒醅样品1#与2#。
综上所述,堆积结束后酒醅样品3#中细菌总数、霉菌总数对数值和芽孢杆菌生物量均高于样品1#和2#,说明添加麸皮和中温大曲Ⅱ的添加有利于堆积过程中酒醅微生物的网罗与预培养。
2.3 酒醅堆积过程中微生物α多样性分析
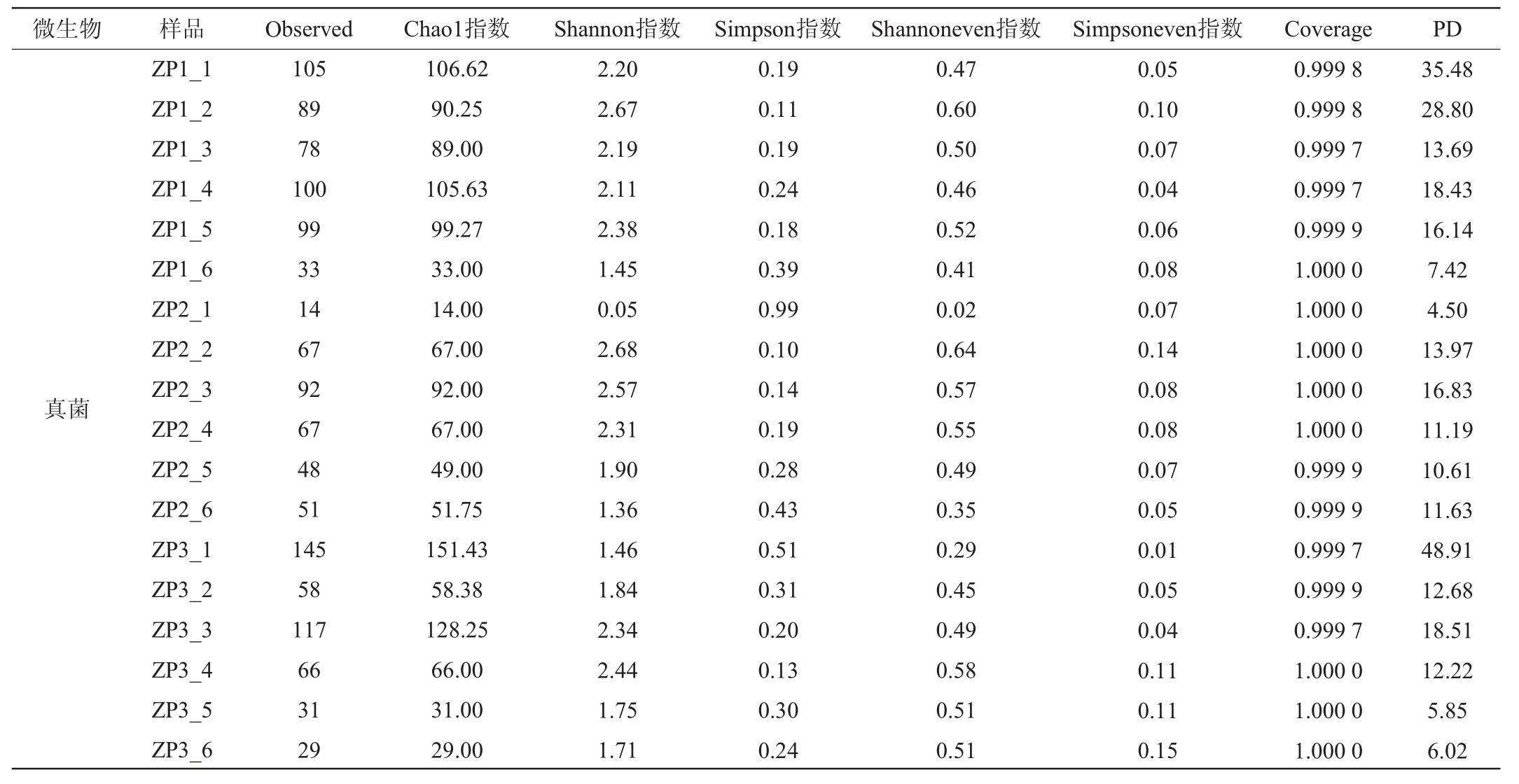
酒醅样品中微生物的α多样性分析结果见表2。Observed指实际测到的特征数,与Chao1指数均反映群落丰富度;香农(Shannon)指数和辛普森(Simpson)指数表示群落多样性;Shannoneven指数和Simpsoneven指数反映群落均匀度情况;系统发育多样性(phylogenetic diversity,PD)反映谱系多样性[30]。各样本覆盖度(Coverage)均>99%,说明测序结果可以全面准确地反映物种信息。总体来看,酒醅堆积过程中细菌菌群的丰富度和多样性均高于真菌,且酒醅样品1#和3#在整个堆积过程中原核和真核群落多样性和均匀度均高于酒醅样品2#。
表2 酒醅样品中微生物的α多样性分析结果
Table 2 Alpha diversity analysis results of microorganism in fermented grain samples

续表
2.4 酒醅堆积过程中微生物群落结构分析
2.4.1 酒醅堆积过程中细菌群落组成分析
细菌16S rDNA测序共检测出27个门、65个纲、149个目、259个科和538个属。根据物种注释结果,选取样品在属水平上相对丰度≥1%的物种(相对丰度<1%合并为Others)生成物种相对丰度堆积图,以便直观分析酒醅堆积过程中各细菌的相对丰度。基于目水平酒醅堆积发酵过程中细菌群落结构见图3,基于属水平酒醅堆积发酵过程中细菌群落结构见图4。
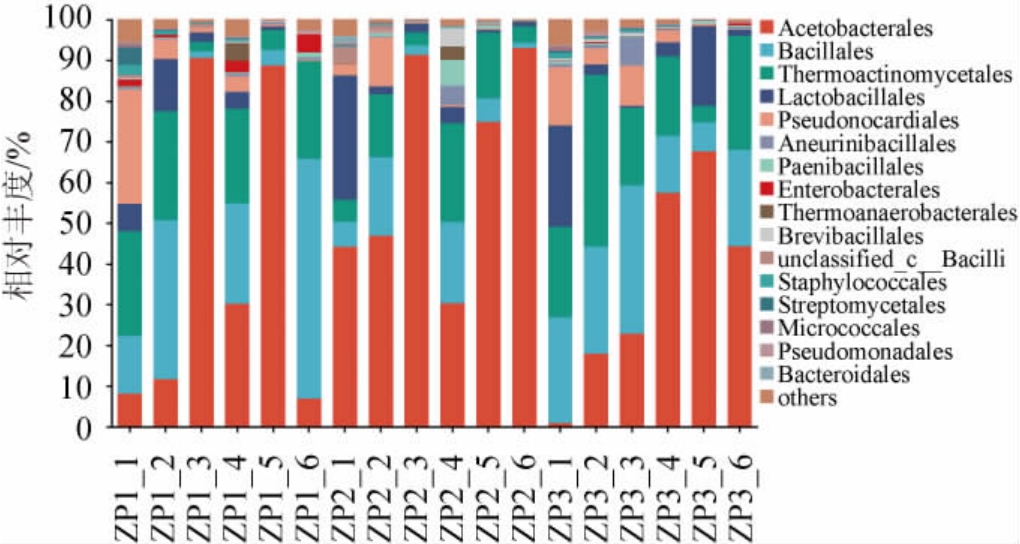
图3 基于目水平酒醅堆积过程中细菌群落结构
Fig.3 Bacterial community structure of fermented grains during the stacking process based on order level
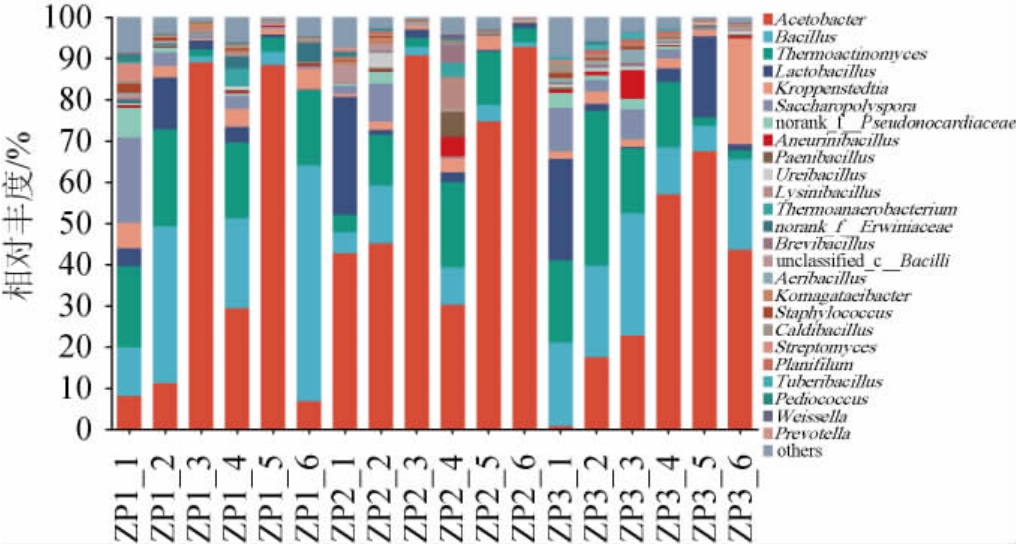
图4 基于属水平酒醅堆积过程中细菌群落结构
Fig.4 Bacterial community structure of fermented grains during the stacking process based on genus level
由图3可知,酒醅堆积过程中主要细菌为醋杆菌目(Acetobacterales)、芽孢杆菌目(Bacillales)、高温放线菌目(Thermoactinomycetales)、乳杆菌目(Lactobacillales)和假诺卡氏菌目(Pseudonocardiales),其中,醋杆菌目在酒醅样品1#、2#、3#平均相对丰度分别为39.37%、63.43%、35.16%,芽孢杆菌目在酒醅样品1#、2#、3#平均相对丰度分别为23.70%、9.11%、22.25%,高温放线菌目在酒醅样品1#、2#、3#平均相对丰度分别为17.80%、11.44%、22.51%,乳杆菌目在酒醅样品1#、2#、3#平均相对丰度分别为4.51%、6.77%、8.74%,假诺卡氏菌目在酒醅样品1#、2#、3#平均相对丰度分别为6.48%、2.62%、5.29%,表明醋杆菌目在酒醅样品1#和3#的相对丰度明显低于酒醅样品2#,芽孢杆菌目和高温放线菌目在酒醅样品1#和3#平均相对丰度明显高于酒醅样品2#。在堆积过程中细菌丰度随堆积时间的延长而变化,乳杆菌目和假诺卡氏菌目在堆积过程中整体均呈下降的趋势,与其他研究者的结论具有一致性[31]。由此可知,醋杆菌目、芽孢杆菌目、高温放线菌目、乳杆菌目和假诺卡氏菌目构成了特定酿造堆积中的细菌基本微生态。
由图4可知,酒醅堆积过程中检出25种优势细菌属,且各样品细菌菌群组成相似,但丰度存在差异。其中,优势细菌属主要为醋酸杆菌属(Acetobacter)、芽孢杆菌属(Bacillus)、高温放线菌属(Thermoactinomyces)和乳杆菌属(Lactobacillus),这4种细菌属在酒醅样品1#、2#、3#中的平均相对丰度分别为79.25%、83.93%、77.57%,该结果与前期研究报道的基本一致[14]。堆积前后,醋酸杆菌属在酒醅样品1#、2#、3#相对丰度分别由8.16%下降至6.91%、42.75%上升至92.85%、0.83%上升至43.62%,芽孢杆菌属在酒醅样品1#、2#、3#相对丰度分别由11.87%上升至57.20%、5.15%下降至1.04%、20.20%上升至21.95%,高温放线菌在酒醅样品1#、2#、3#相对丰度分别由19.54%、4.17%、19.88%下降至18.32%、3.47%、2.21%,乳杆菌属大幅在酒醅样品1#、2#、3#相对丰度分别由4.46%、28.67%、24.68%下降至0.04%、1.29%、1.37%。堆积结束后,酒醅样品3#中优势细菌微生物占比更均衡。醋酸杆菌属、乳杆菌属的代谢产物主要分别为乙酸、乳酸,可结合入窖发酵过程中产生的乙醇等物质,丰富酒体中乙酸乙酯、乳酸乙酯等风味成分[32]。芽孢杆菌属和高温放线菌属均可适应较高的温度,普遍存在于堆积酒醅中,具有分泌蛋白酶、淀粉酶等多种功能酶的能力,催化大分子物质分解形成风味物质,具有产酶和产香的功能[33-35]。与酒醅样品1#相比,酒醅样品3#在堆积前后醋酸杆菌属相对丰度的增长幅度明显高于酒醅样品1#堆积前后,其可能的原因在于中温大曲Ⅱ比中温大曲Ⅰ更适宜酒醅中醋酸杆菌属的快速繁殖;与酒醅样品2#相比,酒醅样品3#在堆积过程中芽孢杆菌属的相对丰度显著高于酒醅样品2#(P<0.05),且堆积结束后酒醅样品3#中芽孢杆菌属的相对丰度明显高于酒醅样品2#,与实时荧光定量结果(图2D)一致,其可能的原因在于麸皮提高了酒醅疏松度和含氧量,更有利于酒醅中芽孢杆菌属的富集[36]。酒醅样品3#中糖多孢菌属(Saccharopolyspora)和克罗彭斯特菌属(Kroppenstedtia)的丰度增加,这两种菌属也是芝麻香型白酒高温大曲中的优势菌属[37]。研究表明,糖多孢菌属与氨基酸合成、脂肪酸合成等有重要联系,且能产生多种功能酶与生物活性物质,在发酵过程中扮演着重要的角色[38];克罗彭斯特菌属是一种嗜热菌属,主要存在于高温大曲和堆积酒醅中[39-40]。因此,复合香白酒堆积过程实现了对耐热细菌的富集,促进后续发酵中酱香、芝麻香风味的形成并提供发酵动力[12,14]。
2.4.2 酒醅堆积过程中真菌群落组成分析
真菌ITS分析共检测出6个门、16个纲、34个目、73个科和132个属。根据物种注释结果,选取样品在目水平、属水平上相对丰度≥1%的物种(<1%合并为Others),生成物种相对丰度堆积图,以便直观分析酒醅堆积过程中各真菌的相对丰度。基于目水平酒醅堆积发酵过程中真菌群落结构见图5,基于属水平酒醅堆积发酵过程中真菌群落结构见图6。
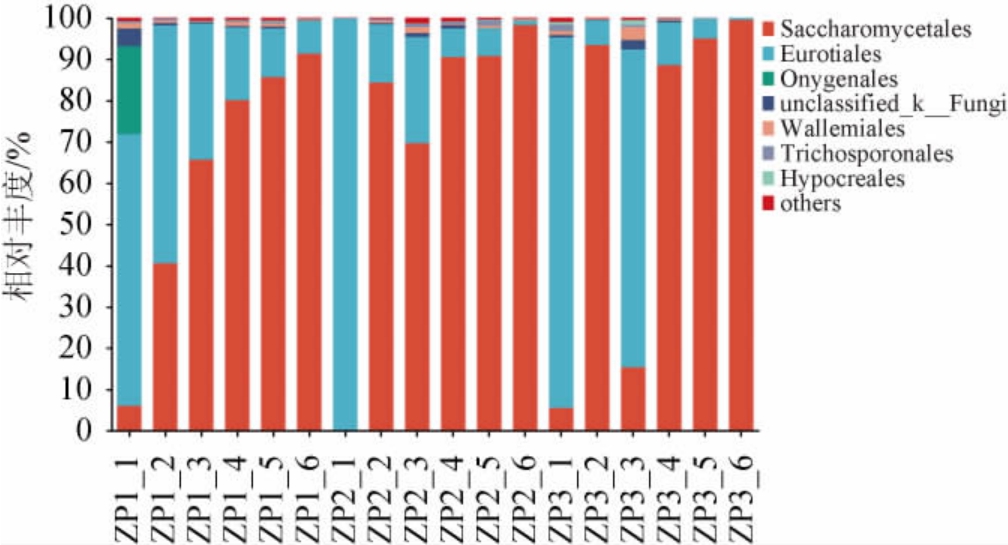
图5 基于目水平酒醅堆积过程中真菌群落结构
Fig.5 Fungal community structure of fermented grains during the stacking process based on order level
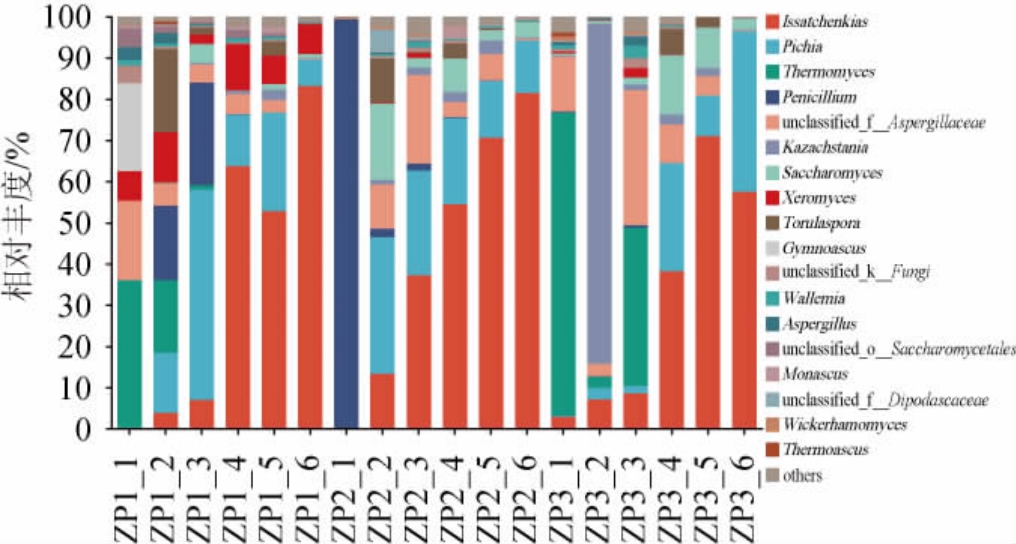
图6 基于属水平酒醅堆积发酵过程中真菌群落结构
Fig.6 Fungal community structure of fermented grains during the stacking process based on genus level
由图5可知,酒醅堆积过程中主要真菌为酵母菌目(Saccharomycetales)和散囊菌目(Eurotiales),酵母菌目在酒醅样品1#、2#、3#中的平均相对丰度分别为61.62%、72.37%、66.27%,散囊菌目在酒醅样品1#、2#、3#中的平均相对丰度分别为32.27%、25.70%、31.39%。随着堆积时间的增加,酵母菌目相对丰度增加,散囊菌目相对丰度降低。堆积开始时散囊菌目比例最高,表明其能在该酿造环境条件下生长代谢,将原料中的淀粉与蛋白质分解成葡萄糖与氨基酸。而堆积后期,散囊菌目比例大幅下降,酵母菌目开始大量增多,堆积结束时酵母菌目在样品1#、2#、3#相对丰度分别为91.46%、98.38%、99.52%,占据了酒醅堆积过程中真菌的绝对优势,说明酵母菌在堆积过程中大量富集,促进了后续乙醇发酵的高活性。经过堆积过程的富集和驯化,有益微生物得到了最大程度的繁殖,为后续发酵提供了充足的发酵动力,也促进白酒风味物质的形成。
由图6可知,在属水平上,主要优势真菌属包括伊萨酵母属(Issatchenkia)、毕赤酵母属(Pichia)和嗜热真菌属(Thermomyces),这3种真菌属在酒醅样品1#、2#、3#中的平均相对丰度分别为62.32%、60.54%、63.35%。随着堆积时间的增加,伊萨酵母属在整个堆积过程呈上升趋势,堆积结束时,伊萨酵母属在酒醅样品1#、2#、3#相对丰度分别为83.10%、81.07%、57.22%,这主要是因为伊萨酵母属具有耐高温能力[41],且其对酒醅的香气形成具有重要作用。其次,毕赤酵母属在酒醅堆积过程整体呈先上升后下降趋势,且堆积结束后其在酒醅样品3#的相对丰度最高。有研究表明,伊萨酵母属和毕赤酵母属这两种酵母是芝麻香型白酒发酵过程的主要酵母菌[42]。嗜热真菌属未在酒醅样品2#中检出,且仅存在酒醅样品1#和3#的堆积阶段前期,堆积结束后的酒醅中未检出。unclassified_f__Aspergillaceae在堆积前期丰度较高,随后相对丰度占比降低,堆积结束后所占比例非常小,说明部分不耐温的霉菌被死亡。因此,伊萨酵母属和毕赤酵母属为酒醅堆积过程中优势真菌,这2种真菌属有利于后期入窖发酵形成更多风味物质。
2.5 理化因子与优势微生物属关联性分析
由酒醅样品堆积过程中理化指标与优势细菌属及真菌属与理化因子相关性分析结果见图7,微生物与理化因子相关性网络分析见图8。
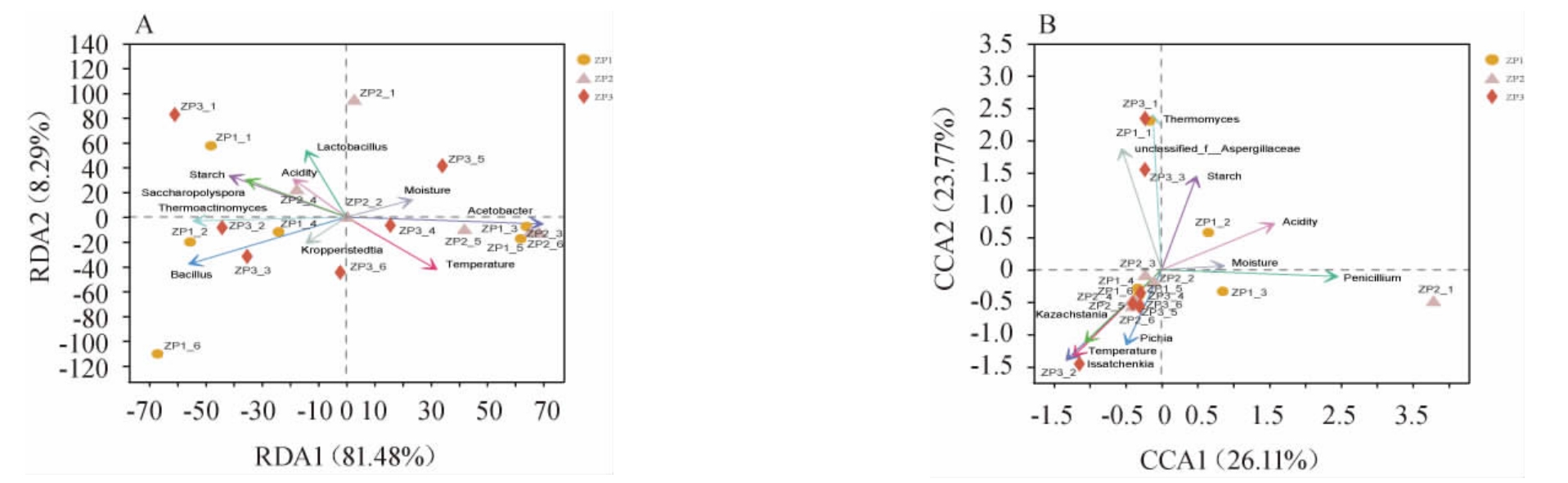
图7 酒醅堆积过程中基于属水平细菌与理化因子冗余分析(A)、真菌与理化因子典型相关分析(B)
Fig.7 Redundancy analysis of bacteria and physicochemical factors (A),canonical correlation analysis of fungi and physicochemical factors (B)based on genus level during the stacking fermentation
不同种属的微生物与理化因子箭头之间的夹角为锐角时呈正相关,为钝角时呈负相关[43]。
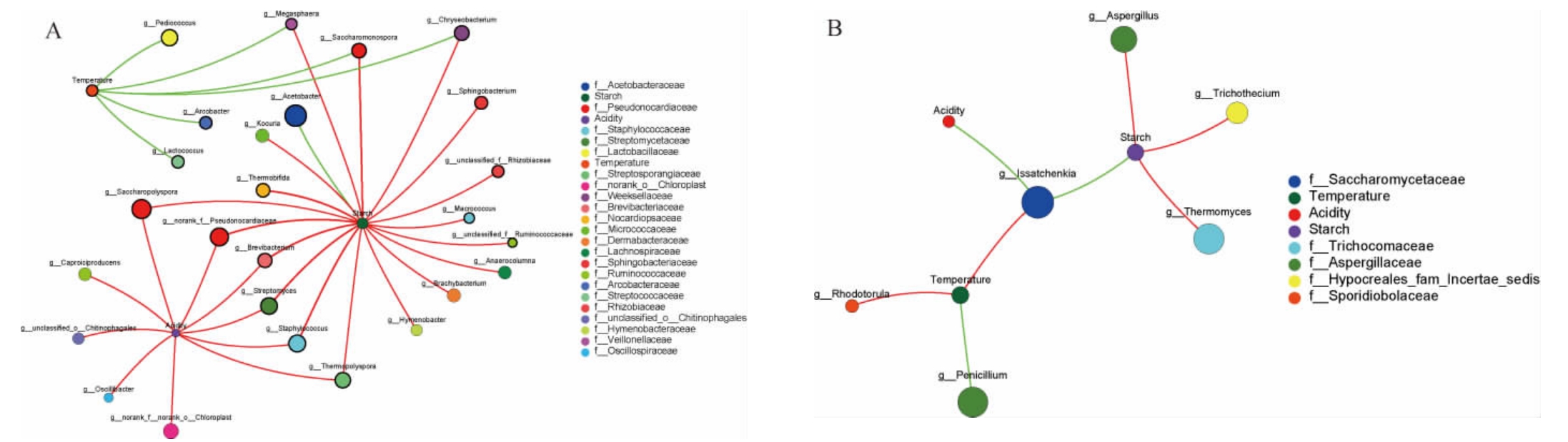
图8 酒醅堆积过程中基于属水平细菌(A)、真菌(B)与理化因子相关性网络分析
Fig.8 Correlation network analysis between bacteria (A),fungi (B) and physicochemical factors based on genus level during the stacking fermentation
红线代表正相关,绿线代表负相关[44]。
由图7A可知,酸度与Lactobacillus呈显著正相关,水分与Acetobacter呈显著正相关,温度与Lactobacillus呈负相关;由图7B可知,酸度与Saccharopolyspora呈显著正相关,水分与Penicillium呈显著正相关,淀粉与Thermoactinomyces、Saccharopolyspora和Thermomyces呈显著正相关,温度与Issatchenkia呈正相关。由图8A可知,淀粉与绝大多数细菌呈正相关,酸度与Staphylococcus、Streptomyces等细菌呈正相关,温度与Saccharopolyspora、Pediococcus等细菌呈负相关;由图8B可知,淀粉与Aspergillus等真菌呈正相关,水分和酸度与Issatchenkia、Pichia等呈正相关,与Monascus等呈负相关。从图中可以明显看出,白酒发酵过程中在胁迫因子的推动下,微生物之间通过相互制约、相互促进影响群落结构的演替。
3 结论
本研究考察了糖化发酵剂及麸皮对复合香调味酒堆积过程中理化指标及微生物指标的影响,并基于高通量测序技术探究堆积过程中微生物群落结构变化。结果表明,3组酒醅样品的理化指标在堆积过程中均处于适宜的范围内,且堆积结束后,酒醅样品3#中细菌总数、霉菌总数对数值和芽孢杆菌生物量均高于样品1#和2#,醋酸杆菌属、芽孢杆菌属、高温放线菌属和乳杆菌属为酒醅堆积过程中主要优势细菌属;伊萨酵母属、毕赤酵母属和嗜热真菌属为堆积过程中主要优势真菌属。酒醅堆积过程中微生物群落结构的变化与理化因子有显著相关关系,酸度与Lactobacillus、Saccharopolyspora呈显著正相关,水分与Acetobacter、Penicillium呈显著正相关,淀粉与Thermoactinomyces、Saccharopolyspora和Thermomyces呈显著正相关,温度与Lactobacillus呈负相关、与Issatchenkia呈正相关。综合考虑,采用3#的组合方式更适于酿造复合香调味酒。本研究通过不同糖化发酵剂及麸皮组合,结合续糟配料与高温堆积工艺,创新性酿造复合香型调味酒,为实际生产上的堆积调控与工艺提供科学指导与理论依据,同时为白酒企业发展个性化风味产品提供思路。
[1]曹静,余有贵,曹智华,等.中国复合香型白酒研究进展[J].食品与机械,2017,33(7):200-204.
[2]韩兴林,张五九,李红.从标准简单分析白酒香型的发展[J].中国酿造,2015,34(5):1-6.
[3]张治刚,张彪,赵书民,等.中国白酒香型演变及发展趋势[J].中国酿造,2018,37(2):15-18.
[4]苏金兰,徐柏田,林培.中国白酒香型发展的进展研究[J].酿酒科技,2017(8):102-110.
[5]李增,程伟,张倩,等.金种子中国力量复合香白酒的生产工艺分析与探讨[J].酿酒科技,2019(8):48-53.
[6]葛向阳,何惠昭,徐岩,等.杜康复合香酿酒堆积与发酵过程中酒醅理化变化趋势研究[J].酿酒科技,2019(12):89-92.
[7]高传强,阳飞,张华山.芝麻香型白酒微生物菌群及风味物质研究进展[J].微生物学通报,2017,44(4):940-948.
[8]赵志平,陈泓帆,韩煦,等.浓香型白酒发酵结束后不同层酒醅的微生物多样性分析[J].食品与发酵科技,2021,57(5):17-21.
[9]田德雨,闫子茹,危晶晶,等.清香型白酒酿造微生物和风味物质的研究进展[J].中国酿造,2021,40(4):20-25.
[10]陈雪,张永利,闫宗科,等.凤香型酒醅微生物群落演替及其与理化指标的相关性分析[J].食品科学,2020,41(22):200-205.
[11]姜明慧,许玲,于文娟,等.高通量测序技术分析酱香型白酒下沙轮次堆积过程的微生物多样性[J].酿酒,2020,47(4):51-55.
[12]张春林,杨亮,李喆,等.酱香型白酒二轮次堆积酒醅微生物群落与理化指标相关性研究[J].中国酿造,2021,40(11):31-36.
[13]张瀚之,席德州,王欢,等.酱香型白酒机械化酿造不同轮次堆积发酵酒醅真菌群落结构多样性研究[J].中国酿造,2021,40(12):64-69.
[14]曹润洁,何宏魁,葛向阳,等.芝麻香型酒醅堆积过程中微生物结构变化分析[J].微生物学通报,2019,46(6):1371-1380.
[15]TANG H L,LIANG H B,SONG J K,et al.Comparison of microbial community and metabolites in spontaneous fermentation of two types Daqu starter for traditional Chinese vinegar production[J].J Biosci Bioeng,2019,128(3):307-315.
[16]GAO L,YU H X,HAN W S,et al.Development of a SCAR marker for molecular detection and diagnosis of Tilletia controversa Kühn,the causal fungus of wheat dwarf bunt[J].World J Microbiol Biotechnol,2014,30(12):3185-3195.
[17]CHEN S F,ZHOU Y Q,CHEN Y R,et al.fastp:an ultra-fast all-in-one FASTQ preprocessor[J].Bioinformatics,2018,34(17):i884-i890.
[18]BOLYEN E,RIDEOUT J R,DILLON M R,et al.Reproducible,interactive,scalable and extensible microbiome data science using QIIME 2[J].Nat Biotechnol,2018,37(8):852-857.
[19]BARBERÁN A,BATES S T,CASAMAYOR E O,et al.Using network analysis to explore co-occurrence patterns in soil microbial communities[J].ISME J,2012,6(2):343-351.
[20]杨国棋,曹利丰,潘春芳,等.芝兰香型白酒堆积过程状态研究[J].发酵科技通讯,2021,50(2):82-85.
[21]陈向田.芝麻香型白酒堆积过程中关键功能微生物变化的研究[J].酿酒,2018,45(6):39-41.
[22]张温清.芝麻香型白酒四甲基吡嗪形成及其高产TTMP酿造工艺研究[D].合肥:合肥工业大学,2020.
[23]郭桂梅,杨军山,杨帅宇,等.高温堆积生产浓香调味酒研究与应用[J].酿酒科技,2021(12):90-94.
[24]山其木格,梁慧珍,张长霞,等.糟醅堆积过程中微生物种群的变化规律[J].食品与生物技术学报,2016,35(3):330-335.
[25]TAYLOR P M,NAPIER E J,FLEMING I D.Some properties of a glucoamylase produced by the thermophilic fungus Humicola lanuginosa[J].Carbohydr Res,1978,61(1):301-308.
[26]宋瑞滨,邵泽良,宋军,等.黄山头包包曲发酵过程中部分理化指标与微生物群落演替规律的研究[J].酿酒,2019,46(5):42-46.
[27]王佳丽,朱丹,孙列雄,等.山西老陈醋大曲霉菌生理代谢特征及优良菌株间的互作[J].中国食品学报,2021,21(4):79-89.
[28]胡晓龙,冯大鸿,田瑞杰,等.中温大曲发酵过程中细菌群落结构解析及功能预测[J].轻工学报,2023,38(2):23-32.
[29]张昭寰,娄阳,杜苏萍,等.分子生物学技术在预测微生物学中的应用与展望[J].食品科学,2017,38(9):248-257.
[30]梁宗林,秦亚玲,王沛,等.云南省蒙自酸性矿山排水微生物群落结构和功能[J].生物工程学报,2019,35(11):2035-2049.
[31]赵才贤.酱香型白酒大曲和酒醅的微物群落结构特征及其变化规律研究[D].贵阳:贵州大学,2021.
[32]韩兴林,崔磊,常强,等.浓香白酒发酵过程中酒醅微生物结构分析[J].酿酒,2021,48(1):81-86.
[33]汤涵岚.基于宏基因组分析增温对食醋大曲固态发酵过程中微生物菌群的影响[D].广州:华南理工大学,2019.
[34]SU Y,YANG L,HUI L,et al.Bacterial communities during the process of high-temperature Daqu production of roasted sesame-like flavour liquor[J].J I Brewing,2015,121(3):440-448.
[35]李豆南,黄魏,王晓丹,等.酱香型大曲中高温放线菌的筛选及风味成分分析[J].食品科学,2018,39(6):171-176.
[36]于有伟,闫可婧,陶玮红,等.小麦麸皮对枯草芽孢杆菌发酵花生饼粕的影响[J].农产品加工,2023,567(1):5-8.
[37]姚粟,葛媛媛,李辉,等.利用非培养技术研究芝麻香型白酒高温大曲的细菌群落多样性[J].食品与发酵工业,2012,38(6):1-6.
[38]吴冬冬,张建丽.糖多孢菌属的多相分类[J].微生物学杂志,2007,27(1):55-58.
[39]张颖,李霄霄,李景辉,等.高产四甲基吡嗪芽孢杆菌的筛选及其对酱香型白酒堆积过程的影响[J].食品工业科技,2022,43(2):142-149.
[40]ZHANG H X,WANG L,TAN Y W,et al.Effect of Pichia on shaping the fermentation microbial community of sauce-flavor Baijiu[J].Int J Food Microbiol,2021,336(2):108898.
[41]彭俊,杨团元,刘蒲临,等.白酒酿造酒醅与周边土壤中酵母菌多样性及东方伊萨酵母发酵特性比较[J].中国酿造,2019,38(8):77-84.
[42]WU Q,LING J,XU Y.Starter culture selection for making Chinese sesame-flavored liquor based on microbial metabolic activity in mixedculture fermentation[J].Appl Environ Microbiol,2014,80(14):4450-4459.
[43]ZHENG X W,YAN Z,NOUT M J R,et al.Microbiota dynamics related to environmental conditions during the fermentative production of Fen-Daqu,a Chinese industrial fermentation starter[J].Int J Food Microbiol,2014,182-183:57-62.
[44]LI Z Y,FENG C X,LUO X G,et al.Revealing the influence of microbiota on the quality of Pu-erh tea during fermentation process by shotgun metagenomic and metabolomic analysis[J].Food Microbiol,2018,76:405-415.