白酒是我国最具代表性的传统发酵食品之一,其酿造是由多种微生物驱动的群体代谢过程,在此过程中多菌系的水解酶协同辅助谷物中的大分子物质(淀粉、蛋白质、纤维素、多糖等)转变成小分子营养物质(单糖、氨基酸等),这些小分子物质被发酵微生物利用,最终形成白酒中的风味物质[1]。所以微生物对于白酒生产的重要性不言而喻,因此明确酿造过程中微生物组成和变化有助于明晰发酵中微生物相互作用的本质,对保证白酒发酵系统生产性能和稳定具有重要作用。
目前解析白酒酿造中“菌系”的主要方法包括传统可培养分离法、微生物多样性分析(16S rDNA和ITS rDNA)、宏基因组学、宏转录组学等组学技术。其中传统可培养法在菌株水平上分析微生物的生理代谢,可直接通过不同筛选分离条件获得纯菌进行应用[2-4],但这种方法无法掌握样品中绝大部分菌种信息[5-6];微生物多样性分析可免培养且进行群落分析,主要基于扩增子测序技术,二代测序技术具有通量高、准确率高、可定性及相对定量及价格低廉等优点[7-8],但由于只可识别部分高变区扩增和测序靶点,导致对微生物的鉴定仅到属水平[9];宏基因组可以精确的测量微生物的群落结构及基因组,使确定关键微生物及群落核心生物成为可能,但其相对于扩增子测序的通量较低[10-11];宏转录组可以精确表达微生物的代谢活性,但其基因表达水平容易受到样品自身因素的影响[12-13]。综合各方面来看,微生物多样性分析因其价格便宜且通量高进而被广泛应用在白酒微生物群落多样性及其演替规律中[14]。
与工业液态发酵不一样,白酒固态发酵多采用边糖化边发酵的双边发酵方法[15],其中原料自身影响对于发酵影响较大,因此如何有效富集微生物体系同时排除原料的干扰至关重要。目前对于白酒酒醅预处理的方法主要集中于应用磷酸盐缓冲液(phosphate buffer solution,PBS)离心洗涤酒醅[16-17],在此基础上研究者通过化学法和物理法不断改良,如会添加溶菌酶、溶壁酶、蛋白酶等多种酶系来去除酒醅中大量多糖、粗粮蛋白等抑制剂的干扰[18-20],还会通过增加离心次数及改变离心转速去除干扰[21-22]。目前的文献大多都是基于处理后的样品进行多样性分析,但未有文献报道表明预处理对于酒醅菌系解析的影响。因此,本试验利用扩增子测序技术探究PBS预处理对酱香型和浓香型酒醅菌群结构测定的影响,为全面解析白酒机理提供理论基础。
1 材料与方法
1.1 材料与试剂
酱香型白酒酒醅(第7轮次):贵州某酱香型酒厂;浓香型白酒酒醅:天津某浓香型酒厂;0.1 mol/L磷酸盐缓冲液(PBS):0.1 mol/L KH2PO4和0.1 mol/L Na2HPO4按照1∶9(V/V)比例配制。
1.2 仪器与设备
TD-C/K型电子天平:天津天马衡基仪器有限公司;CT88A型高压灭菌锅:上海驰通仪器有限公司;DW-86L578J型超低温冷冻储存箱:青岛海尔股份有限公司;17R型低温高速离心机:广州立诺自动化设备有限公司。
1.3 方法
1.3.1 样品采集
选取生产质量稳定的窖池,取酱香型第7轮次出窖酒醅及浓香型出窖酒醅,对每口窖池的上层(距窖顶0.5 m)、中层(距窖顶1 m)、下层(距窖底0.5 m)的酒醅分别进行5点取样并充分混匀备用。PBS预处理前的浓香型酒醅样品编号为N1,处理后样品编号为N2;酱香型酒醅PBS预处理前样品编号为M1,处理后样品编号为M2。共4组样品,每组设置3个重复。
1.3.2 样品预处理
称取30 g酒醅溶于100 mL 0.1 mol/L磷酸盐缓冲液(pH 8.0)中,充分振荡混匀,用布氏漏斗过滤去除高粱、麸皮等大颗粒谷物杂质,利用高速冷冻离心机4 ℃、10 000 r/min离心10 min,去除滤液,收集沉淀物于-80 ℃保存。
1.3.3 扩增子测序及数据处理
脱氧核糖核酸(deoxyribonucleic acid,DNA)提取及聚合酶链式反应(polymerase chain reaction,PCR)扩增均由华大基因公司承担。选择通用引物338F(5'-ACTCCTACGGGAGGCAGCA-3')和806R(5'-GGACTACHVGGGTWTCTAAT-3')对细菌的16S r RNA的V3-V4区域基因序列进行PCR扩增分析细菌菌群结构[23]。选择通用引物18S rRNA基因与5.8S rRNA基因间的内转录间隔1区(internal transcribed spacer-1,ITS-1)(5'-CTTGGTCATTTAGAGGAAGTAA-3')和ITS2(5'-GCTGCGTTCTTCATCGATGC-3')对真菌的ITS区域基因序列进行PCR扩增分析真菌菌群结构[24]。依托深圳华大基因公司进行Illumina HiSeq高通量测序,使用Flash对每个样品reads进行拼接获取原始数据,通过Qiime 2进行数据过滤后得到高质量数据[25],之后利用Usearch[26]软件进行操作分类单元(operational taxonomic units,OTU)聚类;用Uclust[27]软件将全部序列按照97%相似度聚类,进行OTU划分;通过OTU与数据库比对,原核微生物与Greengenes数据库中的菌种信息进行比对[28],真核微生物与Unite数据库中的菌种信息比对[29],得到物种注释分析。
1.3.4 统计分析
使用Mothur 1.32.0软件对样本进行Alpha多样性分析,使用Origin 9.0软件绘制群落组成图,使用SPSS 22.0软件进行结构差异性分析,使用Simca 13.0软件进行聚类分析。
2 结果与分析
2.1 酒醅α多样性分析
2.1.1 OTU分析
为研究处理前后浓香和酱香型酒醅的物种组成,使用Usearch软件对有效序列进行OTU聚类,两种酒醅样品中原核及真核微生物OTU分布韦恩图见图1。
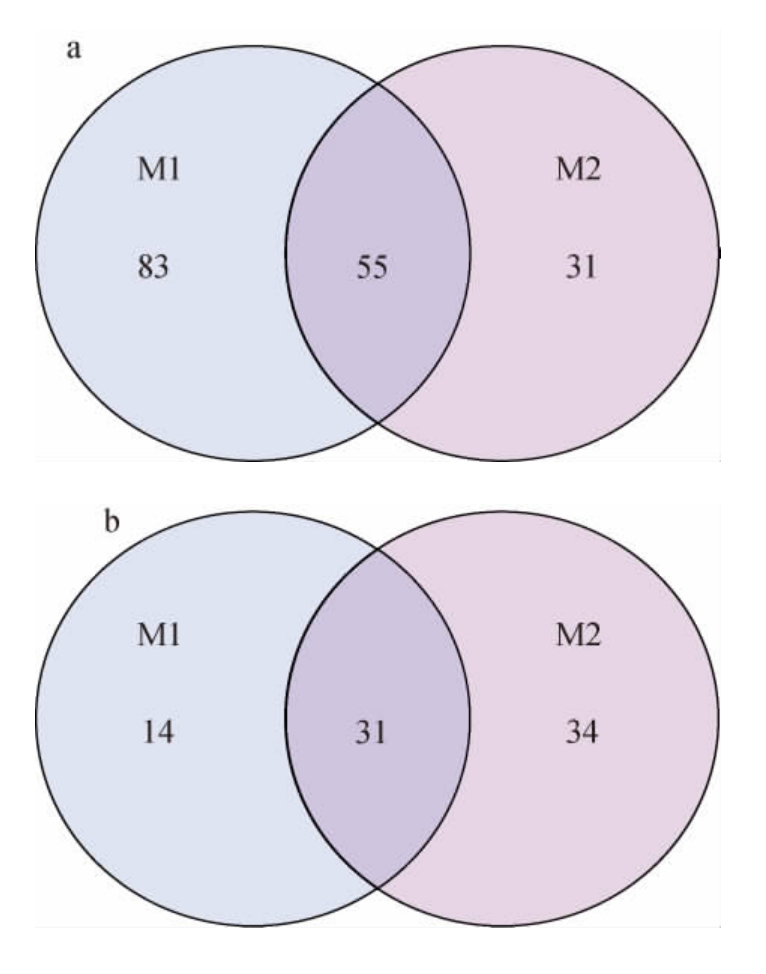
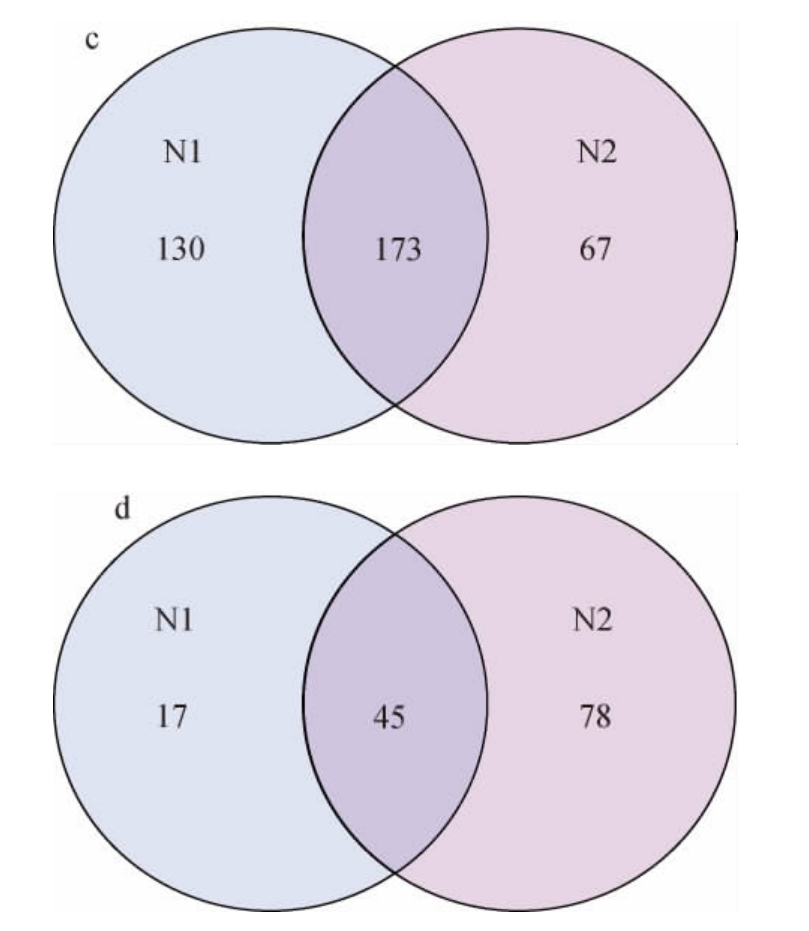
图1 酱香型(a、c)及浓香型(b、d)白酒酒醅样品中原核及真核微生物OTU分布韦恩图
Fig.1 Venn diagram of OTU distribution of prokaryotic and eukaryotic microorganisms in fermented grains samples of sauce-flavor (a and c) and strong-flavor (b and d)Baijiu
由图1可知,酱香型白酒酒醅M1和M2注释到的原核微生物OTU数量分别为138个、86个,共有OTU 55个;注释到的真核微生物OTU数量分别为45个、65个,共有OTU 31个。浓香型酒醅N1和N2注释到的原核微生物OTU数量分别为303个、240个,共有OTU 173个;注释到的真核微生物OTU数量分别为62个、123个,共有OTU 45个。表明经PBS处理后,浓香型及酱香型白酒酒醅真核微生物的特有OTU数量均上升,而原核微生物的特有OTU数量均下降。对预处理前后reads进行比对后发现,差异大于1 000 reads的OTU均在共有OTU内,也是构成样品物种组成的优势菌属。这说明预处理改变了OTU数量但对OTU种类影响较小。
2.1.2 Alpha多样性指数分析
Alpha多样性分析是对单个样品中物种多样性的分析,可以反映物种丰富程度,主要包括Chao1指数和Shannon指数两个评判指标。其中,Chao1指数常用来估计物种总数,Chao1值越大代表样品中微生物群落丰度越高;Shannon指数则用来估算样品中微生物的多样性,数值越高,表明微生物群落的多样性越高。两种酒醅样品Chao1(a)和Shannon(b)指数小提琴图见图2。由图2a可知,PBS预处理后,两种酒醅真核微生物的Chao1指数显著提高(P<0.05),表明经过预处理的两种酒醅真核微生物物种丰度增加。而两种酒醅原核微生物Chao1指数均显著减少(P<0.05),说明预处理后两种酒醅原核微生物的物种丰度减少。由图2b可知,预处理后的两种香型酒醅真核生物和原核生物Shannon指数无显著性差异(P>0.05),表明预处理对两种酒醅微生物群落多样性影响较小。总体来看,Alpha多样性指数表明两种酒醅预处理前后微生物丰度上具有显著差异(P<0.05),而在微生物种类上无显著差异(P>0.05)。从而在扩增子角度显示预处理主要会影响样品微生物的丰度,但对样品的组成影响较小。
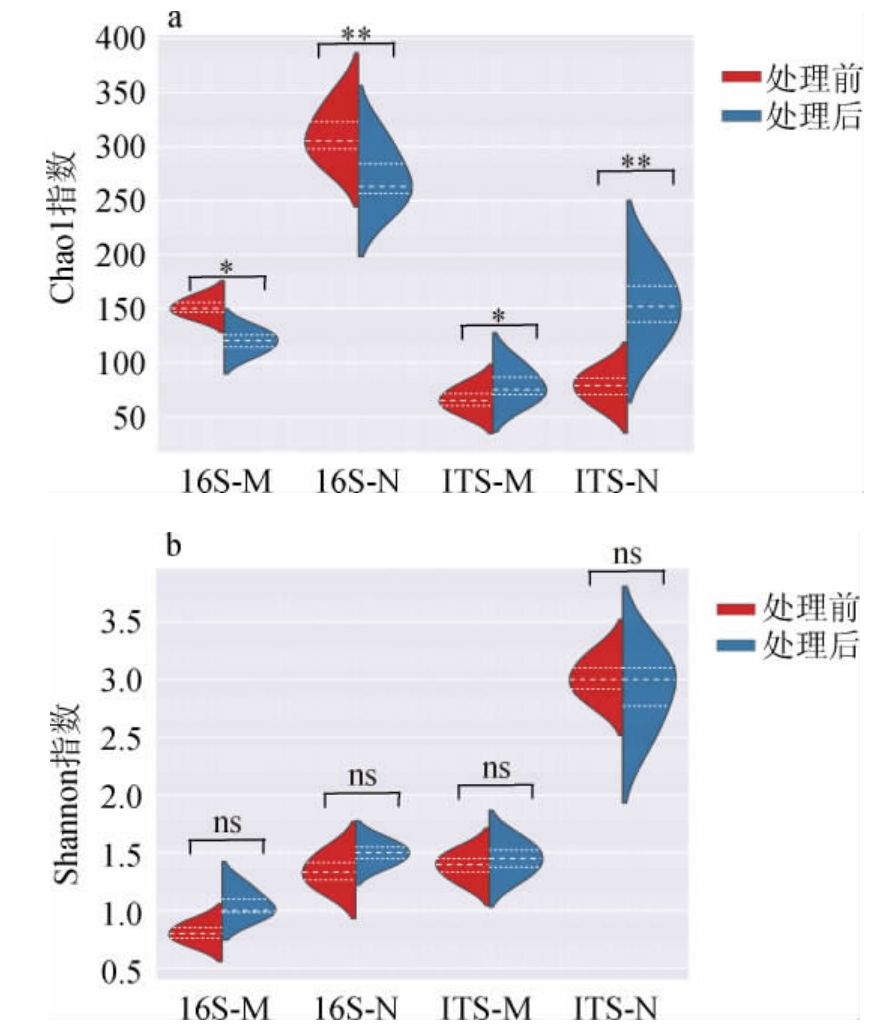
图2 两种酒醅样品Chao1(a)和Shannon(b)指数小提琴图
Fig.2 Violin plot of Chao1 (a) and Shannon (b) indexes of two fermented grain samples
“*”表示差异显著(P<0.05),“**”表示差异极显著(P<0.01),“ns”表示差异不显著(P>0.05)。
2.2 酒醅微生物群落组成分析
2.2.1 门水平上真核微生物物种分析
基于酒醅样品中微生物ITS rDNA基因序列,以97%序列相似性将有效序列在门水平上进行聚类,选取相对丰度<0.01%的微生物归为Others。将相对丰度>10%的微生物定义为绝对优势微生物门(或属),相对丰度>1%的微生物定义为优势门(或属)。
由表1可知,酱香型白酒酒醅所检出的真菌群落归属于3个优势真菌门,分别为子囊菌门(Ascomycota)、担子菌门(Basidiomycota)和接合菌门(Zygomycota)。PBS预处理前后酱香型白酒酒醅中绝对优势真菌门均为子囊菌门,相对丰度均>99%,占主导地位,而其余菌门预处理前后变化幅度不大。浓香型白酒酒醅预处理前后绝对优势真菌门均为子囊菌门和担子菌门(Basidiomycota),处理后优势真菌门增加了接合菌门,相对丰度增加0.68%。总体来看,两种香型白酒酒醅预处理前后真核微生物门水平上物种组成及丰度上变化较小。
表1 基于门水平酒醅样品中真核微生物相对丰度及变化
Table 1 Relative abundance and variation of eukaryotic microorganisms in fermented grain samples based on phylum level
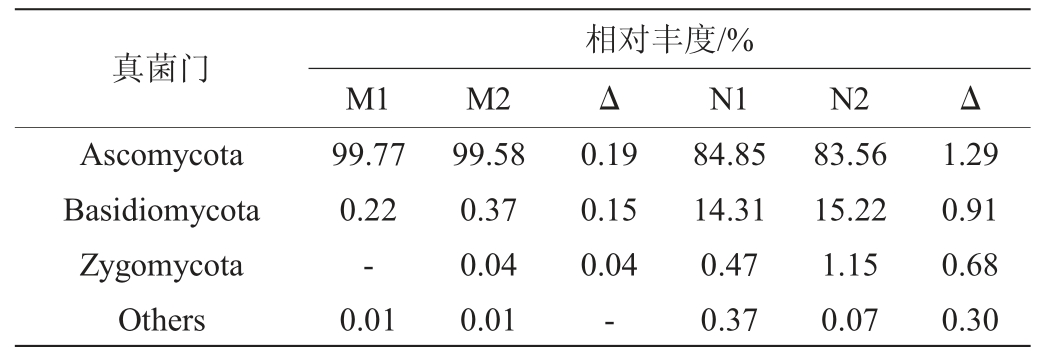
注:“Δ”表示处理前后相对丰度变化的绝对值;“-”表示未检出。下同。
2.2.2 属水平上真核微生物物种分析
由表2可知,处理前酱香型白酒酒醅优势菌属为假丝酵母属(Candida)、接合酵母属(Zygosaccharomyces)、曲霉属(Aspergillus)、红曲霉属(Monascus)、裂殖酵母属(Schizosaccharomyces)和毕赤酵母属(Pichia),相对丰度分别为70.66%、10.02%、5.64%、3.90%、2.59%、1.11%。处理后酱香型白酒酒醅优势菌属为假丝酵母属、接合酵母属、红曲霉属、曲霉属和枝孢属(Cladosporium),相对丰度分别为67.88%、13.65%、6.68%、4.14%、1.53%,预处理前后假丝酵母属和结合酵母属均占主导地位。经过PBS预处理后,接合酵母属和红曲霉属相对丰度升高,分别提高了3.63%和2.78%,假丝酵母属、裂殖酵母属、曲霉属相对丰度相对降低,分别降低了2.78%、2%、1.5%。
表2 基于属水平酒醅样品中真核微生物相对丰度及变化
Table 2 Relative abundance and variation of eukaryotic microorganisms in fermented grain samples based on genus level
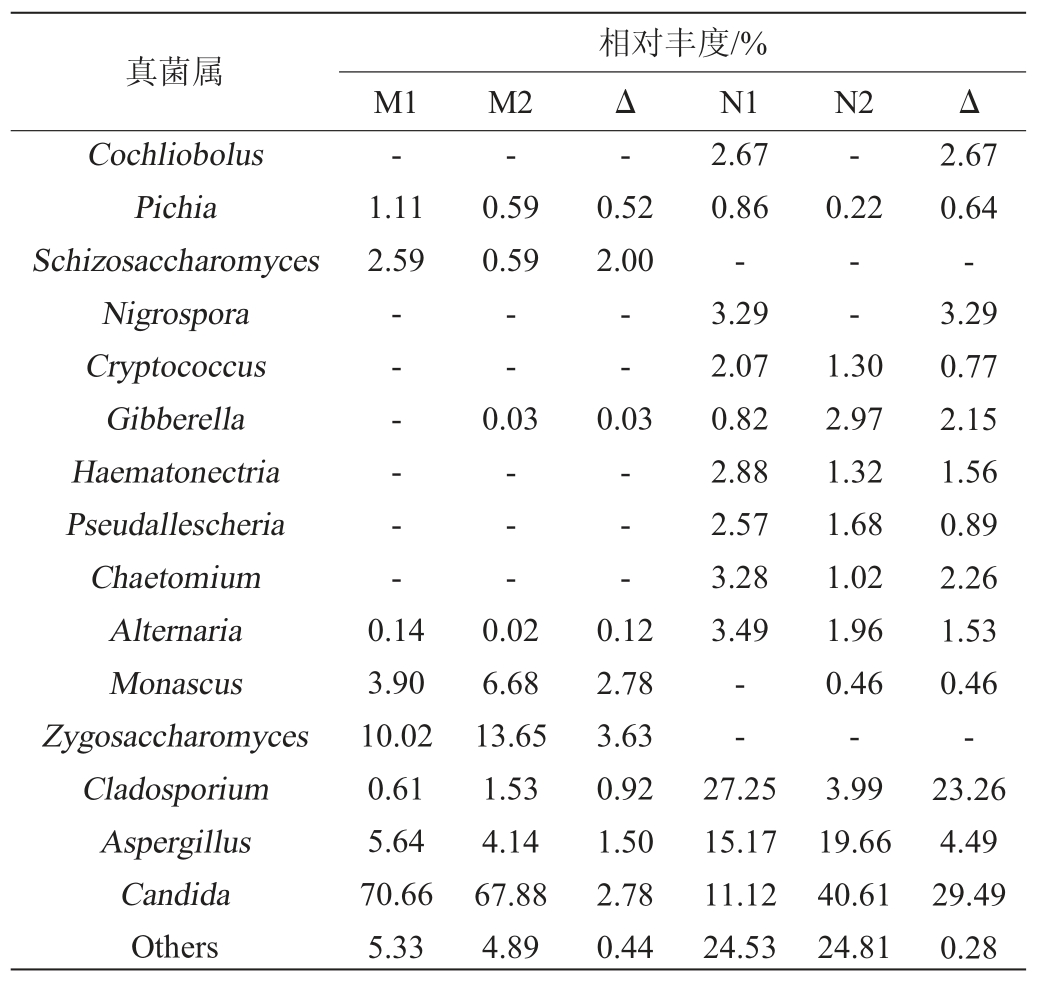
浓香型白酒酒醅PBS预处理前的优势菌属为枝孢属(Cladosporium)、曲霉属(Aspergillus)、假丝酵母属(Candida)、交链孢属(Alternaria)、黑孢属(Nigrospora)、毛壳菌属(Chaetomium)、血赤壳属(Haematonectria)、旋孢腔菌属(Cochliobolus)、假霉样真菌属(Pseudallescheria)和隐球菌属(Cryptococcus),相对丰度分别为27.25%、15.17%、11.12%、3.49%、3.29%、3.28%、2.88%、2.67%、2.57%、2.07%,其中,枝孢属、曲霉属和假丝酵母属占主导地位;预处理后的优势菌属为假丝酵母属、曲霉属、枝孢属、赤霉菌属(Gibberella)、交链孢属、假霉样真菌属、血赤壳属、隐球菌属和毛壳菌属,相对丰度分别为40.61%、19.66%、3.99%、2.97%、1.96%、1.68%、1.32%、1.3%和1.02%,假丝酵母属和曲霉属占主导地位。其中假丝酵母属、曲霉属和赤霉菌属处理后相对丰度增加,分别增加了29.49%、4.49%、2.15%,枝孢属、毛壳菌属、血赤壳属、交链孢属在处理后相对丰度降低,分别降低了23.26%、2.26%、1.56%、1.53%。
总体来看,预处理前后酱香型白酒酒醅属水平上组成和比例上差异不明显,而预处理前后浓香型白酒酒醅属水平上物种组成差异不大,但枝孢属和假丝酵母属这两个属的相对丰度发生较大改变。
2.2.3 门水平上原核微生物物种分析
由表3可知,PBS预处理前酱香型白酒酒醅中优势细菌菌门为厚壁菌门(Firmicutes)、拟杆菌门(Bacteroides),相对丰度分别为97.75%、1.87%,其中厚壁菌门占主导地位。PBS预处理后优势细菌菌门为厚壁菌门和变形菌门(Proteobacteria),相对丰度分别为72.19%和27.64%。经处理后变形菌门相对丰度增长了27.36%,而厚壁菌门和拟杆菌门相对丰度明显降低,分别下降了25.56%和1.87%。
表3 基于门水平酒醅样品中原核微生物相对丰度及变化
Table 3 Relative abundance and variation of prokaryotic microorganisms in fermented grain samples based on phylum level
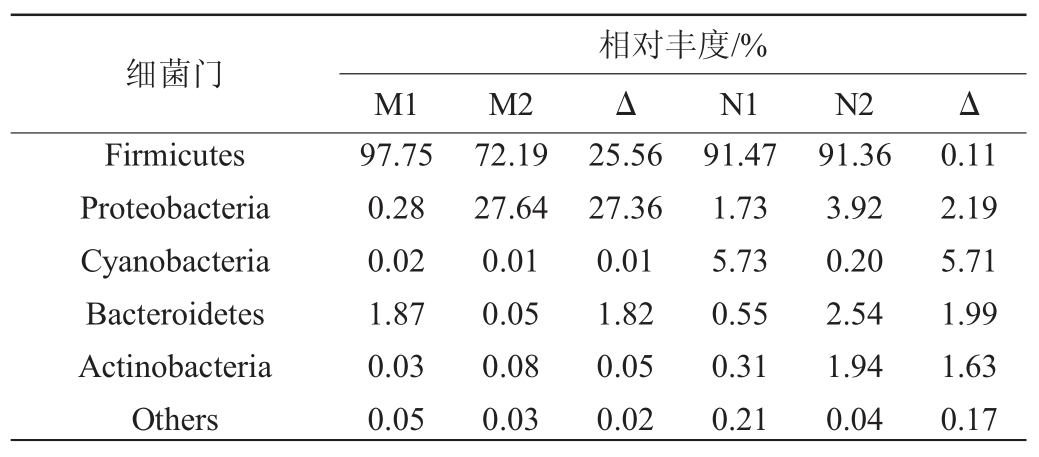
浓香型白酒酒醅所检出的细菌群落主要归属于5个门,分别为厚壁菌门、变形菌门、拟杆菌门、放线菌门(Actinobacteria)和蓝细菌门(Cyanobacteria),这与李泽等[30]运用扩增子测序分析浓香型不同层次酒醅优势细菌门结果一致。PBS预处理前优势菌门为厚壁菌门、蓝细菌门、变形菌门,相对丰度分别为91.47%、5.73%、1.73%,其中厚壁菌门占主导地位。PBS预处理后优势菌门为厚壁菌门、变形菌门、拟杆菌门及放线菌门,相对丰度分别为91.36%、3.92%、2.54%、1.94%,这4个门与肖辰等[31]分析泸型酒酒醅优势细菌门结果一致。处理后的浓香型白酒酒醅中变形菌门、拟杆菌门和放线菌门丰度相对增加,分别增长2.19%、2.54%、1.94%,厚壁菌门相对丰度变化不大,而蓝细菌门丰度相对减少,减少了5.73%。总的来说,酱香型白酒酒醅预处理前后原核微生物门水平上组成无变化但比例有差异,而浓香型酒醅预处理前后组成和丰度上均无较大变化。厚壁菌门和变形菌门预处理前后物种丰度均发生较大改变,后续若有研究对这两个门运用扩增子测序技术检测其丰度时,应采用其他方法来对样品进行合适的富集。
2.2.4 属水平上原核微生物物种分析
由表4可知,PBS预处理前酱香型白酒酒醅优势细菌属为乳杆菌属(Lactobacillus)、高温放线菌属(Thermoactinomyces)、拟杆菌属(Bacteroides),相对丰度分别为86.98%、3.98%、1.60%,乳杆菌属主导地位;PBS预处理后优势细菌菌属为乳杆菌属、寡养单胞菌属(Stenotrophomonas)、丛毛单胞菌属(Comamonas)、假苍白杆菌属(Pseudochrobactrum)和高温放线菌属,相对丰度分别为70.17%、21.63%、3.03%、1.96%、1.02%,乳杆菌属和寡养单胞菌属占主导地位。经过PBS预处理后,寡养单胞菌属、丛毛单胞菌属和假苍白杆菌属相对丰度含量大幅提升,分别增长了21.63%、3.03%、1.96%,乳杆菌属和高温放线菌属相对丰度均降低,分别降低了16.81%和2.96%。
表4 基于属水平酒醅样品中原核微生物相对丰度及变化
Table 4 Relative abundance and variation of eukaryotic microorganisms in fermented grain samples based on genus level
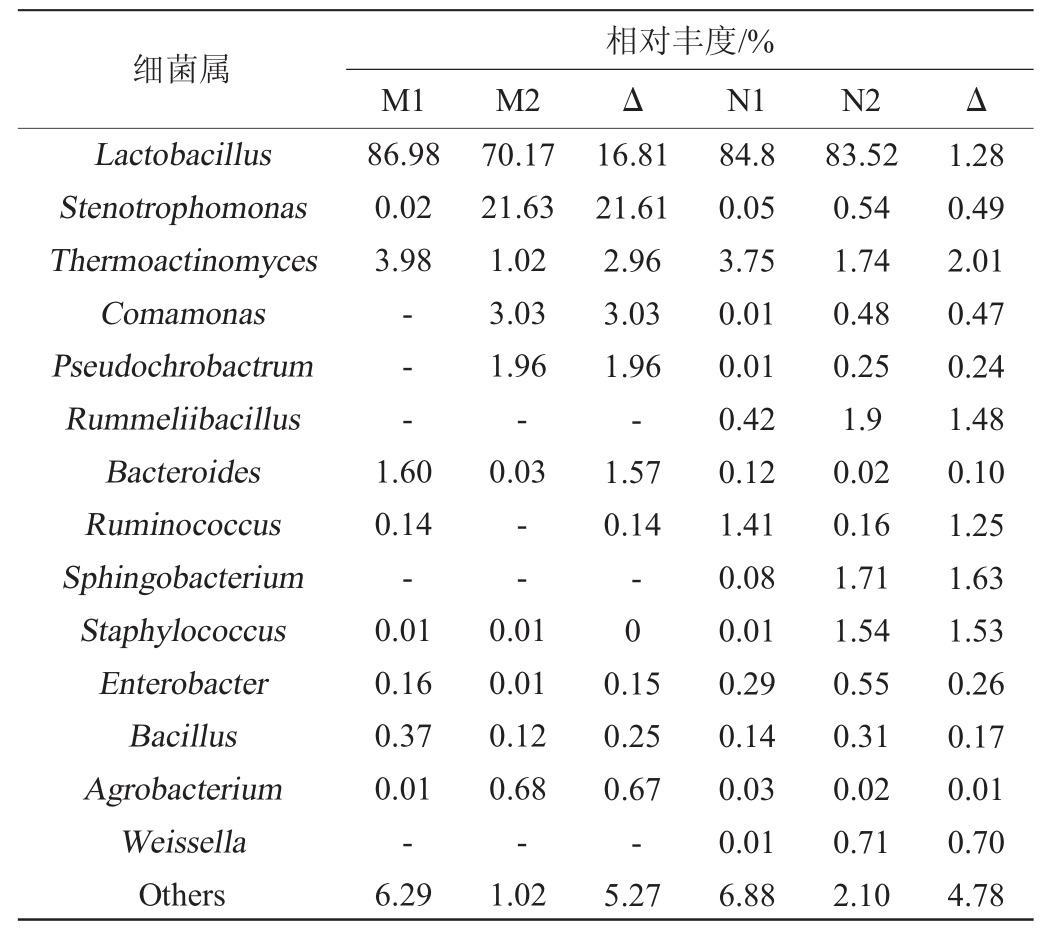
PBS预处理前浓香型白酒酒醅优势菌属为乳杆菌属、高温放线菌属、瘤胃球菌属(Ruminococcus),相对丰度分别为84.8%、3.75%、1.41%,乳杆菌属占主导地位;而PBS预处理后优势细菌属为乳杆菌属、拉梅尔芽孢杆菌属(Rummeliibacillus)、高温放线菌属、鞘氨醇杆菌属(Sphingobacterium)和葡萄球菌属(Staphylococcus),相对丰度分别为83.52%、1.90%、1.74%、1.71%、1.54%,乳杆菌属依旧占主导地位。经处理后拉梅尔芽孢杆菌属、鞘氨醇杆菌属和葡萄球菌属相对丰度明显增加,分别增加了1.9%、1.74%和1.71%,乳杆菌属和放线菌属相对丰度降低,分别降低了1.28%和3.75%。瘤胃球菌属在预处理后的两种酒醅中相对丰度均降低,分别降低了1.25%和0.14%。瘤胃菌属(Clostridia)是梭菌纲微生物,是一类专性厌氧且能产芽孢的细菌,而在预处理过程中接触大量氧气致氧中毒代谢受阻,核酸被裂解至PBS缓冲液中离心后被去除[32-33]。另有研究表明,pH值为8的磷酸盐缓冲液会对厌氧菌产甲烷过程产生抑制,在大量氧气和不适宜的pH环境中,厌氧菌属相对丰度对会大幅下降[34]。
总的来看,与真菌相比,PBS预处理对细菌的菌群结构影响较大,推测可能因真核生物本身构造比原核生物复杂,真核生物具有细胞壁的保护,而原核生物只有细胞膜[35],经振荡、高速离心等操作后进而裂解至PBS缓冲液中离心后被去。
2.3 样品聚类分析
主成分分析(principal components analysis,PCA)可直观展示样品和微生物之间的相关性。相关性强则在图中距离较近,表示此菌种在样品中相对丰度较大;相关性若弱则在图上距离较远,表示此菌种在样品中相对丰度占比较小。
由图3a可知,酱香型白酒酒醅在预处理前后聚在一起,浓香型白酒酒醅在预处理前后则分开,表明预处理对酱香型白酒酒醅真菌属菌群组成影响较小,而对浓香型白酒酒醅真菌属菌群结构影响较大。裂殖酵母属、接合酵母属和红曲霉属距离M1、M2较近,距离N1、N2较远,表明这3个菌属为酱香型酒醅代表真菌属;旋孢腔菌属、黑孢属、枝孢属、毛壳菌属、假霉样真菌属、隐球菌属、交链孢属和血赤壳属距离N1较近,距离M1、M2较远,这表明这8个菌属是浓香型白酒酒醅代表真菌属。
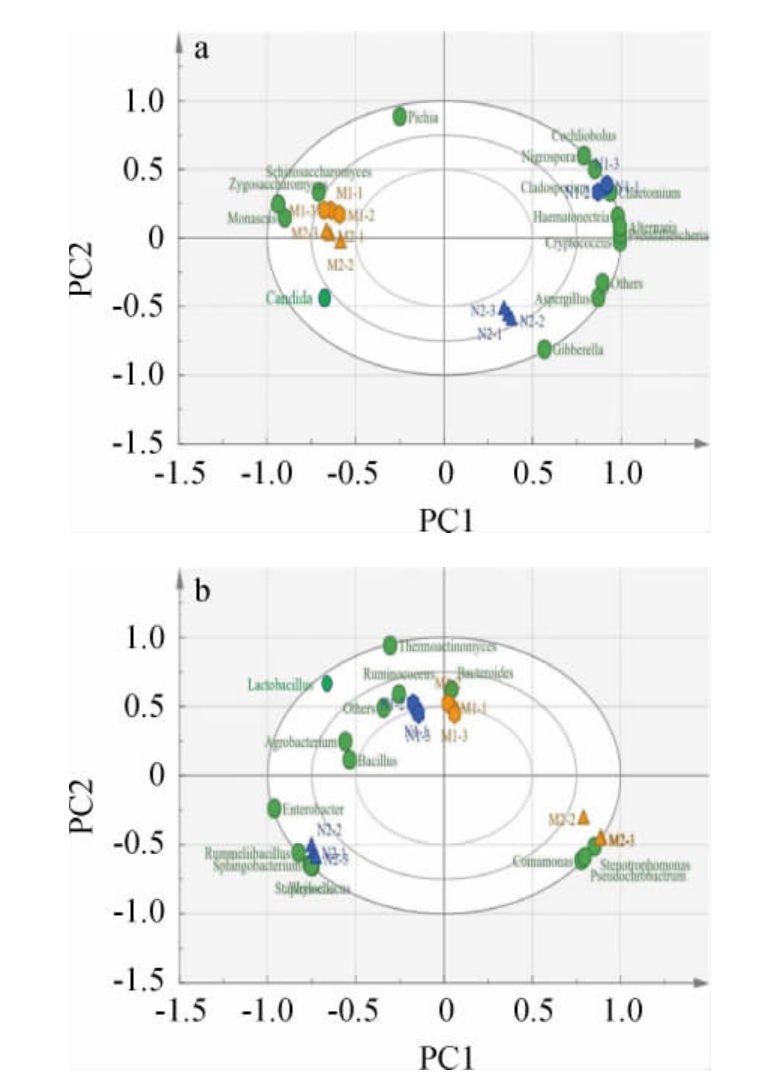
图3 两种酒醅真菌(a)和细菌(b)群落基于属水平主成分分析结果
Fig.3 Principal components analysis results of fungal (a) and bacterial (b) communities based on genus level
由图3b可知,酱香型和浓香型白酒酒醅的原核微生物在预处理前后均分开,表明预处理对酱香型白酒酒醅及浓香型白酒酒醅细菌属菌群结构均有较大影响。假苍白杆菌属、寡养单胞菌属和丛毛单胞菌属距离预处理后的酱香型白酒酒醅M2较近,离未处理的酱香型白酒酒醅M1较远,表明其是预处理后酱香型白酒酒醅代表菌群。拉梅尔芽孢杆菌属、葡萄球菌属、魏斯特菌属及鞘氨醇杆菌属距离预处理后的浓香型白酒酒醅N2较近,且这4个属在预处理后相对丰度均增加,表明其预处理后浓香型白酒酒醅的代表菌群。
3 结论
本研究采用扩增子测序技术对PBS预处理前后的酱香型第7轮次出窖酒醅及浓香型出窖酒醅中的菌群结构进行解析。结果表明,OTU分析表明预处理后两种香型白酒酒醅真核微生物的特有OTU数量均上升,而原核微生物的特有OTU数量均下降;阿尔法多样性分析表明,预处理后两种香型白酒酒醅真核物种丰度均增加,原核物种丰度均减少,且预处理前后两种酒醅真核和原核物种组成均无显著差异(P>0.05)。物种组成分析表明,预处理前后真核和原核微生物在门水平相对丰度有改变但组成差别不大,酱香型白酒酒醅原核微生物属水平上相对丰度及组成均发生较大改变,浓香型白酒酒醅则相反;两种香型白酒酒醅真核微生物属水平上相对丰度和组成均无较大改变。聚类分析表明,预处理前后酱香型白酒酒醅真菌属组成相似而细菌属组成具有差异;浓香型白酒酒醅真菌属和细菌属均具有差异。综上,预处理会对样品的群落结构造成影响,且主要影响样品微生物的丰度,对其组成影响不大。
[1]MARCO M,HEENEY D,BINDA S.Health benefits of fermented foods:microbiota and beyond[J].Curr Opin Biotech,2017,44:94-102.
[2]李景辉,郭莹,张颖,等.高产酯菌株的筛选及其在酱香型白酒堆积发酵中的应用[J].中国酿造,2021,40(10):168-173.
[3]卢君,山其木格,唐平,等.耐酸酵母菌株的筛选及其在酱香白酒酿造过程中的应用研究[J].酿酒科技,2019(10):106-111.
[4]赵微,赵璇,崔美林,等.清香型白酒酒醅中乳酸高产菌株的筛选及乳酸的分离纯化[J].中国酿造,2022,41(6):63-68.
[5]潘虎,卢向阳,董俊德,等.未培养微生物研究策略概述[J].生物学杂志,2012,29(1):79-83.
[6]卢萌萌,任聪,聂尧,等.白酒酿造窖泥未培养微生物菌群的可培养化策略[J].食品与发酵工业,2020,46(19):9-16.
[7]赵亮,王莉,汪地强,等.白酒微生物群落研究技术现状与二代测序数据分析方略[J].酿酒科技,2016(7):88-96.
[8]BOKULICH N A,LEWIS Z T,BOUNDY-MILLS K,et al.A new perspective on microbial landscapes within food production[J].Curr Opin Biotech,2016,37:182-189.
[9]WU L Y,WEN C Q,QIN Y J,et al.Phasing amplicon sequencing on Illumina Miseq for robust environmental microbial community analysis[J].BMC Microbiol,2015,15(1):125.
[10]麻颖垚.宏基因组学对酱香型白酒第四轮次窖内发酵微生物代谢通路的研究[D].贵阳:贵州大学,2020.
[11]古兴波,胡小霞,左乾程,等.宏基因组学在白酒酿造领域中的研究及应用[J].酿酒科技,2020(6):17-25,36.
[12]ZHOU W,LIAO Z M,WU Z Y,et al.Analysis of the difference between aged and degenerated pit mud microbiome in fermentation cellars for Chinese Luzhou-flavor Baijiu by metatranscriptomics[J].J Sci Food Agr,2021,101(11):4621-4631.
[13]田瑞杰,张勇,冯大鸿,等.基于宏转录组学的浓香型白酒酒醅活性微生物群落空间异质性研究[J].轻工学报,2022,37(1):1-11.
[14]吴成,王春晓,王晓丹,等.高通量测序技术在酿酒微生物多样性研究中的应用[J].食品科学,2019,40(3):348-355.
[15]高银涛,何璇,余博文,等.白酒固态双边发酵糖化机理及其对发酵过程的影响[J].食品与发酵工业,2021,47(13):92-97.
[16]沈才萍,李德林,沈才洪,等.酒醅微生物DNA提取预处理方法研究[J].四川理工学院学报(自然科学版),2013,26(3):16-20.
[17]闫亮珍,李晓然,全哲学,等.汾酒大曲和酒醅样品DNA提取方法的优化[J].食品与发酵工业,2011,37(3):32-36.
[18]郭小蛟,唐玉明,田新惠,等.基于两种预处理方法的白酒酒醅微生物宏蛋白质组学比较[J].食品科学,2022,43(20):216-221.
[19]赵鑫锐,张清玫,陈坚,等.从白酒大曲或酒醅中提取微生物总DNA的方法及用途:CN113215146A[P].2021-08-06.
[20]宋哲玮.酱香型白酒核心酿造菌群及群体代谢机制的研究[D].无锡:江南大学,2020.
[21]王欢,席德州,黄永光,等.酱香型白酒机械化酿造不同轮次堆积发酵细菌菌群结构多样性分析[J].食品科学,2020,41(2):188-195.
[22]郭敏,黄永光,邱树毅,等.高通量测序在酱香白酒微生态多样性研究中的应用[J].中国酿造,2017,36(5):146-151.
[23]DU H,WANG X S,ZHANG Y H,et al.Exploring the impacts of raw materials and environments on the microbiota in Chinese Daqu starter[J].Int J Food Microbiol,2019,297:32-40.
[24]李斌,陈亚楠,孙旭,等.Illumina MiSeq高通量测序分析红谷黄酒酒曲微生物菌群多样性[J].中国酿造,2022,41(9):86-92.
[25]SONG Z W,DU H,ZHANG Y,et al.Unraveling core functional microbiota in traditional solid-state fermentation by high-throughput amplicons and metatranscriptomics sequencing[J].Frontiers Microbiol,2017,8:1294.
[26]DEGRUNE F,DUMACK K,FIORE-DONNO A M,et al.Distinct communities of Cercozoa at different soil depths in a temperate agricultural field[J].FEMS Microbiol Ecol,2019,95(4):fiz041.
[27]STEINEGGER M,SÖDING J.Clustering huge protein sequence sets in linear time[J].Nat Commun,2018,9(1):2542.
[28]DESANTIS T Z,HUGENHOLTZ P,LARSEN N,et al.Greengenes,a chimera-checked 16S rRNA gene database and workbench compatible with ARB[J].Appl Environ Microbiol,2006,72(7):5069-5072.
[29]KÕLIALG U,LARSSON K H,ABARENKOV K,et al.UNITE:a database providing web-based methods for the molecular identification of ectomycorrhizal fungi[J].New Phytol,2005,166(3):1063.
[30]李泽,邱磊.浓香型白酒发酵过程中酒醅的原核生物群落演替过程[J].齐鲁工业大学学报,2021,35(4):26-31.
[31]肖辰,陆震鸣,张晓娟,等.泸型酒酒醅细菌群落的发酵演替规律[J].微生物学报,2019,59(1):195-204.
[32]胡晓龙,冯大鸿,张勇,等.浓香型酒醅中梭菌群落演替及其与理化性质的相关性分析[J].食品与发酵工业,2022,47(8):14-20.
[33]LU Z,IMIAY J A.When anaerobes encounter oxygen: mechanisms of oxygen toxicity,tolerance and defence[J].Nat Rev Microbiol,2021,19(12):774-785.
[34]张冬凌,黄晓婷,袁海平,等.磷酸盐缓冲液初始pH值对高含固污泥厌氧消化性能影响[J].环境工程学报,2017,11(11):6028-6033.
[35]汪慧慧,高俊杰,李庆腾,等.浓香型酒醅发酵过程中真核微生物群落的研究[J].食品科技,2022,47(8):14-20.