作为白酒酿造的主要发酵剂之一,麸曲以麸皮为主要原料,通过接种纯种根霉(Rhizopus)菌种培养而成,在酿酒过程中主要起糖化作用[1]。目前对麸曲的研究大多集中在麸曲工艺优化、理化特性及发酵性能等方面,如郑自强等[2]以产纤维素酶的菌种枯草芽孢杆菌(Bacillus subtilis)制作麸曲,通过响应面法优化制曲工艺,并对所制麸曲的产酶特性进行了分析,实现了成品曲生物量的扩培。陈雪玲等[3]对黄水生香麸曲的制备工艺进行了优化,得到生香麸曲最佳工艺为:麸皮细粉36%,初始水分含量50%,孢子悬液接种量4%,32 ℃培养84 h后每克曲添加0.6 mL酵母菌液,最后35 ℃烘干4 h。亦有研究指出,在白酒的酿造过程中,麸曲为酒体提供了大量的功能微生物,且其所含风味物质与前体物质也可直接或间接进入酒体中,赋予了白酒独特风味[4-5]。
本研究通过常规理化分析和电子鼻技术相结合的手段,对麸曲理化指标、发酵特性和香气物质进行了评价,采用MiSeq高通量测序技术解析麸曲群落结构,并采用纯培养技术对麸曲中乳酸菌和芽孢杆菌进行了分离鉴定,旨在为后续酿酒微生物资源的挖掘提供理论参考和菌株支持。
1 材料与方法
1.1 材料与试剂
1.1.1 材料麸曲:从山东省济宁市某制曲厂采集同一批次的3个麸曲样品,分别命名为FQSX1、FQSX2和FQSX3。
1.1.2 试剂
Ex Taq酶、脱氧核糖核酸(deoxyribonucleic acid,DNA)聚合酶(5 U/μL)、10×聚合酶链式反应(polymerase chain reaction,PCR)缓冲液、脱氧核糖核苷三磷酸(deoxy-ribonucleoside triphosphate,dNTPs)混合液:北京全式金生物技术有限公司;DNeasymericon FoodKitDNA基因组提取试剂盒:德国QIAGEN公司;Axygen清洁试剂盒:康宁生命科学吴江有限公司;引物338F/806R、M13F/M13R、ITS3F/ITS4R和27F/1495R:天一辉远(武汉)生物科技有限公司。
1.1.3 培养基
MRS、LB、营养琼脂(nutrient agar,NA)培养基:青岛海博生物技术有限公司。
1.2 仪器与设备
DYY-12电泳仪:北京六一仪器厂;DG250厌氧工作站:英国Don Whitley公司;Veriti FAST梯度PCR仪:美国ABI公司;MiSeq PE300高通量测序平台:美国Illumina公司;R930机架式服务器:美国DELL公司;PEN3电子鼻:德国Airsense公司;K1100全自动凯氏定氮仪:济南海能仪器股份有限公司。
1.3 方法
1.3.1 麸曲样品的制备
麸曲制作是以麸皮、谷糠、酒糟和豆饼为原料,原料按比例混合,调整混合原料的水分含量大约在50%;将配制好的原料边投料边蒸煮,达到圆气后继续蒸煮40 min,蒸煮完成后冷却至40 ℃,按原料的0.25%~0.35%进行接种,接种后将曲料投入制曲箱中,控制温度在33 ℃左右,通过机械式通风使曲料中水分含量在25%以下,制得麸曲。
1.3.2 麸曲各理化指标的测定
麸曲蛋白质含量测定:凯氏定氮法[6];麸曲水分含量、水分活度、酸度、灰分、淀粉含量、酯化力、氨基酸态氮、发酵力、糖化力和酯化力测定:参考轻工行业标准QB/T 4257—2011《酿酒大曲通用分析方法》。
液化力单位定义:在35 ℃、pH 4.6条件下,1 g绝干曲1 h能液化淀粉的克数为一个单位(U),以g/(g·h)表示。
发酵力单位定义:在30 ℃条件下,72 h内0.5 g大曲利用可发酵糖类所产生的二氧化碳的克数为一个单位(U),以g/(0.5 g·72 h)表示。
酯化力单位定义:每50 g大曲在35 ℃,经过7 d催化己酸和乙醇合成乙酸乙酯的毫克数为一个单位(U),以mg/(50 g·7 d)表示。
糖化力单位定义:在35 ℃,pH 4.6条件下,1 g大曲1 h转化可溶性淀粉生成葡萄糖的毫克数为一个单位(U),以mg/(g·h)表示。
1.3.3 宏基因组DNA提取、PCR扩增及高通量测序
DNA提取:取麸曲样品2.0 g,按基因组提取试剂盒的方法提取麸曲微生物的宏基因组DNA。
细菌16S rRNA V3-V4区序列扩增:根据HOU Q C等[7]的PCR扩增体系和扩增条件进行序列扩增。
真菌ITS2区序列扩增:根据WANG Y R等[8]的PCR扩增体系和扩增条件进行真菌序列扩增。
MiSeq高通量测序:将扩增产物经1.0%琼脂糖凝胶电泳检测后送往上海美吉生物医药科技有限公司进行MiSeq高通量测序。
1.3.4 序列拼接、质控及生物信息学分析
下机数据参照CAI W C等[9]的方法进行质控后,基于QIIME(v1.90)平台使用PyNAST软件校准排齐序列[10],应用两步UCLUST算法进行序列划分并建立操作分类单元(operational taxonomic units,OTU)[11]。使用核糖体数据库项目(ribosomal database project,RDP)[12]和Greengenes[13]数据库进行序列比对确定细菌分类学地位,使用UNITE数据库进行序列比对确定真菌分类学地位[14]。
1.3.5 乳酸菌及芽孢杆菌分离鉴定
分离:使用倍比稀释涂布法对麸曲样品中疑似乳酸菌和芽孢杆菌的菌株进行初步分离,经3 次平板划线将菌株纯化后,使用30%甘油将其于-80 ℃冷冻保藏,其中疑似乳酸菌于37 ℃厌氧培养2 d[15],芽孢杆菌于30 ℃好氧培养1 d[16]。
鉴定:将疑似乳酸菌[15]和芽孢杆菌[15]的菌株进行DNA提取、PCR扩增和清洁、连接、转化和鉴定,挑选阳性克隆子送至上海桑尼生物科技有限公司进行测序。
1.3.6 基于电子鼻技术麸曲挥发性风味物质的测定
取10 mL麸曲样品于顶空瓶中,在50 ℃水浴锅中平衡30 min,室温静置10 min后使用PEN3电子鼻进行麸曲挥发性风味物质测定,测定方法及参数设置参考CAI W C等[17]的方法。
1.3.7 数据处理
使用Origin2018绘制雷达图;使用R(v4.1.0)绘制气泡图;使用Mega7.0构建系统发育树。
2 结果与分析
2.1 麸曲理化指标及发酵特性分析
对麸曲样品的理化指标和发酵特性分析结果见表1。
表1 麸曲样品的理化指标和发酵特性测定结果
Table 1 Determination results of physicochemical indexes and fermentation characteristics of Fuqu samples

由表1可知,麸曲样品FQSX1~FQSX3的水分含量分别为9.52%、8.70%和8.18%,变异系数为7.7%;酸度分别为0.6 mmol/10 g、0.6 mmol/10 g和0.5 mmol/10 g,变异系数为5.0%;水分活度分别为0.55、0.55和0.49,变异系数为6.4%;灰分含量分别为7.43g/100g、6.73g/100g和6.53g/100 g,变异系数为6.9%;氨基酸态氮含量分别为3.49 g/kg、2.76 g/kg和3.75 g/kg,变异系数为15.5%;淀粉含量分别为42.31%、39.15%和42.64%,变异系数为4.7%;蛋白质含量分别为18.05%、17.98%和18.32%,变异系数为1.0%。结果表明,3个麸曲样品间氨基酸态氮含量的差异偏大。氨基酸态氮的含量与麸曲中微生物的种类和数量以及蛋白酶的活力有关[18],因此本研究中麸曲样品的氨基酸态氮含量的差异偏大,可能是因为麸曲样品的微生物多样性和蛋白酶的活力具有一定的差异而引起。
糖化力、酯化力和发酵力是评价酒曲品质的重要指标,其中糖化力的活力高低影响酿酒过程中淀粉的转化率,酯化力是酒曲发酵后催化生酯的能力,发酵力是将酒曲中的糖类发酵产生酒精和二氧化碳的能力[19]。由表1可知,麸曲FQSX1~FQSX3的糖化力分别为123 mg/(g·h)、66 mg/(g·h)和10 mg/(g·h),变异系数为85.2%;酯化力分别为294.79 mg/(50 g·7 d)、98.81 mg/(50 g·7 d)和101.61 mg/(50 g·7 d),变异系数为68.1%;发酵力分别为3.99 g/(0.5 g·72 h)、1.08 g/(0.5 g·72 h)和4.37 g/(0.5 g·72 h),变异系数为57.2%;液化力分别为4.78g/(g·h)、4.61 g/(g·h)和4.57 g/(g·h),变异系数为2.4%。结果表明,3个麸曲样品的糖化力、酯化力和发酵力有明显差异。成品曲在储藏过程中应保证曲库定期通风排湿,麸曲储藏的时间及环境均会对酒曲的品质造成一定的影响[20]。麸曲中的酵母可将淀粉转化为酒精,本研究的3个麸曲样品中麸曲FQSX3的发酵力最高,而麸曲FQSX2最低,由此发现,麸曲发酵力的差异可能是与其淀粉含量及微生物的生长代谢有关[21]。
2.2 麸曲香气物质分析
进一步采用电子鼻技术对麸曲样品的香气物质进行分析,结果见图1。
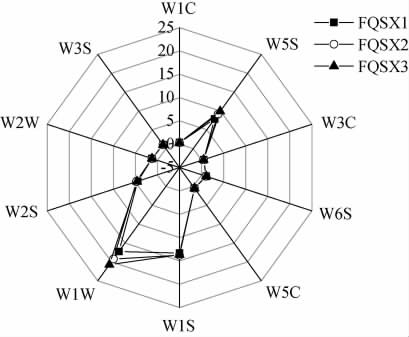
图1 麸曲样品的电子鼻各传感器响应值相对强度的雷达图
Fig.1 Radar plot of the relative intensity of response values of each sensor in electronic nose of Fuqu samples
W1C对芳香类物质灵敏,W5S对氢氧化物灵敏,W3C对氨气、芳香类物质灵敏,W6S对氢气有选择性,W5C对烷烃、芳香类物质灵敏,W1S对甲烷灵敏,W1W对有机硫化物、萜类物质灵敏,W2S对乙醇灵敏,W2W对有机硫化物灵敏,W3S对烷烃类物质灵敏。
由图1可知,传感器W1W和W5S对3 个麸曲样品的响应值存在明显差异。结果表明,3个麸曲样品在风味方面的差异主要体现在氢氧化物、有机硫化物和萜类物质上。本研究中有机硫化物是麸曲中含量较高的风味物质,SONG X等[22]研究发现,二甲基二硫醚可用于根据香气类型和酒曲来区分白酒。
2.3 麸曲微生物多样性分析
2.3.1 细菌群落多样性分析
经MiSeq高通量测序分析发现,3 个麸曲样品共产生155 065 条16S rRNA序列和145 125 条ITS序列,在97%相似性下16S rRNA和ITS序列分别划分为1 944 个和334 个OTU,其中FQSX1~FQSX3分别含有1 619、1 044和1 486 个细菌类群OTU和294、203和207个真菌类群OTU。经序列比对发现,3 个麸曲样品细菌类群分别隶属于5 个门、10 个纲、22 个目、44 个科和85 个属,其中相对含量>1.0%的门和属见图2。
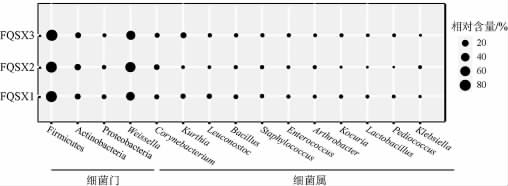
图2 基于门和属水平麸曲样品细菌群落多样性分析
Fig.2 Diversity analysis of bacterial community of Fuqu samples based on phylum and genus levels
由图2可知,3个相对含量>1.0%的细菌门分别为厚壁菌门(Firmicutes)(80.81%)、放线菌门(Actinobacteria)(14.15%)、变形菌门(Proteobacteria)(4.17%),12个相对含量>1.0%的细菌属分别为魏斯氏菌属(Weissella)(51.59%)、棒杆菌属(Corynebacterium)(8.28%)、库特菌属(Kurthia)(7.76%)、明串珠菌属(Leuconostoc)(4.75%)、芽孢杆菌属(Bacillus)(4.14%)、葡萄球菌属(Staphylococcus)(3.72%)、肠球菌属(Enterococcus)(2.71%)、节杆菌属(Arthrobacter)(2.50%)、考克氏菌属(Kocuria)(2.12%)、乳杆菌属(Lactobacillus)(1.34%)、片球菌属(Pediococcus)(1.34%)和克雷伯氏菌属(Klebsiella)(1.01%)。麸曲样品中均含有较为丰富 的Bacillus、Weissella、Leuconostoc、Enterococcus、Lactobacillus和Pediococcus,其中Weissella、Leuconostoc和Lactobacillus作为功能微生物在发酵过程中能够产生乳酸等有机酸类物质,具有减少酒体刺激性的作用[23]。GOU M等[24]研究发现,Bacillus、Lactobacillus和Pediococcus为麸曲中的主要细菌类群,这与本研究结果一致。作为优势细菌属,Bacillus是麸曲中重要的功能微生物,该菌株在白酒的酿造过程中产生的芳香类、吡嗪和酚类等物质能够增强挥发性化合物的丰度,有助于提升白酒的风味品质[25]。ZHANG W Q等[26]利用解淀粉芽孢杆菌(Bacillus amyloliquefaciens)对麸曲的生产进行了强化,结果发现该菌株可以产生四甲基吡嗪,而该物质是白酒中重要的功能物质和关键特征香气成分。
2.3.2 真菌群落多样性分析
麸曲样品真菌类群分别隶属于4 个门、7 个纲、8 个目、19 个科和25 个属,其中相对含量>1.0%的门和属见图3。
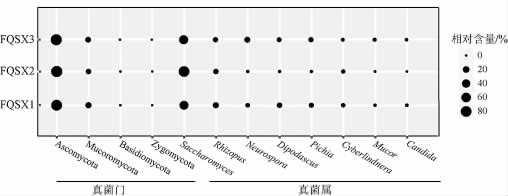
图3 基于门和属水平麸曲样品真菌群落多样性分析
Fig.3 Diversity analysis of fungal community of Fuqu samples based on phylum and genus levels
由图3可知,3 个麸曲样品中检测到的4 个真菌门为子囊菌门(Ascomycota)(86.83%)、毛霉门(Mucoromycota)(13.13%)、担子菌门(Basidiomycota)(0.04%)和接合菌门(Zygomycota)(0.002%);基于属水平分析发现,麸曲样品中相对含量>1.0%的真菌属有8个,分别为酵母菌属(Saccharomyces)(59.74%)、根霉菌属(Rhizopus)(11.46%)、脉孢菌属(Neurospora)(6.83%)、双足囊菌属(Dipodascus)(6.26%)、毕赤酵母属(Pichia)(5.92%)、塞伯林德纳氏酵母属(Cy-berlindnera)(3.77%)、毛霉菌属(Mucor)(1.68%)和假丝酵母属(Candida)(1.16%)。由此可知,Saccharomyces和Rhizopus为麸曲的主要真菌群落。酵母菌作为白酒酿造过程中产香气和产酒精的主要微生物,在麸曲样品的真菌群落中占绝对的优势[27];霉菌在酿酒过程中产生大量酯化酶、蛋白酶和糖化酶等水解酶,提高了麸曲的降解发酵[28]。GOU M等[24]研究发现,Saccharomyces和Rhizopus为麸曲中的主要真菌类群,这与本研究所得到的结果一致。麸曲主要是接入纯种霉菌制备而成,但MiSeq结果显示,麸曲中含有较高含量的Pichia、Candida以及Staphylococcus、Enterococcus和Klebsiella等,这些微生物类群可能来源于环境,因而在后续生产过程中,加强制曲及储存环境的管理是极为必要的。
2.4 麸曲中乳酸菌的分离鉴定
本研究对3个麸曲样品中的乳酸菌进行了分离鉴定并构建了系统发育树,结果见图4。
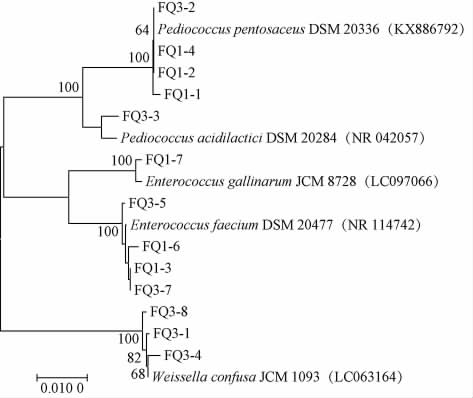
图4 基于16S rRNA基因序列乳酸菌分离菌株的系统发育树
Fig.4 Phylogenetic tree of isolated lactic acid bacteria strains based on 16S rRNA gene sequences
由图4可知,分离出的13株乳酸菌被鉴定为5个种,其中菌株FQ1-1、FQ1-2、FQ1-4和FQ3-2被鉴定为戊糖片球菌(Pediococcus pentosaceus),菌株FQ3-3被鉴定为酸性乳酸片球菌(Pediococcus acidilactici),菌株FQ1-7被鉴定为鸡肠球菌(Enterococcus gallinarum),菌株FQ1-3、FQ1-6、FQ3-5和FQ3-7被鉴定为屎肠球菌(Enterococcus faecium),菌株FQ3-1、FQ3-4和FQ3-8被鉴定为融合魏斯氏菌(Weissella confuse)。由此可见,P.pentosaceus、E.faecium和W.confuse为本研究麸曲中的优势乳酸菌,其分离株分别占分离株总数的30.77%、30.77%和23.08%。
MiSeq分析结果表明,Lactobacillus在麸曲样品中的相对含量高达1.34%,但是本研究使用纯培养法并未从麸曲中分离得到乳酸杆菌,这可能与一些未培养微生物虽然可以通过分子生物学的方法检测到,但是并不能实现纯培养有关[29]。通过综合多重培养条件、基质辅助激光解吸电离飞行时间质谱(matrix-assisted laser desorption/ionization time of flight mass spectrometry,MALDI-TOF-MS)及16S rRNA基因测序技术,培养组学(culturomics)技术极大地优化了培养条件,使可培养微生物的种类和数量得以明显增加[30],有研究表明利用培养组学技术可使从肠道中分离出的微生物种类至少增加了一倍[31]。由此可见,在后续研究中将培养组学技术应用于麸曲微生物菌株的分离培养中,实现未培养微生物菌株的可培养具有极强的可行性。
2.5 麸曲中芽孢杆菌的分离鉴定
本研究对3个麸曲样品中的芽孢杆菌进行了分离鉴定并构建了系统发育树,结果见图5。
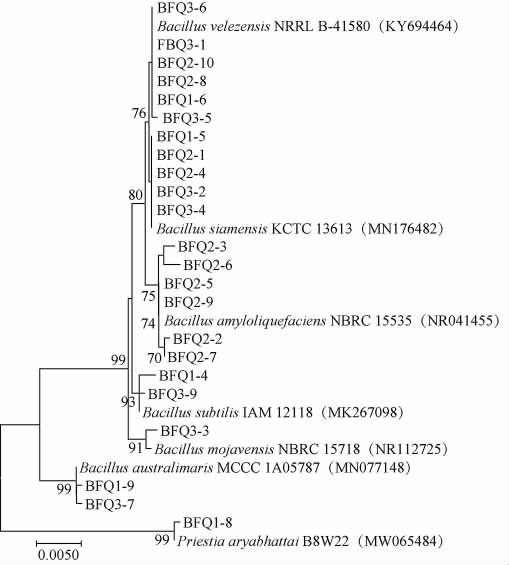
图5 基于ITS基因序列芽孢杆菌分离菌株的系统发育树
Fig.5 Phylogenetic tree of isolated Bacillus strains based on ITS gene sequences
由图5可知,分离出的23株芽孢杆菌被鉴定为7个种,其中菌株BFQ2-2、BFQ2-3、BFQ2-5、BFQ2-6、BFQ2-7和BFQ2-9被鉴定为解淀粉芽孢杆菌(Bacillus amyloliquefaciens),菌株BFQ1-8被鉴定为阿氏芽孢杆菌(Priestia aryabhattai),菌株BFQ1-9和BFQ3-7被鉴定为澳大利亚芽孢杆菌(Bacillus australimaris),菌株BFQ3-3被鉴定为漠海威芽孢杆菌(Bacillus mojavensis),菌株BFQ1-5、BFQ2-1、BFQ2-4、BFQ3-2和BFQ3-4被鉴定为暹罗芽孢杆菌(Bacillus siamensis),菌株BFQ1-4和BFQ3-9被鉴定为枯草芽孢杆菌(Bacillus subtilis),菌株BFQ1-6、BFQ2-8、BFQ2-10、BFQ3-1、BFQ3-5和BFQ3-6被鉴定为苏云金芽孢杆菌(Bacillus velezensis)。由此发现,B.amyloliquefaciens、B.velezensis和B.siamensis为麸曲中的优势芽孢杆菌,其分离株分别占分离株总数的26.07%、26.07%和21.74%。
3 结论
本研究对同一批次生产的3 个麸曲样品理化指标、发酵特性、挥发性风味物质和微生物类群进行了解析,对其中蕴含的乳酸菌和芽孢杆菌进行了分离鉴定。结果发现,麸曲样品的氨基酸态氮含量、糖化力、酯化力和发酵力存在一定的差异,香气物质的差异主要体现在氢氧化物、有机硫化物和萜类上,Weissella为其优势细菌属,Saccharomyces和Rhizopus为其优势真菌属,乳酸菌类群主要为P.pentosaceus、E.faecium和W.confuse,芽孢杆菌类群主要为B.amyloliquefaciens、B.velezensis和B.siamensis。
[1]罗惠波,谢军,黄治国,等.纯种根霉麸曲制曲工艺优化研究[J].四川理工学院学报(自然科学版),2015,28(5):7-11.
[2]郑自强,卫春会,邓杰,等.一株产纤维素酶枯草芽孢杆菌的麸曲制作及其产酶特性研究[J].食品与机械,2021,37(10):12-17.
[3]陈雪玲,兰小艳,廖诚.黄水生香麸曲的制备工艺优化[J].安徽农学通报,2021,27(12):108-110.
[4]LIU H L,SUN B G.Effect of fermentation processing on the flavor of Baijiu[J].J Agr Food Chem,2018,66(22):5425-5432.
[5]DONG W W,YANG Q,LIAO Y X,et al.Characterisation and comparison of the microflora of traditional and pure culture xiaoqu during the Baijiu liquor brewing process[J].J I Brewing,2020,126(2):213-220.
[6]HOU Q C,WANG Y R,CAI W C,et al.Metagenomic and physicochemical analyses reveal microbial community and functional differences between three types of low-temperature Daqu[J].Food Res Int,2022,156:111167.
[7]HOU Q C,WANG Y R,NI H,et al.Deep sequencing reveals changes in prokaryotic taxonomy and functional diversity of pit muds in different distilleries of China[J].Ann Microbiol,2022,72(1):1-14.
[8]WANG Y R,XIANG F S,ZHANG Z D,et al.High-throughput sequencingbased analysis of fungal diversity and taste quality evaluation of Douchi,a traditional fermented food[J].Food Sci Nutr,2020,8(12):6612-6620.
[9]CAI W C,WANG Y R,LIU Z J,et al.Depth-depended quality comparison of light-flavor fermented grains from two fermentation rounds[J].Food Res Int,2022,159:111587.
[10]CAPORASO J G,KUCZYNSKI J,STOMBAUGH J,et al.QIIME allows analysis of high-throughput community sequencing data[J].Nat Methods,2010,7(5):335-336.
[11]EDGAR R C.Search and clustering orders of magnitude faster than BLAST[J].Bioinformatics,2010,26(19):2460-2461.
[12]COLE J R,CHAI B,FARRIS R J,et al.The ribosomal database project(RDP-II): introducing myRDP space and quality controlled public data[J].Nucleic Acids Res,2007,35(1):169-172.
[13]DESANTIS T Z,HUGENHOLTZ P,LARSEN N,et al.Greengenes,a chimera-checked 16S rRNA gene database and workbench compatible with ARB[J].Appl Environ Microbiol,2006,72(7):5069-5072.
[14]NILSSON R H,LARSSON K H,TAYLOR A F S,et al.The UNITE database for molecular identification of fungi: handling dark taxa and parallel taxonomic classifications[J].Nucleic Acids Res,2019,47(D1):D259-D264.
[15]TALIB N,MOHAMAD N E,YEAP S K,et al.Isolation and characterization of Lactobacillus spp.from kefir samples in Malaysia[J].Molecules,2019,24(14):2606.
[16]DUAN Y N,CHEN R,ZHANG R,et al.Isolation,identification,and antibacterial mechanisms of Bacillus amyloliquefaciens QSB-6 and its effect on plant roots[J].Front Microbiol,2021,12(9):746799.
[17]CAI W C,TANG F X,GUO Z,et al.Effects of pretreatment methods and leaching methods on jujube wine quality detected by electronic senses and HS-SPME-GC-MS[J].Food Chem,2020,330:127330.
[18]崔东琦,罗小叶,班世栋,等.强化曲的制备及其在酱香型白酒中的应用[J].食品与发酵工业,2022,48(7):78-84.
[19]FU G M,DENG M F,CHEN Y,et al.Analysis of microbial community,physiochemical indices,and volatile compounds of Chinese te-flavor Baijiu Daqu produced in different seasons[J].J Sci Food Agr,2021,101(15):6525-6532.
[20]关玉权,程伟,张杰,等.金种子浓香型强化大曲制备工艺分析与探讨[J].酿酒科技,2018(2):49-54,61.
[21]李媛媛,罗碧霞,张兰兰,等.不同粮食曲的理化特性及其微生物群落结构[J].食品与发酵工业,2020,46(10):74-79.
[22]SONG X,WANG G,ZHU L,et al.Comparison of two cooked vegetable aroma compounds,dimethyl disulfide and methional,in Chinese Baijiuby a sensory-guided approach and chemometrics[J].LWT-Food Sci Technol,2021,146:111427.
[23]刘延波,王琳琳,金尚萍,等.不同储藏期浓香型白酒大曲的微生物多样性分析[J].中国酿造,2022,41(4):105-110.
[24]GOU M,WANG H Z,YUAN H W,et al.Characterization of the microbial community in three types of fermentation starters used for Chinese liquor production[J].J I Brewing,2015,121(4):620-627.
[25]ZHANG R,WU Q,XU Y.Aroma characteristics of Moutai-flavour liquor produced with Bacillus licheniformis by solid-state fermentation[J].Lett Appl Microbiol,2013,57(1):11-18.
[26]ZHANG W Q,SI G R,RAO Z M,et al.High yield of tetramethylpyrazine in functional Fuqu using Bacillus amyloliquefaciens[J].Food Biosci,2019,31(10):100435.
[27]ZHANG W,LI J L,RAO Z M,et al.Sesame flavour Baijiu:A review[J].J I Brewing,2020,126(3):224-232.
[28]SAKANDAR H A,HUSSAIN R,KHAN Q F,et al.Functional microbiota in Chinese traditional Baijiu and Mijiu Qu(starters):A review[J].Food Res Int,2020,138:109830.
[29]VARDI R,MITTERMEIER J C,ROLL U.Combining culturomic sources to uncover trends in popularity and seasonal interest in plants[J].Conserv Biol,2021,35(2):460-471.
[30]LAGIER J C,DUBOURG G,MILLION M,et al.Culturing the human microbiota and culturomics[J].Nat Rev Microbiol,2018,16(9): 540-550.
[31]LAGIER J C,KHELAIFIA S,ALOU M T,et al.Culture of previously uncultured members of the human gut microbiota by culturomics[J].Nature Microbiol,2016,1(12):1-8.