蒙古族奶酪因独特的发酵方式与风味,深受人民喜爱[1],与西方奶酪相比口感更加细腻柔和,更能满足国内消费者的需求[2]。在不添加发酵剂依靠自然环境成熟的情况下,奶酪中的微生物对特有风味的形成起着关键作用[3],目前,国外对西方奶酪成熟过程中的微生物多样性已经做了大量深入细致的研究[4-7],而国内对于少数民族传统奶酪的研究却相对较少。内蒙古地区不同地域、不同微生物群落结构使得风味存在较大波动,导致传统发酵奶酪的品质不稳定,质量安全无法得到控制,同时也极大影响了传统发酵乳制品的批量化生产。因此,深入研究奶酪样品中微生物多样性,探究不同成熟阶段样品中微生物群落差异,对解析奶酪风味质构形成具有重要的参考价值[8]。
目前,奶酪中微生物群落多样性研究主要依靠培养法和免培养法,然而传统培养法受技术局限,无法对奶酪中微生物群落进行深入研究[9]。本研究采用高通量测序技术对蒙古族奶酪自然成熟过程中的微生物群落多样性进行解析,了解天然奶酪在不同成熟过程中的微生物多样性、探讨微生物的群落构成和特征差异,为进一步解析微生物与奶酪风味质构之间的关系提供基础数据,为发掘蒙古族传统奶酪中微生物菌种资源奠定基础。
1 材料与方法
1.1 材料与试剂
1.1.1 材料
从内蒙古锡林郭勒盟西乌珠穆沁旗牧民家,分别采集样品:发酵凝乳、成熟0 d、10 d、20 d和30 d的奶酪各3份,分别标记为Yni、Y0d~Y30d,样品采集方式为随机采样,样品采集后,立即送往实验室-20 ℃保存。
1.1.2 试剂
Fast脱氧核糖核酸(deoxyribonucleic acid,DNA) Spin Kit for Soil:美国MP Biomedicals公司;琼脂糖(生化试剂):西班牙Biowest公司;AxyPrep DNA Gel Extraction Kit:美国Axygen公司;NEXTFLEX
Spin Kit for Soil:美国MP Biomedicals公司;琼脂糖(生化试剂):西班牙Biowest公司;AxyPrep DNA Gel Extraction Kit:美国Axygen公司;NEXTFLEX Rapid DNA-Seq Kit:美国Bioo Scientific公司;MiSeq Reagent Kit v3/NovaSeq Reagent Kits:美国Illumina公司。其他试剂均为国产分析纯。
Rapid DNA-Seq Kit:美国Bioo Scientific公司;MiSeq Reagent Kit v3/NovaSeq Reagent Kits:美国Illumina公司。其他试剂均为国产分析纯。
1.2 仪器与设备
mySPIN 6小型离心机、SPIN 12小型离心机:美国赛默飞科技公司;M1324R高速台式冷冻离心机:深圳瑞沃德公司;UV5Nano超微量分光光度计:瑞士METTLER TOLEDO公司;HBS-ScanY酶标仪:南京德铁生物科技有限公司;Fluo-380微型荧光计:北京原平皓生物技术有限公司;DYY-2C电泳仪:南京普阳科学仪器研究所;KS96G型聚合酶链式反应(polymerase chain reaction,PCR)仪:冠森生物科技(上海)有限公司;Miseq DX基因测序仪:美国Illumina公司。
1.3 方法
1.3.1 DNA提取及PCR扩增
根据FastDNA Spin Kit for Soil说明书对微生物群落总DNA进行抽提,使用NanoDrop2000测定DNA浓度和纯度,使用1%的琼脂糖凝胶电泳检测DNA的完整性[10];细菌使用338F(5'-ACTCCTACGGGAGGCAGCAG-3)'和806R(5'-GGACTACHVGGGTWTCTAAT-3)'对16S rRNA基因V3-V4可变区进行PCR扩增,真菌使用ITS1F(5'-CTTGGTCATTTAGAGGAAGTAA-3')和ITS2R(5'-GCTGCGTTCTTCATCGATGC-3')对ITS基因进行PCR扩增。PCR扩增程序:95 ℃预变性3 min;95 ℃变性30 s,55 ℃退火30 s,72 ℃延伸30 s,共27个循环;然后72 ℃稳定延伸10 min[11]。
Spin Kit for Soil说明书对微生物群落总DNA进行抽提,使用NanoDrop2000测定DNA浓度和纯度,使用1%的琼脂糖凝胶电泳检测DNA的完整性[10];细菌使用338F(5'-ACTCCTACGGGAGGCAGCAG-3)'和806R(5'-GGACTACHVGGGTWTCTAAT-3)'对16S rRNA基因V3-V4可变区进行PCR扩增,真菌使用ITS1F(5'-CTTGGTCATTTAGAGGAAGTAA-3')和ITS2R(5'-GCTGCGTTCTTCATCGATGC-3')对ITS基因进行PCR扩增。PCR扩增程序:95 ℃预变性3 min;95 ℃变性30 s,55 ℃退火30 s,72 ℃延伸30 s,共27个循环;然后72 ℃稳定延伸10 min[11]。
1.3.2 Illumina Miseq高通量测序
将同一样本的PCR扩增产物混合后使用2%琼脂糖凝胶电泳检测产物,利用AxyPrep DNA Gel Extraction Kit进行产物纯化,并按照测序量的要求对产物进行相应比例的混合。使用NEXTFLEX RapidDNA-SeqKit进行建库:(1)接头连接;(2)使用磁珠筛选去除接头自连片段;(3)利用PCR扩增进行文库模板的富集;(4)磁珠回收PCR产物得到最终的文库。利用Illumina公司的Miseq PE300平台测序[12]。
RapidDNA-SeqKit进行建库:(1)接头连接;(2)使用磁珠筛选去除接头自连片段;(3)利用PCR扩增进行文库模板的富集;(4)磁珠回收PCR产物得到最终的文库。利用Illumina公司的Miseq PE300平台测序[12]。
1.3.3 数据处理
使用fastp[13]软件对原始测序序列进行质控,使用FLASH[14]软件进行拼接。使用UPARSE软件,根据97%的相似度对序列进行操作分类单元(operationaltaxonomicunits,OTU)聚类并剔除嵌合体[15-16]。利用RDP classifier对每条序列进行物种分类注释[17],比对Silva 16S rRNA数据库(version 138),设置比对阈值为70%。利用Mothur进行多样性分析,利用R语言(version 3.3.1)进行主坐标分析(principal coordinate analysis,PCoA)和作图。
2 结果与分析
2.1 发酵凝乳及奶酪成熟过程中的微生物多样性
Coverage值均>0.99,表明细菌和真菌测序的序列覆盖率对后续的微生物分析是可靠的。Alpha多样性包括4个指标,其中Chao1和ACE指数衡量物种丰富度[18],Shannon指数和Simpson指数作为多样性参数[19]。
由表1可知,奶酪成熟10 d时,细菌群落丰度和多样性指数最高,细菌数量和种类最多,说明此时奶酪的内部微生物相互作用的环境与外部自然环境均有利于微生物的生长;成熟20 d时,丰度和多样性下降,可能是乳酸的逐渐积累,pH值的下降,部分不耐酸细菌死亡导致;成熟30 d时,丰度下降但多样性上升。由表2可知,发酵凝乳与奶酪成熟0 d时,真菌群落丰度和多样性指数较低;成熟10 d时,真菌群落丰度和多样性指数最高;成熟20 d时,真菌群落丰度和多样性指数下降;成熟30 d时,丰度和多样性指数均升高。细菌和真菌的发酵特征表明,奶酪成熟10 d时微生物多样性达到最大,10 d后随着发酵时间延长,细菌和真菌多样性均呈现先减小后增大趋势。
表1 细菌群落多样性分析结果
Table 1 Diversity analysis results of bacterial community

表2 真菌群落多样性分析结果
Table 2 Diversity analysis results of fungal community
2.2 发酵凝乳及奶酪成熟过程中微生物群落组成的差异
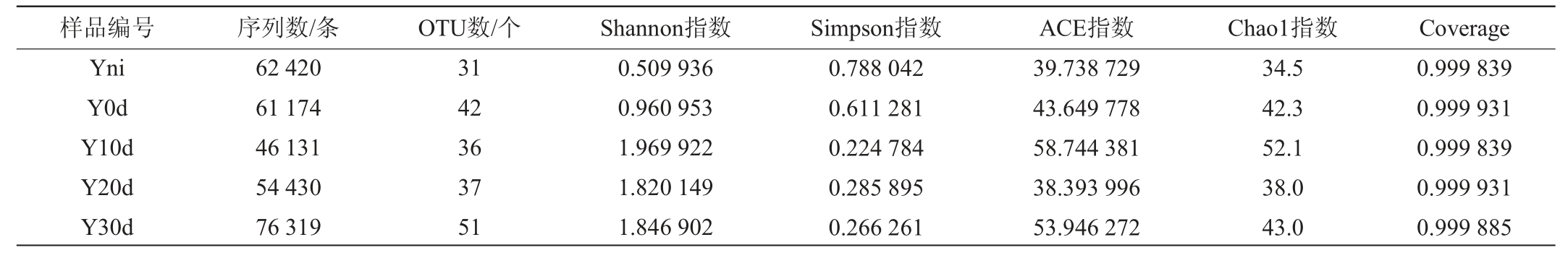
韦恩图能更好地观察奶酪成熟过程中微生物群落的动态变化,细菌和真菌群落OTU韦恩图见图1。由图1a可知,5种样品细菌菌群的OTU总数为307个,共有的OTU数为18个,占OTU总数的5.86%;各个样品特有的OTU数分别为2个、0个、7个、9个、13个。由图1b可知,真菌菌群的OTU总数为203个,共有的OTU数为14个,占OTU总数的6.90%;各个样品特有的OTU数分别为2个、8个、4个、2个、19个,说明成熟30 d时奶酪特有细菌和真菌OTU数均最多。综上可知,奶酪成熟30 d时产生较多特有细菌与真菌,与其他发酵阶段具有较大差别。
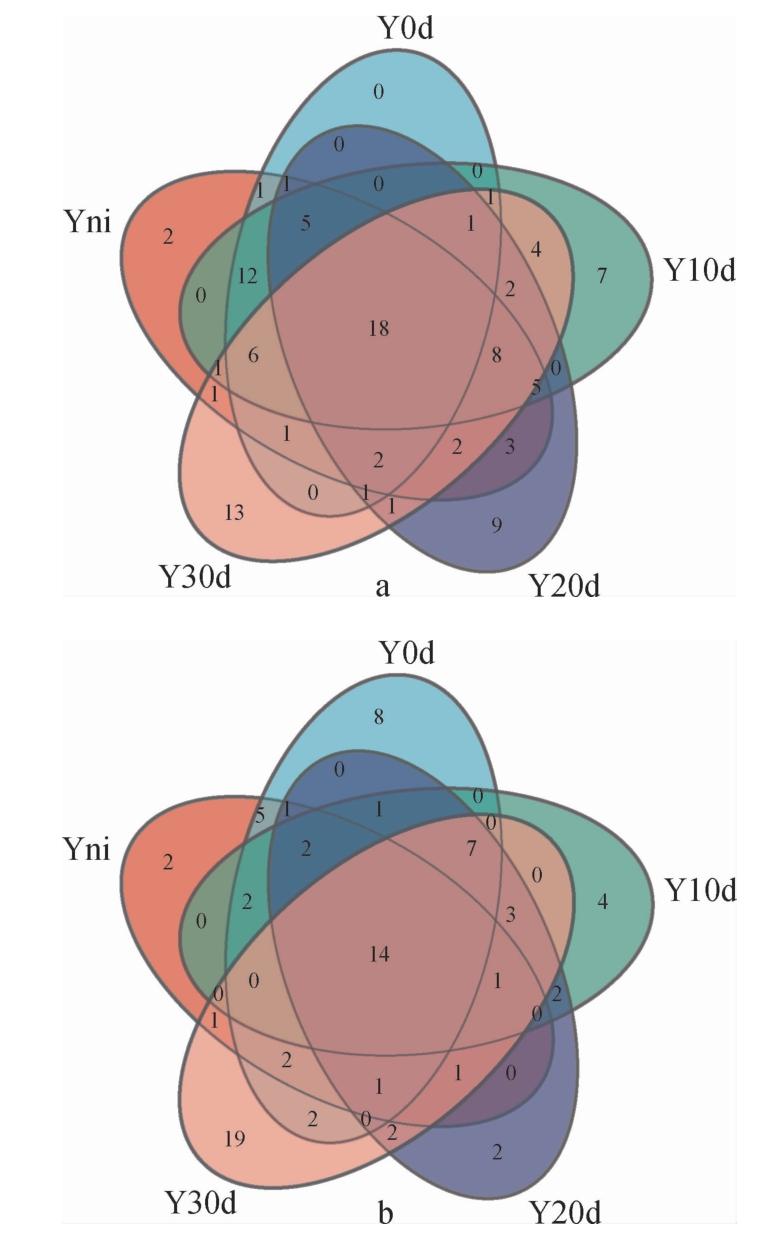
图1 细菌(a)和真菌(b)群落OTU韦恩图
Fig.1 OTU Venn diagram of bacterial (a) and fungal (b) communities
为了进一步分析样品群落结构的差异性,根据OTUs的数量对不同样品中细菌和真菌群落进行PCoA[20],结果见图2。由图2可知,细菌菌群PC1和PC2的方差贡献率分别为72.14%和15.85%,累计方差贡献率为87.99%;真菌菌群的PC1和PC2的方差贡献率分别为67.97%和31.01%,累计方差贡献率为98.98%,说明细菌菌群和真菌菌群的两个主成分均能解释原变量的绝大部分信息。发酵凝乳及成熟0 d和30 d奶酪样品在图中的分布较为集中,说明这3种样品中的细菌和真菌群落结构较为相似,成熟10 d和20 d的奶酪样品在图中的分布较为分散,说明这2种样品的细菌和真菌群落结构差异较大。
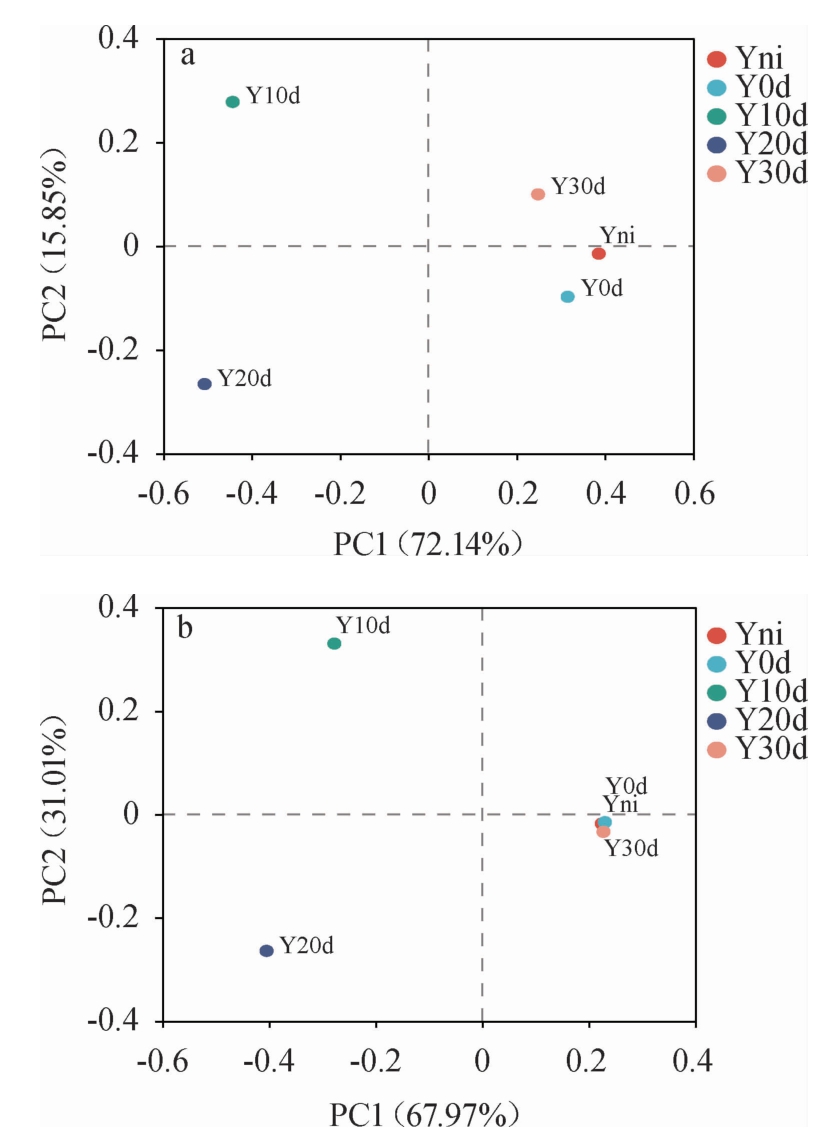
图2 基于OTUs数发酵凝乳及奶酪成熟过程中细菌(a)和真菌(b)群落的PCoA结果
Fig.2 PCoA results of bacterial (a) and fungal (b) communities of fermented curds and cheese during ripening process based on OTUs number
2.3 发酵凝乳及奶酪成熟过程中微生物群落结构分析
基于属水平,发酵凝乳及奶酪成熟过程中细菌和真菌菌群的结构见图3。
由图3a可知,乳球菌属(Lactococcus)、乳杆菌属(Lactobacillus)、片球菌属(Pediococcus)和明串珠菌属(Leuconostoc)存在于所有样品中,为发酵过程中的主要优势细菌属,其中Lactococcus相对丰度较高,且在奶酪成熟10 d和20 d时波动性较大,分布不均匀,说明其易受内外环境的影响[21-23]。奶酪成熟10 d和20 d时细菌菌群结构发生明显变化,成熟10 d时,除Lactococcus外Pediococcus和Leuconostoc大量出现;成熟20 d时Lactobacillus占主导作用。成熟时间延长到30 d时,Lactococcus相对丰度达到95.31%,与发酵凝乳接近。说明奶酪成熟过程中,成熟10 d和20 d时细菌菌落结构变化明显,这与文献[24]报道的结果一致。此外,在奶酪成熟过程中首次发现拉恩氏菌属(Rahnella)和拉乌尔菌属(Raoultella),这些菌属的存在可能会引起奶酪风味的变化,并对其安全性产生影响[25]。
图3 基于属水平发酵凝乳及奶酪成熟过程中细菌(a)和真菌(b)群落组成
Fig.3 Composition of bacterial (a) and fungal (b) communities of fermented curds and cheese during ripening process based on genus level
others:所有样品中相对丰度<0.01%的属。
由图3b可知,发酵凝乳及奶酪成熟过程中真菌属以地霉属(Geotrichum)、孢圆酵母属(Torulaspora)、毕赤酵母属(Pichia)、伊萨酵母菌(Issatchenkia)、克鲁维酵母属(Kluyveromyces)为主。从发酵凝乳到奶酪成熟0 d时,Geotrichum的相对丰度由88.43%降低至77.37%;成熟0~10 d时真菌菌群结构发生变化,Torulaspora的相对丰度由0.01%升高至39.73%,克鲁维酵母属(Kluyveromyces)的相对丰度由0.37%升高至22.77%;成熟10~20 d时,Pichia的相对丰度由5.94%升高至49.55%,Torulaspora的相对丰度由39.73%降低至13.59%;成熟20~30 d时,Issatchenkia的相对丰度由4.60%升高至39.82%,Geotrichum的相对丰度由2.32%升高至52.45%,可见奶酪成熟10 d和20 d的真菌菌落结构与其他时间点显著不同。地霉属(Geotrichum)在奶酪中具有可代谢乳酸、降低奶酪pH值的作用,有效改善奶酪风味和口感,对奶酪香味的形成起主要作用[26]。孢圆酵母属(Torulaspora)是发酵食品尤其是奶酪成熟过程中一类重要的酵母[27],能够起到提高芳香族化合物产量的效果,对奶酪的香味起主要作用。毕赤酵母属(Pichia)是食品中较为常见的酵母菌,其在许多传统发酵食品的风味形成中起着重要作用[28-31],在原料乳中具有较强降解脂肪能力[32]。伊萨酵母菌(Issatchenkia)与克鲁维酵母属(Kluyveromyces)是葡萄酒和中国酒风味形成的主要贡献者[33]。
为更好地探究奶酪成熟过程中的优势细菌与真菌,进一步基于总相对丰度排名前30的微生物属对样品进行层次聚类分析,结果见图4。
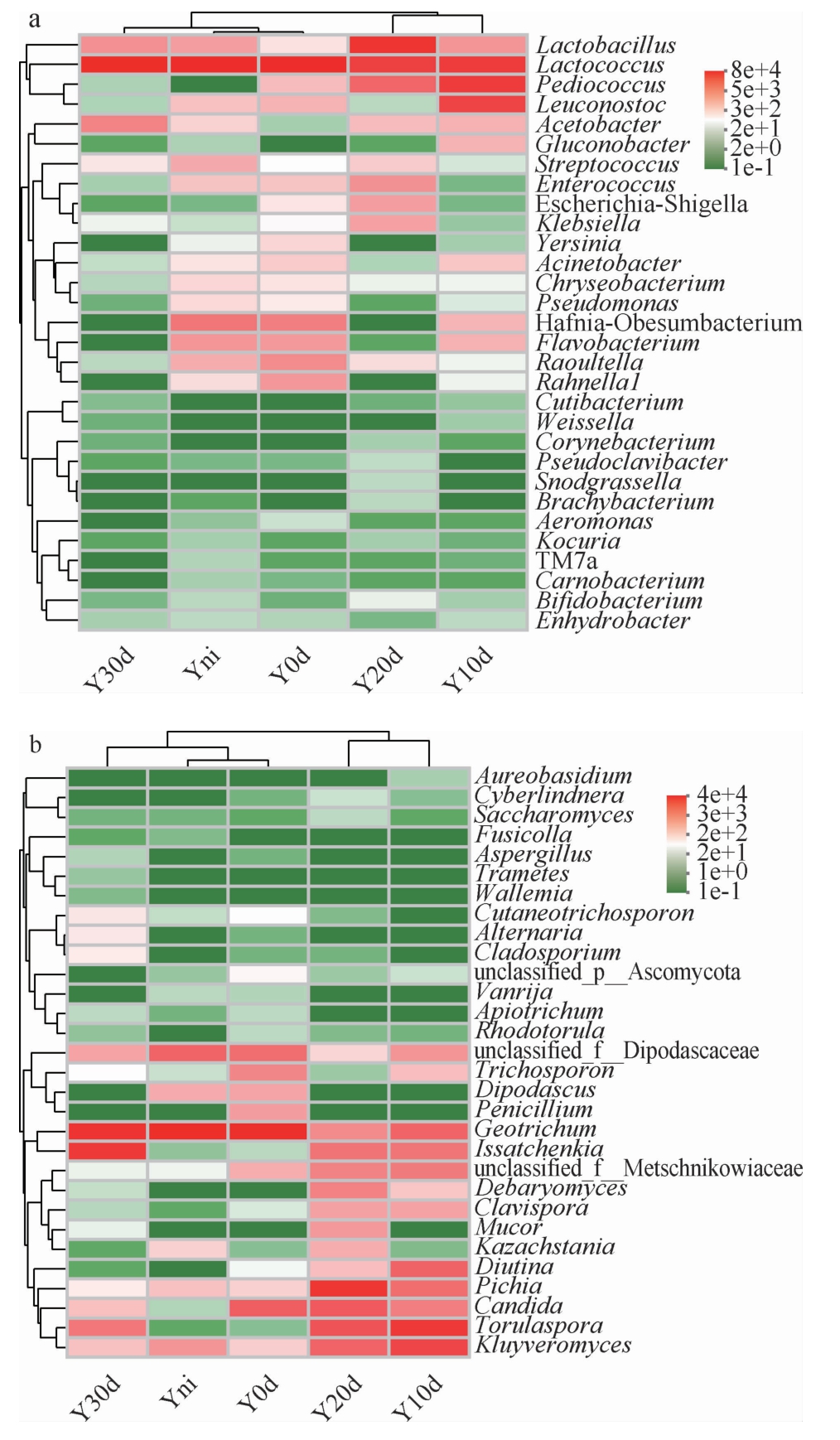
图4 基于主要细菌属(a)和真菌属(b)发酵凝乳及奶酪样品成熟过程中的层次聚类分析热图
Fig.4 Hierarchical cluster analysis heat map of fermented curds and cheese samples during the ripening process based on main bacterial (a) and fungal (b) genera
由图4可知,奶酪成熟过程中细菌和真菌菌群变化一致。其中发酵凝乳和奶酪成熟0 d的样品菌群组成相似,说明奶酪成熟过程中主导微生物主要来自凝乳。奶酪成熟10 d和20 d的样品的菌落构成与前一发酵阶段相比发生明显变化,可能原因是由于奶酪酸度降低引起了菌群结构变化。由图4a可知,相对丰度较高的乳球菌属(Lactococcus)、乳杆菌属(Lactobacillus)、片球菌属(Pediococcus)和明串珠菌属(Leuconostoc)聚为一类,且均属于乳酸菌,区别于其他聚类较为分散的低丰度细菌属。由图4b可知,相对丰度较高的酵母菌克鲁维酵母属(Kluyveromyces)、孢圆酵母属(Torulaspora)、假丝酵母菌属(Candida)、毕赤酵母属(Pichia)和链状念珠菌属(Diutina)聚为一类,当奶酪成熟30 d时,地霉属(Geotrichum)、伊萨酵母菌(Issatchenkia)等酵母菌聚为一类,说明发酵过程中细菌群落以乳酸菌为主,真菌群落以酵母菌为主,且真菌群落结构变化差异较大。
3 结论
本研究应用Illumina Miseq高通量测序技术,对蒙古族自然发酵奶酪不同成熟过程中的微生物群落进行多样性分析。基于Alpha多样性分析结果表明,在成熟过程中细菌与真菌菌群的丰度以及多样性发生较大变化,奶酪成熟10 d时微生物多样性及丰度均达到最大。通过Venn图和PCoA表明,成熟30 d时奶酪特有细菌和真菌OTU数均最多。通过群落结构及热图分析表明,奶酪在整个成熟过程中细菌属以乳酸菌为主,包括乳球菌属(Lactococcus)、乳杆菌属(Lactobacillus)、片球菌属(Pediococcus)和明串珠菌属(Leuconostoc),其中乳球菌属(Lactococcus)是奶酪成熟过程中的主要发酵剂,参与整个发酵过程;真菌属以酵母菌为主,优势细菌地霉菌属(Geotrichum)、孢圆酵母属(Torulaspora)、伊萨酵母菌(Issatchenkia)、毕赤酵母菌属(Pichia)和克鲁维酵母属(Kluyveromyces)。此外,在奶酪成熟过程中首次发现拉恩氏菌属(Rahnella)和拉乌尔菌属(Raoultella),其可能会引起奶酪风味的变化,并对奶酪安全性产生影响。本研究结果将为发掘和分离奶酪中的食源性微生物资源及了解蒙古族自然发酵奶酪安全性奠定基础。
[1]秦渤智,孙万成.中国奶酪行业发展现状和趋势[J].乳业科学与技术,2021,44(3):37-42.
[2]高磊,王超,高岩松.酸凝奶豆腐产品开发及其品质评价[J].食品与发酵工业,2023,49(3):105-111.
[3]郑晓吉,刘飞,任全路.基于SPME-GC-MS法比较新疆哈萨克族不同居住区奶酪风味差异[J].食品科学,2018,39(8):83-85.
[4]HAUFF S,VETTER W.Quantification of fatty acids as methyl esters and phospholipids in cheese samples after separation of triacylglycer-ides and phospholipids[J].Anal Chim Acta,2009,636(2):229-235.
[5]DElGADO F J,GONZLEZ-CRESPO J,CAVA R,et al.Characterisation by SPME-GC-MS of the volatile profile of a Spanish soft cheese P.D.O.Torta del Casar during ripening[J].Food Chem,2010,118(1):182-189.
[6]RIU-AUMATELL M,VARGAS L,VICHI S,et al.Characterisation of volatile composition of white salsify(Tragopogon porrifolius L.)by headspace solid-phase microextraction(HS-SPME)and simultaneous distillation-extraction(SDE)coupled to GC-MS[J].Food Chem,2011,129(2):557-564.
[7]KURTOVIC I,MARSHALL S N,MILLER M R,et al.Flavour development in dairy cream using fish digestive lipases from Chinook salmon(Oncorhynchus tshawytscha)and New Zealand hoki(Macruronus novaezealandiae)[J].Food Chem,2011,127(4):1562-1568.
[8]曾椿淋,朱琳,高凤.不同天然奶酪发酵剂和非发酵剂微生物多样性的高通量测序研究[J].现代食品科技,2018,34(10):118-121.
[9]达林,苏馨.奶豆腐制作过程中细菌多样性与风味物质研究[J].食品与发酵工业,2022,48(14):168-169.
[10]METZKER M L.Sequencing technologies-the next generation[J].Nat Rev Genet,2010,11(1):31-46.
[11]王馨蕊,史巧,刘毕琴.诺邓火腿加工过程中细菌群落的动态变[J].食品工业科技,2020,42(2):84-86.
[12]谢芳,谢华德,唐振华.基于高通量测序分析不同品种水牛生乳中细菌多样性[J].食品安全质量检测学报,2020,11(20):7529-7530.
[13]牟建平,滕宝霞,史中飞.基于高通量测序考察甘肃黄芪种植区根际土壤微生物群落结构及多样性[J].中国野生植物资源,2022,41(3):16-18.
[14]CHEN S F,ZHOU Y Q,CHEN Y R,et al.fastp:an ultra-fast all-in-one FASTQ preprocessor[J].Bioinformatics,2018,34(17):i884-i890.
[15]MAGOCˇT,SALZBERG SL.FLASH: fast length adjustment of short reads to improve genome assemblies[J].Bioinformatics,2011,27(21):2957-2963.
[16]EDGAR R C.UPARSE:highly accurate OTU sequences from microbial amplicon reads[J].Nat Methods,2013,10(10):996-998.
[17]于佳琦,夏亚男,乔晓宏.锡林郭勒牧区鲜马奶风味物质与微生物多样性分析[J].中国乳品工业,2021,49(5):8-9.
[18]赵凤琴.凉州熏醋传统酿造过程主要功能微生物群落演替对四甲基吡嗪的影响[D].兰州:甘肃农业大学,2016.
[19]曾椿淋.内蒙古手工奶酪微生物多样性和功能性研究[D].镇江:江苏大学,2020.
[20]RIVAS M N,BURTON O T,WISE P,et al.A microbiota signature associated with experimental food allergy promotes allergic sensitization and anaphylaxis[J].J Allergy Clin Immun,2013,131(1):201-212.
[21]肖芳,朱建军,翁庭钰.贮藏过程中锡林郭勒盟传统奶豆腐和奶皮子的微生物数量变化分析[J].农产品加工,2021,539(11):63-64.
[22]BONETTA S,BONETTA S,CARRARO E,et al.Microbiological characterisation of Robiola di Roccaverano cheese using PCR-DGGE[J].Food Microbiol,2008,25(6):786-792.
[23]SCHORNSTEINER E,MANN E,BEREUTER O,et al.Cultivation-independent analysis of microbial communities on Austrian raw milk hard cheese rinds[J].Int J Food Microbiol,2014,180(2):88-97.
[24]郑晓吉.新疆哈萨克族奶酪微生物菌群结构及特征[D].无锡:江南大学,2018.
[25]王鸿,谢斯超,周广敏.拉乌尔菌Raoultella sp.Pan22x菌株群体感应抑制活性的培养基及发酵条件的优化[J].浙江工业大学学报,2018,46(1):101-103.
[26]高代微,王琳琳,张岩.相对湿度对牦牛毛霉霉菌奶酪成熟过程中品质的影响[J].食品工业科技,2020,41(5):23-25.
[27]刘飞,郭孝敬,陈欢.新疆哈萨克族奶酪中潜在益生酵母菌的筛选[J].食品与发酵工业,2018,44(7):128-130.
[28]洋洋,乌有娜,双全.奶豆腐中乳酸菌的分离鉴定及风味特性研究[J].中国乳品工业,2021,49(9):19-22.
[29]MOUNIER J,MONNET C,JACQUES N,et al.Assessment of the microbial diversity at the surface of Livarot cheese using culture-dependent and independent approaches[J].Int J Food Microbiol,2009,133(1-2):31-37.
[30]李王强,樊哲新,刘月.新疆伊犁地区发酵驼乳中酵母菌多样性分析[J].中国食品学报,2020,20(7):189-190.
[31]ESCOBAR-ZEPEDA A,SANCHEZ-FLORES A,BARUCH M Q.Metagenomic analysis of a Mexican ripenedcheese reveals a unique complex microbiota[J].Food Microbiol,2016,57(7):116-127.
[32]COCOLIN L,AGGIO D,MANZANO M,et al.An application of PCRDGGE analysis to profile the yeast populations in raw milk[J].Int Dairy J,2002,12(5):407-411.
[33]董琦楠,李莹,叶冬青.耐热克鲁维酵母在葡萄酒酿造中的研究进展[J].微生物学通报,2022,49(5):1941-1954.