甜面酱是以小麦粉为原料,经过蒸煮灭菌、接种制曲、加盐水,半开放日晒夜露的方式自然发酵,形成具有咸中带甜具有独特滋味的发酵制品[1]。目前工业上生产甜面酱多以此种方式,自然发酵能够接触到更多的微生物,有着复杂的微生态[2],不同的外部因素如气候、地域、原辅料、发酵方法等均能够影响发酵体系的微生物群落结构[3-4],不同种类的微生物产生的酶系不同以及代谢产物不同,从而导致风味的差异,对于贵州传统发酵甜面酱中微生物还有待研究。甜面酱发酵过程中菌相变化复杂,传统可培养方法很难了解到参与发酵过程的微生物组成全貌及动态变化[1]。宏基因组技术可以分析生产发酵过程中微生物菌群结构动态变化规律以及核心功能微生物[5],可将此技术运用于贵州传统甜面酱中微生物的研究。
甜面酱品质的提高,在改进生产工艺的同时,应当更多地从菌相的研究着手,研究微生物的组成及变化与产品质量的关系,作为一个珍贵的天然菌库,甜面酱中微生物资源开发还有很大的空间,可以适当地进行人为控制微生物组成,筛选高产菌株用以制曲与发酵[6]。目前优良的菌种资源仍然比较匮乏,在发酵食品中筛选微生物可作为获取优质发酵剂的重要来源。蛋白酶作为最重要的工业酶之一,市场需求量极大,已知的蛋白酶产生菌很多来源于土壤或者海洋,可能是潜在的致病菌或条件致病菌[7],在食品、饲料等领域使用受到限制,仍需不断筛选可用于食品领域的蛋白酶高产菌株。
现阶段对甜面酱的研究中,人们大多关注其制作工艺和风味的研究[8],对于其中微生物较多关注到霉菌和酵母菌[9],对细菌的研究相对较少,忽略了其重要的产蛋白酶能力。因此,本研究以自然发酵甜面酱为对象,运用宏基因组测序技术对其中的微生物多样性进行分析,分离、筛选高产蛋白酶菌株,并采用单因素及响应面试验优化其产酶条件。旨在明确在不同发酵阶段中的优势菌群,有利于挖掘功能性菌株,缩短发酵时间,改善传统甜面酱的风味,为甜面酱的生产与质量控制提供理论依据,为菌种资源开发应用奠定基础,为发酵工业提供有利的菌种资源。
1 材料与方法
1.1 材料与试剂
1.1.1 样品采集
本实验所用甜面酱样品(后熟发酵阶段前期(1月)、中期(3月)、末期(8月)):贵州某调味品公司。
1.1.2 化学试剂
E.Z.N.ATMMag-BindSoil脱氧核糖核酸(deoxyribonucleic acid,DNA)Kit试剂盒:美国OMEGA公司;干酪素、福林酚、果糖(均为分析纯):北京索莱宝科技有限公司;三氯乙酸、碳酸钠、硼酸(均为分析纯):天津致远化学试剂有限公司;牛肉膏、酵母浸膏、蛋白胨、琼脂粉(均为生化试剂):上海生工生物工程有限公司;硫酸铵、尿素(均为分析纯):成都金山化学试剂有限公司。
1.1.3 培养基
Luria-Bertani(LB)肉汤培养基:上海生工生物工程有限公司。
蛋白酶筛选固体培养基:25 g/L LB肉汤培养基,10 g/L干酪素,18 g/L琼脂,水1 000 mL。121 ℃灭菌20 min。
1.2 仪器与设备
LDZM-60L 型立式高压蒸汽灭菌器:上海申安医疗器械厂;SW-CJ-ID超净工作台:上海力辰邦西仪器科技有限公司;SPX-150BⅢ型生化培养箱:天津市泰斯特仪器有限公司;SHZ气浴恒温振荡器:常州金坛良友仪器有限公司;TGL-16M 型台式高速冷冻离心机:湖南平凡科技有限公司;Multiskan Sky High全波长酶标仪:美国Thermo Fisher公司。
1.3 方法
1.3.1 样品中微生物多样性分析
参照万宁威等[10]的方法,具体操作流程:采用E.Z.N.ATM Mag-Bind Soil DNA Kit试剂盒对样品进行宏基因组微生物总DNA的提取,琼脂糖凝胶电泳进行样本质检,以其为模板,使用通用引物341F/806R对细菌16S rRNA V3-V4区域[11]进行聚合酶链反应(polymerase chain reaction,PCR)扩增来鉴定细菌[12]。使用通用引物ITS1F/ITS2R对真菌内部转录间隔区(internal transcribed spacer,ITS)ITS1-ITS2区域进行PCR扩增来鉴定真菌[13]。PCR扩增体系及PCR扩增条件参照罗爱国等[14]的方法。
采用凝胶回收试剂盒回收PCR产物,用Tris-HCl洗脱,并将PCR产物用DNA荧光定量系统进行检测定量。在Illumina MiSeq平台上对其进行测序,委托生工生物工程(上海)股份有限公司完成。
数据分析与处理:测序得到的序列首先根据高精度循环共识测序(circular consensus sequencing,CCS)自我矫正模式可获得高质量的数据,再根据双端barcode进行样本区分,然后去噪分析。将其进行聚类,其中相似性≥97%的序列归为1个操作分类单元(operational taxonomic unit,OTU),将OTU代表序列与Silva数据库[15]进行比对和物种注释,最后对有效数据进行OTUs丰度、Alpha多样性指数和物种组成等分析。
1.3.2 产蛋白酶菌株的分离与筛选
称取甜面酱样品于无菌水中进行梯度稀释,涂布于蛋白酶筛选固体培养基上,产生蛋白酶的菌株能够利用培养基中的干酪素从而产生透明的蛋白水解圈,故挑取产生透明圈的单菌落进行分离纯化,保存备用。
初筛:采用滤纸片法[16]。用无菌接种环挑取保存在试管中的菌株接种至LB肉汤培养基中,于37 ℃、180 r/min条件下摇瓶培养72 h,发酵液于4 ℃条件下12 000 r/min离心15 min后过0.22 μm滤菌膜,取7 μL发酵液于干酪素平板的无菌滤纸片上,37 ℃静置24~48 h后测量水解圈直径。在相同时间内蛋白酶活力与其产生蛋白水解圈直径呈正比。
复筛:采用福林酚法。选取产生蛋白水解圈直径最大10株菌,将目的菌株在LB肉汤培养基中活化,于37 ℃、180 r/min条件下摇瓶培养24 h作为种子液(控制活菌数为107~108 CFU/mL),将种子液按接种量2%(V/V)接种至LB肉汤培养基中,相同条件下摇瓶培养72 h,发酵液在4 ℃、12 000 r/min条件下离心5 min,测定上清液蛋白酶活力,选取酶活力最高的菌株。
1.3.3 菌株鉴定
采用形态学观察、生理生化试验及分子生物学方法对筛选菌株进行鉴定,具体参考《伯杰细菌鉴定手册》[17]和《常见细菌系统鉴定手册》[18]。利用通用引物27F和1492R扩增菌株16S rDNA片段[19]。将测序结果与美国国家生物信息中心(national center for biotech nology information,NCBI)的GenBank数据库中已知的序列进行基本局部比对搜索工具(basic local alignment search tool,BLAST)同源性比对,采用MEGA7.0中邻接(neighbor joining,NJ)法构建系统发育树。
1.3.4 菌株生长曲线的测定
采用比浊法测定生长曲线[20]。前24 h每2 h、后24 h每4 h测定OD600nm值。
1.3.5 产蛋白酶条件的优化
产酶培养基优化单因素试验:以LB肉汤培养基为基础,分别考察碳源(葡萄糖、果糖、蔗糖、乳糖、可溶性淀粉,添加量均为1%)、有机氮源(黄豆饼粉、玉米粉、豆粕、麦芽浸粉、酵母浸膏、牛肉浸膏,添加量均为1%)+0.5%硫酸铵[19]对蛋白酶活力的影响。初始培养条件为接种量2%(V/V),初始pH值为7,在37 ℃、180 r/min条件下培养72 h,离心测定上清液蛋白酶活力,每组3个平行,确定最佳碳氮源。
产酶条件优化单因素试验:分别考察发酵温度(25 ℃、28 ℃、31 ℃、34 ℃、37 ℃)、接种量(2%、3%、4%、5%、6%)、发酵时间(1 d、2 d、3 d、4 d、5 d)及初始pH(7、8、9、10、11)4个因素对蛋白酶活力的影响,确定每个因素最优水平。
产酶条件优化响应面试验:在单因素试验基础上,选取发酵温度(A)、发酵时间(B)及初始pH(C)为自变量,以蛋白酶活力(Y)为响应值,进行响应面试验。
1.3.6 蛋白酶活力测定方法
粗酶液的制备:发酵液在4 ℃、12 000 r/min条件下离心5 min。由前期实验可知,菌株PC9产酶类型为碱性蛋白酶,故用pH=10.5硼酸缓冲液将其稀释适宜浓度作为待测酶液。
碱性蛋白酶测定:按照GB/T 23527—2009《蛋白酶制剂》中的福林法。
碱性蛋白酶酶活定义:1 g固体酶粉(或1 mL液体酶),在pH 10、40 ℃的条件下,每分钟水解酪蛋白能产生微克酪氨酸,定义为一个酶活力单位(U/mL)。
1.3.7 数据处理
利用SPSS20.0对数据进行处理与分析,Origin2017、GraphPad Prasm8.0软件制图,Design-Expert 10.0.4软件分析响应面试验数据。
2 结果与分析
2.1 甜面酱中微生物多样性分析
2.1.1 测序数据统计及Alpha 多样性统计
对甜面酱自然发酵过程中3个阶段样品的微生物群落进行测序分析,统计去除嵌合体后序列量,得到高质量序列16S rRNA 52 790条,ITS 140 814条。微生物群落α多样性指数详情见表1。
覆盖率(Coverage)测序深度指数均>99.9%,可以真实反映样品中的绝大多数微生物。通过Chao1指数和Ace指数来评价物种丰富度,由表1可知,随着发酵的进行,真菌Chao1指数和Ace指数都逐渐升高,真菌的物种丰富度呈逐渐上升的趋势,在发酵末期(8月)达到最高。而细菌的物种丰富度则呈先升高再降低的趋势。
表1 甜面酱发酵过程中的微生物菌群Alpha多样性分析结果
Table 1 Alpha diversity analysis results of microbial community in the fermentation process of sweet flour sauce
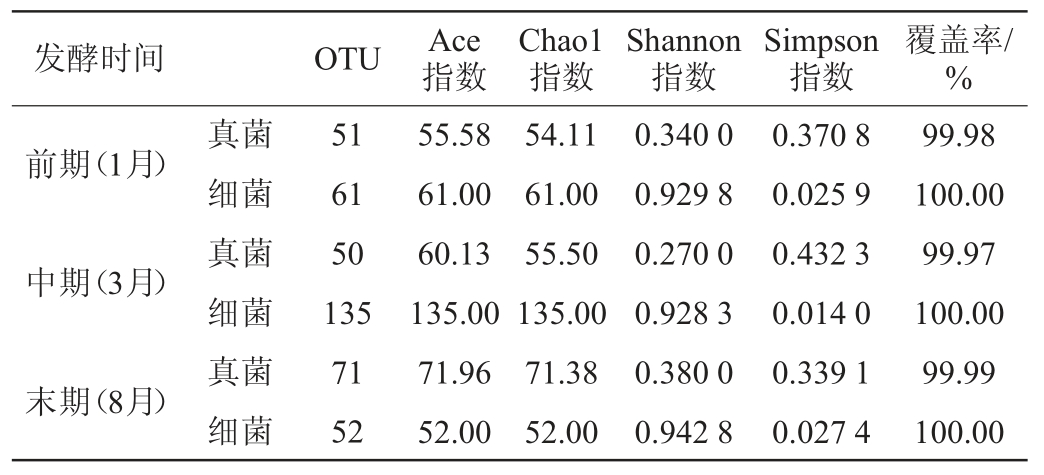
菌群多样性通过Shannon指数和Simpson指数来评价,在发酵过程中真菌物种多样性呈先降低再上升的趋势;细菌的Shannon指数并无显著变化,Simpson指数呈先降低再上升的趋势,结合样本OTUs总数,说明样品中的细菌多样性先上升再降低,且在自然发酵过程中细菌物种丰富度远高于真菌。
结果表明,在发酵前期(1月)样品中微生物多样性相对较低,随着发酵的进行,微生物丰富度有所升高,推测原因可能与其发酵工艺有关。发酵甜面酱在前期采用纯种制曲,在刚进入后发酵日晒夜露阶段前期,刚加入盐水后许多细菌和丝状真菌因无法适应酱醪的高盐度或高水分而相继死亡[22],随后甜面酱经历在酱缸中的自然发酵,耐盐乳酸菌和酵母菌开始大量繁殖,可发酵糖类被转化成乙醇、乳酸乙酯和乙酸乙酯等香气物质[23],赋予传统甜面酱独特的风味。
2.1.2 传统甜面酱不同发酵阶段微生物多样性Venn图分析
OTU分布Venn图可以直观展现不同发酵阶段微生物在OTU构成上的相似性、特异性和交叠情况。如图1所示,在发酵过程中微生物菌群组成在发酵不同时期均具有明显的差异性,Venn图中不同的颜色代表不同的分组。由图1a可知,不同分组间真菌群落共有OTU数为27,说明有27种真菌在发酵前中末期都有被检出,在发酵前期(1月)、中期(3月)、末期(8月)独有的OTU数量分别为13、5和29。由图1b可知,不同分组间细菌群落共有OTU数为8,独有的OTU数量分别为50、126和40。其中,真菌在发酵中期(3月)时独有的OTU数最少,在末期(8月)时最多。细菌不同时期菌种组成存在较大差异,细菌独有OTU数在发酵初期末期时较少,在中期时最多达到126。
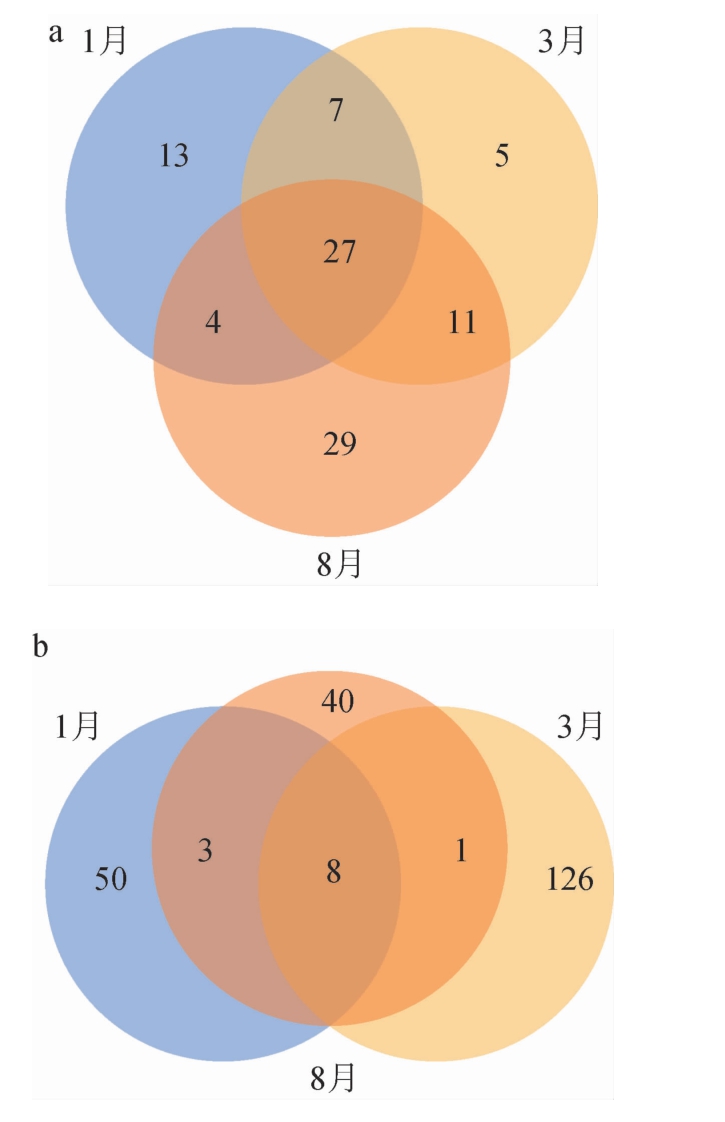
图1 传统甜面酱中真菌(a)和细菌(b)操作分类单元数的Venn图
Fig.1 Venn diagram of operational taxonomic unit number of fungi (a) and bacteria (b) in traditional sweet flour sauce
在保温发酵前期,发酵体系中真菌历经高渗(拌盐)、低氧(密闭)等环境的限制,水分活度降低,OTU数量较少,一些抗逆性强的细菌适应了环境细菌迅速增长[23],在发酵中期细菌在其中占有极大优势。
2.1.3 传统甜面酱不同发酵时期微生物群落结构分析
传统甜面酱不同发酵时期微生物群落结构分析结果见图2。由图2a可知,3个样品中真菌群落组成在属水平上主要共检测出9个真菌属和其他未分类菌属,分别为曲霉属(Aspergillus)、接合酵母属(Zygosaccharomyces)、威克汉姆酵母属(Wickerhamomyces)、米勒酵母属(Millerozyma)、酵母菌属(Saccharomyces)、镰刀菌属(Fusarium)、伊萨酵母属(Issatchenkia)、根毛霉属(Rhizomucor)、巴德利酵母属(Debaryomyces)及其他未分类菌属。
由图2a可知,曲霉属和接合酵母属贯穿整个发酵过程,在发酵前期(1月)和末期(8月)时,优势菌群皆为曲霉属,相对丰度达到了55.67%和51.36%,但在发酵中期(3月)时,相对丰度降低至36.78%,接合酵母属相对丰度由31.12%上升至58.52%,成为了优势菌群;威克汉姆酵母属在发酵末期相对丰度上升至27.52%。米勒酵母属在发酵前期和末期中的相对丰度分别为4.63%和6.41%,但在发酵中期时分布较少。
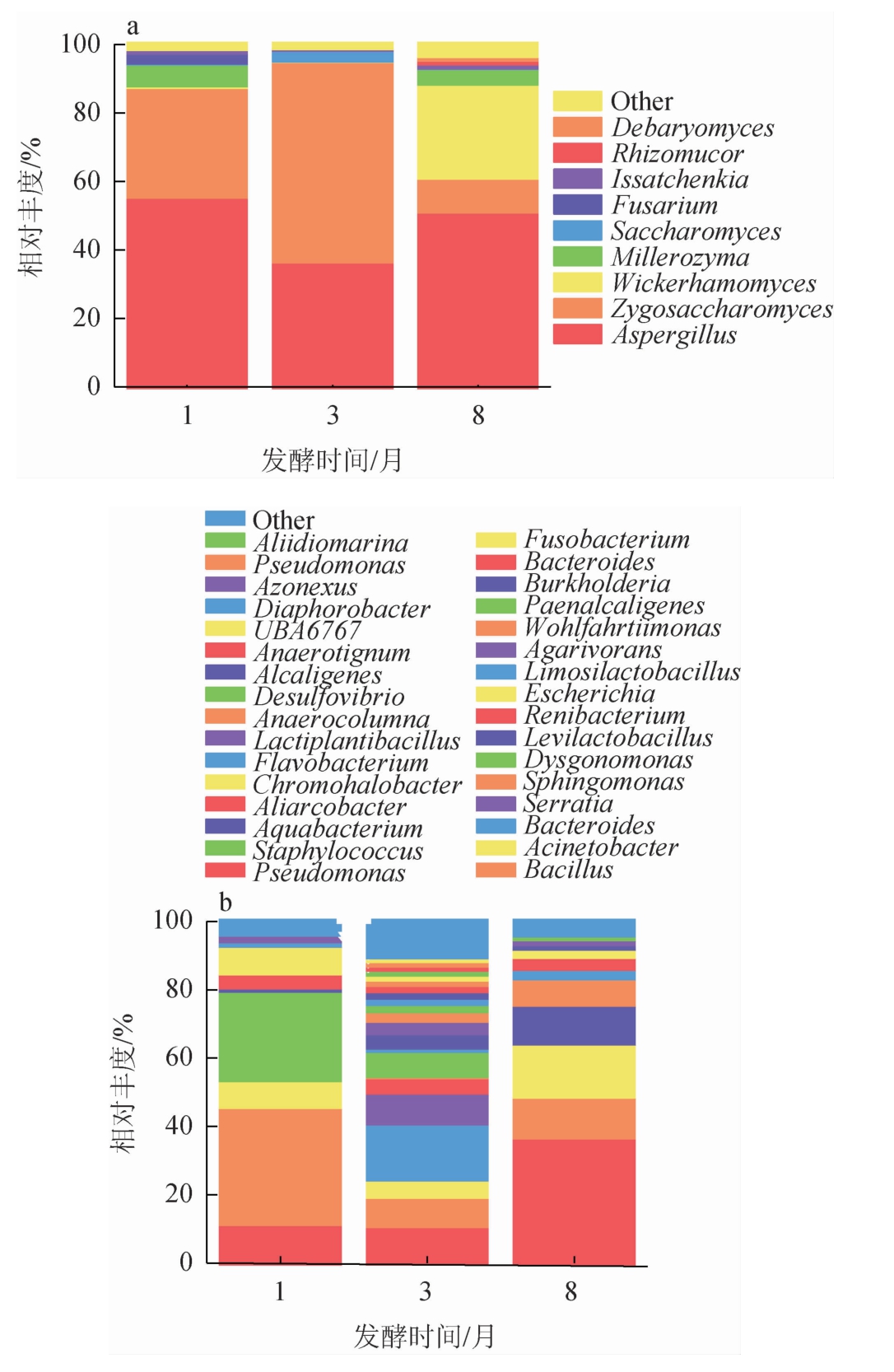
图2 传统甜面酱发酵过程中真菌(a)和细菌(b)在属水平群落结构变化
Fig.2 Changes of community structure of fungi (a) and bacteria (b)in the fermentation process of traditional sweet flour sauce in genus level
结合甜面酱生产工艺,在制曲阶段采用纯种制曲,菌种为米曲霉(Aspergillus oryzae)沪酿3.042,结果显示曲霉属是其中优势菌群,能产生丰富的淀粉酶和蛋白酶,将物料中营养物质降解为小分子的糖和氨基酸等;酵母菌主要作用在发酵繁殖阶段,发酵产物乙醇和细菌发酵所产生的有机酸在酯化酶作用下生成酯类物质,构成甜面酱风味成分的主体[6]。在发酵后期,部分酵母菌发生自溶,菌体内核酸类物质释放,有助于提升发酵食品的鲜味[24],推断这可能也是导致发酵末期酵母菌群相对丰度降低的原因之一。自然发酵开放的环境也可能接触到一些不利于人体健康的真菌属,如镰刀菌属,有些可产生真菌毒素,食用后可能会造成食物中毒[25],但只在发酵前期被检出,推测其可能受到其中一些拮抗细菌的抑制,故在中期(3月)和末期(8月)样品中未检出。
传统发酵甜面酱中细菌群落组成明显丰富于真菌。由图2b可知,3个样品中主要共检测出32个细菌属和其他未分类菌属,不同发酵阶段的优势菌群有所差别。其中贯穿发酵过程的优势菌属主要有假单胞菌属(Pseudomonas)、芽孢杆菌属(Bacillus)、不动杆菌属(Acinetobacter)、黄杆菌属(Flavobacterium)。假单胞菌属在发酵前中期的相对丰度分别为11.58%和11.03%,到发酵末期显著提升至36.75%;芽孢杆菌属在发酵前期的相对丰度最高达到34.29%,到发酵中期下降至8.55%,到末期有所升高,达到11.96%。除此之外,发酵前期的优势菌属主要还包括葡萄球菌属(Staphylococcus)(26.24%)、色盐杆菌属(Chromohalobacter)(8.00%)、不动杆菌属(Acinetobacter)(7.76%);在发酵中期的主要优势菌还包括拟杆菌属(Bacteroides)(16.36%)、赛氏杆菌属(Serratia)(9.08%)、营发酵单胞菌属(Dysgonomonas)(7.37%)、利维拉菌属(Levilactobacillus)(4.27%)、乳植杆菌属(Lactiplantibacillus)(3.62%);发酵末期的主要优势菌群还有不动杆菌属(Acinetobacter)(15.45%)、水杆菌属(Aquabacterium)(11.24%)、氨醇单胞菌属(Sphingomonas)(7.76%)等。
MAO P等[26]采用PCR-Denature梯度凝胶电泳法研究表明,甜面酱发酵过程中分离到的真菌大部分属于两种接合酵母,芽孢杆菌、葡萄球菌及乳酸菌为优势细菌。AN F等[27]通过高通量测序得出东北豆酱主要的细菌为四联球菌属、乳酸菌属、芽孢杆菌属及肠球菌属,主要优势真菌为曲霉属、毛霉属及根霉属。余丹等[22]研究发现,传统甜面酱后发酵120~180 d的优势真菌为接合酵母属(≥85.42%),芽孢杆菌属在整个发酵阶段一直是最主要的优势细菌,与本研究结果具有一致性,曲样和酱醪中的优势菌属还包括葡萄球菌属、雷尔氏菌属、泛菌属、伯克霍尔德氏菌属、片球菌属、鞘氨醇单胞菌属等,与本试验结果存在一定的差异。芽孢杆菌属在整个发酵周期中都占据较大的相对丰度,因其对某些逆境耐受阈值较其他许多发酵细菌更高[28],且具有强大的产酶能力,分泌的代谢产物会促进甜面酱发酵后熟,有助于发酵的进行,此外芽胞杆菌可以产生具酱香风味的吡嗪类物质[24],是发酵食品中风味和呈色的重要来源,在发酵食品较为常见,通过其代谢功能,促进了发酵食品风味形成及品质提升。
甜面酱的独特滋味通常为游离氨基酸、还原糖、有机酸、核苷酸以及小分子肽等,其主要作用的是霉菌、乳酸菌、芽孢杆菌以及酵母菌。通过以上结果发现,这些微生物为甜面酱中的优势菌群,丰富的酶系能有效分解原料中营养物质,既能为其他微生物的生长代谢提供物质基础,又能分解蛋白质产生的氨基酸、多肽等代谢产物,可为发酵食品特征风味的形成奠定基础。从传统发酵食品中分离和筛选优势微生物可能是获得功能发酵菌株的一种重要研究手段,直接添加筛选得到的真菌和细菌到原料中作为发酵起始菌株去制作发酵食品也已经形成一种突破,可以达到高度控制发酵过程和得到标准化产品的目的。
2.2 产蛋白酶菌株的筛选
采用酪素平板法从样品中挑选出细菌47株(编号为PC1~PC30、pr01a~pr01g、pr02a~pr02j)。通过滤纸片法进行初筛,产生明显蛋白水解圈的有27株,结果见表2。由表2可知,菌株PC9水解圈直径最大达到2.467 cm。选取其中水解圈直径最大的10株进行复筛,结果见图3。由图3可知,菌株PC9产蛋白酶活力显著高于其他菌株(P<0.05),达到48 U/mL。因此,对菌株PC9进行进一步鉴定。
表2 不同菌株在酪素平板上产蛋白水解圈直径测定结果
Table 2 Determination results of proteolytic circle diameters produced by different strains on casein plate cm
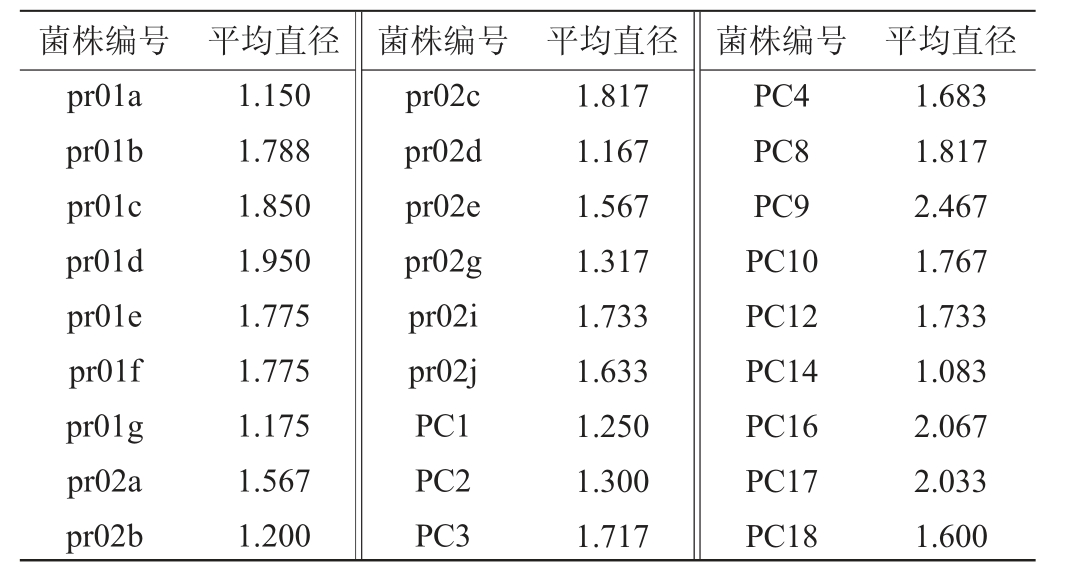
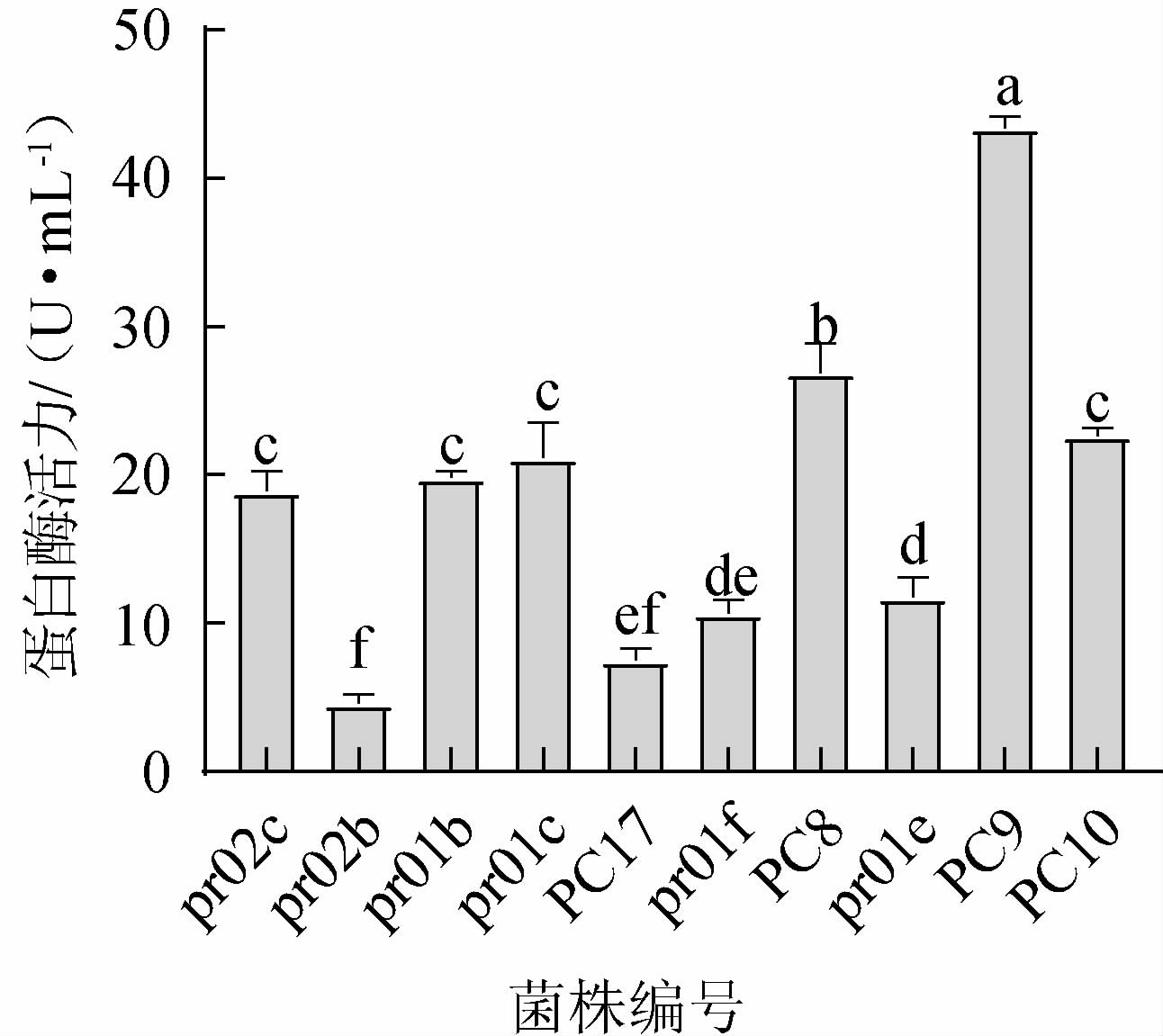
图3 筛选菌株产蛋白酶活力测定结果
Fig.3 Determination results of protease activity produced by screened strains
不同小写字母表示差异显著(P<0.05)。下同。
2.3 菌株PC9的鉴定
2.3.1 形态学与生理生化鉴定
菌株PC9的形态观察结果见图4。由图4a可知,菌株PC9菌落形态为乳白色,表面干燥,中间凹陷,边缘粗糙不规则,菌落直径为2~5 mm。由图4b可知,菌株PC9细胞形态呈杆状,单细胞,革兰氏染色和芽孢染色均为阳性。由图4c可知,菌株PC9在干酪素平板上产生蛋白水解圈较大,说明其具有较强产蛋白酶能力。
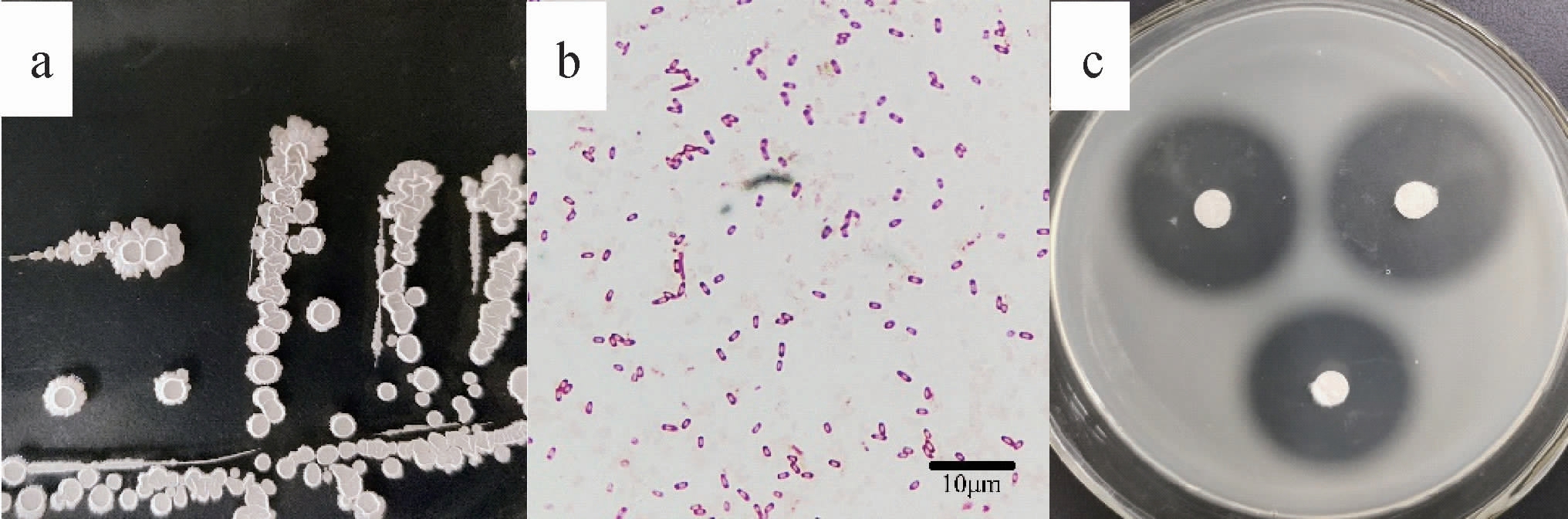
图4 菌株PC9的菌落形态(a)、细胞形态(b)和蛋白水解圈(c)
Fig.4 Colony morphology (a),cell morphology (b) and proteolytic circle (c) of strain PC9
菌株PC9的生理生化特征见表3。由表3可知,菌株PC9具有较强的耐盐能力,利用乳糖、麦芽糖、木糖不产酸,具有淀粉水解能力,不能水解油脂、纤维素。接触酶、明胶液化和甲基红试验、柠檬酸盐利用、VP试验结果呈阳性,其他试验结果均为阴性。因此,初步鉴定菌株PC9为芽孢杆菌属(Bacillus)。
表3 菌株PC9的生理生化特征
Table 3 Physiobiochemical characteristics of strain PC9
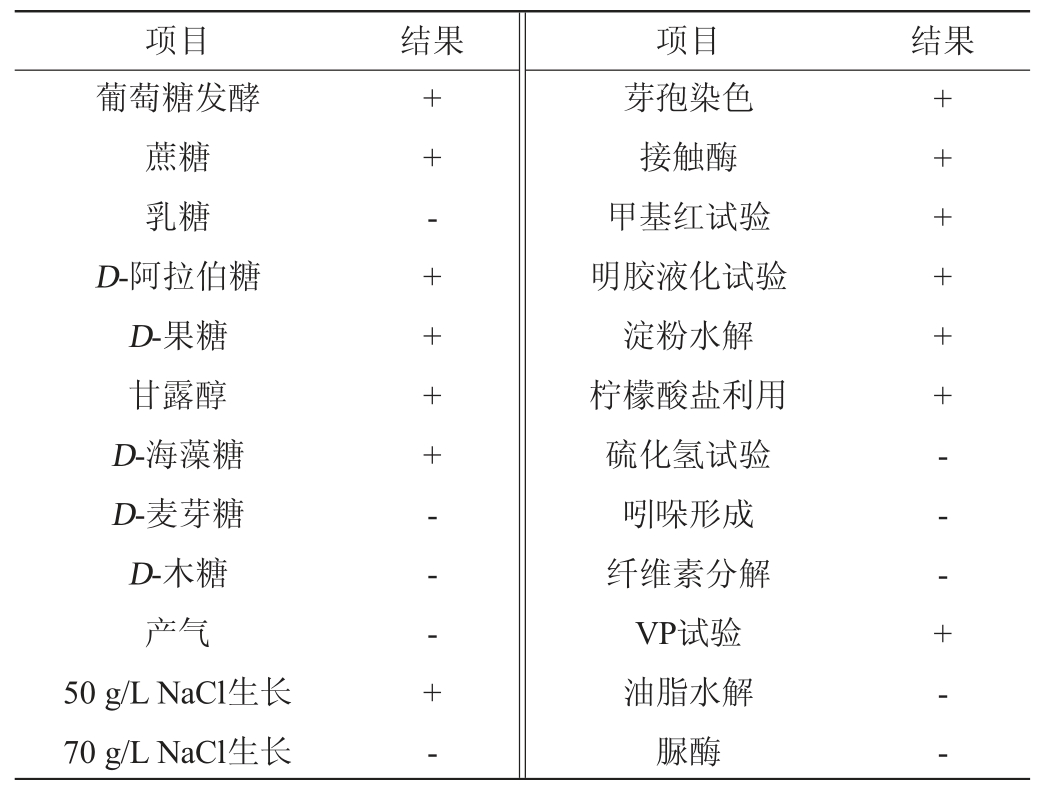
注:“+”表示结果呈阳性;“-”表示结果呈阴性。
2.3.2 分子生物学鉴定
经扩增、纯化和测序,菌株PC9的16S rDNA长度在1 449 bp左右。根据同源序列搜索结果构建系统发育树,如图5所示,菌株PC9扩增序列与菌株Bacillus mojavensis(登录号:KF811046.1)形成一个分枝,亲缘关系最近。结合菌株的形态特征、生理生化性[29-31],鉴定菌株PC9为莫海威芽孢杆菌(Bacillus mojavensis)。

图5 基于16S rDNA基因序列菌株PC9的系统发育树
Fig.5 Phylogenetic tree of strain PC9 based on16S rDNA gene sequences
任菲等[16]在传统发酵食品中筛选产蛋白酶菌株,芽孢杆菌属丰度最高,碱性蛋白酶活性普遍较高,生长最适温度一般在30~40 ℃、最适pH一般在7~9,其中也包括莫海威芽孢杆菌(B.mojavensis)。芽孢杆菌具有很强的产酶能力,但少有人关注到莫海威芽孢杆菌的产蛋白酶能力。DIABANKANA R G C等[32]筛选出的生防菌莫海威芽孢杆菌(B.mojavensis)PS17能产生几丁质酶、β-葡聚糖酶、纤维素酶、脂肪酶等多种水解酶,且在-2.2 MPa的渗透压下表现出耐旱性和中度耐盐性。ADIGÜZEL A O[33]分离出嗜热和耐盐酶/嗜盐细菌莫海威芽孢杆菌TH309,表现出优异的酯酶分泌能力和产壳聚糖酶能力[34]。且ANISSA H等[35]研究表明,其对人红细胞无溶血活性,支持其作为功能性食品加工添加剂的适宜性。AMAL H等[36]利用莫海威芽孢杆菌SA生产蛋白酶,并将其用于肉类副产品,生产生物活性蛋白质水解物,所得产物显示出重要的抗氧化活性。
2.4 菌株PC9的生长曲线
生长曲线的测定可以直观反映出细菌生长和代谢的规律,菌株PC9的生长曲线结果见图6。
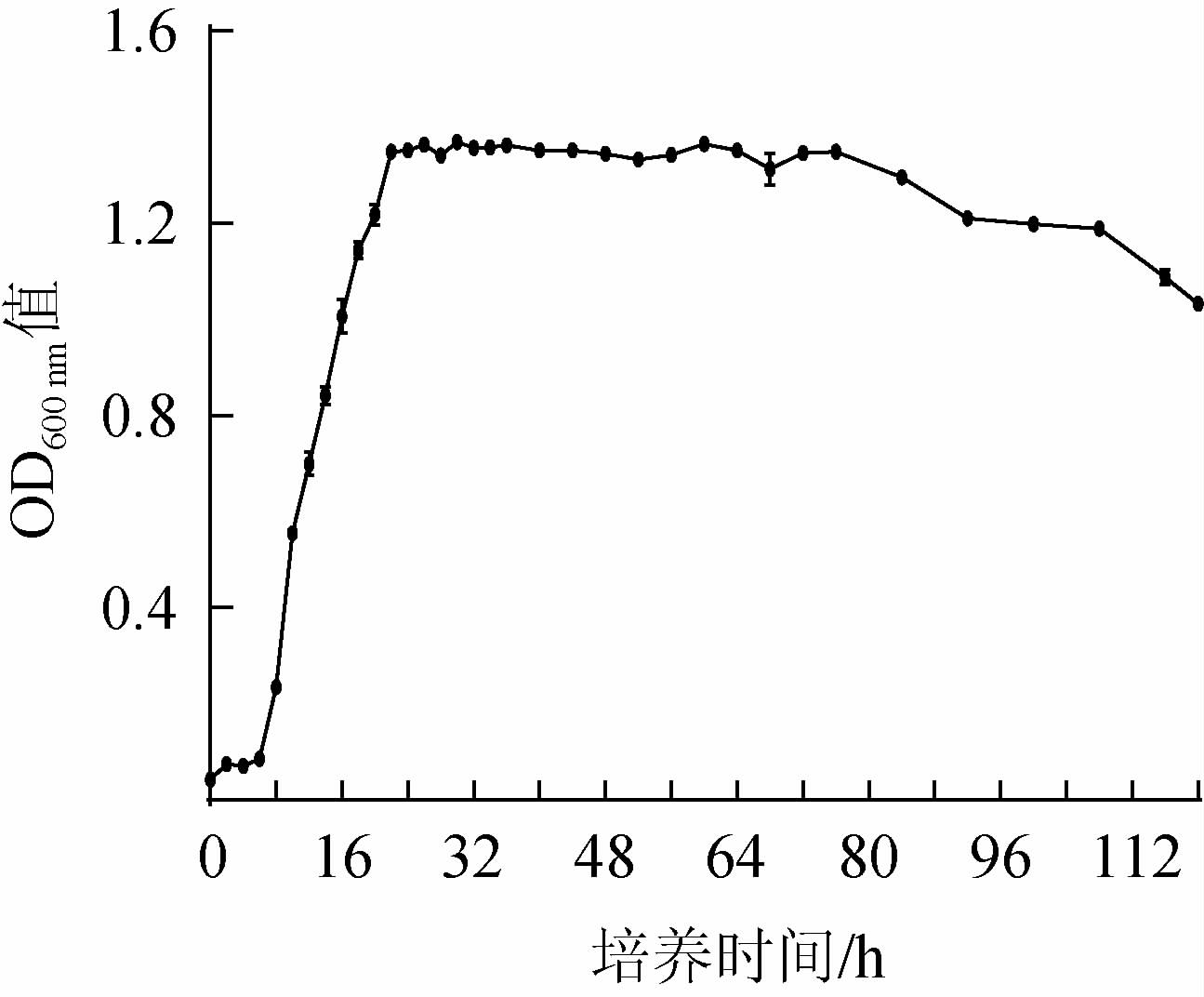
图6 菌株PC9的生长曲线
Fig.6 Growth curve of strain PC9
由图6可知,菌株在0~6 h生长迟缓,6~24 h进入对数生长期,24~72 h为稳定期,到72 h为衰亡期。菌株PC9可以长时间保持在稳定期直至72 h后才缓慢进入衰亡期,某些代谢产物的次级代谢产物在稳定期产生,可能包括抗生素、多肽、某些蛋白或代谢分泌到胞外的酶类[37],所以稳定期是发酵生产的最适收获期。
2.5 菌株PC9产蛋白酶培养基优化单因素试验
碳源种类、有机氮源+硫酸铵对菌株PC9产蛋白酶活力的影响结果见图7。
由图7a可知,以果糖为唯一碳源时,菌株PC9产蛋白酶酶活力显著高于其他碳源(P<0.05)。因此,选择果糖作为发酵培养基中碳源。
由于不同来源的氮源对菌体生长和产酶促进作用不同,当使用复合氮源时,对菌株产酶有进一步的促进作用,菌株PC9可以利用硫酸铵,采用有机氮源加硫酸铵的方式,探究其对产酶的影响[38]。由图7b可知,当氮源为酵母浸膏加硫酸铵时,酶活力显著高于其他水平(P<0.05),且价格较为低廉,故以酵母浸膏加硫酸铵作为最佳氮源。
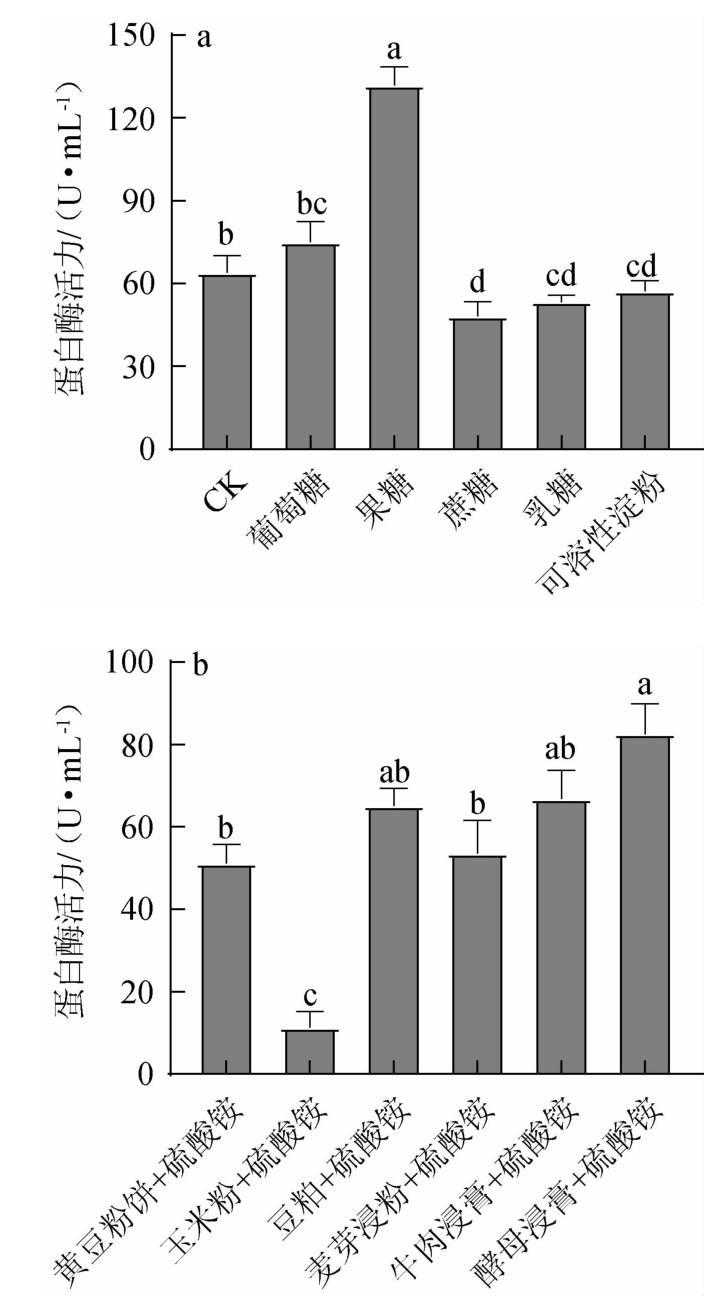
图7 碳源种类(a)、有机氮源+硫酸铵(b)对菌株PC9产蛋白酶活力的影响
Fig.7 Effect of carbon source type (a),organic nitrogen source+(NH4)2SO4(b) on protease activity produced by strain PC9
2.6 菌株PC9产蛋白酶发酵条件优化单因素试验
由图8a可知,当发酵温度为25~28 ℃时,蛋白酶活力随之增高;当发酵温度为28 ℃时,蛋白酶活力达到最高为418.31 U/mL,此时菌体代谢旺盛,致使蛋白酶大量表达;当发酵温度高于28 ℃之后,酶活力反而有所下降。因此,选择最佳发酵温度为28 ℃。
由图8b可知,不同接种量对蛋白酶活力影响差异不显著(P>0.05),当接种量为2%~6%时,蛋白酶活力随之增加;当接种量在6%时,蛋白酶活力达到最大值,为143.24 U/mL;当接种量>6%时,接种量继续增加酶活力不在继续升高。这可能是因为接种量过大,则会造成菌体密度过大,营养物质和氧含量有限,影响生长,进而限制了菌株的产酶能力[39]。因此,选择最佳接种量为6%。
由图8c可知,当发酵时间为1~2 d时,蛋白酶活力随之增加;当发酵时间为2 d时,蛋白酶活力达到最高为203.96 U/mL;当发酵时间>2 d之后,蛋白酶活力有所下降。可能是因为随着时间的延长,培养基中营养成分逐渐耗尽和次级代谢产物的不断积累,抑制了该菌株产蛋白酶活力。因此,选择最佳发酵时间为2 d。
由图8d可知,当初始pH值为为7~9时,蛋白酶活力逐渐增加;当初始pH值为9时,蛋白酶活力最高为244.12 U/mL;但当初始pH值>9之后,蛋白酶活力有所下降,原因可能为过高的pH影响了菌体的增值从而影响菌体产酶能力。因此,选择最佳初始pH值为9。
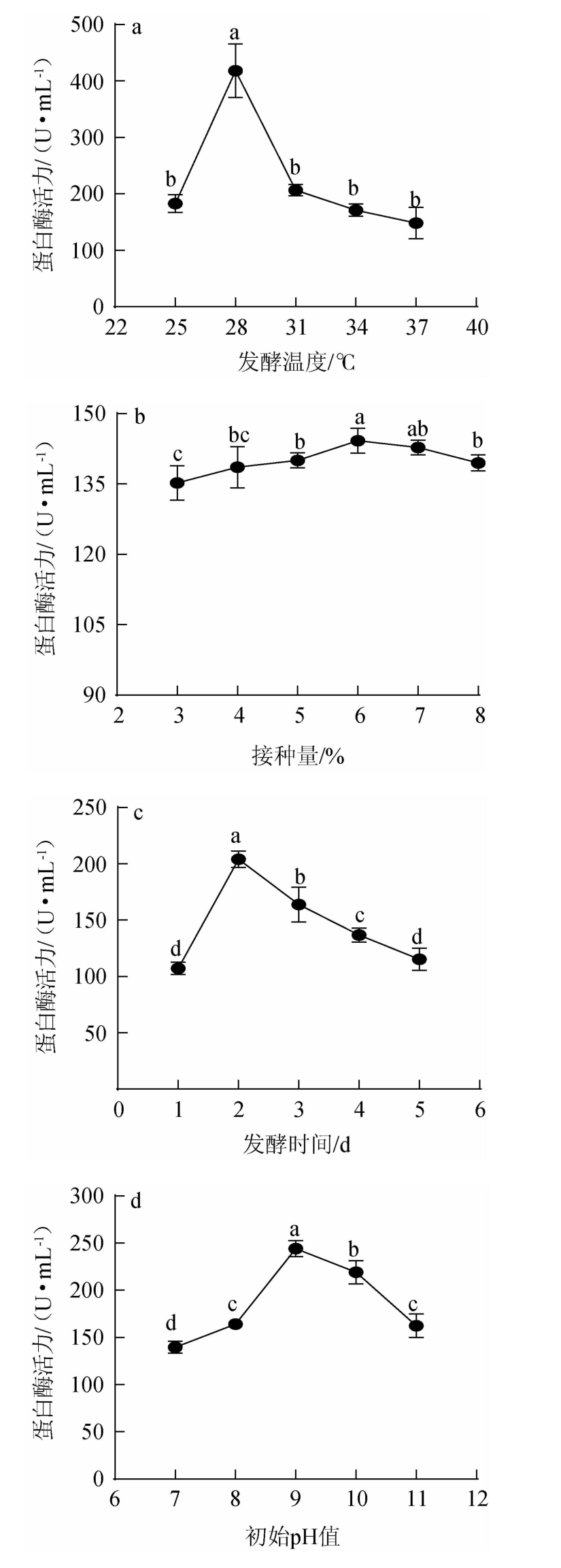
图8 发酵温度(a)、接种量(b)、发酵时间(c)、初始pH值(d)对蛋白酶活力的影响
Fig.8 Effect of fermentation temperature (a),inoculum (b),fermentation time (c) and initial pH value (d) on protease activity
2.7 菌株PC9产蛋白酶条件优化响应面试验
在单因素试验基础上,选取发酵温度(A)、发酵时间(B)及初始pH值(C)为自变量,以蛋白酶活力(Y)为响应值,采用响应面试验优化发酵条件,Box-Behnken试验设计及结果见表4,回归模型方差分析见表5。
表4 Box-Behnken试验设计及结果
Table 4 Design and results of Box-Behnken experiments
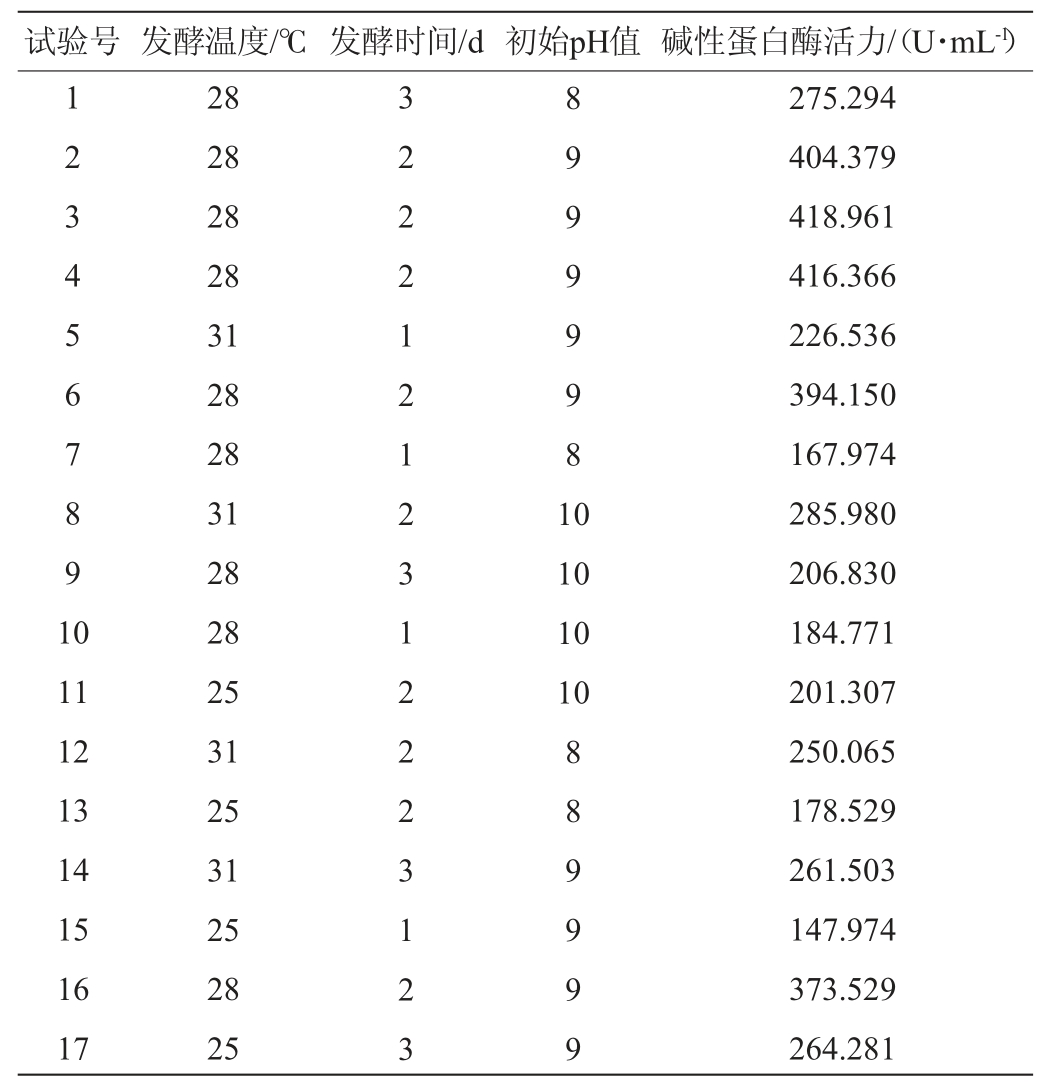
表5 回归模型方差分析
Table 5 Analysis of variance of regression model
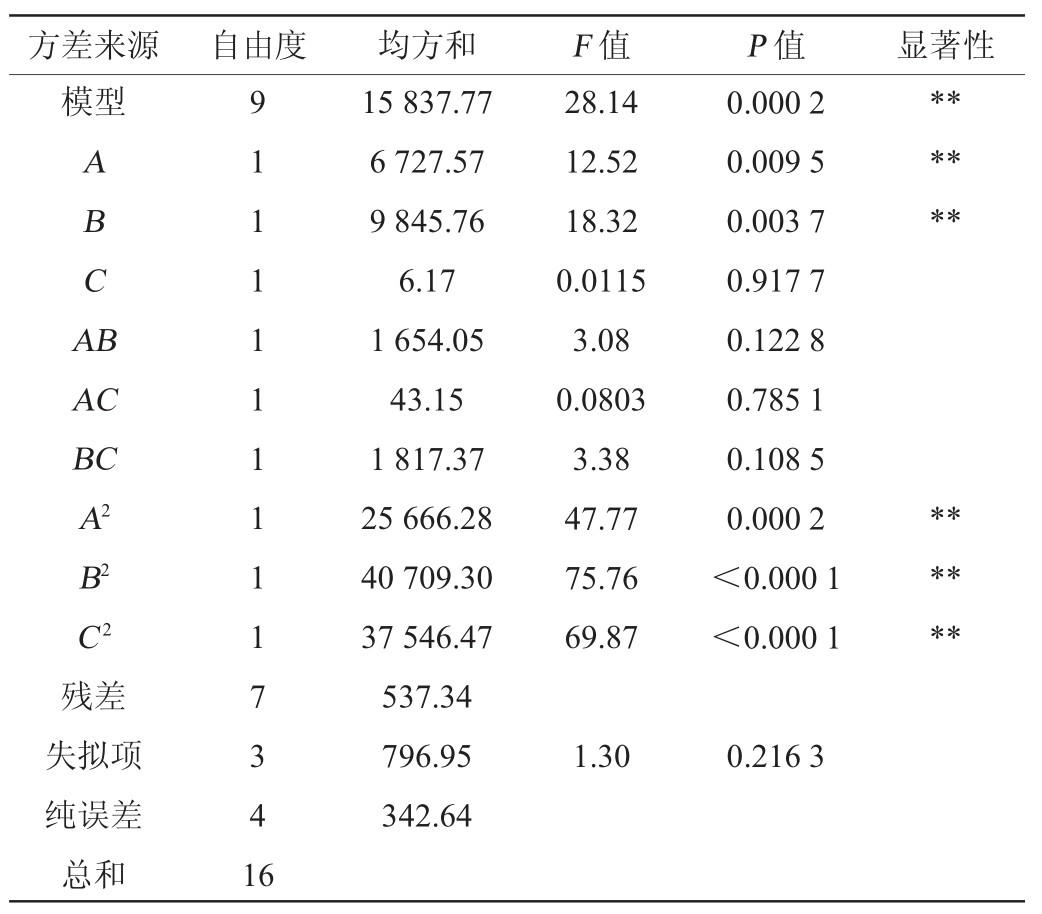
注:“*”表示对结果影响显著(P<0.05);“**”表示对结果影响极显著(P<0.01)。
由表5可知,模型P值=0.000 2<0.01,极显著,失拟项P值=0.390 3>0.05,不显著,决定系数R2=0.967 1,说明模型预测值与实际值的拟合度较高,适用于菌株PC9发酵产蛋白酶的预测分析。由P值可知,一次项A、B、二次项A2、B2、C2对结果影响极显著(P<0.01),其余项对结果影响不显著(P>0.05)。
利用Design-Expert 10.0.4软件,分析3种显著性因素发酵温度、发酵时间、初始pH的响应面和等高线,对回归方程进行求解,进行二次线性回归拟合,得到回归方程:Y=401.48+29A+35.08B+0.8783C-20.34AB+3.28AC-21.32BC-78.08A2-98.33B2-94.43C2。
得到最佳产酶条件为:发酵温度28.48 ℃、发酵时间2.2 d、初始pH8.9,此优化条件下模型预测蛋白酶活力理论值为411.254 U/mL。为了便于实际操作,将最佳产酶条件修正为:发酵温度28.5 ℃、发酵时间2.0 d、初始pH 9.0。为验证模型的准确性及重复性,利用上述最佳工艺参数进行3次平行验证试验,蛋白酶活力实际值为404.153 U/mL,与理论值接近,表明该模型可信度较高,能够很好预测莫海威芽孢杆菌PC9产蛋白酶实际情况。
3 结论
本研究采用宏基因组测序技术解析贵州传统甜面酱保温阶段不同发酵时期的微生物多样性。真菌菌群中在发酵前期和末期的优势菌群为曲霉属(Aspergillus),在发酵中期接合酵母属(Zygosaccharomyces)成为优势菌群,发酵前、中、末期的优势细菌分别为芽孢杆菌属(Bacillus)、拟杆菌属(Bacteroides)、假单胞菌属(Pseudomonas)。在传统发酵甜面酱中分离获得一株莫海威芽孢杆菌(Bacillus mojavensis),其最佳产酶条件为1%果糖,1%酵母浸膏,0.5%硫酸铵,1%NaCl,发酵温度28.5 ℃,发酵时间2.0 d、初始pH 9.0、接种量6%。在此最优条件下,菌株PC9产碱性蛋白酶活力为404.153 U/mL,是优化前的8.42倍。
本研究揭示了参与传统甜面酱自然发酵过程中的微生物群落结构及其动态变化规律,可以确定不同阶段传统甜面酱中优势菌属,为工业制得高品质甜面酱发酵条件的控制及开发利用微生物资源提供依据,探究了莫海威芽孢杆菌PC9的产蛋白酶条件,但对于其他水解酶活性还有待研究。根据现有研究报道,具有一定的安全性,且在食品中具有一定的应用价值,因此菌株PC9在发酵食品中有作为发酵菌种和益生菌剂潜力,为菌种资源及酶制剂的开发提供基础。
[1]叶碧霞,左勇,赖琳,等.甜面酱保温发酵过程中微生物的研究[J].中国调味品,2017,42(4):80-84.
[2]周袁璐,师苑,陆跃乐,等.传统发酵过程微生物互作研究进展[J].中国食品学报,2021,21(11):349-358.
[3]刘巧,罗强,张明,等.利用16S rDNA分析不同地区传统发酵泡菜的细菌多样性[J].食品与发酵工业,2020,46(22):91-97.
[4]侯强川,王玉荣,王文平,等.茅台和尧治河高温大曲细菌群落结构差异及功能预测[J].食品与发酵工业,2022,48(1):36-44.
[5]CAI W,TANG F,WANG Y,et al.Bacterial diversity and flavor profile of Zha-Chili,a traditional fermented food in China[J].Food Res Int,2021,141:110112.
[6]左勇,叶碧霞,杨小龙,等.甜面酱风味物质的研究进展[J].中国调味品,2016,41(2):139-142.
[7]李绮雯,邓毛程,李静,等.豆豉中蛋白酶高产菌筛选及产酶条件的研究[J].广东轻工职业技术学院学报,2021,20(1):1-6.
[8]王沛.甜面酱的风味提升工艺研究[D].天津:天津科技大学,2020.
[9]谢光杰,叶碧霞,左勇,等.甜面酱中黑曲霉的分离及制曲条件的优化[J].中国调味品,2017,42(7):76-81.
[10]万宁威,雷帮星,李彪,等.白酸汤发酵过程中微生物多样性及有机酸动态变化研究[J].食品与发酵工业,2022,48(9):65-70.
[11]MANCABELLI L,MILANI C,LUGLI G A,et al.The impact of primer design on amplicon-based metagenomic profiling accuracy:detailed insights into bifidobacterial community structure[J].Microorganisms,2020,8(1):131.
[12]宫路路,赵志敏,杨颜,等.丹江镇辣椒红酸汤细菌菌群多样性分析及抗食源性致病菌乳酸菌筛选[J].中国酿造,2022,41(9):93-98.
[13]张慧林,王永胜,李冲伟.传统发酵豆酱的微生物群落结构和游离氨基酸组成及其相关性分析[J].食品科学,2019,40(14):192-197.
[14]罗爱国,王家彦,郝建伟,等.山西老陈醋丟糟微生物群落多样性分析[J].中国调味品,2022,47(9):53-58.
[15]QUAST C,PRUESSE E,YILMAZ P,et al.The SILVA ribosomal RNA gene database project: improved data processing and web-based tools[J].Nucleic Acids Res,2013,41:590-596.
[16]任菲,刘玉春,王超,等.高效降解玉米蛋白粉益生菌筛选及生长特性研究[J].粮油食品科技,2021,29(3):183-191.
[17]BUCHANAN R E,GIBBONS N E.伯杰细菌鉴定手册[M].北京:科学出版社,1984:729-758.
[18]东秀珠,等.常见细菌系统鉴定手册[M].北京:科学出版社,2001:62-65.
[19]王屈祎,钟倩,闫如玉,等.淀粉酶产生菌的筛选、鉴定及其发酵条件优化[J].微生物学通报,2022,49(1):173-188.
[20]张宸瑞,薛桥丽,白彬阳,等.云南牟定酸浆水中优势产酸菌的分离鉴定及生长特性[J].食品科学,2021,42(20):75-82.
[21]侯泽林.从土壤中筛选碱性蛋白酶产生菌及产酶条件优化研究[D].哈尔滨:东北农业大学,2021.
[22]余丹,毛娉,宋颀,等.基于高通量测序的传统甜面酱自然发酵过程中的微生物群落结构及其动态演替[J].微生物学通报,2018,45(5):1061-1072.
[23]沈弘洋,邓微,赵云珠,等.传统大豆酱不同发酵阶段微生物多样性变化[J].食品与发酵工业,2021,47(23):118-124.
[24]董玺梅,阮志强,蒋雪薇.酱香风味传统发酵食品中微生物与风味形成的相关性[J].中国食品学报,2022,22(7):397-406.
[25]董蕴,张苗苗,邓子文,等.基于Illumina MiSeq高通量测序技术解析利川酱豆真菌多样性[J].中国酿造,2022,41(4):137-141.
[26]MAO P,HU Y,LIAO T,et al.Microbial diversity during fermentation of sweet paste,a Chinese traditional seasoning,using PCR-denaturing gradient gel electrophoresis[J].J Microbiol Biotechn,2017,27(4):678-684.
[27]AN F,LI M,ZHAO Y,et al.Metatranscriptome-based investigation of flavor-producing core microbiota in different fermentation stages of dajiang,a traditional fermented soybean paste of Northeast China[J].Food Chem,2021,343:128509.
[28]赵文鹏,李浩,杨慧林,等.曲霉型豆豉发酵阶段细菌群落的演替及其与环境因子的关系[J].食品科学,2021,42(4):138-144.
[29]吴振超.洛氏鱥肠道产酶益生菌的筛选及其粘附特性分析[D].长春:吉林农业大学,2020.
[30]檀迎会.层出镰孢拮抗菌的分离筛选及发酵条件优化[D].保定:河北农业大学,2015.
[31]冯志彬,薛钰,陈国忠,等.1株产壳聚糖酶细菌的分离、鉴定和发酵条件优化[J].食品科学,2016,37(19):171-176.
[32]DIABANKANA R G C,AFORDOANYI D M,SAFIN R I,et al.Antifungal properties,abiotic stress resistance,and biocontrol ability of Bacillus mojavensis PS17[J].Curr Microbiol,2021,78(8):3124-3132.
[33]ADIGÜZEL A O.Production and characterization of thermo-,halo-and solvent-stable esterase from Bacillus mojavensis TH309[J].Biocatal Biotransfor,2020,38(3):210-226.
[34]LIAQAT F,SÖZER B P,ELIBOL M,et al.Optimization of chitosanase production by Bacillus mojavensis EGE-B-5.2i[J].J Basic Microb,2018,58(10):836-847.
[35]ANISSA H,AMIR B,RIM M,et al.Rheological and thermal properties of levan from Bacillus mojavensis[J].J Polym Environ,2022,30:741-751.
[36]AMAL H,NAHED F,OLA A,et al.Proteolytic and amylolytic enzymes from a newly isolated Bacillus mojavensis SA:Characterization and applications as laundry detergent additive and in leather processing[J].Int J Biol Macromol,2021,248(30):745-751.
[37]刘亚楠,彭丹丹,王敏,等.枯草芽孢杆菌Q125降解黄曲霉毒素B1发酵条件优化及活性物质分析[J].河南工业大学学报(自然科学版),2021,42(4):9-15.
[38]冒鑫哲,彭政,周冠宇,等.枯草芽孢杆菌高产角蛋白酶发酵条件优化[J].食品与发酵工业,2020,46(17):138-144.
[39]李文静,李春生,李来好,等.鱼露中高产蛋白酶耐盐菌株的筛选、鉴定及产酶条件优化[J].食品与发酵工业,2021,47(23):134-142.