谷氨酸棒杆菌(Corynebacterium glutamicum)作为一种新型的底盘菌株和异源蛋白表达宿主,是一种绿色高效的底盘微生物,可用于生产大宗氨基酸、精细化学品、营养保健品等众多高附加值产品,创造巨大的经济价值[1-2]。传统的菌株改良采用随机诱变的方式,在菌种发育过程中会产生生长缺陷、低耐受性或积累副产物等问题[3]。基因组编辑技术作为分子生物学的重要技术之一,是一种有效的调控菌株代谢网络的手段,在谷氨酸棒杆菌合成生物学研究和生产菌株改造等方面发挥巨大潜力[4]。谷氨酸棒杆菌的基因修饰最早可追溯到1984年[5],早期通过脱氧核糖核酸(deoxyribonucleic acid,DNA)重组的方式,为生物进化创造条件,促进了新物种的产生和发展。
近年来,新兴的规律成簇的间隔短回文重复(clustered regularly interspaced short palindromic repeats,CRISPR)技术是一种革命性的基因编辑工具,可对目标序列进行识别,完成DNA双链断裂及原生DNA修复等操作。CRISPR及其相关的蛋白(CRISPR-associated proteins,Cas)系统已成功应用于大肠杆菌(Escherichia coli)、人类、鼠等物种的基因工程改造[6]。随着分子遗传学和全基因组测序的发展,应用理性代谢工程的方法,借助基因编辑技术,对菌株基因组中的特定基因进行修饰,以提高菌株代谢物的产量。然而,谷氨酸棒杆菌的遗传操作仍存在一些挑战[7-8],远远落后于大肠杆菌。本文综述了谷氨酸棒杆菌常用基因编辑技术,详细阐述了传统基因编辑技术中基于等位基因交换系统和Cre/loxP位点特异性重组系统、CRISPR/Cas介导的基因编辑技术中CRISPR/Cas9系统和CRISPR/Cpf1系统及CRISPRi技术的作用机制及其在谷氨酸棒杆菌中的应用,旨在为基因编辑技术在谷氨酸棒杆菌菌株设计和生产高附加值产品方面提供依据,对目标菌株进行代谢工程定向改造对大规模生产附加值产品具有重要的意义。
1 传统基因编辑技术的作用机制及应用
谷氨酸棒杆菌的传统基因编辑技术主要包括基于等位基因交换系统和Cre/loxP位点特异性重组系统(见图1)。基于等位基因交换系统的特点主要依赖于同源重组,将含有目的基因同源序列的外源基因导入宿主菌,借助筛选标记进行反向筛选,通过等位替换的方式,实现基因敲除、定点突变和基因整合,筛选获得谷氨酸棒杆菌突变体[9];Cre/loxP系统则在谷氨酸棒杆菌中主要用于基因敲除,该系统的特点也是基于自杀质粒,通过Cre催化两个loxP的重组,但其需要经历两轮的同源重组导致获得重组体的时间较长[10]。目前,基于等位基因交换的pk18mobsacB系统依然是常用的菌株改造工具。
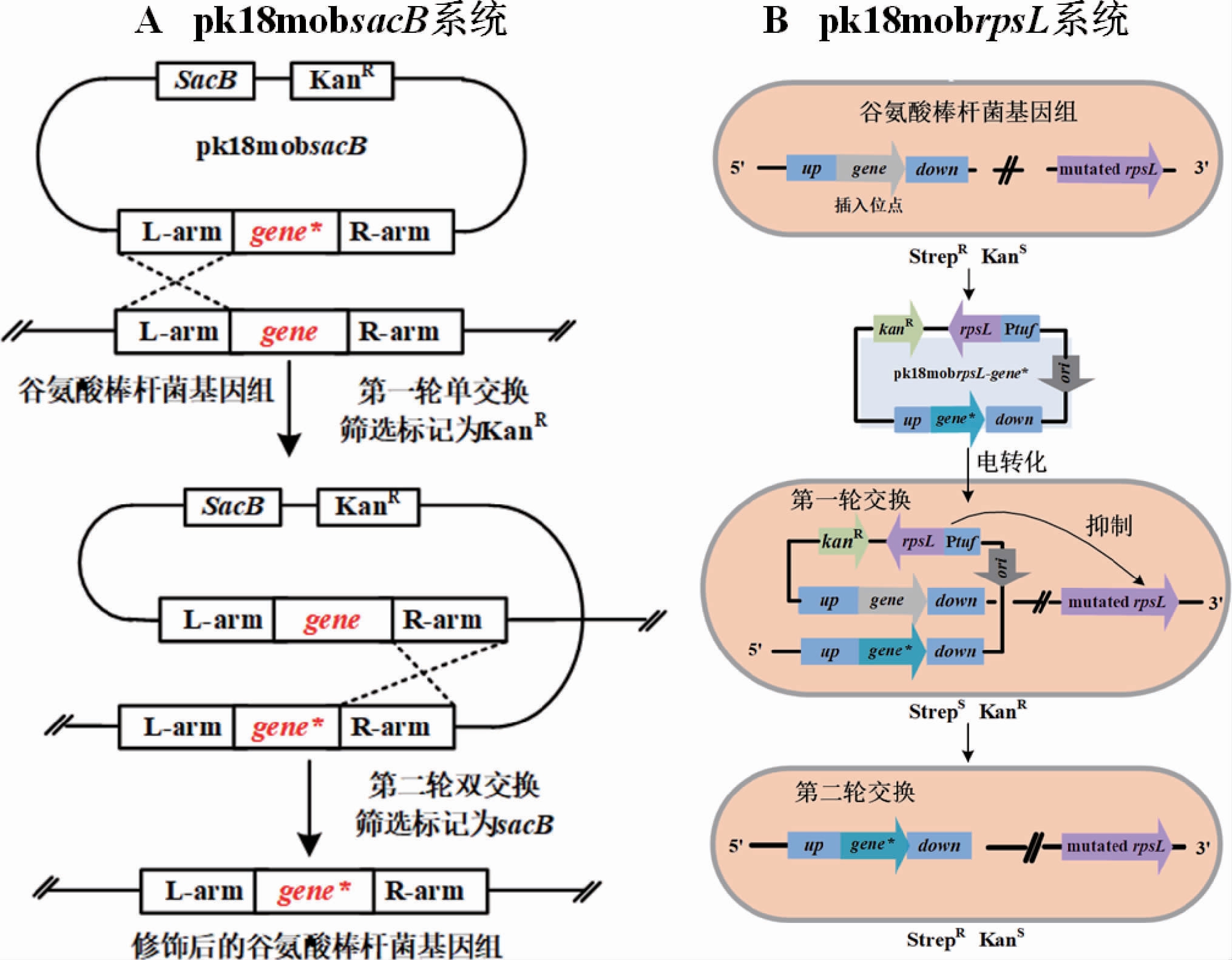
图1 基于等位基因交换的传统基因编辑系统示意图
Fig.1 Schematic diagram of traditional gene editing system based on allele exchange
A为pk18mobsacB系统在编辑位点引入KanR选择标记和sacB反向筛选标记,经两轮同源重组的编辑过程;B为pk18mobrpsL系统包涵KanR和野生型rpsL表达盒,用于构建基因编辑的重组质粒;gene*表示插入基因。
1.1 pk18mobsacB系统
pk18mobsacB系统是由自杀质粒介导的二次交换同源重组,通过核酸链间等位替换的方式对目的基因修饰。该系统的sacB基因作为筛选标记,可将蔗糖转化为有毒代谢物,对菌株产生蔗糖致死作用。当第一次交换时,整个质粒插入基因组,菌体显示为卡那霉素抗性(KanR);当第二次交换时,插入菌体基因组上的KanR和sacB被移除,因此蔗糖致死作用消失,之后就开始用sacB作为筛选标记(图1A)。有研究者利用该系统对六个涉及草酰乙酸和α-酮戊二酸分支的基因过表达或基因敲除,通过动态调节优化三羧酸(tricarboxylic acid,TCA)循环和碳通量,降低了副产物的合成,获得了高产4-羟基异亮氨酸(34.21 g/L)的菌株[11]。虽然,pk18mobsacB系统在实际应用中存在回复突变频率高、sacB易失活导致转化子筛选耗时长、编辑效率低、遗传操作复杂等问题,在一定程度上限制了谷氨酸棒杆菌代谢工程的改造进度,但该系统目前依然是研究者们常用的菌株改造手段[12]。
1.2 pk18mobrpsL系统
为了解决pk18mobsacB系统的诸多问题,在前期研究中作者团队对传统的pk18mobsacB系统进行了优化,寻找到了另一种可替代sacB且更严格的筛选标记——rpsL基因[13]。rpsL的突变基因可使谷氨酸棒杆菌对链霉素产生耐药性,研究结果显示采用rpsL标记和链霉素用于反向筛选是高效的,且自发突变频率较小,成功构建了一种新型的谷氨酸棒杆菌反向筛选基因组编辑方法——pk18mobrpsL系统(图1B)。该系统借助强大的遗传筛选标记rpsL,将菌株两步验证程序简化为一步验证,并通过优化筛选双交换菌的培养方式,即采用先在含有卡那霉素的脑心浸出液肉汤(brain heart infusion broth,BHIB)摇管中培养,再涂布培养于含有链霉素的脑心浸液琼脂(brain heart infusion agar,BHIA)平板的方法显示编辑效率为100%。此后,团队成员利用pk18mobrpsL系统筛选到一株高产L-茶氨酸的谷氨酸棒杆菌。为了促进菌株L-茶氨酸的合成,分别在野生型C.glutamicum ATCC 13032和L-谷氨酸生产菌株GDK-9中过表达了不同类型的γ-谷氨基甲酰胺合成酶(γ-glutamylmethylamide synthetase,GMAS),然后,敲除基因Ncgl1221阻断了宿主细胞中L-谷氨酸的输出,使GDK-9菌株细胞中L-茶氨酸含量显著升高,最终获得了产量为42 g/L的L-茶氨酸工程菌株[14]。可见,优化后的pk18mobrpsL系统显著提高了反向筛选基因组编辑技术的筛选效率,摒弃了sacB系统进行两轮双交换对点平板和菌落聚合酶链式反应(polymerase chain reaction,PCR)验证的方法,使链霉素筛选成为了一种快速高效的方法,简化了操作,提高了编辑效率,缩短了筛选目的菌株的时间。
1.3 Cre/loxP位点特异性重组系统
Cre/loxP特异位点重组系统常用于目的基因的表达和无痕敲除[15]。环化重组酶(cyclization recombination enzyme,Cre)是一种可逆性重组酶,loxP是一段34 bp的碱基序列。Cre可识别loxP位置,将两个loxP位点之间的DNA切除,把DNA二聚体分解成单体[16-17],进行两轮同源重组完成基因组修饰。
为了提高重组效率,有研究利用RecET介导Cre/loxP系统,提高了C.glutamicum ATCC 14067菌株基因编辑效率,通过一轮重组即可获得目标突变体[18];之后研究发现基因组重排是一项越来越重要的技术,有助于理解基因组的功能,研究者们通过对比分析C.glutamicum R和C.glutamicum ATCC 13032基因组,识别出了与细胞生存无关的14.5 kb和56 kb区域,并利用建立的谷氨酸棒杆菌基因组大规模重排的Cre/loxP基因敲除系统对该区域进行了删除,通过同源重组技术将loxP位点整合在靶区的两端,在Cre的作用下两个染色体loxP位点的基因敲除效率很高[17];此外,有研究者利用建立的RecET-Cre/loxP系统对C.glutamicum ATCC 14067进行了启动子替换,增强了菌株L-亮氨酸合成能力[19]。鉴于Cre/loxP系统的第二轮重组突变体的筛选耗时较长,且Cre特异性识别两个loxP序列之间DNA剪切是可逆的。所以,这种方法的基因效率不高,也存在外源基因残留等问题。
2 CRISPR/Cas基因编辑技术的作用机制及应用
在CRISPR/Cas系统的基因编辑过程中,CRISPR先被转录成核糖核酸(ribonucleic acid,RNA),然后变成更短的CRISPR RNA(crRNAs),后者引导Cas蛋白的活性,切割目的DNA[20]。该系统的遗传组件包括一个核酸内切酶、一个靶向RNA表达元件和一个供体DNA分子。当RNA与核酸内切酶(Cas9或Cpf1等)结合后靶向基因组目标位点,实现DNA断裂,然后以供体DNA分子为模板对基因组修复[21]。CRISPR/Cas9系统和CRISPR/Cpf1系统作为较简单的CRISPR/Cas系统,已被成功应用于多种微生物的基因组编辑[22]。然而,CRISPR技术在谷氨酸棒杆菌中的应用仍存在基因编辑效率不稳定和筛选效率低等问题,科研工作者投入了大量的人力物力进行研发改进,部分研究进展结果见表1。
表1 谷氨酸棒杆菌CRISPR/Cas系统基因编辑研究进展
Table 1 Research progress on CRISPR/Cas system gene editing of Corynebacterium glutamicum

2.1 CRISPR/Cas9系统
来自化脓性链球菌(Streptococcus pyogenes)II型的CRISPR/Cas9系统存在一些缺点与应用限制:对于那些离染色体最近NGG(PAM序列)[21](>20 bp)以外区域的无法进行基因修饰;由于靠近protospacer 5'端的核苷酸发生突变,使编辑效率大幅度降低。CRISPR/Cas9系统作为最简单的CRISPR系统,对菌株遗传操作产生了革命性的影响[26]。到目前为止,CRISPR/Cas9系统仅在部分菌株改造中被采用,而在谷氨酸棒杆菌中的高效应用却鲜有报道。
CHO J S等[21]指出Cas9蛋白对谷氨酸棒杆菌产生毒性,原因是由于谷氨酸棒杆菌基因组中缺少一种SpCas9典型的crRNA序列,该研究通过Cas9蛋白密码子优化,解决了菌株细胞的毒性作用。有研究者将Cas9和gRNA质粒共表达转化,解决了Cas9突变产生毒性的问题。并将DNA供体置于载体提高重组效率,基因敲除和基因整合的效率达到30.8%~60.0%与16.7%~62.5%,同时,研究的单链ssDNA编辑效率可达80.0%以上[23];白仲虎等[24]开发了双质粒介导的CRISPR/Cas9系统,两个质粒分别含有sgRNA-donorDNA与Cas9蛋白,并验证了该系统对porB等基因表达的干扰能力,实验结果显示编辑效率为100%;鉴于基因重组是提高编辑效率的关键因素,WANG B等[25]为了增加重组效率,在谷氨酸棒杆菌基因组中插入Cas9-RecET蛋白,这种方法实现了大片段10~20 kb的基因敲除和2.5~7.5 kb的基因插入。
2.2 CRISPR/Cpf1系统
二类V型的CRISPR/Cas系统拥有一个源自新凶手弗朗西斯菌(Francisella novicida)(Fn)的Cpf1大蛋白(1 300个氨基酸)。与Cas9相比,Cpf1具有更强的靶向特异性,可以作为Cas9基因组编辑的替代选择。Cpf1是一个缺乏tracrRNA的单链RNA引导内切酶,利用富含T的默认突变几率(percent accepted mutation,PAM),在末端交错切割,使DNA双链断裂[29]。由于Cas9蛋白对细胞产生毒性作用,谷氨酸棒杆菌的基因组编辑并不能简单地用Cas9或dCas9来实现。
鉴于Cpf1蛋白的优势,JIANG Y等[26]成功构建了Allin-one单质粒的CRISPR/Cpf1系统,当利用单链ssDNA重组时,微小DNA修饰的效率为86%~100%。当没有重组酶时,大片段DNA的基因编辑效率为5%~15%;此后,有研究者利用CRISPR/Cpf1单链DNA重组,编辑效率为86%~100%。虽然进行了一系列参数优化(PAM序列、间隔序列、修复模板),但该系统缺失重组酶的相关研究,编辑效率仅在有限的范围内得到了提升;有研究针对C.glutamicum ATCC 14067菌株,通过优化PAM序列和可编辑位点范围区域,开发了一种多重基因组同步编辑策略的CRISPR/Cpf1-RecT系统,结果可同时编辑3个基因,2个基因时的编辑效率达到93.6%,利用CRISPR/Cpf1-RecET系统进行大片段DNA敲除,敲除基因组中1 kb、5 kb和20 kb DNA片段的效率分别为79.6%、91.3%和36.4%[27]。鉴于CRISPR/Cpf1系统更加精准的编辑效果,基本没有脱靶效应,显示其在微生物合成生物学领域的应用潜力。
3 CRISPR干扰技术的作用机制及应用
CRISPR干扰(CRISPR interference,CRISPRi)技术采用缺乏核酸酶活性的Cas9或Cpf1失活蛋白(dCas9或dCpf1),是CRISPR/Cas9或CRISPR/Cpf1系统的失活版本。例如,与活跃的Cas9核酸酶不同,dCas9仍然保留了与sgRNA复合体的能力,可结合到同源位点。只是该复合体没有切割DNA的能力,而是在空间上阻止RNA聚合酶的进程,最终导致转录抑制,此过程不会引入永久性的DNA编码突变,可通过关闭CRISPRi系统逆转其所诱导的表型突变,从而实现基因组调控[30]。为了增加目的产物的通量,传统基因敲除方法是中断消耗中间体到最终产品的途径[31]。由于细胞代谢的复杂性,需要对多个代谢工程目标进行操作,以平衡代谢通量并最大化所需产物的生物合成[23]。然而,连续删除多个基因不仅费时费力,而且对于那些与细胞生长和代谢密切相关的基因,基因敲除并不是最好的选择。因此,利用CRISPRi系统作为一种替代方法优化代谢途径,进行基因转录抑制,成为平衡细胞生长和产物合成的首选方法[32]。
目前,谷氨酸棒杆菌中CRISPRi/dCas9介导的单基因转录抑制[33-36]和CRISPRi/dCpf1介导的多基因下调已经实现[31,37],但几乎没有关于转录激活的报道。WANG Q Z等[8,21]将dCas9和sgRNA载体引入谷氨酸棒杆菌,通过CRISPRi的空间位阻效应调控特定基因的转录水平,快速弱化目标基因,利用基因下调,避免了基因失活,高效获得生产菌株;也有研究者利用CRISPRi/dCas9系统,分别使pgi和pck两个基因的表达量降低了98%,同时,pyk基因的表达量降低了97%,使L-赖氨酸和L-谷氨酸的产量的提高率与基因缺失水平相当[38],但该研究中dCas9和sgRNA载体仍然保留在生产菌株中,限制了菌体细胞兼容其他载体的数量。LIU J等[23]研究揭示了组成型表达dCas9是不可行的,利用异丙基-β-D-硫代半乳糖苷(isopropyl-β-D-thiogalactoside,IPTG)诱导的启动子Ptac和丙酸盐诱导的启动子PprpD2表达dCas9,揭示了Ptac表达gRNA可促进谷氨酸棒杆菌CRISPRi系统干扰作用;进一步有研究构建了双质粒CRISPRi载体,成功对4个单基因(pyc、gltA、idsA、glgC)和双基因(idsA、glgC)进行干扰,通过降低靶位点柠檬酸合成酶活性,使L-赖氨酸产量提高了1.39倍[39];有研究构建了单质粒和LacI/TetR为基础的双诱导的单基因和多基因靶向CRISPRi系统,包含由一四环素ATc诱导的tetR/tetA启动子,及由IPTG诱导的tac启动子控制的sgRNA-cs,有效下调L-精氨酸合成途径中argH和pgi基因[40];为了提高编辑效率,有研究在CRISPRi/dCpf1系统中,优化了dCpf1(E1006A,D917A)和单crRNA阵列,建立了可同时抑制多个目的基因表达CRISPRi干扰系统,该系统用单一阵列抑制了与赖氨酸生物合成有关的4个基因(gltA、pck、pgi和hom),使赖氨酸滴度和产量提高了约4.0倍,转录均被抑制约90%。因此,CRISPRi系统作为一种干扰技术,提供了调控特定基因表达水平的方法,是一种非常适合用于多基因原核代谢工程的工具。
4 结语
目前,谷氨酸棒杆菌最常用的编辑技术依然是基于等位基因单双交换的传统基因编辑系统,尽管取得了较大改进,但与大肠杆菌相比,谷氨酸棒杆菌的基因改造方法非常有限,研发工作相对滞后,仍存在一些缺陷,如pk18mobsacB技术也存在sacB易失活、筛选假阳性高及筛选过程费时低效等诸多问题。正确基因编辑效率并不高,不能用于大片段基因组整合,研究者们实验结果差异较大且不能重复等问题,限制了CRISPR技术的应用。因此,亟待开发高效便捷的基因编辑方法,用于谷氨酸棒杆菌的代谢工程改造。
[1]谭海,顾阳,卢南巡,等.代谢工程改造谷氨酸棒状杆菌促进L-异亮氨酸发酵合成的研究进展[J].中国酿造,2021,40(9):1-6.
[2]王婷,蔡柠匀,张德志,等.高产L-精氨酸谷氨酸棒状杆菌的构建[J].中国酿造,2019,38(5):169-173.
[3]VERTÈS A A,INUI M,YUKAWA H.Manipulating Corynebacteria,from individual genes to chromosomes[J].Appl Environ Microbiol,2005,71(12):7633-7642.
[4]王婷,马洪坤,赵桂红,等.谷氨酸棒杆菌CRISPR-Cpf1/ssDNA基因组编辑系统优化[J].食品与发酵工业,2019,45(19):1-7.
[5]OZAKI A,KATSUMATA R,OKA T,et al.Functional expression of the genes of Escherichia coli in gram-positive Corynebacterium glutamicum[J].Mol Gen Genet,1984,196:175-178.
[6]ZHAO D,XU F,ZHU X N,et al.CRISPR/Cas9-assisted gRNA-free onestep genome editing with no sequence limitations and improved targeting efficiency[J].Sci Rep,2017,7(1):1-9.
[7]杨娟娟,马晓雨,王晓蕊,等.谷氨酸棒杆菌基因编辑的研究进展[J].生物工程学报,2020,36(5):820-828.
[8]WANG Q Z,ZHANG J,AL MAKISHAH N H,et al.Advances and perspectives for genome editing tools of Corynebacterium glutamicum[J].Front Microbiol,2021,12:654058.
[9]SCHÄFER A,TAUCH A,JÄGER W,et al.Small mobilizable multi-purpose cloning vectors derived from the Escherichia coli plasmids pk18 and pk19:selection of defined deletions in the chromosome of Corynebacterium glutamicum[J].Gene,1994,145(1):69-73.
[10]李涛,卢圣栋.Cre/loxP系统与时空特异性基因打靶[J].国际遗传学杂志,2001,24(4):177-180.
[11]ZHANG C L,LI Y J,MA J,et al.High production of 4-hydroxyisoleucine in Corynebacterium glutamicum by multistep metabolic engineering[J].Metab Eng,2018,49:287-298.
[12]SUZUKI N,INUI M.Genome engineering of Corynebacterium glutamicum[M].Berlin:Springer,2013:89-105.
[13]WANG T,LI Y J,LI J,et al.An update of the suicide plasmid-mediated genome editing system in Corynebacterium glutamicum[J].Microb Biotechnol,2019,12(5):907-919.
[14]MA H K,FAN X G,CAI N Y,et al.Efficient fermentative production of L-theanine by Corynebacterium glutamicum[J].Appl Microbiol Biotechnol,2020,104:119-130.
[15]KOS C H.Methods in nutrition science:Cre/loxP system for generating tissue-specific knockout mouse models[J].Nutr Rev,2004,62(6):243-246.
[16]SUZUKI E,NAKAYAMA M.VCre/VloxP and SCre/SloxP: new sitespecific recombination systems for genome engineering[J].Nucleic Acids Res,2011,39(8):e49.
[17]SUZUKI N,TSUGE Y,INUI M,et al.Cre/loxP-mediated deletion system for large genome rearrangements in Corynebacterium glutamicum[J].Appl Microbiol Biotechnol,2005,67(2):225-233.
[18]HUANG Y Y,LI L,XIE S,et al.Recombineering using RecET in Corynebacterium glutamicum ATCC14067 via a self-excisable cassette[J].Sci Rep,2017,7(1):7916.
[19]LUO G J,ZHAO N N,JIANG S B,et al.Application of RecET-Cre/loxP system in Corynebacterium glutamicum ATCC14067 for L-leucine production[J].Biotechnol Lett,2021,43:297-306.
[20]HSU P D,LANDER E S,ZHANG F.Development and applications of CRISPR-Cas9 for genome engineering[J].Cell,2014,157(6):1262-1278.
[21]CHO J S,CHOI K R,PRABOWO C P S,et al.CRISPR/Cas9-coupled recombineering for metabolic engineering of Corynebacterium glutamicum[J].Metab Eng,2017,42:157-167.
[22]CHOI K R,LEE S Y.CRISPR technologies for bacterial systems:current achievements and future directions[J].Biotechnol Adv,2016,34(7):1180-1209.
[23]LIU J,WANG Y,LU Y J,et al.Development of a CRISPR/Cas9 genome editing toolbox for Corynebacterium glutamicum[J].Microb Cell Fact,2017,16(1):1-17.
[24]PENG F,WANG X Y,SUN Y,et al.Efficient gene editing in Corynebacterium glutamicum using the CRISPR/Cas9 system[J].Microb Cell Fact,2017,16(1):1-13.
[25]WANG B,HU Q T,ZHANG Y,et al.A RecET-assisted CRISPR-Cas9 genome editing in Corynebacterium glutamicum[J].Microb Cell Fact,2018,17(1):1-16.
[26]JIANG Y,QIAN F H,YANG J J,et al.CRISPR-Cpf1 assisted genome editing of Corynebacterium glutamicum[J].Nat Commun,2017,8(1):15179.
[27]ZHAO N,LI L,LUO G J,et al.Multiplex gene editing and large DNA fragment deletion by the CRISPR/Cpf1-RecE/T system in Corynebacterium glutamicum[J].J Ind Microbiol Biotechnol,2020,47(8):599-608.
[28]ZHANG J,YANG F Y,YANG Y P,et al.Optimizing a CRISPR-Cpf1-based genome engineering system for Corynebacterium glutamicum[J].Microb Cell Fact,2019,18(1):1-13.
[29]CONG L,RAN F A,COX D,et al.Multiplex genome engineering using CRISPR/Cas systems[J].Science,2013,339(6121):819-823.
[30]NA D,YOO S M,CHUNG H,et al.Metabolic engineering of Escherichia coli using synthetic small regulatory RNAs[J].Nat Biotechnol,2013,31(2):170-174.
[31]LI M Y,CHEN J Z,WANG Y,et al.Efficient multiplex gene repression by CRISPR-dCpf1 in Corynebacterium glutamicum[J].Front Bioeng Biotechnol,2020,8:357.
[32]LV L,REN Y L,CHEN J C,et al.Application of CRISPRi for prokaryotic metabolic engineering involving multiple genes,a case study: Controllable P(3HB-co-4HB)biosynthesis[J].Metab Eng,2015,29:160-168.
[33]LEE S S,SHIN H,JO S,et al.Rapid identification of unknown carboxyl esterase activity in Corynebacterium glutamicum using RNA-guided CRISPR interference[J].Enzyme Microb Technol,2018,114:63-68.
[34]YOON J,WOO H M.CRISPR interference-mediated metabolic engineering of Corynebacterium glutamicum for homo-butyrate production[J].Biotechnol Bioeng,2018,115(8):2067-2074.
[35]ZHANG B,LIU Z Q,LIU C,et al.Application of CRISPRi in Corynebacterium glutamicum for shikimic acid production[J].Biotechnol Lett,2016,38(12):2153-2161.
[36]LI Z,LIU J Z.Transcriptomic changes in response to putrescine production in metabolically engineered Corynebacterium glutamicum[J].Front Microbiol,2017,8:1987.
[37]LIU W,TANG D D,WANG H J,et al.Combined genome editing and transcriptional repression for metabolic pathway engineering in Corynebacterium glutamicum using a catalytically active Cas12a[J].Appl Microbiol Biotechnol,2019,103(21):8911-8922.
[38]CLETO S,JENSEN J V K,WENDISCH V F,et al. Corynebacterium glutamicum metabolic engineering with CRISPR interference(CRISPRi)[J].ACS Synth Biol,2016,5(5):375-385.
[39]PARK J,SHIN H,LEE S M,et al.RNA-guided single/double gene repressions in Corynebacterium glutamicum using an efficient CRISPR interference and its application to industrial strain[J].Microb Cell Factories,2018,17(1):1-10.
[40]GAUTTAM R,SEIBOLD G M,MUELLER P,et al.A simple dual-inducible CRISPR interference system for multiple gene targeting in Corynebacterium glutamicum[J].Plasmid,2019,103:25-35.