作为一种具有地方特色的传统发酵食品,鲊广椒常以玉米粉或大米粉为主要原料,通过添加红辣椒、花椒粉、胡椒粉和盐等辅料,放于陶罐中密封发酵而成。经焙熟后的鲊广椒常被用作蒸肉、扣肉的底料或伴料,可使做出来的蒸肉不油腻,爽口且易消化吸收[1]。CAI W C等[2]采用Illumina MiSeq高通量测序技术对重庆、湖北和湖南地区鲊广椒的细菌多样性进行分析,发现乳酸杆菌属(Lactobacillus)在所有鲊广椒样品中均存在,且含量高达74.17%。在湖北省当阳市和恩施土家族苗族自治州的鲊广椒样品中,ZHANG Z D等[3-4]分别研究发现并命名了鲊广椒乳杆菌(L.zhachilii)和恩施乳杆菌(L.enshiensis)2个乳酸杆菌新物种。由此可见,鲊广椒中蕴含的乳酸杆菌比较丰富且独特。湖北省恩施土家族苗族自治州巴东县,境内山峦起伏,峡谷幽深,光、热、水分布垂直差异明显,其土家族和苗族等少数民族人口占全县常住人口的半数以上,当地人民具有制作并食用鲊广椒的饮食习惯,然而目前关于巴东地区鲊广椒细菌多样性分析及乳酸菌分离鉴定的研究报道尚少。
通过实现对数百万个脱氧核糖核酸(deoxyribo nucleic acid,DNA)分子同时进行测序,Illumina MiSeq高通量测序技术具有测序通量大、精确度高和信息量丰富等优点[5],实现了对样本中微生物类群多样性的有效解析。通过采用Illumina MiSeq高通量测序和传统微生物纯培养手段相结合的方法,GUO Z等[6]解析了湖南省怀化地区和贵州省松桃地区鲊广椒细菌的多样性并对其蕴含的乳酸菌资源进行了挖掘,结果发现在所有鲊广椒样品中共存在4个优势细菌门和12个优势细菌属,虽然经纯培养分离到的乳酸菌不如高通量测序丰富,但其可在种水平上鉴定优势分类群。将Illumina MiSeq高通量测序技术与传统纯培养技术相结合用于鲊广椒中细菌类群的解析是可行和具有一定优势的。
本研究采用高通量测序技术对湖北恩施巴东地区鲊广椒的细菌类群进行解析,并对其细菌的基因功能进行了预测,同时采用纯培养方法对其蕴含的乳酸菌菌株进行了分离、鉴定和保存,通过本研究的开展以期为后续鲊广椒的工业化生产及相关研究提供菌种支持。
1 材料与方法
1.1 材料与试剂
1.1.1 材料
14个鲊广椒样品:均采自湖北省恩施土家族苗族自治州巴东县(位于东经110°04′~110°32′,北纬30°28′~31°28′之间)的张富菜市场和大顺批零菜市场,记为Z1~Z14。采集时间为2018年11月下旬,所有样品均以当地自产玉米和辣椒为主要原料制作,无霉斑,无异味,发酵时间为20~25 d。样品采集时,从鲊广椒发酵坛中取出约500 g的样品,放于20 cm×20 cm的自封袋中密封,并于低温条件下送回实验室立即进行乳酸菌菌株的分离。
1.1.2 试剂
十六烷基三甲基溴化铵、乙酸钠酚、氯仿、异戊醇、乙醇(均为分析纯):国药集团化学试剂有限公司;AXGEN聚合酶链式反应(polymerase chain reaction,PCR)清洁试剂盒:北京科博汇智生物科技发展有限公司;引物27F/1495R和M13F(-47)/M13R(-48):武汉天一辉远生物科技有限公司;QIAGEN DNeasy maricon Food Kit提取试剂盒:德国Qiagen公司;PCR buffer、脱氧核糖核苷三磷酸(deoxy-ribonucleoside triphosphate,dNTPs)Mix、DNA聚合酶、缓冲溶液I、pMD18-T载体、10×PCR缓冲液和上样缓冲液(Loading buffer):宝生物工程(大连)有限公司;大肠杆菌(Escherichia coli)top10:湖北省食品配料工程技术研究中心制作并保存;Illumina MiSeq测序配套试剂:美国Illumina公司。
1.1.3 培养基
MRS培养基、LB培养基:青岛海博生物技术有限公司。
1.2 仪器与设备
Veriti FAST梯度PCR仪:美国ABI公司;UVPCDS8000凝胶成像分析系统:美国Protein Simple公司;DG250厌氧工作站:英国Don Whitley公司;MiSeq PE250高通量测序平台:美国Illumina公司;R920机架式服务器:美国DELL公司。
1.3 实验方法
1.3.1 宏基因组DNA提取、PCR扩增和MiSeq高通量测序
使用试剂盒提取鲊广椒中微生物的宏基因组DNA,参考CHEN Y等[7]的方法进行16S rRNA V3-V4区聚合酶链式反应(PCR)扩增,并将通过1.0%琼脂糖凝胶电泳检测的扩增产物寄往上海美吉生物医药科技有限公司进行高通量测序。
1.3.2 生物信息学分析
参考郭壮等[8]的方法去除序列中的核苷酸标签与引物,使用QIIME分析平台[9]对原始序列进行PyNAST对齐[10],分别在100%和97%相似度条件下进行分类操作单元(operational taxonomic units,OTU)的划分[11]。使用ChimeraSlayer检查去除含有嵌合体的OTU[12],通过与Greengene[13]、核糖体数据库(ribosomal database project,RDP)[14]和SILVE[15]数据库比对明确各分类学地位。在使用FastTree软件构建系统发育树的基础上计算β和α多样性,最后使用功能预测软件PICRUSt预测巴东鲊广椒中细菌的基因功能[16],并参照蛋白质直系同源簇(clusters of orthologous groups of proteins,COGs)数据库注释样品中细菌的菌群功能[17]。
1.3.3 乳酸菌的分离鉴定
参考李娜等[18]的方法对巴东地区鲊广椒中乳酸菌进行分离、纯化和保藏。参考PEREIRA G A等[19]的方法提取疑似乳酸菌的DNA,参照肖石高等[20]的方法对提取到的DNA进行PCR扩增,通过1.0%琼脂糖凝胶电泳检测的PCR产物经清洁、连接和转化后选取阳性克隆子寄往上海桑尼生物科技有限公司进行测序,对返回序列进行拼接和去引物后于美国国家生物技术信息中心(national center of biotechnology information,NCBI)网站进行对比,以确定乳酸菌分离株的分类学地位。
1.3.4 数据处理
基于UniFrac距离使用非加权组平均法(unweighted pair-group method with arithmetic means,UPGMA)聚类进行β多样性分析;使用Past3软件中的Moon-Median检验进行2个聚类的显著性分析;使用R软件(v4.1.2)的“ggpubr”和“ggplot2”包绘制小提琴图、“waterfall”包绘制优势OTU的瀑布图、“pheatmap”包绘制乳酸菌分离株的热图;使用MEGA5.0和R软件的“tidyverse”、“treeio”与“ggtree”包构建系统发育树;使用Origin 2017软件绘制其他图。
2 结果与分析
2.1 巴东地区鲊广椒细菌类群的β和α多样性分析
基于Illumina MiSeq测序平台,本研究对恩施土家族苗族自治州巴东地区14个鲊广椒样品进行了高通量测序,共获得了609 196条高质量序列,平均每个样品测序深度为43 514条。本研究首先对其细菌类群进行了β多样性分析,基于UniFrac加权距离的UPGMA聚类分析见图1。
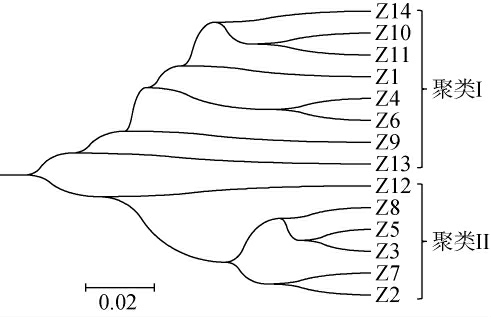
图1 基于UniFrac加权的非加权组平均法聚类分析结果
Fig.1 Unweighted group mean cluster analysis results based on UniFrac weighting
由图1可知,在14个鲊广椒样品中形成较为明显的聚类趋势,其中Z1、Z4、Z6、Z9、Z10、Z11、Z13和Z14等8个样品形成聚类I,Z2、Z3、Z5、Z7、Z8和Z12等6个样品形成聚类II。当测序深度为25 010条序列时,本研究采用Moon-Median检验对2个聚类鲊广椒样本的α多样性指数进行了显著性分析,结果见图2。
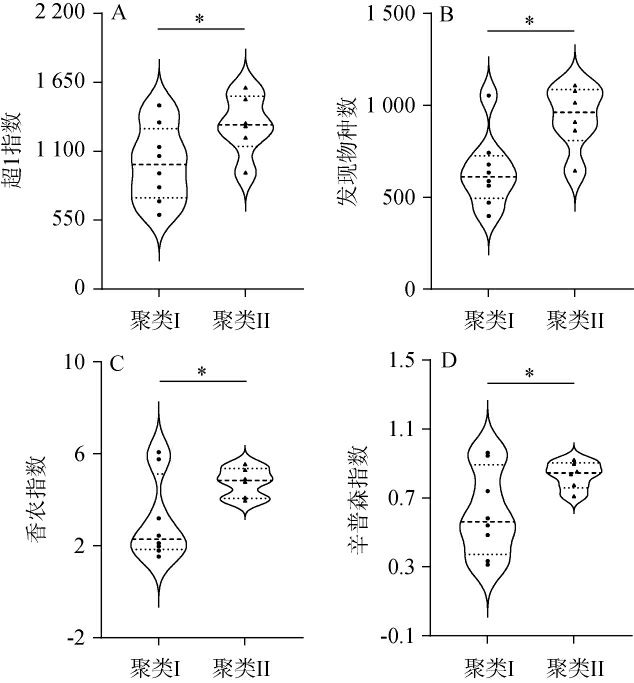
图2 两个聚类鲊广椒α多样性指数的比较分析
Fig.2 Comparative analysis of alpha diversity indexes of two clusters of Zha-chili
“*”代表差异性显著(P<0.05)。
由图2可知,用以评估样本中微生物丰度的超1指数和发现物种数在2个聚类样品中均存在显著差异(P<0.05),这表明聚类II鲊广椒样品的细菌菌群丰度较之聚类I样本含量更为丰富[21];同时用以评估样本中微生物多样性的香农指数和辛普森指数在2个聚类样品中亦都存在显著差异(P<0.05),这表明聚类II鲊广椒样品的细菌菌群种类较之聚类I样本更为多样化[21]。综上所述,聚类II样品的细菌类群在丰富度和多样性上均显著高于聚类I的鲊广椒样品。
2.2 基于门、属和OTU水平巴东地区鲊广椒细菌类群组成分析
本研究将鲊广椒中平均相对含量>1.0%的细菌门和属定位为优势门和属,巴东地区鲊广椒优势细菌门和属的含量见图3。
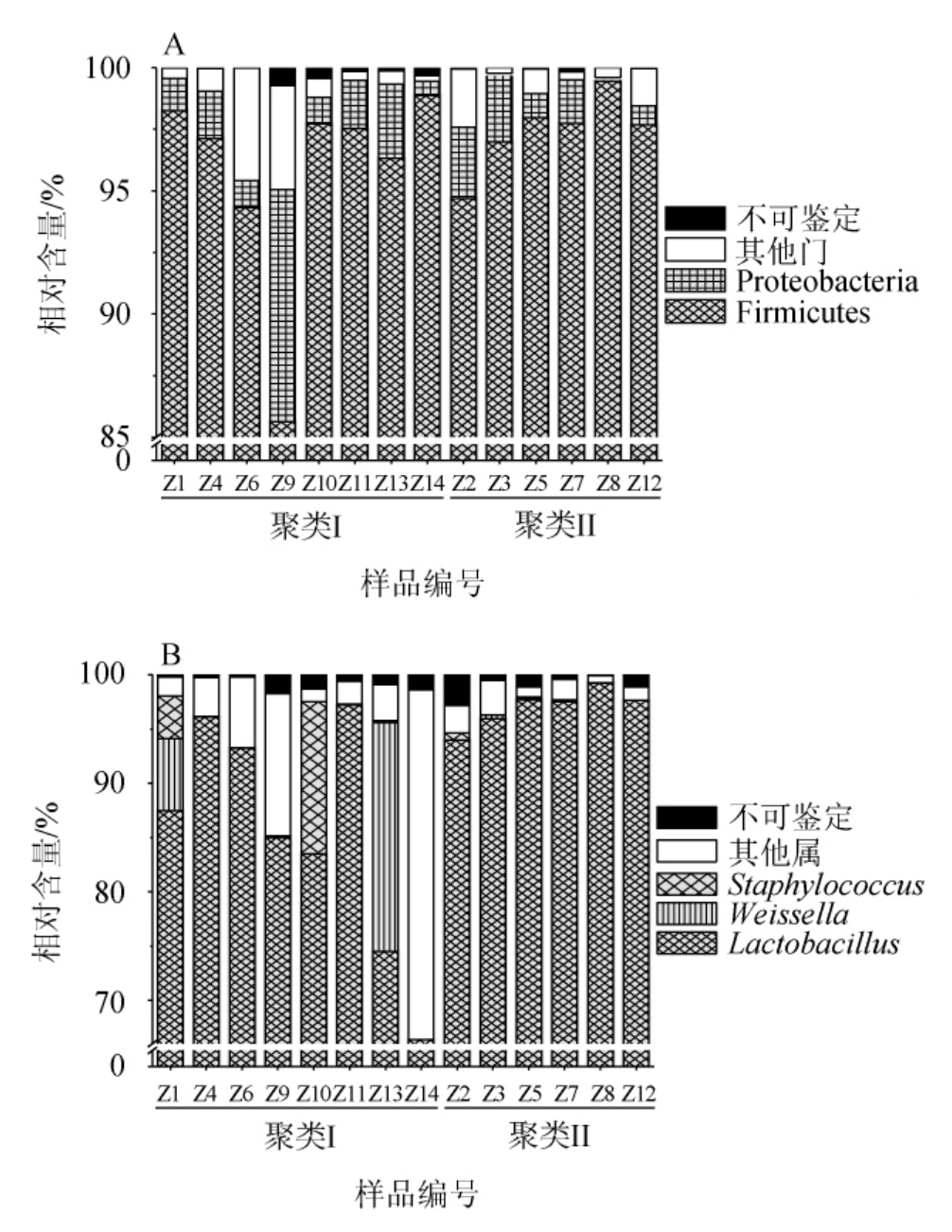
图3 巴东地区鲊广椒样品优势细菌门(A)和属(B)水平上相对含量
Fig.3 Relative content of dominant bacteria phylum (A) and genus (B) of Zha-chili samples in Badong region
由图3可知,巴东地区鲊广椒中细菌隶属于23个门下的304个属,其中平均相对含量>1.0%的门为厚壁菌门(Firmicutes,96.45%)和(Proteobacteria,2.12%),平均相对含量>1.0%的属为乳酸杆菌属(Lactobacillus,92.22%)、魏斯氏菌属(Weissella,2.19%)和葡萄球菌属(Staphylococcus,1.48%)。由图3亦可知,优势细菌属在各鲊广椒样品中的含量分布存在较大差异,作为优势细菌属的Weissella在Z2、Z6、Z8、Z10、Z12和Z14共6个样品中均不存在,而其在样品Z11和Z1中的相对含量高达21.10%和6.64%。有研究人员发现Weissella的菌株在酸面团的发酵中非常突出,且部分Weissella在发酵过程中可产生大量的葡聚糖和具有潜在益生元活性的新型胞外多糖[22]。值得一提的是,Staphylococcus虽然亦为本研究的优势细菌属,但其在样品Z3中未被发现,而在Z10和Z1样品中的含量高达14.01%和3.91%。有研究发现,Staphylococcus广泛存在于自然界中,其多数具有蛋白水解和脂肪分解活性,还有少数为条件致病菌,属中最突出的病原物种是金黄色葡萄球菌(S.aureus),这类菌株寄居在人类的鼻腔和皮肤中,会引起皮肤损伤和感染[23]。故而,在分离、鉴定和保藏鲊广椒中乳酸菌的基础上,筛选具有优良发酵特性和较高安全性的菌株显得尤为重要。
为进一步了解巴东鲊广椒各样品细菌菌群的差异,本研究将平均相对含量>1.0%的OTU定义为优势OTU,并对14个样品中OTU的出现次数及优势OTU的平均相对含量进行了分析,结果见图4。
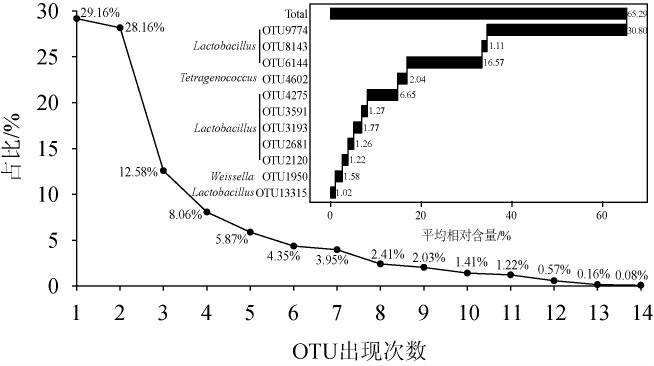
图4 巴东鲊广椒样品细菌的丰度等级曲线
Fig.4 Abundance grade curve of bacteria of Zha-chili samples in Badong region
由图4可知,在根据97%的相似性划分和剔除嵌合体后,可划分出3 697个OTU,其中在所有样品中均存在的OTU仅有3个,其包含的序列数高达190 932条,占总质控合格序列数的31.87%;所有鲊广椒样品中的独特OTU有多达1 078个,但其包含的序列数仅有8 586条,占总质控合格序列数的1.43%,由此可见,巴东鲊广椒样品中存在大量的共有细菌菌群,虽然样品中特有细菌类群的物种数较多,但其含量并不多。由图4亦可知,从鲊广椒样品中甄别出11个优势OTU,分别为隶属于Lactobacillus的OTU9774、OTU6144、OTU4275、OTU3193、OTU3591、OTU2681、OTU 2120、OTU8143和OTU13315,隶属于Tetragenococcus(四联球菌属)的OTU4602与隶属于Weissella的OTU1950,平均相对含量分别为30.80%、16.57%、6.65%、1.77%、1.27%、1.26%、1.22%、1.11%、2.04%和1.58%。值得一提的是,YAO S J等[24]的研究表明,Tetragenococcus适合在高盐浓度的基质中生长,对高盐浓度发酵食品的生产具有重要意义,而巴东鲊广椒主要是由当地农户选用玉米粉添加食盐和辣椒等辅料混合固态发酵制成,其盐浓度较高,较适于Tetragenococcus的生长。
2.3 巴东地区鲊广椒细菌类群的基因功能预测
本研究进一步使用PICRUSt软件对巴东地区鲊广椒中细菌类群的基因功能进行了预测,发现所有序列共注释到4 145个COG,分别隶属于23个功能大类,其潜在功能基因及相对含量见图5。

图5 巴东地区鲊广椒细菌序列中潜在功能基因及其相对含量分析结果
Fig.5 Analysis results of potential functional genes and their relative contents in bacterial sequences of Zha-chili samples in Badong region
由图5可知,巴东地区鲊广椒样品中的细菌除了在转录、核糖体结构和生物发生以及复制、重组和修复等维持细胞基本生命活动的功能上表达较高外,在碳水化合物运输和代谢功能以及氨基酸转运与代谢功能上的占比也相对较高,平均相对含量分别为8.73%和7.88%。KLAENHAMMER T R 等[25]研究表明,Lactobacillus具有的特异性基因包括参与细胞粘附的粘液结合蛋白,以及碳水化合物和氨基酸的几种运输系统。在本研究中占比92.22%的Lactobacillus为巴东地区鲊广椒中的绝对优势细菌属,这可能是造成巴东地区鲊广椒在碳水化合物和氨基酸转运与代谢功能上具有较高表达的一个主要原因。
2.4 基于纯培养方式巴东地区鲊广椒中乳酸菌菌株的分离鉴定
本研究进一步采用传统微生物学方法对鲊广椒中的乳酸菌进行了分离鉴定,将分离株的序列提交到Genbank进行比对后,寻找模式菌株进行了系统发育树的构建,结果见图6。
由图6可知,14个巴东地区鲊广椒样品共分离出58 株乳酸菌,经鉴定其隶属于Lactobacillus和肠球菌属(Enterococcus)2个属下的7个种。其中HBUAS51495等30株菌被鉴定为植物乳杆菌(L.plantarum),HBUAS51476等15株菌被鉴定为消化乳杆菌(L.alimentarius),HBUAS51520等5株菌被鉴定为戊糖乳杆菌(L.pentosus),HBUAS51506被鉴定为仙人掌乳杆菌(L.senmaizukei),HBUAS51507被鉴定为短乳杆菌(L.brevis),HBUAS51463被鉴定为副干酪乳杆菌(L.paracasei),HBUAS51471等5株菌被鉴定为屎肠球菌(Enterococcus faecium)。综上所述,在分离出的58株乳酸菌中,有高达91.38%的分离株隶属于Lactobacillus,为巴东地区鲊广椒中的优势乳酸菌。虽然高通量测序结果表明Weissella是巴东地区鲊广椒中的优势细菌属,但本研究采用纯培养方式并未获得该菌株的分离株。向凡舒等[26]的研究亦得出相似结论,其研究发现Weissella是建始地区酸豇豆盐水中的优势乳酸菌属,相对含量为9.33%,但通过纯培养方法分离出的13株乳酸菌中并不存在隶属于Weissella的菌株。该研究指出,由于Weissella耐酸性能较弱仅存活于发酵前期,而基质中酸度随着发酵的进行逐渐提高,导致菌株进入休眠或者死亡状态,因而无法进行分离培养。虽然菌株随着发酵的进行进入了休眠或者死亡状态,但其核苷酸序列依旧存在于发酵基质中,而MiSeq高通量测序技术是基于核苷酸序列进行测序,菌株是否存活、休眠或者死亡对其结果影响不大,这就导致了虽然MiSeq高通量测序技术可以检测出Weissella,但采用纯培养技术无法实现该菌株的培养[26]。
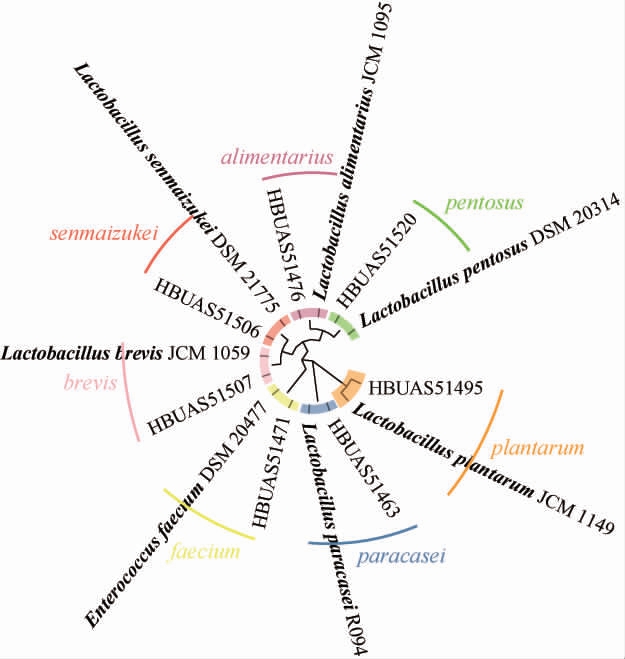
图6 部分优势乳酸菌分离株及其模式株构建的系统发育树
Fig.6 Phylogenetic tree of some dominant lactic acid bacteria isolates and their model strains
本研究进一步比较分析了2个聚类样品中乳酸菌分离株种类与数量的差异,结果见图7。
由图7可知,比较2个聚类鲊广椒样品的乳酸菌类群发现,L.alimentarius、L.plantarum、L.pentosus和E.faecium在2个聚类样品中均存在,而L.paracasei仅存在于聚类I的样品中,L.brevis和L.senmaizukei仅存在于聚类II的样品中。值得一提的是,L.pentosus和E.faecium虽然在2个聚类中均存在,但它们均只在聚类I的一个样品中被分离鉴定出,且分离株数量较少。由此可见,2个聚类鲊广椒样品的乳酸菌组成存在较明显的差异,聚类II样品的鉴定物种数较之聚类I更为丰富,这与高通量测序结果一致。
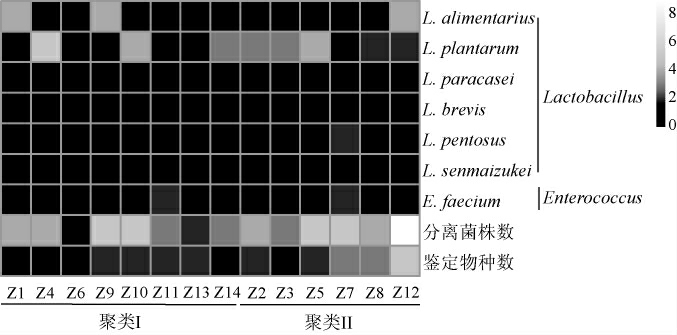
图7 2个聚类鲊广椒样品中乳酸菌分离株的热图
Fig.7 Heat map of lactic acid bacteria isolates in two cluster of Zha-chili samples
3 结论
采用高通量测序和传统纯培养技术相结合的方法对巴东地区鲊广椒细菌多样性进行了解析,结果发现其细菌类群多样性较高,优势菌门为Firmicutes和Proteobacteria,优势菌属为Lactobacillus、Weissella和Staphylococcus,且乳酸杆菌主要以L.plantarum和L.alimentarius为主。从14个鲊广椒样品中共分离出58株乳酸菌,其中53株被鉴定为植物乳杆菌(L.plantarum)、消化乳杆菌(L.alimentarius)、戊糖乳杆菌(L.pentosus)、仙人掌乳杆菌(L.senmaizukei)、短乳杆菌(L.brevis)和副干酪乳杆菌(L.paracasei),5株被鉴定为屎肠球菌(E.faecium)。L.plantarum和L.alimentarius占分离株总数的51.72%和25.86%。由此可见,巴东地区鲊广椒细菌多样性较高,且其乳酸杆菌以L.plantarum和L.alimentarius为主。
[1]DONG W W,SHEN H Y,LIU H M,et al.Unraveling the microbial community and succession during zha-chili fermentation and their relationships with flavor formation[J].Food Res Int,2022,157:111239.
[2] CAI W C,TANG F X,WANG Y R,et al.Bacterial diversity and flavor profile of Zha-Chili,a traditional fermented food in China[J].Food Res Int,2021,141:110112.
[3] ZHANG Z D,HOU Q C,WANG Y R,et al. Lactobacillus zhachilii sp.nov.,a lactic acid bacterium isolated from Zha-Chili[J].Int J Syst Evol Micr,2019,69(8):2196-2201.
[4]ZHANG Z D,WANG Y R,HOU Q C,et al. Lactobacillus enshiensis sp.nov.,a novel arsenic-resistant bacterium[J].Int J Syst Evol Micr,2020,70(4):2580-2587.
[5]SCHIRMER M,IJAZ U Z,D'AMORE R,et al.Insight into biases and sequencing errors for amplicon sequencing with the Illumina MiSeq platform[J].Nucleic Acids Res,2015,43(6):e37.
[6]GUO Z,WANG Y R,XIANG F S,et al.Evaluating the flavor and divergent bacterial communities in corn-based zha-chili[J].Food Biosci,2022,46(4):101563.
[7]CHEN Y,TIAN W,SHAO Y,et al. Miscanthus cultivation shapes rhizosphere microbial community structure and function as assessed by Illumina MiSeq sequencing combined with PICRUSt and FUNGUIld analyses[J].Arch Microbiol,2020,202:1157-1171.
[8]郭壮,葛东颖,尚雪娇,等.退化和正常窖泥微生物多样性的比较分析[J].食品工业科技,2018,39(22):93-98.
[9] LÓPEZ-GARCÍA A,PINEDA-QUIROGA C,ATXAERANDIO R,et al.Comparison of Mothur and QIIME for the analysis of rumen microbiota composition based on 16S rRNA amplicon sequences[J].Front Microbiol,2018,9:3010.
[10] CAPORASO J G,BITTINGER K,BUSHMAN F D,et al.PyNAST: a flexible tool for aligning sequences to a template alignment[J].Bioinformatics,2010,26(2):266-267.
[11] EDGAR R C.Search and clustering orders of magnitude faster than BLAST[J].Bioinformatics,2010,26(19):2460-2461.
[12] EDGAR R C,HAAS B J,CLEMENTE J C,et al.UCHIME improves sensitivity and speed of chimera detection[J].Bioinformatics,2011,27(16):2194-2200.
[13]CAN M.Annotation of bacteria by Greengenes classifier using 16S rRNA gene hyper variable regions[J].Southeast E J Soft Comput,2019,8(2):35-45.
[14]BACCI G,BANI A,BAZZICALUPO M,et al.Evaluation of the performances of ribosomal database project (RDP) classifier for taxonomic assignment of 16S rRNA metabarcoding sequences generated from Illumina-Solexa NGS[J].J Genomics,2015,3:36-39.
[15]GLÖCKNER F O.The SILVA database project:an ELIXIR core data resource for high-quality ribosomal RNA sequences[J].Biodivers InformSci Stand,2019,3:e36125.
[16]WILKINSON T J,HUWS S A,EDWARDS J E,et al.CowPI: a rumen microbiome focussed version of the PICRUSt functional inference software[J].Front Microbiol,2018,9:1095.
[17] GALPERIN M Y,KRISTENSEN D M,MAKAROVA K S,et al.Microbial genome analysis:the COG approach[J].Brief Bioinform,2019,20(4):1063-1070.
[18]李娜,崔梦君,马佳佳,等.基于Illumina MiSeq测序和传统可培养方法的洪湖鲊广椒乳酸菌多样性研究[J].食品与发酵工业,2021,47(4):110-115.
[19]PEREIRA G A,SANTOS E M,ARA JO G G L,et al.Isolation and identification of lactic acid bacteria in fresh plants and in silage from Opuntia and their effects on the fermentation and aerobic stability of silage[J].J Agr Sci,2019,157(9-10):684-692.
JO G G L,et al.Isolation and identification of lactic acid bacteria in fresh plants and in silage from Opuntia and their effects on the fermentation and aerobic stability of silage[J].J Agr Sci,2019,157(9-10):684-692.
[20]肖石高,何梦雪,张振东,等.长寿老人肠道乳酸菌多样性及其分离株对发酵乳品质的影响[J].中国乳品工业,2021,49(12):8-12.
[21]向凡舒,张婷,董蕴,等.糯米酸细菌多样性解析及其表型和基因功能预测[J].中国粮油学报,2021,36(1):14-20.
[22]TEIXEIRA C G,FUSIEGER A,MILIÃO G L,et al.Weissella:An emerging bacterium with promising health benefits[J].Probiotics Antimicro,2021,13(4):915-925.
[23] CHEUNG G Y C,BAE J S,OTTO M.Pathogenicity and virulence of Staphylococcus aureus[J].Virulence,2021,12(1):547-569.
[24]YAO S J,HAO L Y,ZHOU R Q,et al.Co-culture with Tetragenococcus halophilus improved the ethanol tolerance of Zygosaccharomyces rouxii by maintaining cell surface properties[J].Food Microbiol,2021,97:103750.
[25]KLAENHAMMER T R,ALTERMANN E,PFEILER E,et al.Functional genomics of probiotic Lactobacilli[J].J Clin Gastroenterol,2008,42:S160-S162.
[26]向凡舒,黄怡,陈小艳,等.建始地区酸豇豆盐水细菌多样性及乳酸菌分离鉴定[J].中国酿造,2020,39(10):61-65.