米酒,又称为醪糟、甜酒,是我国民间传统的小曲酒,因其香气浓郁,口感独特,酒精度低且不易上头[1]而深受人们的喜爱。至今为止,米酒已传承几千年,受地域环境和人们饮食习惯的影响,逐渐呈现出多元化的趋势,其总体制作工艺较为简单,多以大米或糯米为主料,蒸煮降温后加入酒曲和水发酵而成[2]。米酒中含有丰富的糖类、蛋白质、维生素和矿物质等营养物质[3],具有独特的微酸甜口感及淡淡的酒香,不仅可以直接饮用,还可以作为调味品,赋予食品独特的香气[4]。大竹米酒是四川省达州市大竹县的著名特产,又称“东汉醪糟、东柳醪糟”,是将糯米淘洗浸泡3~5 h后蒸熟摊凉,加入适量凉开水和酒曲拌匀后搭窝保温发酵3 d左右制作而成的[5]。成品具有色白汁清、甜浓爽口,酒香四溢的特点,同时还有补气、生血、活络、通经、润肺之功,夏日更兼清爽。黄酒历史悠久,源于汉,盛于清,是中国原产地地理标志保护产品和生态原产地保护产品[6],《中国土特名产辞典》亦有其相关记录,2006年其酿造工艺列入四川省非物质文化遗产项目。米酒属于半开放式液态发酵型酒精饮料[7],其品质除了与米酒曲中微生物结构息息相关,与原料的选择和地理环境密不可分[8]。
米酒的制作是多种微生物混合发酵完成的,发酵过程中存在微生物群落结构的变化,主要包含酵母菌、霉菌以及细菌三大类微生物[9-10]。其中真菌微生物参与了米酒发酵的全过程,在米酒发酵中的作用不可替代。MiSeq高通量测序技术具有成本低、通量高、测序周期短的优势,已广泛应用于米酒及米酒曲微生物群落结构的研究中[11]。宁亚丽等[12]以吉林延边朝鲜族地区酒曲和米酒为研究对象,采用高通量测序技术分析其细菌和真菌微生物群落结构的多样性,表明在一定测序深度下该技术可以有效地覆盖样品中微生物种类。周书楠等[13]以湖北恩施地区米酒为研究对象,采用MiSeq高通量测序技术分析其样品中的真菌微生物,结果显示该地区米酒中存在大量真菌资源且含有多种未被鉴定的微生物结构。MiSeq高通量测序技术的使用对米酒中微生物群落的研究及其产业化的发展具有重要的意义。
本研究以四川省达州市大竹地区的米酒为研究对象,采用MiSeq高通量测序技术研究米酒中真菌微生物的多样性,同时使用传统微生物技术确定米酒中酵母菌的微生物学分类地位,从而为大竹地区米酒中微生物的多样性研究及其产业化的进一步发展提供一定的理论基础。
1 材料与方法
1.1 材料与试剂
1.1.1 原料
米酒样品(编号为DZ1~DZ7):采集自四川省达州市大竹县。
1.1.2 化学试剂
脱氧核糖核酸(deoxyribonucleic acid,DNA)基因组提取试剂盒:德国QIAGEN公司;5×FastPfu Buffer、脱氧核糖核苷三磷酸(deoxy-ribonucleoside triphosphates,dNTPs)和FastPfu Fly DNA Polymerase:北京全式金生物技术有限公司;DL2000Marker、10×聚合酶链式反应(polymerase chain reaction,PCR)Buffer、rTaq DNA聚合酶、pMD18-T克隆载体:宝生物工程(大连)有限公司;MiSeq高通量测序配套试剂:美国Illumina公司。
1.1.3 培养基
马铃薯葡萄糖琼脂(potato dextrose agar,PDA)培养基、酵母浸出粉胨葡萄糖(yeast extract peptone dextrose,YEPD)培养基:青岛海博生物技术有限公司。
1.1.4 引物及其序列
引物ITS3F/ITS4R、M13F(-47)/M13R(-48)、NS1/NL4:武汉天一辉远生物科技有限公司合成。引物及其对应的序列见表1。
表1 本研究所用引物名称及其对应引物序列
Table 1 Names of primers and its corresponding primer sequences used in the study
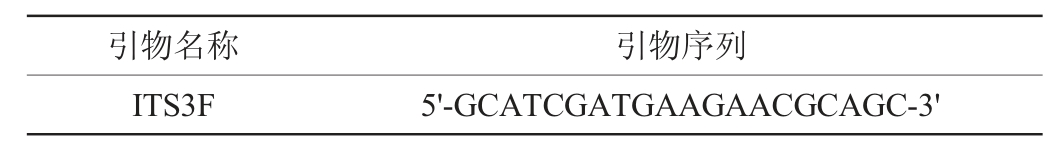
续表

1.2 仪器与设备
ND-2000C微量紫外分光光度计:美国Thermo公司;VeritiFAST96 PCR:美国ABI公司;DYY-12电泳仪:北京六一仪器厂;UVPCDS8000凝胶成像分析系统:美国Protein Simple公司;Illumina MiSeq高通量测序平台:美国Illumina公司;5810R型台式高速冷冻离心机:德国Eppendorf公司;R930型机架式服务器:美国DELL公司。
1.3 方法
1.3.1 样品采集
本研究中7份米酒样品(编号为DZ1~DZ7)于2021年4月24日采集自四川省达州市大竹县的菜市场,均为农户手工制作自然发酵而成,纳入研究的样本均未经过杀菌处理。将采集的米酒样品暂放于无菌自封袋中,置于放有冰袋的采样箱中运回实验室。在实验室条件下,取一定量的样品于无菌50 mL离心管中备用。
1.3.2 宏基因组DNA提取、PCR扩增及测序
参考文献[14]中的方法,采用基因组DNA提取试剂盒提取米酒中微生物的宏基因组DNA,使用1%琼脂糖凝胶电泳对其进行检测,将检测合格的DNA作为模板进行后续实验。使用引物ITS3F和ITS4R对宏基因组DNA的真菌ITS2区进行PCR扩增[15],使用2%琼脂糖凝胶电泳对扩增产物进行检测,将检测合格的扩增产物寄往上海美吉生物医药科技有限公司进行MiSeq高通量测序。
1.3.3 序列质控
参照文献[16]中的方法将下机序列拼接并进行质控:①重叠区>10 bp;②最大错配比率<0.2;③核苷酸标签(barcode)碱基无错配;④引物碱基错配数<2 bp。将满足上述四个条件的拼接序列保留下来,并去除其中的barcode和引物得到合格的序列。
1.3.4 生物信息学分析[17]
采用QIIME(v1.9.5)平台对7份米酒样品进行真菌物种的生物信息学分析。基本流程为:①使用PyNAST软件对序列进行同源性比对;②采用UCLUST法划分操作分类单元(operational taxonomic units,OTU);③使用ChimeraSlayer去除含有嵌合体的OTU序列;④使用UNITE数据库进行物种注释;⑤使用FastTree软件基于OTU水平绘制系统发育进化树,并对α多样性和β多样性进行统计分析。本研究中使用的序列数据提交至MG-RAST数据库,获取编号为mgp103191。
1.3.5 酵母菌分离纯化
用0.85%的无菌生理盐水对样品进行梯度稀释,稀释梯度依次为10-1、10-2、10-3、10-4、10-5和10-6,取100 μL各个梯度的稀释液涂布于PDA固体培养基上,置于28 ℃静置培养3~5 d,取菌落形态和颜色不同的单菌落进行划线,纯化三次后收集菌体,使用30%的甘油保存菌体并置于-80 ℃冰箱中保藏[18]。
1.3.6 酵母菌鉴定
根据参考文献[19]的方法提取酵母菌DNA并略做调整:①使用组织破碎仪破除菌株细胞壁;②加入10%十二烷基硫酸钠(sodium dodecylsulfate,SDS)和20 mg/mL蛋白酶K破坏细胞结构;③加入5 mol/L NaCl析出蛋白质;④使用苯酚-氯仿-异戊醇(25∶24∶1,V/V)溶剂抽提2次;⑤使用氯仿-异戊醇(24∶1,V/V)溶剂抽提1次;⑥加入异丙醇和3 mol/L NaAc沉淀DNA;⑦加入体积分数为70%乙醇去除杂质;⑧加入TE缓冲液溶解DNA,-20 ℃贮藏备用。
参照文献[20]进行酵母菌内转录间隔区(internal transcribed spacer,ITS)扩增,经过清洁、连接和转化操作后将阳性单克隆产物送往上海桑尼生物科技有限公司进行测序,将测序结果与美国国家生物信息中心(national center for biotechnology information,NCBI)的GenBank数据库中已知的序列进行基本局部比对工具(basic local alignment search tool,BLAST)同源性比对,应用MEGA7.0中邻接(neighbor joining,NJ)法构建系统发育树。
1.3.7 数据分析与图形绘制
使用Excel2016整理数据,Origin2021b绘制α多样性曲线和优势真菌门和属的堆积柱状图,R4.0.0绘制OTU花瓣图和核心OTU瀑布图,MEGA7绘制真菌ITS区扩增的系统发育树。
2 结果与分析
2.1 米酒中真菌群落α多样性分析
微生物群落α多样性主要与物种丰富度和多样性有关,其中发现物种数[21]能够反映物种丰富度,香农(Shannon)指数[22]能够反映物种多样性,利用各样本的测序量在不同测序深度时的α多样性构建发现物种数曲线和Shannon指数曲线,反映各样本在不同测序深度下的物种丰富度和多样性,结果见图1。
由图1(a)可知,随着测序量的增加,发现物种数不断升高,物种丰富度逐渐升高。由图1(b)可知,随着测序量的增加,Shannon指数逐渐升高,当测序量达到5 000 bp后,Shannon指数曲线趋于稳定,物种多样性曲线进入平台期。同一测序深度下,样品DZ7中发现物种数和Shannon指数最高,样品DZ6中发现物种数和Shannon指数最低,且不同样品中真菌群落的丰富度和多样性存在较为明显的差异。综上所述,随着测序量的增加在米酒中依旧会发现新的真菌物种,但真菌微生物的多样性已趋于稳定,表明本研究的测序深度可以有效反映样品中绝大部分的微生物信息。
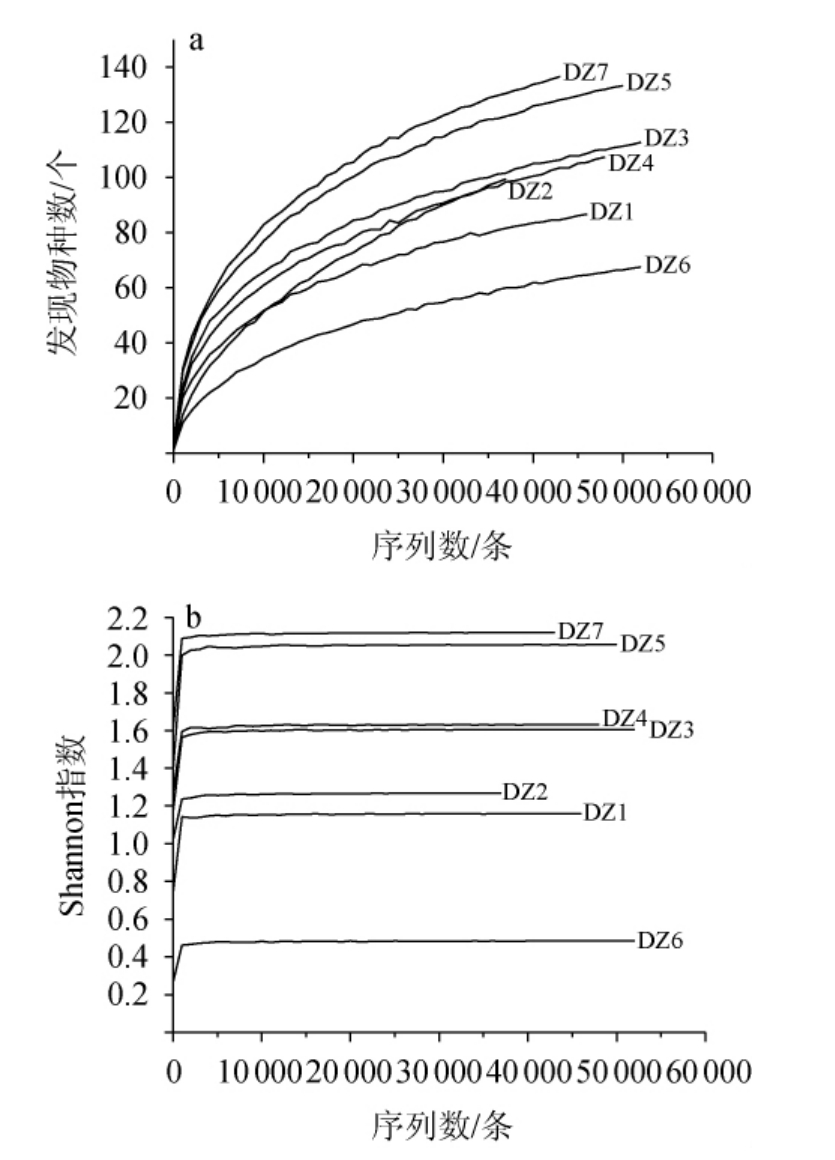
图1 米酒样品中真菌群落的发现物种数(a)和Shannon指数(b)曲线
Fig.1 Observed number of species (a) and Shannon index curves(b) of fungi community in rice wine samples
2.2 米酒中真菌群落结构分析
基于门和属水平对米酒样品中真菌微生物结构进行揭示,其中将平均相对含量≥1%的真菌门和真菌属定义为优势真菌门和优势真菌属[23],将平均相对含量<1%的门和属定义为other,将未鉴定部分定义为unclassified,对各样品中真菌群落结构进行生物信息学注释,结果见图2。
由图2a可知,在真菌门水平上,子囊菌门(Ascomycota)(50.11%)和毛霉亚门(Mucoromycota)(49.81%)是7份米酒样品的优势菌门,样品DZ1中Ascomycota相对含量最高(96.85%),样品DZ6中Mucoromycota相对含量最高(94.90%)。由图2b可知,在真菌属水平上,根霉属(Rhizopus)(49.64%)、复膜孢酵母属(Saccharomycopsis)(19.93%)、酿酒酵母属(Saccharomyces)(16.58%)和威克汉姆酵母属(Wickerhamomyces)(11.53%)为米酒中的优势真菌属。样品DZ1以威克汉姆酵母属(Wickerhamomyces)(80.67%)为主,样品DZ2、DZ3、DZ4、DZ5、DZ6和DZ7以根霉属(Rhizopus)为主,相对含量分别为56.07%、59.76%、60.63%、34.22%、94.52%和39.88%。此外,部分样品中存在丰富的复膜孢酵母属(Saccharomycopsis)和酿酒酵母属(Saccharomyces)的微生物群落。
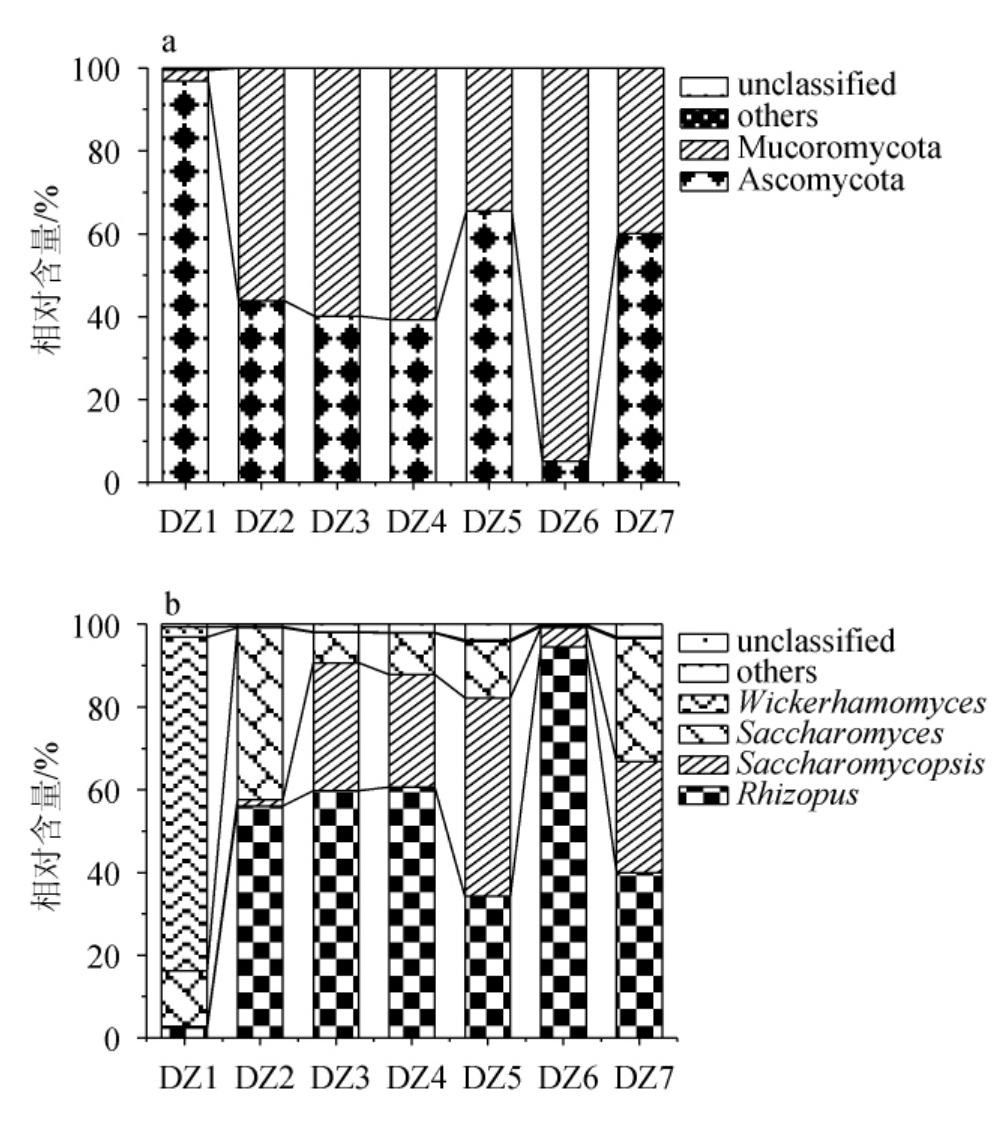
图2 基于门(a)及属(b)水平米酒样品中真菌菌群结构分析结果
Fig.2 Results of fungal community structure analysis in rice wine samples based on phylum (a) and genus (b) level
湖北恩施地区米酒与四川大竹地区米酒相比,在门水平上,均是以Ascomycota和Mucoromycotina为其优势真菌门,但Saccharomycopsis、Rhizopus和淀粉菌属(Amylomyces)为恩施地区米酒的优势真菌属,Rhizopus、Saccharomycopsis、Saccharomyces和Wickerhamomyces为大竹米酒的优势真菌属[13]。由此可知,恩施地区和大竹地区米酒中真菌微生物群落结构存在相似性,亦含有其特有的优势真菌属群落结构。同时湖北恩施地区位于109°4'~109°58'E,29°50'~30°39'N之间,属于亚热带季风和季风性湿润气候;四川大竹地区位于106°59'~107°32'E,30°20'~31°00'N之间,属于中亚热带湿润季风气候,地理位置和气候条件的差异对微生物的生长繁殖具有不可忽视的影响[24]。
2.3 米酒中真菌群落OTU水平分析
本研究进一步在OTU水平对米酒中真菌群落结构进行分析,若某个OTU在所有样品中均存在则将其定义为核心OTU,结果分别见图3和图4。
由图3可知,米酒样品中总OTU为747个,总序列数为332 860条。其中真菌核心OTU仅有13个,但包含287 412条序列,占总序列数的86.35%。尽管各样品中存在多个非核心OTU,但非核心OTU中仅含有少量的序列,大部分序列分布于核心OTU中,表明大竹米酒中存在大量核心真菌微生物。
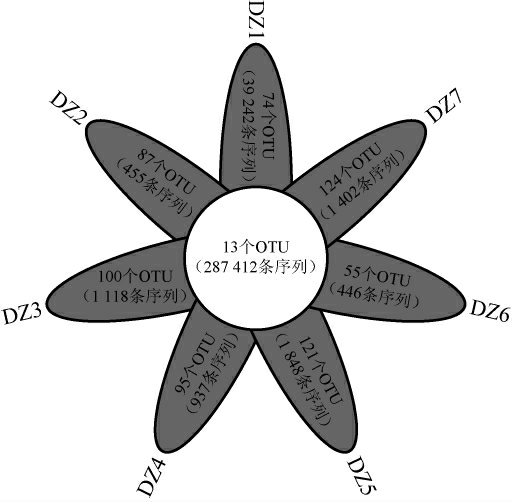
图3 米酒样品中OTU分布的花瓣图
Fig.3 Petal plot of OTU distribution in rice wine samples
由图4可知,核心OTU334、OTU398隶属于根霉属(Rhizopus),累积平均相对含量为49.48%。核心OTU313、OTU486隶属于扣囊腹膜孢酵母属(Saccharomycopsis),累积平均相对含量为19.16%。核心OTU114、OTU199、OTU945隶属于酿酒酵母属(Saccharomyces),累积平均相对含量为16.21%。核心OTU129隶属于毛霉属(Mucor),平均相对含量为0.17%。核心OTU56、OTU873、OTU660、OTU857、OTU697均隶属于酵母目(Saccharomycetales),累积平均相对含量为1.10%。由此可知,核心OTU以Rhizopus、Saccharomycopsis和Saccharomyces为主,进一步证明大竹米酒样品中存在大量核心真菌群落。
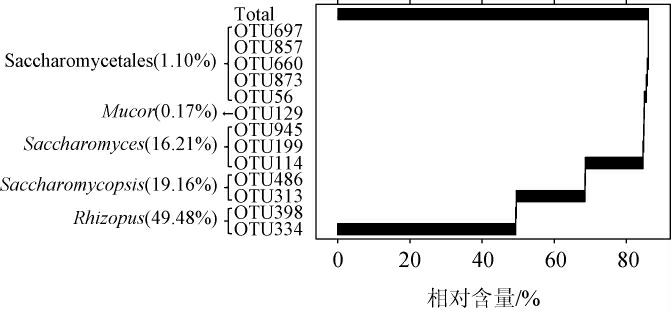
图4 米酒样品中核心OTUs相对含量的瀑布图
Fig.4 Waterfall plot of the relative content of core OTUs in rice wine samples
2.4 米酒中酵母菌的分离及鉴定
2.4.1 米酒中酵母菌分离纯化及形态观察
由上述真菌多样性分析显示,米酒中微生物结构丰富且存在大量优势真菌属,使用传统微生物分离纯化方法对来自大竹地区的米酒样品进行酵母菌的分离纯化,并对分离株进行菌落形态和显微镜形态的观察,并在种水平上明确其分离株的生物学地位。从米酒样品中共分离纯化出16株菌株,部分分离菌株菌落及细胞形态见图5。
由图5A、5B、5C可知,在PDA培养基上的分离菌株菌落呈乳白色或白色,大小适中、边缘整齐;由图5a、5b、5c可知,细胞形态呈现大小不一的椭圆形。
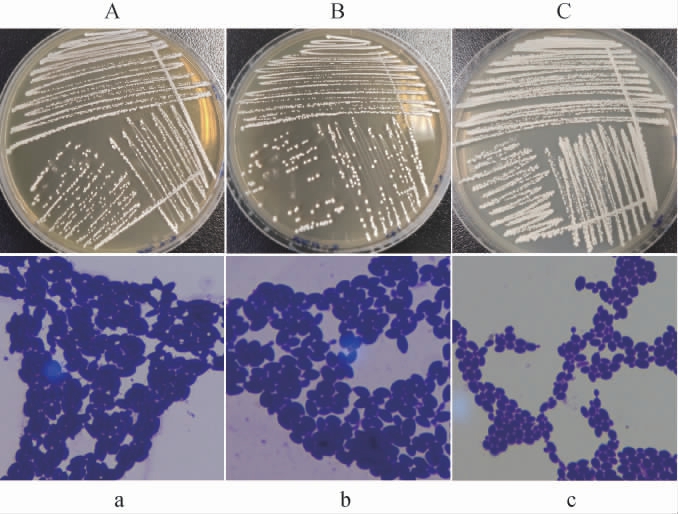
图5 米酒样品中部分分离菌株的菌落(A,B,C)及细胞(a,b,c)形态
Fig.5 Morphologies of colony (A,B,C) and cell (a,b,c) of some isolated strains from rice wine samples
2.4.2 米酒中分离菌株的分子生物学鉴定
16株菌株的系统发育树见图6。由图6可知,从米酒中分离的16 株菌株聚为两类,其中菌株DZ1-1和DZ1-2与异常威克汉姆酵母(Wickerhamomyces anomalus)聚为一类,同源性达99%以上,故将其鉴定为异常威克汉姆酵母(W.anomalus)。其余14株菌株与酿酒酵母(Saccharomyces cerevisiae)聚为一类,同源性均在99%以上,故将其鉴定为酿酒酵母(S.cerevisiae)。CHEN L H等[25]研究表明,W.anomalus和S.cerevisiae共同发酵对米酒的理化特性及风味质量有重要影响。大竹米酒中存在大量有益于产生良好的米酒风味和品质的酵母菌资源,可对后续大竹米酒品质与真菌群落结构之间的相关性研究以及米酒的进一步发展提供可靠的研究基础。
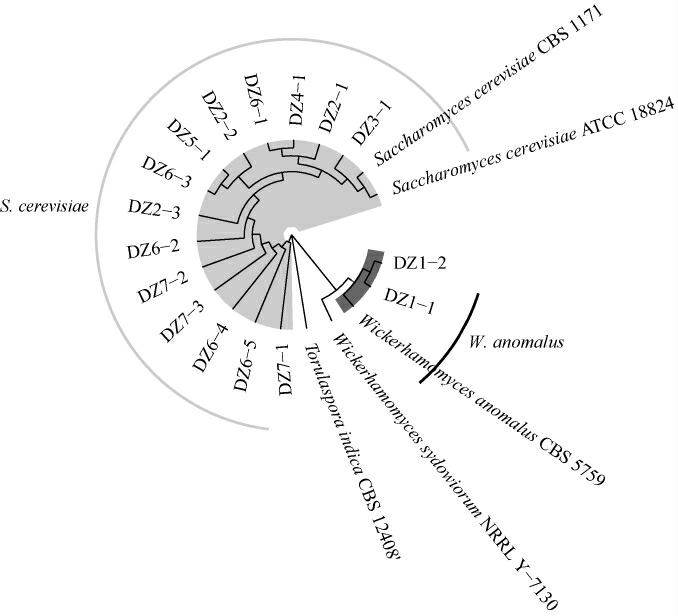
图6 米酒样品中分离菌株的系统发育树
Fig.6 Phylogenetic tree of strains isolated from rice wine samples
酵母菌分离频率结果表明,大竹米酒中可分离菌株以S.cerevisiae为主,相对含量达87.5%[26]。有研究显示,使用传统方法对四川大竹米酒曲中酵母菌进行分析,其分离株中扣囊复膜酵母(Saccharomycopsis fibuligera)的相对含量约为66.67%,S.cerevisiae的相对含量仅为8.33%[27]。虽然米酒的品质与米酒曲中微生物的群落结构密切相关,但由于米酒的酿造工艺属于液态发酵型,因此米酒与米酒曲的微生物结构亦存在差异。
3 结论
本研究采集的大竹地区米酒中存在大量核心真菌类群,多样性分析表明,该地区米酒中真菌微生物群落主要由隶属于Ascomycota的Saccharomycopsis、Saccharomyces和Wickerhamomyces,以及隶属于Mucoromycota的Rhizopus构成。酵母菌分离频率显示,米酒中可分离株由S.cerevisiae和W.anomalus构成,且S.cerevisiae相对含量高达87.5%。本研究旨在对米酒产业的工业化发展提供较为可靠的理论依据,同时对进一步探究米酒中微生物群落结构亦具有积极作用。
[1]张洋洋.米酒液态发酵的研究[D].无锡:江南大学,2021.
[2]李宁宁,王敏.四川传统米酒酿造工艺研究[J].现代农村科技,2020,49(11):79-80.
[3]JIAO A Q,XU X M,JIN Z Y.Research progress on the brewing techniques of new-type rice wine[J].Food Chem,2017,215:508-515.
[4]陈樱,傅生生.文旅康养产业视角下的闽西特色饮食文化研究[J].农村经济与科技,2021,32(20):87-89.
[5]黄晓庆,王晓英,谢强,等.四川·达州“农”腾巴山达天下[J].经营管理者,2022,38(1):44-47.
[6]郑伟.四川调味品类非物质文化遗产保护传承现状及对策研究[J].中国调味品,2021,46(12):189-192.
[7] HUANG Y Y,LIANG Z C,LIN X Z,et al.Fungal community diversity and fermentation characteristics in regional varieties of traditional fermentation starters for Hong Qu glutinous rice wine[J].Food Res Int,2021,141:110146.
[8]CAI H Y,ZHANG T,ZHANG Q,et al.Microbial diversity and chemical analysis of the starters used in traditional Chinese sweet rice wine[J].Food Microbiol,2018,73:319-326.
[9] REN Q,SUN L P,WU H J,et al.The changes of microbial community and flavor compound in the fermentation process of Chinese rice wine using Fagopyrum tataricum grain as feedstock[J].Sci Rep,2019,9:3365.
[10]HUANG Z R,GUO W L,ZHOU W B,et al.Microbial communities and volatile metabolites in different traditional fermentation starters used for Hong Qu glutinous rice wine[J].Food Res Int,2019,121:593-603.
[11]PU D,XIAO P F.A real-time decoding sequencing technology-new possibility for high throughput sequencing[J].RSC Adv,2017,7: 40141-40151.
[12]宁亚丽,吴跃,何嫱,等.基于高通量测序技术分析朝鲜族传统米酒及其酒曲中微生物群落多样性[J].食品科学,2019,40(16):107-114.
[13]周书楠,王玉荣,周亚澳,等.基于MiSeq高通测序技术的米酒真菌多样性分析[J].食品工业科技,2019,40(8):85-89,96.
[14]JIANG L,SU W,MU Y C,et al.Major metabolites and microbial community of fermented black glutinous rice wine with different starters[J].Front Microbiol,2020,11:593.
[15] QIAN W,LU Z M,CHAI L J,et al.Cooperation within the microbial consortia of fermented grains and pit mud drives organic acid synthesis in strong-flavor Baijiu production[J].Food Res Int,2021,147:110449.
[16]OBAFEMI Y D,ORANUSI S U,AJANAKU K O,et al.African fermented foods:overview,emerging benefits,and novel approaches to microbiome profiling[J].Npj Sci Food,2022,6:15.
[17]ZANG J H,XU Y S,XIA W S,et al.Dynamics and diversity of microbial community succession during fermentation of Suan yu,a Chinese traditional fermented fish,determined by high throughput sequencing[J].Food Res Int,2018,111:565-573.
[18]SIMÕES L A,CRISTINA D S A,FERREIRA I,et al.Probiotic properties of yeasts isolated from Brazilian fermented table olives[J].J Appl Microbiol,2021,131(4):1983-1997.
[19]徐文欢,吴若菡,李采婵,等.传统虾酱中酵母菌分离鉴定及碳源利用特性[J].中国食品学报,2021,21(4):303-309.
[20]CHOW B,GROESCHEL M,CARSON J,et al.Development and evaluation of a novel fast broad-range PCR and sequencing assay(FBR-PCR/S)using dual priming oligonucleotides targeting the ITS/LSU gene regions for rapid diagnosis of invasive fungal diseases:multi-year experience in a large Canadian healthcare zone[J].BMC Infect Dis,2022,22:375.
[21] JI X A,YU X W,WU Q,et al.Initial fungal diversity impacts flavor compounds formation in the spontaneous fermentation of Chinese liquor[J].Food Res Int,2022,155:110995.
[22] ULLRICH S,THRIENE K,BINDER N,et al.Influence of fermented vegetable consumption on gut microbiome diversity[J].Curr Dev Nutr,2021,5(2):1188.
[23]DAI Y J,TIAN Z Q,MENG W N,et al.Microbial diversity and physicochemical characteristics of the Maotai-flavored liquor fermentation process[J].J Nanosci Nanotechnol,2020,20(7):4097-4109.
[24] WEI R T,DING Y T,GAO F F,et al.Community succession of the grape epidermis microbes of cabernet sauvignon(Vitis vinifera L.)from different regions in China during fruit development[J].Int J Food Microbiol,2022,362:109475.
[25]CHEN L H,LI D N,REN L X,et al.Effects of simultaneous and sequential cofermentation of Wickerhamomyces anomalus and Saccharomyces cerevisiae on physicochemical and flavor properties of rice wine[J].Food Sci Nutr,2020,9(1):71-86.
[26]巫婷玉,徐芳玲,叶蔚歆,等.乡村自然发酵豆腐乳的真菌多样性研究[J].食品工业科技,2021,42(18):112-118.
[27]雷炎,刘梦琦,易秦振,等.扣囊复膜酵母在红枣酒中的应用[J].食品研究与开发,2021,42(5):131-136.