由于所使用的高温大曲微生物类群丰度和多样性均较低[1-2],因而酱香型白酒酒醅的发酵通常包含高温堆积发酵和入窖密封发酵两部分[3]。通过高温堆积发酵,酒醅不仅可以富集环境中的微生物[4],同时又可以代谢产生有机酸、醇类和酯类等香味物质及前体物质,为后续的入窖密封发酵提供良好的原材料[5]。在堆积过程中,随着醅堆内部的温度不断上升,醅堆表层或内部的部分酒醅会由黑褐色逐渐变成白色或微黄色,并成团状或带霉味,这种酒醅被称之为腰线酒醅[6]。腰线部位的酒醅是一个局部封闭的发酵体系,其形成会对醅堆中微生物与微生物和微生物与环境的关系造成破坏[7],邵明凯等[8]研究亦指出,腰线的形成会导致酒醅中具有优良发酵特性的酿酒微生物减少,最终降低酱香型白酒的品质,因而腰线形成的机制和处理对策一直是酱香型白酒生产企业比较关心的问题。要明确腰线的形成机制,首要问题是对腰线和正常部位酒醅的微生物类群差异进行甄别,然而目前关于这方面的研究尚少。
本研究从某酱香型白酒生产企业选取了4个正在进行第四轮次堆积发酵的醅堆,从腰线部位和正常部位各采集了4个样本,在对其菌落总数、乳酸菌及霉菌和酵母进行计数的基础上,采用Illumina MiSeq高通量测序技术对不同部位酒醅微生物类群的构成进行了解析,以期为腰线形成机制的揭示和处理对策的提出提供一定的数据支持,进而最终实现酱香型白酒品质的提升。
1 材料与方法
1.1 材料与试剂
基因组脱氧核糖核酸(deoxyribonucleic acid,DNA)提取试剂盒:德国QIAGEN公司;rTaq酶、10×Buffer、DNA聚合酶、脱氧核糖核苷三磷酸(deoxy-ribonucleoside triphosphate,dNTP):宝生物工程(大连)有限公司;正向引物338F(5'-ACTCCTACGGGAGGCAGCA-3')、反向引物806R(5'-GGACTACHVGGGTWTCTAAT-3')、正向引物ITS3F(5'-GCATCGATGAAGAACGCAGC-3')、反向引物ITS4R(5'-TCCTCCGCTTATTGATATGC-3'):上海桑尼生物科技公司;平板计数琼脂培养基、MRS琼脂培养基和马铃薯葡萄糖琼脂培养基:青岛海博生物技术有限公司;高通量测序配套试剂:美国Illumina公司。其他试剂均为国产分析纯。
1.2 仪器与设备
MiSeq PE300高通量测序平台:美国Illumina公司;UVPCDS8000凝胶成像分析系统:美国Protein Simple公司;R930机架式服务器:美国Dell公司;ND-2000C微量紫外分光光度计:美国Nano Drop公司;Veriti FAST梯度聚合酶链式反应(polymerase chain reaction,PCR)仪:美国ABI公司;DG250型厌氧工作站:英国Don Whitley公司;LRH-250F生化培养箱:上海一恒科学仪器有限公司。
1.3 方法
1.3.1 样品采集
从湖北省襄阳市某酒厂选取4个正在进行第四轮次堆积发酵的醅堆,各醅堆高160 cm左右,已堆积发酵8 d且即将入池。各醅堆中腰线上限距表面约10~12 cm,宽约5~10 cm,成弧形分布,与其他位置酒醅呈现的黑褐色不同,腰线位置酒醅呈明显的白色且有一定板结。样品采集时,首先使用吊车抓机沿醅堆中线位置将一半酒醅投入窖池,进而露出醅堆的腰线,然后从醅堆截面的正中垂直方向与腰线的交界处采取200 g样品,将其定义为腰线样品,编号依次为YX1~YX4。从腰线样品采集的正下方5~10 cm处,继续采集200 g样品,定义为正常样品,编号依次为ZC1~ZC4,其中样品YX1和ZC1来自1号醅堆,以此类推。样品采集时,腰线位置的酒醅温度平均为55 ℃,正常位置酒醅的温度平均为49 ℃。
1.3.2 酒醅中微生物计数
菌落总数的测定:参照GB 4789.2—2022《食品安全国家标准食品微生物学检验菌落总数测定》;乳酸菌计数:参照GB 4789.35—2016《食品安全国家标准食品微生物学检验乳酸菌检验》;霉菌和酵母计数:参照GB 4789.15—2016《食品安全国家标准食品微生物学检验霉菌和酵母计数》。
1.3.3 酒醅中微生物宏基因组DNA的提取和Illumina MiSeq测序
使用基因组DNA提取试剂盒对酒醅中的微生物宏基因组DNA进行提取,以其为模板,使用引物对338F/806R和ITS3F/ITS4R对细菌和真菌的核苷酸序列进行PCR扩增,PCR扩增条件和PCR扩增体系参照文献[9-10],PCR产物送至上海美吉生物医药科技公司进行高通量测序,测序平台为Illumina MiSeq PE300。
1.3.4 序列质控和生物信息学分析
下机数据参照董蕴等[11]的方法进行质控,质控后的序列合并为一个fna文件后,基于QIIME(v1.9.0)平台进行生物信息学分析[12-13],其主要步骤如下:①使用PyNAST软件对高质量序列与标准数据库比对和对齐[14];②分别按照100%和97%的相似度进行UCLUST划分[15],构建非冗余数据集和操作分类单元(operational taxonomic unit,OTU);③使用ChimeraSlayer软件对OTU中的嵌合体序列进行识别[16],并删除含嵌合体序列的OTU;④从OTU中选取代表性序列,细菌基于GREENGENE[17]、SILVA[18]和核糖体数据库项目(ribosomal database project,RDP)[19]数据库进行同源性比对,只有3个数据库结果一致时才认可鉴定结果,否则取上一分类学地位鉴定结果,直到3个数据库结果一致,真菌基于UNITE数据库进行同源性比对[20];⑤使用FastTree软件绘制系统发育进化树[21],并基于此对酒醅中微生物的α多样性指数进行计算[22];⑥基于UniFrac距离进行主坐标分析(principal coordinate analysis,PCoA)[23],进而完成腰线和正常部位酒醅中细菌和真菌的β多样性分析。
1.3.5 多元统计学分析
使用Wilcoxon秩和检验对不同种类酒醅微生物的活菌数、属水平平均相对含量和α多样性指数进行显著性分析;使用Spearman相关性分析对酒醅中优势菌属之间的相关性进行计算,并使用Cytoscape软件进行相关性网络图的可视化。本研究分析和可视化使用R软件或Origin 2017软件。
2 结果与分析
2.1 基于纯培养技术腰线和正常部位酒醅各微生物类群活菌数比较分析
本研究首先采用纯培养计数的方法对腰线和正常部位酒醅中乳酸菌、细菌及酵母菌和霉菌的活菌数进行比较分析,结果见图1。
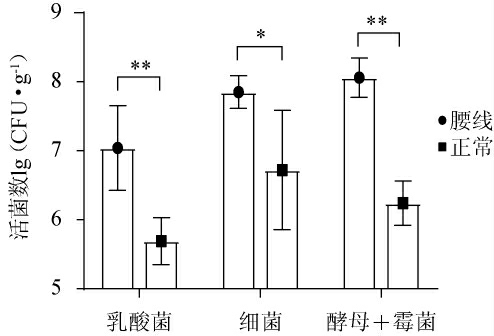
图1 基于纯培养技术腰线和正常部位酒醅各微生物类群活菌数的比较分析
Fig.1 Comparative analysis of the viable counts of each microbial community in "waistline" and normal parts of fermented grains based on pure culture technique
“*”代表差异显著(P<0.05);“**”代表差异极显著(P<0.01)。下同。
由图1可知,腰线和正常部位酒醅中的乳酸菌、细菌以及酵母和霉菌的活菌数均存在显著性差异(P<0.05,P<0.01),且在腰线部位酒醅中明显偏高,这可能与腰线形成而导致的发酵环境改变有关。因此,进一步解析腰线部位酒醅中微生物的类群则显得极为必要。
2.2 基于高通量测序腰线和正常部位酒醅的α和β多样性分析
通过对16S rRNA基因序列的MiSeq高通量测序,8个样品共产生348 216条高质量序列,平均每个样品43 527条,其中896条比对失败,共347 320条序列参与了OTU的划分,在97%的序列相似度下划分为9 698个OTU,且并未在OTU中识别到嵌合体序列,经序列比对鉴定为15个门、31个纲、48个目、96个科和152个属,8.34%的序列没有鉴定到属水平。通过对ITS序列的MiSeq高通量测序,8个样品共产生655 724条高质量序列,平均每个样品81 965条,共655 724条序列参与了OTU的划分,在97%的序列相似度下划分为1 715个OTU,且并未在OTU中识别到嵌合体序列,经序列比对鉴定为4个门、13个纲、23个目、38个科和52个属,0.36%的序列没有鉴定到属水平。通过对8个样品的细菌和真菌菌群进行高通量测序,其中样品ZC3获得的16S rRNA基因序列最少,为36 010条,因而为了使α多样性数据具有可比性,从细菌和真菌序列集中随机抽取36 010条序列进行发现物种数指数和香农指数计算,结果见图2。
由图2可知,经Wilcoxon test检验发现,腰线和正常部位酒醅细菌和真菌类群的发现物种数和香农指数均无显著差异(P>0.05)。而通过对比酒醅中细菌和真菌的α多样性发现,酒醅中细菌的发现物种数显著高于真菌(P<0.01),但香农指数无显著性差异(P>0.05)。发现物种数常用来表征微生物的丰富度,而香农指数则常用于评估微生物的多样性[24]。由此可见,腰线部位酒醅细菌和真菌的丰富度和多样性与正常部位酒醅均无显著差异(P>0.05)。
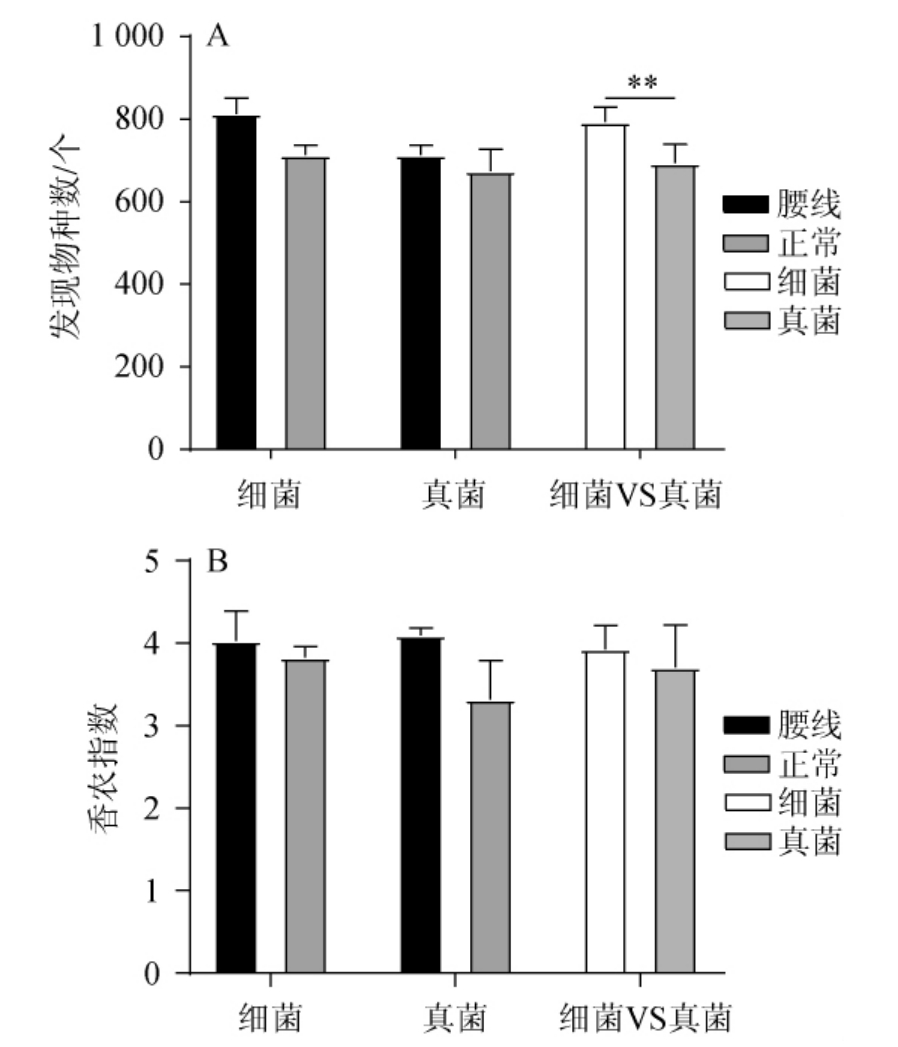
图2 腰线和正常部位酒醅微生物发现物种数(A)和香农指数(B)的比较分析
Fig.2 Comparative analysis of microbial observed species (A) and Shannon indexes (B) in "waistline" and normal parts of fermented grains
本研究进一步基于OTU水平对不同部位酒醅微生物类群的差异性进行揭示,基于加权UniFrac距离对不同部位酒醅细菌和真菌菌落结构的差异性进行分析,结果见图3。
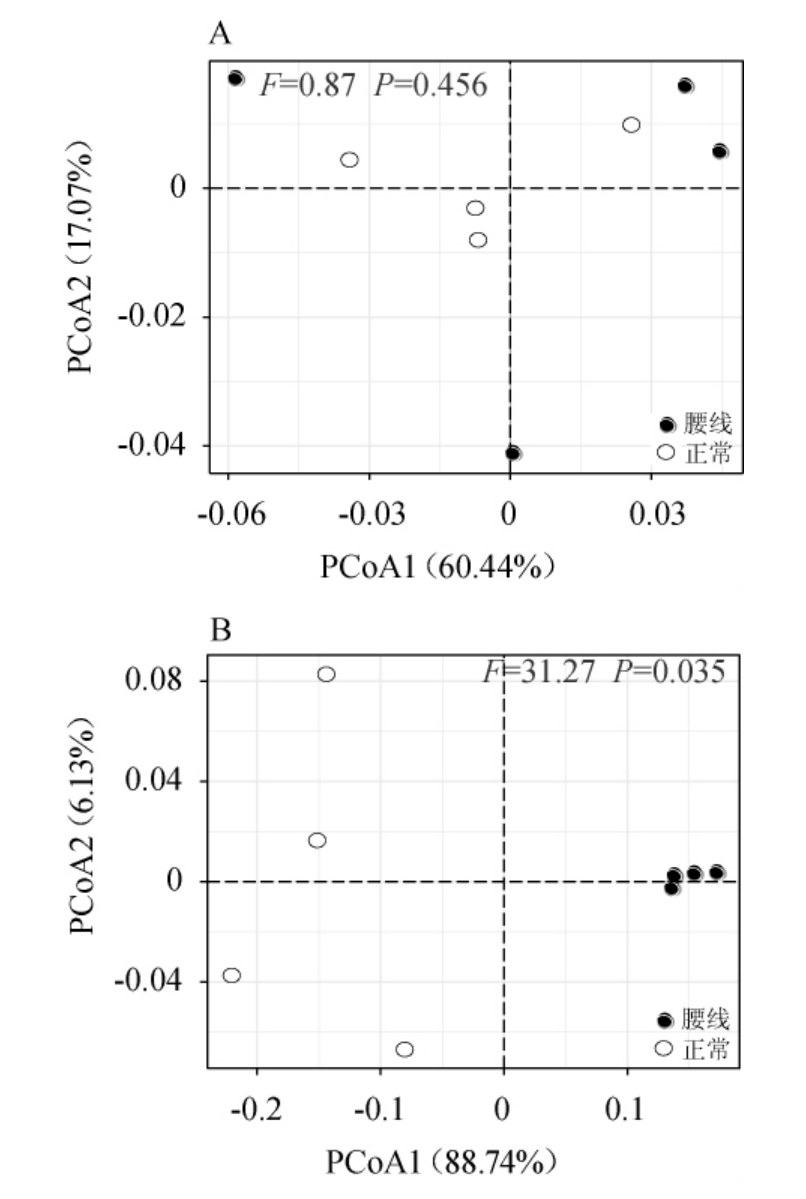
图3 基于加权UniFrac距离腰线和正常部位酒醅细菌(A)和真菌(B)类群的主坐标分析结果
Fig.3 Principal coordinate analysis results of bacterial (A) and fungal (B) communities in "waistline" and normal parts of fermented grains based on weighted UniFrac distances
由图3A可知,腰线处酒醅样品在空间上分布较为分散,而正常部位酒醅在空间分布上较为收敛,经多元统计分析发现,两组样品细菌类群差异不显著(P>0.05);由图3B可知,腰线处酒醅样品均位于X轴的正方向,而正常部位的酒醅样品则全部位于X轴的负方向,两组样品在空间分布上呈现明显的分离趋势,多元统计分析结果亦显示两组样品之间真菌类群差异显著(P<0.05)。由此可见,腰线和正常部位酒醅细菌菌群结构较为相似,而真菌菌群结构存在较大差异。
2.3 基于门和属水平腰线和正常部位酒醅微生物菌群结构解析
腰线部位酒醅中的优势细菌门(平均相对含量>1.0%)为硬壁菌门(Firmicutes),平均相对含量为98.83%,正常部位酒醅中的优势细菌门为Firmicutes和放线菌门(Actinobacteria),其平均相对含量分别为96.78%和2.04%。经Wilcoxon test检验发现,腰线部位酒醅中Actinobacteria的平均相对含量显著低于正常部位(P<0.05),其平均相对含量仅为0.22%。腰线和正常部位酒醅中的优势真菌门均为子囊菌门(Ascomycota),平均相对含量分别为99.87%和99.86%,经Wilcoxontest检验发现两者差异不显著(P>0.05)。基于属水平,腰线和正常部位酒醅微生物菌群构成见图4。

图4 基于属水平腰线和正常部位酒醅细菌(A)和真菌(B)类群的比较分析
Fig.4 Comparative analysis of bacterial (A) and fungal (B)communities in "waistline" and normal parts of fermented grains based on genus level
other表示平均相对含量<1%的门或属。
由图4A可知,热放线菌属(Thermoactinomyces)、克罗彭施泰特氏菌(Kroppenstedtia)和芽孢杆菌(Bacillus)为酒醅中的优势细菌属(平均相对含量>1.0%),其在腰线和正常部位酒醅中平均相对含量分别为66.75%和68.96%、13.62%和4.54%、8.25%和14.84%,经Wilcoxon test检验发现,Bacillus在不同部位酒醅中的平均相对含量差异显著(P<0.05)。由图4B可知,腰线部位酒醅中的优势真菌属为丝衣霉属(Byssochlamys)、毕赤酵母属(Pichia)和酵母属(Saccharomyces),其平均相对含量分别为70.10%、27.44%和1.21%,而正常部位酒醅中的优势真菌属为毕赤酵母属(Pichia)、丝衣霉属(Byssochlamys)、酵母属(Saccharomyces)、有孢圆酵母属(Torulaspora)和接合酵母属(Zygosaccharomyces),其平均相对含量分别为60.52%、15.18%、14.85%、3.00%和2.07%。经Wilcoxon test检验发现,较之正常部位酒醅,腰线部位酒醅Byssochlamys的平均相对含量显著偏高(P<0.05),而Pichia、Saccharomyces、Torulaspora和Zygosaccharomyces的平均相对含量显著偏低(P<0.05),尤其是Torulaspora和Zygosaccharomyces,其在腰线部位酒醅中的平均相对含量仅为0.72%和0.05%。有研究指出,Byssochlamys的有性繁殖会导致食品的腐败,这也可能是导致腰线处出现“霉味”的主要原因之一[25-26]。此外,科研人员发现随着腰线处霉菌的大量繁殖会对酵母菌的繁殖产生一定的抑制作用[27]。本研究进一步对腰线和正常部位酒醅优势菌属之间的相关性进行分析,结果见图5。
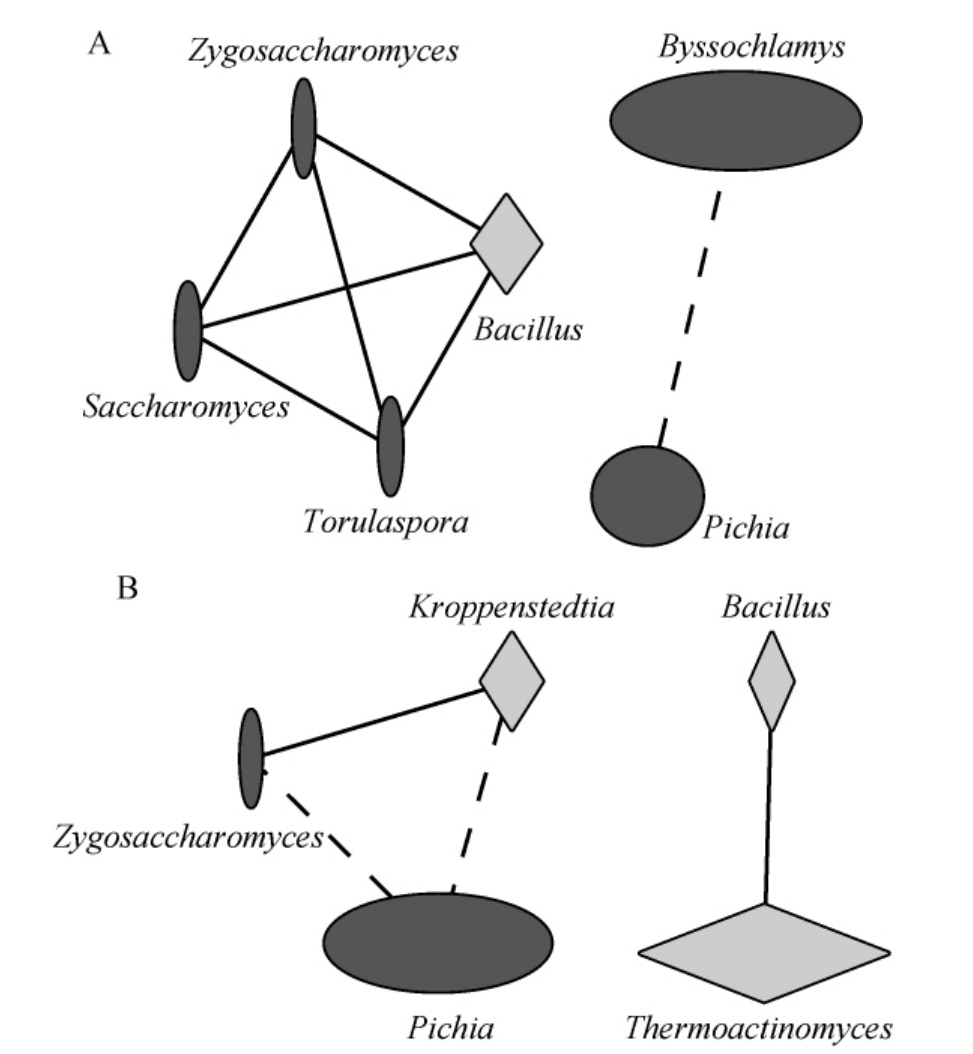
图5 腰线(A)和正常(B)部位酒醅优势微生物菌属间相关性分析网络图
Fig.5 Correlation analysis network diagram of the dominant microbial genera in "waistline" (A) and normal(B) parts of fermented grains
实线表示显著正相关(P<0.05),虚线表示显著负相关(P<0.05);连线的粗细表示相关性的大小;点的大小表示平均相对含量的大小。
由图5A可知,腰线部位酒醅5个优势真菌属和1个优势细菌属之间存在显著相关性(P<0.05),其中Zygosaccharomyces、Saccharomyces、Torulaspora与Bacillus 两 两 之 间均呈现显著正相关(P<0.05),而Byssochlamys与Pichia呈现显著负相关(P<0.05)。由图5B可知,正常部位酒醅2个优势真菌属和3个优势细菌属之间存在显著相关性(P<0.05),其中Pichia与Zygosaccharomyces和Kroppenstedtia之间呈现显著负相关(P<0.05),而Zygosaccharomyces与Kroppenstedtia,Thermoactinomyces与Bacillus之间存在显著正相关(P<0.05)。值得注意的是,腰线部位酒醅微生物菌群间存在更加密切的相关性,因而使得腰线部位酒醅微生物菌群的群落结构稳定性更强。
3 结论
本研究采用纯培养和高通量测序技术对酱香型白酒第四轮次堆积发酵醅堆腰线部位酒醅微生物菌群多样性进行解析,结果发现,腰线部位酒醅乳酸菌、菌落总数以及酵母和霉菌的活菌数均显著高于正常部位(P<0.05),但两处酒醅细菌及真菌菌群的丰度和多样性差异均不显著(P>0.05);腰线部位酒醅细菌类群与正常部位差异不显著(P>0.05),以Thermoactinomyces、Kroppenstedtia和Bacillus为主,而腰线部位酒醅真菌类群与正常部位差异显著(P<0.05),腰线部位酒醅中Byssochlamys平均相对含量显著偏高(P<0.05),而Pichia、Saccharomyces、Torulaspora和Zygosaccharomyces显著偏低(P<0.05),而相关性分析表明,腰线部位酒醅微生物菌群间相较于正常部位存在更加密切的联系。由此可见,酱香型白酒第四轮次堆积发酵醅堆腰线与正常部位酒醅的微生物类群差异主要体现在真菌类群上。
[1]WANG Y R,CAI W C,WANG W P,et al.Analysis of microbial diversity and functional differences in different types of high-temperature Daqu[J].Food Sci Nutr,2021,9(2):1003-1016.
[2]JIANG Q R,WU X Y,XU Y Q,et al.Microbial composition and dynamic succession during the Daqu production process of Northern Jiang-flavored liquor in China[J].3 Biotech,2021,11:1-15.
[3]张春林,杨亮,李喆,等.酱香型白酒二轮次堆积酒醅微生物群落与理化指标相关性研究[J].中国酿造,2021,40(11):31-36.
[4]张瀚之,席德州,王欢,等.酱香型白酒机械化酿造不同轮次堆积发酵酒醅真菌群落结构多样性研究[J].中国酿造,2021,40(12):64-69.
[5]张春林,杨亮,李喆,等.酱香型白酒酒醅堆积微生物多样性及其与风味物质的关系[J].食品科技,2022,47(4):111-118.
[6]江鹏,蒋红军,王和玉,等.酱香型白酒堆积发酵过程中“腰线”的形成机理[J].酿酒科技,2004,14(6):43-44.
[7]汪地强,陈良强,涂昌华,等.酱香型白酒堆积过程腰线酒醅浅析[J].中国酿造,2020,39(9):97-100.
[8]邵明凯,王海燕,徐岩,等.酱香型白酒发酵中酵母群落结构及其对风味组分的影响[J].微生物学通报,2014,280(12):2466-2473.
[9]HOU Q C,WANG Y R,NI H,et al.Deep sequencing reveals changes in prokaryotic taxonomy and functional diversity of pit muds in different distilleries of China[J].Ann Microbiol,2022,72(1):1-14.
[10] CAI W C,WANG Y R,NI H,et al.Diversity of microbiota,microbial functions,and flavor in different types of low-temperature Daqu[J].Food Res Int,2021,150:110734.
[11]董蕴,许小玲,代凯文,等.基于MiSeq高通量测序技术对细菌型豆豉细菌类群的评价[J].食品工业科技,2018,39(13):150-154.
[12] YANG C C,ZHAO F Y,HOU Q C,et al.PacBio sequencing reveals bacterial community diversity in cheeses collected from different regions[J].J Dairy Sci,2020,103(2):1238-1249.
[13]CAPORASO J G,KUCZYNSKI J,STOMBAUGH J,et al.QIIME allows analysis of high-throughput community sequencing data[J].Nat Methods,2010,7:335-336.
[14]NOURRISSON C,SCANZI J,BRUNET J,et al.Prokaryotic and eukaryotic fecal microbiota in irritable bowel syndrome patients and healthy individuals colonized with blastocystis[J].Front Microbiol,2021,12:713347.
[15]GORDIN E,GORDIN D,VIITANEN S,et al.Urinary clusterin and cystatin B as biomarkers of tubular injury in dogs following envenomation by the European adder[J].Res Vet Sci,2021,134:12-18.
[16] EDGAR R C,HAAS B J,CLEMENTE J C,et al.UCHIME improves sensitivity and speed of chimera detection[J].Bioinformatics,2011,27(16):2194-2200.
[17] DESANTIS T Z,HUGENHOLTZ P,LARSEN N,et al.Greengenes,a chimera-checked 16S rRNA gene database and workbench compatible with ARB[J].Appl Environ Microb,2006,72(7):5069-5072.
[18] VAN DER POL W J,KUMAR R,MORROW C D,et al.In silico and experimental evaluation of primer sets for species-level resolution of the vaginal microbiota using 16S ribosomal RNA gene sequencing[J].J Infect Dis,2019,219(2):305-314.
[19] SALIH T S,SAEED H K,IBRAHEEM M A.Specialized high-quality 16S ribosomal RNA gene databases for identification of bacterial taxonomic groups:a review[J].Rafidain J Sci,2021,30(2):40-46.
[20] NILSSON R H,LARSSON K H,TAYLOR A F S,et al.The UNITE database for molecular identification of fungi: handling dark taxa and parallel taxonomic classifications[J].Nucleic Acids Res,2019,47(D1):D259-D264.
[21]PIÑEIRO C,ABUÍN J M,PICHEL J C.Very Fast Tree:speeding up the estimation of phylogenies for large alignments through parallelization and vectorization strategies[J].Bioinformatics,2020,36(17):4658-4659.
[22]WANG Y R,XIANG F S,ZHANG Z D,et al.High-throughput sequencing-based analysis of fungal diversity and taste quality evaluation of Douchi,a traditional fermented food[J].Food Sci Nutr,2020,8(12):6612-6620.
[23]WANG L L,ZHANG F Y,DONG W W,et al.A novel approach for the forensic diagnosis of drowning by microbiological analysis with next-generation sequencing and unweighted UniFrac-based PCoA[J].Int J Legal Med,2020,134(6):2149-2159.
[24]姚国强.传统发酵乳中细菌多样性及其功能基因研究[D].呼和浩特:内蒙古农业大学,2017.
[25]HOUBRAKEN J,VARGA J,RICO-MUNOZ E,et al.Sexual reproduction as the cause of heat resistance in the food spoilage fungus Byssochlamys spectabilis(anamorph Paecilomyces variotii)[J].Appl Environ Microbiol,2008,74(5):1613-1619.
[26]LÓPEZ-FERNÁNDEZ S,CAMPISANO A,SCHULZ B J,et al.Viriditins from Byssochlamys spectabilis,their stereochemistry and biosynthesis[J].Tetrahedron Lett,2020,61(5):151446.
[27]庄名扬,孙达孟.酱香型白酒高温堆积糟醅中酵母菌分离、选育及其分类学鉴定[J].酿酒,2003,28(2):12-13.