山西老陈醋历史悠久,因具有绵、酸、甜、香、鲜等特点而位居“四大名醋”之首。但是山西老陈醋生产依然面临着很多问题,如生产机械化程度低、原辅料利用率低,导致食醋成本较高[1]。当地企业为降低成本,进行了一定的工艺改良,形成了多种地方酿造技艺,有回糟固态发酵工艺[2]、生料发酵工艺[3]和多微液态发酵工艺[4]。其中具有代表性的是怀仁醋酿造工艺(团体标准T/SXMYT 0601—2020《怀仁醋酿造工艺规程》),它与山西老陈醋工艺(国标GB/T 19777—2013《地理标志产品山西老陈醋》)的醋酸发酵阶段是一致的,差别主要在酒精发酵阶段,两种工艺虽然都使用了传统大曲,但是用量不同,山西老陈醋工艺中大曲用量仅为62.5%,而怀仁醋酿造工艺除了使用30%大曲以外,还使用了一定量的酶制剂和纯化酵母。众所周知,食醋酿造是特定工艺下的微生物种群动态演替的过程,酿造工艺的不同会直接影响微生物的种群结构,进而导致其代谢产物的不同,最终体现为食醋品质的差异。因此,解析食醋不同酿造工艺下的微生物种群结构,对于食醋酿造工艺的改进至关重要。
目前,针对山西食醋工艺中微生物菌群的研究材料大多是山西老陈醋[5-7],但是对基于山西老陈醋工艺发展起来的其他食醋工艺对微生物菌群影响的研究较少。近些年,研究山西食醋微生物菌群多采用高通量测序技术,该技术具有通量大、快速、高效方便等特点。范三红等[8]对山西陈醋动态酿造过程的细菌菌群进行高通量测序分析,结果显示,魏斯氏菌属(Weissella)和乳杆菌属(Lactobacillus)是酒精发酵阶段的主要优势菌;寇蓉等[9]对山西陈醋酿造微生物的研究也得出相似的结论,酒精发酵阶段乳杆菌属(Lactobacillus)相对含量最高,除此之外是醋酸杆菌属(Acetobacter)、魏斯氏菌属(Weissella)和假单胞菌属(Pseudomonas)。
本研究通过高通量测序手段,比较了山西老陈醋和怀仁醋酒精发酵阶段样品的细菌菌群差异,旨在为怀仁醋工艺的改进提供一定的指导,并在一定程度上能更好地对山西老陈醋工艺进行创新传承。
1 材料与方法
1.1 材料与试剂
1.1.1 样品
从山西省晋中市榆次区某大型醋厂,于酒精发酵第3天分别采集山西老陈醋工艺的发酵样品(编号S)和怀仁醋酿造工艺的发酵样品(编号X),各3个平行,置于-80 ℃备用。
1.1.2 试剂
TGuide S96磁珠法土壤/粪便基因组脱氧核糖核酸(deoxyribonucleic acid,DNA)提取试剂盒:天根生化科技(北京)有限公司;OMEGA DNA纯化试剂盒:美国Bio-Tek公司;琼脂糖凝胶回收试剂盒:日本宝生物工程株式会社;2×Phusion High-Fidelity聚合酶链式反应(polymerase chain reaction,PCR)Master Mix:美国纽英伦生物技术公司;Ion Plus建库试剂盒:美国赛默飞世尔科技公司;其他试剂均为国产分析纯。
1.2 仪器与设备
W5L-1恒温水浴槽:杭州瑞诚仪器有限公司;SL8高速离心机、Veriti Dx PCR热循环仪:美国Thermo Scientific公司;XH-B漩涡振荡仪:无锡沃信仪器制造有限公司;Mini-20雪花制冰机:上海豫明仪器有限公司;DYY-6C型电泳仪:北京六一仪器厂;GelDoc2000UV凝胶成像系统:美国Bio-Rad公司;Nova-Seq6000测序仪:美国Illumina公司。
1.3 方法
1.3.1 样品细菌基因组DNA的提取及检测
将样品用磷酸缓冲液(phosphate buffered solution,PBS)洗涤3次后收集菌体,采用十六烷基三甲基溴化铵(cetyl trimethyl ammonium bromide,CTAB)法提取菌体的基因组DNA,采用0.8%琼脂糖凝胶电泳检测基因组DNA提取效果。
1.3.2 16S rRNA V4区基因的获得、文库的构建及高通量测序
以提取的基因组DNA为模板,采用引物515F(5'-TGCCAGCMGCCGCGGTAA-3')和806R(5'-GGACTACHVGGGTTCTAAT-3')PCR扩增16S rRNA基因V4区基因序列。PCR扩增体系:基因组DNA10μL(5~10ng),515F(2μmol/L)3 μL,806R(2 μmol/L)3 μL,2×Mix 15 μL,双蒸水(ddH2O)补至30 μL。PCR扩增条件:94 ℃预变性5 min;94 ℃变性30 s,55 ℃退火30 s,72 ℃延伸90 s,共35个循环;72 ℃再延伸10 min。通过2%琼脂糖凝胶电泳对PCR扩增产物进行检测。采用合格的扩增产物构建文库,将质检合格的文库委托百迈客生物工程有限公司进行高通量测序。
1.3.3 生物信息分析
使用FLASH v1.2.7软件对测序数据进行拼接,使用Trimmomatic v0.33软件对拼接序列进行过滤,使用UCHIME v4.2软件去除嵌合体序列,获得有效序列。使用Usearch软件,将序列相似性达到97%以上的有效序列归为同一个操作分类单元(operational taxonomic units,OTU),即分子水平的“种”[10]。选取每一个OTU的代表序列,在美国国立生物技术信息中心(national center for biotechnology information,NCBI)的GenBank数据库中通过基本局部比对搜索工具(basic local alignment search tool,BLAST)进行物种注释。
1.3.4 多样性分析
采用Mothur(网址:http://mothur.org/wiki/download_mothur/)计算文库的库容Coverage和样品的α-多样性指数(香农(Shannon)指数、辛普森(Simpson)指数、超1(Chao 1)指数)和β-多样性[11]。
1.3.5 物种组成分析
基于微生物多样性分析软件Qiime 2[12]和核糖体数据库项目(ribosomal database project,RDP)的贝叶斯算法对OTU代表序列进行分类学注释[13],参照SILVA核糖体核糖核酸(ribosomal ribonucleic acid,rRNA)数据库[14],分别在门和属水平统计各样品的群落组成,再利用R语言工具绘制成样品门和属水平下的群落结构柱状图。
1.3.6 LEfSe分析
采用线性判别分析效应大小(line discriminant analysis effect size,LEfSe)(http://huttenhower.sph.harvard.edu/galaxy/root?_id=lefse_upload)获得造成样品间显著性差异的物种[15]。
1.3.7 物种相关性分析
选取属水平总相对丰度排名前50的物种,计算物种之间的斯皮尔曼(Spearman)相关系数,筛选相关性>0.1且P<0.05的数据,利用Gephi 0.9.2软件进行物种相关网络图的绘制[16]。
2 结果与分析
2.1 两种食醋酿造工艺酒精发酵阶段样品中细菌菌群的OTU分析
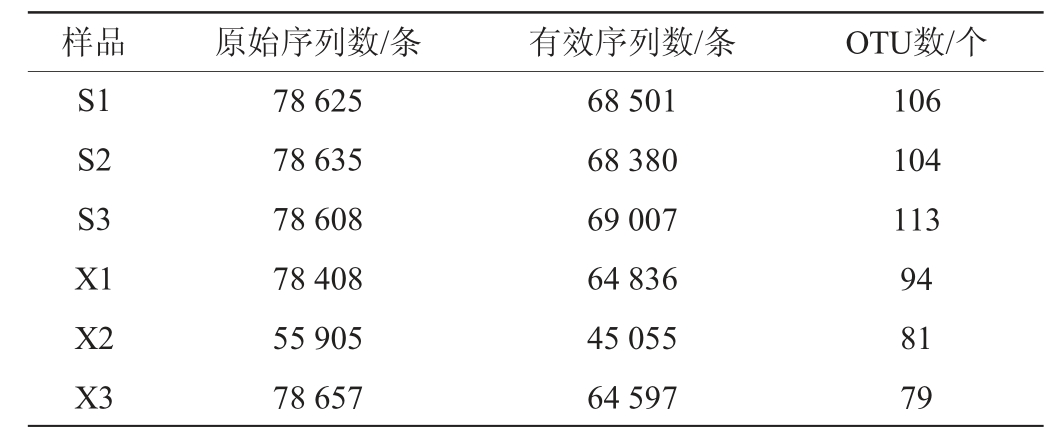
两种样品细菌菌群的原始序列数、有效序列数和OTU数统计结果见表1。由表1可知,山西老陈醋工艺酒精发酵阶段样品的有效序列数及OTU数均高于怀仁醋工艺酒精发酵阶段样品,说明山西老陈醋工艺样品的细菌种类多于怀仁醋工艺样品。
表1 两种食醋酿造工艺样品细菌菌群的高通量测序结果及操作分类单元数
Table 1 High-throughput sequencing results and operational taxonomic unit numbers of bacterial diversity of vinegar samples made by two brewing processes
韦恩(Venn)图可以展示样品之间共有、特有的OTU数目,两种食醋工艺酒精发酵阶段样品细菌菌群的OTU韦恩图见图1。由图1可知,两种食醋酿造工艺酒精发酵阶段样品的共有序列数有93条;山西老陈醋工艺样品的特有序列数为77条,而怀仁醋工艺样品的特有序列数为42条,说明食醋的不同酿造工艺对酒精发酵时期样品中细菌物种数量有较大影响。
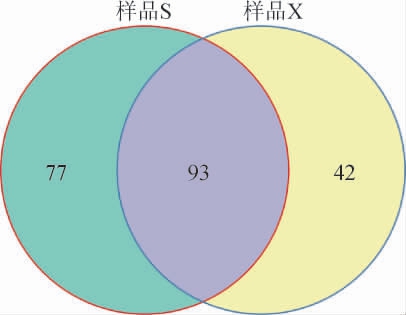
图1 两种食醋酿造工艺样品细菌菌群的操作分类单元韦恩图
Fig.1 Venn figure of operational taxonomic unit of bacterial flora in vinegar samples made by two brewing processes
稀释曲线用于验证测序数据量是否足够反映样品的物种多样性,当曲线逐渐平坦时,说明测序深度基本可以覆盖样品中的所有物种[17]。样品物种的丰富度和均匀度则是通过丰度等级聚类曲线体现的,在横轴方向上,曲线越宽,则物种丰富度越高;在纵轴方向上,曲线越平坦,则物种均匀度越高[18]。两种食醋酿造工艺酒精发酵阶段样品细菌菌群的稀释曲线及丰度等级聚类曲线见图2。

图2 两种食醋酿造工艺样品细菌菌群的稀释曲线(a)和等级聚类曲线(b)
Fig.2 Rarefaction curves (a) and hierarchical clustering curves (b) of vinegar samples made by two brewing processes
由图2a可知,随着测序深度的增加,两种工艺样品的稀释曲线基本逐渐平缓,说明测序量达到基本要求。由图2b可知,与怀仁醋工艺样品相比,山西老陈醋工艺样品的丰度等级聚类曲线跨度更大、更平缓,说明其物种更丰富、分布更均匀。
2.2 两种食醋酿造工艺酒精发酵阶段样品中细菌菌群的多样性分析
2.2.1 α-多样性分析
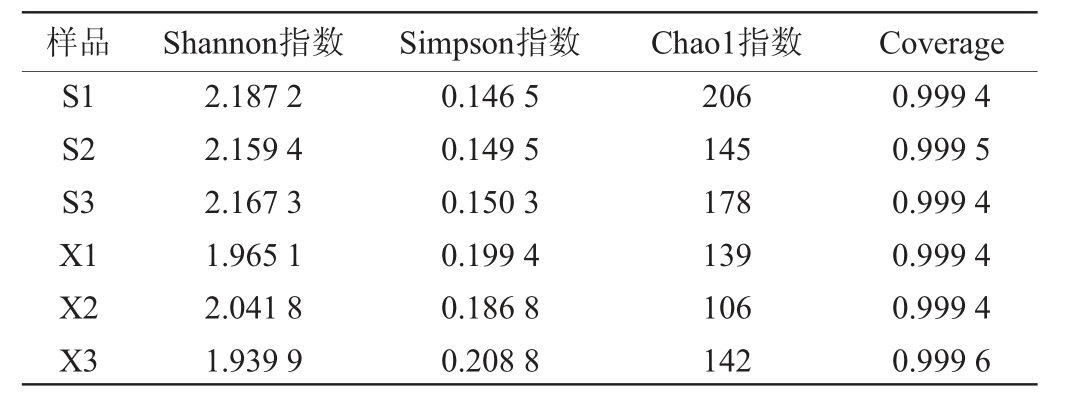
Coverage反映了测序的库容,数值越大,表明库容越大,越能反映样品的真实情况[17]。样品的α-多样性程度通常用Shannon指数和Simpson指数表示,Shannon指数越大,Simpson指数越小,说明样品内物种多样性越高[19-20],而Chao1指数可以预测样品中细菌的总种类数[21]。两种食醋酿造工艺酒精发酵阶段样品中细菌菌群的α-多样性分析结果见表2。由表2可知,山西老陈醋和怀仁醋酿造工艺样品的Coverage均>0.99,说明库容均满足了基本要求。与山西老陈醋工艺的样品相比,怀仁醋工艺样品的Shannon指数更小,Simpson指数更大,说明怀仁醋工艺降低了样品细菌菌群的多样性。由表2亦可知,怀仁醋工艺样品的Chao1指数低于山西老陈醋工艺样品的Chao1指数,表明怀仁醋酿造工艺降低了样品中细菌物种的丰富度。
表2 两种食醋酿造工艺样品细菌菌群的α-多样性分析结果
Table 2 α-diversity analysis results of bacterial flora in vinegar samples made by two brewing processes
2.2.2 β-多样性分析
主成分分析(principal component analysis,PCA)是一种分析和简化数据集的技术,通过将方差进行分解,将多组数据的差异反映在二维坐标图上,两个样品距离越近,则表示这两个样品的组成越相似[22]。基于OTU对两种样品进行主成分分析,结果见图3。由图3可知,在酒精发酵过程中,山西老陈醋工艺的样品与怀仁醋工艺的样品之间的距离很远,说明两种工艺样品的细菌组成差别很大。
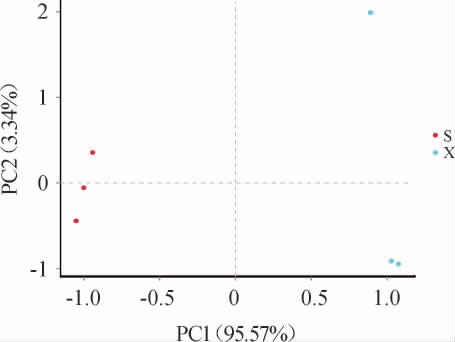
图3 基于操作分类单元两种食醋酿造工艺样品的主成分分析结果
Fig.3 Result of principal component analysis of vinegar samples made by two brewing processes based on operational taxonomic unit
2.3 两种食醋酿造工艺酒精发酵阶段样品中细菌群落结构的组成
2.3.1 细菌菌群在门水平上的组成
在门水平上,两种工艺样品的细菌群落结构见图4。由图4可知,从两个工艺样品中共注释到11个细菌门,在山西老陈醋工艺样品和怀仁醋工艺样品中分别为11个和9个。样品中平均相对丰度排名前10的细菌门分别为厚壁菌门(Firmicutes)、变形菌门(Proteobacteria)、放线菌门(Actinobacteria)、蓝藻门(Cyanobacteria)、拟杆菌门(Bacteroides)、梭杆菌门(Fusobacteria)、埃普西隆杆菌门(Epsilonbacter aeota)、绿弯菌门(Chloroflexi)、脱铁杆菌门(Deferribacteres)和未确定分类地位(unclassified)。在山西老陈醋工艺样品中,除未确定分类地位(unclassified)以外,其他9个门均能检测到,其中,占绝对优势的细菌门为厚壁菌门。除脱铁杆菌门外,其余9个细菌门均存在于怀仁醋工艺样品中,绝对优势细菌门也是厚壁菌门。与范三红等[8-9]的研究结果类似,厚壁菌门在两种工艺样品中均是绝对优势类群,且在怀仁醋工艺样品中的平均相对丰度(88.48%)低于山西老陈醋工艺样品(97.21%)。此外,变形菌门、放线菌门和蓝藻门在山西老陈醋工艺样品中的相对丰度也高于怀仁醋酿造工艺样品。
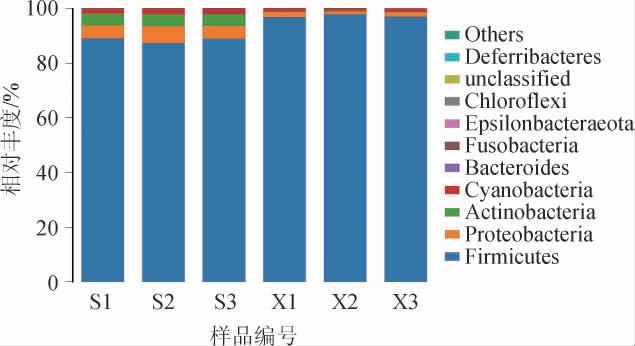
图4 基于门水平两种食醋酿造工艺样品的细菌菌群结构
Fig.4 Bacterial community structure of vinegar samples made by two brewing processes based on phylum level
Others:平均相对丰度排名在10以后的细菌门。
2.3.2 细菌菌群在属水平上的组成
在属水平上,两种工艺样品的细菌群落结构见图5。由图5可知,从两个工艺样品中共注释到123个细菌属,在山西老陈醋工艺样品和怀仁醋工艺样品中分别为123个和98个。样品中平均相对丰度排名前10的细菌属分别为乳杆菌属(Lactobacillus)、片球菌属(Pediococcus)、明串珠菌属(Leuconostoc)、魏斯氏菌属(Weissella)、泛菌属(Pantoea)、链霉菌属(Streptomyces)、乳球菌属(Lactococcus)、短状杆菌属(Brachybacterium)和肠杆菌属(Enterobacter)、未培养细菌(uncultured bacterium)。这10个细菌属在两种工艺样品中均存在。在山西老陈醋工艺样品中,乳杆菌属、片球菌属、明串珠菌属的平均相对丰度较高,分别为41.87%、28.13%、17.03%;而在怀仁醋工艺样品中,乳杆菌属、魏斯氏菌属、片球菌属的平均相对丰度较高,分别为73.74%、15.01%、5.53%。第一优势属乳杆菌属在怀仁醋工艺样品中的平均相对丰度远高于山西老陈醋工艺样品。山西老陈醋工艺样品的第二优势属片球菌属则成为怀仁醋工艺样品中的第三优势属。在山西老陈醋工艺样品中的第三优势属明串珠菌属在怀仁醋工艺样品中的平均相对丰度较低;相反,数量极少的魏斯氏菌属则成为怀仁醋工艺样品中的优势细菌属。另外,在山西老陈醋工艺样品中存在的泛菌属、链霉菌属、短状杆菌属、肠杆菌属(Enterobacter)和非培养细菌(uncultured bacterium)在怀仁醋工艺样品中的数量都有所降低;相反,在怀仁醋工艺样品中较多的乳球菌属则在山西老陈醋工艺样品中极少。
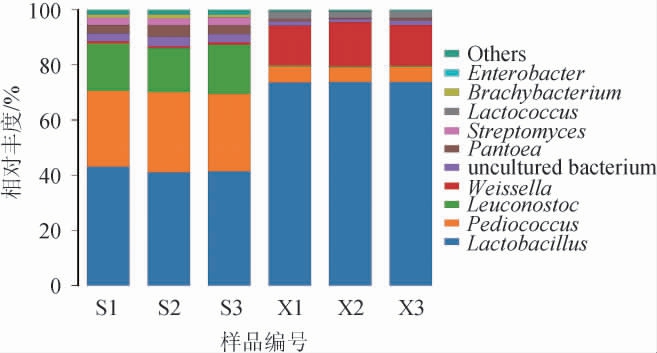
图5 基于属水平两种食醋酿造工艺样品的细菌菌群结构
Fig.5 Bacterial community structure of vinegar samples made by two brewing processes based on genus level
Others:平均相对丰度排名在10以后的细菌属。
综上,山西老陈醋工艺和怀仁醋工艺样品的主要细菌均为乳酸菌。乳酸菌产生的乳酸是老陈醋的第二主体酸[23],不仅能降低乙酸的刺激口感、平衡酸味,使得食醋更加柔和[24-25],还可生成重要功能物质川芎嗪的前体物质—乙偶姻,提高食醋的保健功能[23,26-27]。乳酸菌产生的蛋白酶能水解蛋白质为氨基酸,赋予食醋醇厚风味[28]。另外,乳酸菌还能产生少量的天然抑菌剂—乳酸链球菌素,具有非常好的抑菌效果,有助于延长食醋的保存期[29]。
本研究中,乳杆菌属在山西老陈醋工艺和怀仁醋工艺的样品中都是第一优势菌,这与聂志强等[30-32]的研究报道相一致。通常,醋醅的微生物类群受大曲微生物类群的影响巨大。已有研究表明,山西食醋大曲中优势微生物主要是芽孢杆菌属(Bacillus)和魏斯氏菌属,并不是乳杆菌属[33-34]。结合本研究的两种工艺样品均取自酒精发酵中期,但并未检测到高水平的芽孢杆菌属,反而是乳杆菌属占据优势。这可能是芽孢杆菌属在食醋酿造酒精发酵阶段的前期发挥作用,提供了乳杆菌属生长繁殖的营养物质。尽管乳杆菌属在两种工艺样品中都是第一优势属,但是其在怀仁醋工艺样品中的相对丰度更高。这可能是由于怀仁醋工艺使用了酶制剂,能快速分解高粱原料,为乳杆菌属细菌的迅速生长提供了大量前体物质,从而产生了大量乳酸[35]。但与此同时,怀仁醋工艺的大曲用量减半,致使细菌菌群的多样性降低。
本研究结果与其他文献结果有所差异[30-34],导致这些差异化的因素包括酿造原料、大曲、酿造工艺等。山西老陈醋的酿造主料是高粱,高粱单宁对发酵微生物的生长有一定的影响作用[36];不同高粱子粒的糊化差异较大[37],对润粮和糖化的影响较大[38],进而影响发酵过程中微生物的菌群类型。山西老陈醋酿造的大曲为清香型大曲,来自清徐和榆次周边的大曲基地,微生物类群相对稳定,但其他地区醋厂所用大曲的来源不一定相同;而不同来源的大曲的微生物差异较大[39],从而造成酿造过程中微生物种群的不同。在后续研究中,需要将山西老陈醋工艺和怀仁醋工艺的细菌菌群与各自产品的理化成分、香味物质等指标结合分析,才能较全面、较科学地指导酿造工艺的改进和创新。
2.4 两种食醋酿造工艺酒精发酵阶段样品的LEfSe分析
LEfSe可以发现不同样品间在丰度上有显著差异的物种[15],两种食醋酿造工艺酒精发酵阶段样品的LEfSe分析结果见图6。图6展示了线性判别分析(linear discriminant analysis,LDA)分值大于设定值(4.0)的物种,不同颜色表示不同样品的物种,柱状图的长度代表差异物种的影响大小。由图6可知,在所有细菌属中,导致两种工艺样品有差异的细菌属有7个,在山西老陈醋工艺样品中为片球菌属(Pediococcus)、明串珠菌属(Leuconostoc)、链霉菌属(Streptomyces)和泛菌属(Pantoea),其中片球菌属的影响最大;而在怀仁醋工艺样品中则为乳杆菌属(Lactobacillus)、魏斯氏菌属(Weissella)和乳球菌属(Lactococcus),其中乳杆菌属的影响最大。
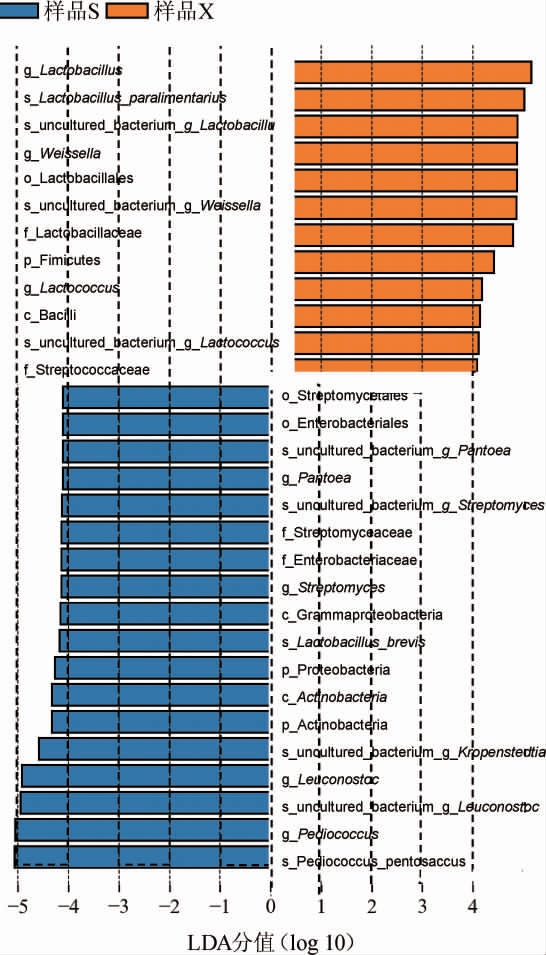
图6 两种食醋酿造工艺样品的LEfSe分析结果
Fig.6 LEfSe analysis results of vinegar samples made by two brewing processes
2.5 两种食醋酿造工艺酒精发酵阶段样品中细菌属间相关性分析
基于各个细菌属在各个样品中的丰度及变化情况,绘制物种相关性网络图,从而可以获得物种在环境样本中的共存关系,得到物种在同一环境下的相互作用情况[40]。本研究选取山西老陈醋和怀仁醋工艺酒精发酵阶段样品中相关性最高的前50个细菌属绘制物种相关性网络图,结果见图7。图7中网络节点大小代表该物种丰度高低;连线颜色表示物种间的相关性(红色表示正相关,绿色表示负相关),线的粗细表示相关性大小(线粗表示相关性高),线的多少表示该物种与其他物种的联系密切程度。
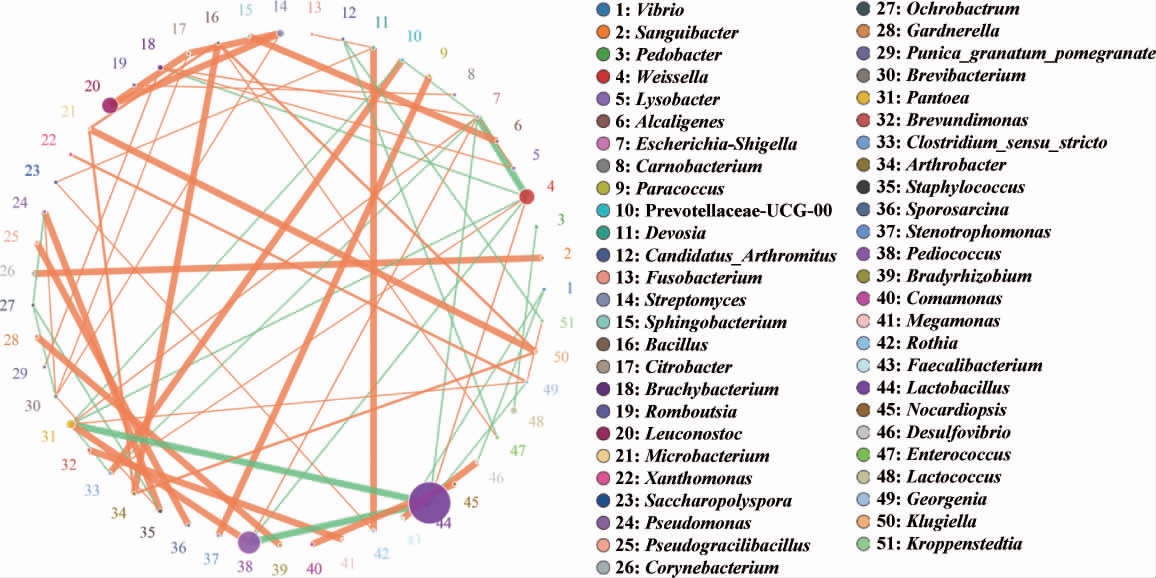
图7 两种食醋酿造工艺样品中细菌属相关性分析网络图
Fig.7 Correlation analysis network diagram of bacterial genera in vinegar samples made by two brewing processes
由图7可知,连接节点最多的是乳杆菌属(Lactobacillus)、泛菌属(Pantoea)和埃希氏菌属-沙门氏菌属(Escherichia-Shigella)。乳杆菌属在山西老陈醋工艺和怀仁醋工艺样品中均是第一优势属,其与Faecalibacterium和脱硫弧菌属(Desulfovibrio)高度正相关,与魏斯氏菌属(Weissella)呈正相关;而与片球菌属(Pediococcus)和泛菌属(Pantoea)高度负相关,与埃希氏菌属-沙门氏菌属(Escherichia-Shigella)和乔治菌属(Georgenia)呈负相关。泛菌属虽然并不是两种工艺样品的主要优势属,而且在怀仁醋工艺样品中的相对丰度少于山西老陈醋工艺样品,但是它却与片球菌属(Pediococcus)高度正相关、与Georgenia和埃希氏菌属-沙门氏菌属(Escherichia-Shigella)正相关;与乳杆菌属(Lactobacillus)高度负相关,与魏斯氏菌属(Weissella)、短波单胞杆菌属(Brevundimonas)和Provotellaceae-UCG-00负相关。
连接节点居第二位的是魏斯氏菌属(Weissella),其在怀仁醋工艺样品是第二优势属,与乳杆菌属(Lactobacillus)和梭菌属(Clostridium)呈正相关;而与埃希氏菌属-沙门氏菌属(Escherichia-Shigella)高度负相关,与短状杆菌属(Brachybacterium)和泛菌属(Pantoea)呈负相关。连接节点居第三位的是短杆菌属(Brevibacterium)和克鲁格氏菌属(Klugiella)。连接节点居第四位的是片球菌属(Pediococcus)和短状杆菌属(Brachybacterium)。片球菌属在山西老陈醋工艺样品中是第二优势属,在怀仁醋工艺样品是第三优势属,其与泛菌属(Pantoea)高度正相关,与埃希氏菌属-沙门氏菌属(Escherichia-Shigella)呈正相关;与乳杆菌属(Lactobacillus)呈高度负相关,与魏斯氏菌属(Weissella)呈负相关。
研究表明,通过网络中微生物间的相互作用可以推测[41-43],处于互利共生关系的微生物呈正相关[44],而处于竞争关系的微生物呈负相关[45]。因此,在山西老陈醋工艺和怀仁醋工艺样品中的第一优势属乳杆菌属(Lactobacillus)与Faecalibacterium和脱硫弧菌属(Desulfovibrio)处于高度互利共生关系,与魏斯氏菌属(Weissella)为互利共生关系。有研究发现,在山西老陈醋的酒精发酵过程中,乳杆菌属及魏斯氏菌属与酸度呈正相关,乳杆菌属与乳酸、乙酸、柠檬酸、丙酮酸、草酸和琥珀酸有较高相关性,魏斯氏菌属与3种酯类相关性较高[35]。本研究中的第一优势属乳杆菌属与片球菌属(Pediococcus)、泛菌属(Pantoea)为高度竞争关系,与埃希氏菌属-沙门氏菌属(Escherichia-Shigella)和Georgenia存在竞争关系。张天震[35]发现,山西老陈醋的酒精发酵中的片球菌属与温度呈正相关,与3种酯类相关性较高;泛菌属与pH呈正相关。由此可见,山西老陈醋发酵过程中细菌菌群的改变会影响发酵产物的种类。而发酵过程中的细菌菌群之间通过何种方式相互影响,有待深入研究。因此,对山西老陈醋工艺的改进,包括大曲的用量和酶制剂的使用等必须要考虑对第一优势属及其相关细菌菌群的影响,再结合产品的性状综合考虑工艺改进的方向。
3 结论
本研究利用高通量测序技术对山西老陈醋工艺(S)和怀仁醋酿造工艺(X)酒精发酵中期样品中的细菌群落结构进行分析。多样性分析结果表明,S样品的细菌菌群多样性更高。两种工艺样品的细菌菌群组成差别较大,与S样品相比,X样品中相对丰度较高的乳杆菌属(Lactobacillus)、魏斯氏菌属(Weissella)、乳球菌属(Lactococcus)均增多,片球菌属(Pediococcus)、明串珠菌属(Leuconostoc)、泛菌属(Pantoea)、链霉菌属(Streptomyces)、短状杆菌属(Brachybacterium)、肠杆菌属(Enterobacter)和非培养细菌(uncultured bacterium)均减少。LEfse分析结果表明,引起两种工艺样品有显著差异的细菌在S样品中为片球菌属、明串珠菌属、链霉菌属和泛菌属,在X样品中则为乳杆菌属、魏斯氏菌属和乳球菌属。物种相关性分析表明,乳杆菌属、泛菌属、魏斯氏菌属和片球菌属与其他物种的联系紧密。
[1]程程.山西老陈醋风味分析及品质改良[D].天津:天津科技大学,2018.
[2]周景丽,闫裕峰,梁楷,等.醋糟续米查回活醋酸发酵生产陈醋工艺的研究[J].中国酿造,2018,37(5):114-119.
[3]张永杰,崔宁波,张丽珍,等.基于DNA高通量测序分析生料酿醋过程中的真菌多样性[J].微生物学报,2020,60(7):1358-1369.
[4]赵文良.老陈醋生产中高粱酒精发酵特性的研究[D].太原:山西大学,2016.
[5]柴鹏飞.山西老陈醋发酵过程潜在的生物催化脱氢反应分析[D].天津:天津科技大学,2020.
[6]朱丹,康帅帅,王宏霞,等.山西老陈醋压醅后熟过程中微生物和风味物质的动态变化规律[J].中国酿造,2022,41(1):48-54.
[7]王家东,刘丹彤,王馨宇,等.山西老陈醋酿造过程四甲基吡嗪形成规律及稳定性研究[J].食品与发酵工业,2022,48(15):62-68.
[8]范三红,薛虎贵,白宝清,等.山西陈醋发酵过程微生物群落动态分析及差异菌属筛选[J].食品工业科技,2022,43(24):171-179.
[9]寇蓉,董弘毅,何永吉,等.山西陈醋酿造微生物群落结构及其互作网络分析[J].中国酿造,2022,41(4):59-64.
[10]MCCCAIG A E,GLOVER L A,PROSSER J I.Molecular analysis of bacterial community structure and diversity in unimproved and improved upland grass pastures[J].Appl Environ Microbiol,1999,65(4):1721-1730.
[11]SCHLOSS P D,WESTCOTT S L,RYABIN T,et al.Introducing mothur:open-source,platform-independent,community-supported software for describing and comparing microbial communities[J].Appl Environ Microbiol,2009,75(23):7537-7541.
[12]HALL M,BEIKO R G.16S rRNA gene analysis with QIIME2[J].Methods Mol Biol,2018,1849:113-129.
[13]QUAST C,PRUESSE E,YILMAZ P,et al.The SILVA ribosomal RNA gene database project:improved data processing and web-based tools[J].Nucleic Acids Res,2013,41(D1):D590-D596.
[14]COLE J R,WANG Q,FISH J A,et al.Ribosomal database project:data and tools for high throughput rRNA analysis[J].Nucleic Acids Res,2014,42(D1):D633-D642.
[15]SEGATA N,IZARD J,WALDRON L,et al.Metagenomic biomarker discovery and explanation[J].Genome Biol,2011,12(6):1-18.
[16]黄博聪,龙健,刘灵飞,等.锑矿周边稻田土壤垂直剖面锑砷形态与细菌群落结构分布及相互关系[J].环境科学学报,2019,39(4):1274-1283.
[17]GOOD I J.The population frequencies of species and the estimation of population parameters[J].Biometrika,1953,40:237-264.
[18]LUNDBERG D S,YOURSTONE S,MIECZKOWSKI P,et al.Practical innovations for high-throughput amplicon sequencing[J].Nat Methods,2013,10(10):999-1002.
[19]张和钰,周华荣,叶琴,等.新疆额尔齐斯河流域典型地区灌木群落多样性[J].生态学杂志,2016,35(5):1188-1196.
[20]郑绍伟,陈泓,黎燕琼,等.岷江上游干旱河谷区植被多样性与环境因子的相关性研究[J].四川林业科技,2009,30(6):21-27.
[21]蒋旭瑶,吉喜燕,黄德英,等.不同植物类型复合垂直流人工湿地根系微生物群落结构的研究[J].农业环境科学学报,2019,38(1):176-183.
[22]DUBOIS P C,TRYNKA G,FRANKE L,et al.Multiple common variants for celiac disease influencing immune gene expression[J].Nat Genet,2010,42(4):295-302.
[23]NIE Z,ZHENG Y,DU H,et al.Dynamics and diversity of microbial community succession in traditional fermentation of Shanxi aged vinegar[J].Food Microbiol,2015,47:62-68.
[24]邝格灵,张洁,孔德华,等.植物乳杆菌与解淀粉芽孢杆菌对食醋风味的影响[J].中国酿造,2018,37(6):25-29.
[25]沈咪娜.镇江香醋微生物群落乳酸代谢途径研究[D].无锡:江南大学,2019.
[26]王丽娟.产乙偶姻功能微生物群落的分离及其在食醋酿造中的应用[D].无锡:江南大学,2018.
[27]任聪,杜海,徐岩.中国传统发酵食品微生物组研究进展[J].微生物学报,2017,57(6):885-898.
[28]ZHAO H L,LI Z X,YANG X M,et al.Vinegar identification byultraviolet spectrum technology and pattern recognition method[J].Sci Magna,2009,5(3):104-111.
[29]刘杨岷,张家骊,王利平,等.食醋风味成分比较研究[J].食品与机械,2005,21(5):40-42.
[30]聂志强,汪越男,郑宇.传统食醋酿造过程中微生物群落的多样性及功能研究进展[J].中国酿造,2012,31(7):1-6.
[31]邓朝霞.永春老醋发酵过程中有机酸和生物胺变化分析及其细菌菌群结构分析[D].福州:福建师范大学,2014.
[32]李沙.传统岐山醋醋酸固态发酵过程中细菌群落动态演变与风味物质形成规律的研究[D].广州:华南理工大学,2016.
[33]李潇.传统固态发酵食醋大曲中的微生物多样性[D].太原:山西大学,2019.
[34]张天震,孙晓琪,刘丹彤,等.山西老陈醋大曲细菌群落结构及多样性研究[J].中国食品学报,2021,21(5):323-330.
[35]张天震.山西老陈醋微生物酿造机理及四大名醋指纹图谱差异性研究[D].晋中:山西农业大学,2019.
[36]郭旭凯,杨玲,刘聪,等.山西老陈醋酿造过程中乳酸菌和醋酸菌的分离鉴定及高粱单宁对其生长的影响[J].中国酿造,2018,37(8):37-44.
[37]毛祥,温雪瓶,黄丹,等.5种常用酿酒高粱的主要成分及淀粉特性差异分析[J].中国酿造,2020,39(3):57-62.
[38]李晓慧.老陈醋生产中高粱糊化特性的研究[D].太原:山西大学,2016.
[39]周森,胡佳音,崔洋,等.应用高通量测序技术解析清香型大曲微生物多样性[J].中国食品学报,2019,19(6):244-250.
[40]DENG Y,JIANG E H,YANG Y F,et al.Molecular ecological network analyses[J].BMC Bioinformatics,2012,13:113.
[41]FAUST K,RAES J.Microbial interactions:from networks to models[J].Nat Rev Microbiol,2012,10(8):538-550.
[42]BARBERÁN A,BATES S T,CASAMAYOR E O,et al.Using network analysis to explore co-occurrence patterns in soil microbial communities[J].ISME J,2012,6(2):343-351.
[43]FUHRMAN J A,CRAM J A,NEEDHAM D M.Marine microbial community dynamics and their ecological interpretation[J].Nat Rev Microbiol,2015,13(3):133-146.
[44]MOLLOY S.Environmental microbiology:Disentangling syntrophy[J].Nat Rev Microbiol,2014,12(1):7.
[45]HIBBING M E,FUQUA C,PARSEK M R,et al.Bacterial competition:surviving and thriving in the microbial jungle[J].Nat Rev Microbiol,2010,8(1):15-25.