大曲是白酒生产过程中重要的糖化发酵剂,是在开放环境下以未经高压灭菌的原料自然接种生产而成的,对白酒产品品质有重要影响[1-2]。生产过程中的微生物主要来自未灭菌的原料和生产环境[3]。大曲的制备需经过谷物润湿、粉碎混合、固定成型、堆积发酵、贮存成熟等多个步骤[4]。通常,大曲的贮存过程所需时间较长,需存放在通风良好的开放环境下经历长达数个月之久[5]。目前,大曲成熟时间仍取决于个体生产者的经验,导致大曲产品品质参差不齐且稳定性差。因此,了解大曲在贮存期间的微生物变化规律极为重要,已成为行业关注重点。
近年来,高通量测序技术已被广泛用于研究大曲的微生物群落组成[6]。陈晓茹等[7]通过高通量测序技术发现,魏斯氏菌属(Weissella)、刺糖多孢菌属(Saccharopolyspora)和乳酸杆菌属(Lactobacillus)是大曲贮存过程中的优势细菌,但细菌群落结构变化显著。GUAN T等[1]分析了浓香型大曲贮存6个月期间的细菌群落变化规律,魏斯氏菌属(Weissella)、乳酸杆菌属(Lactobacillus)、片球菌属(Pediococcus)、芽孢杆菌属(Bacillus)和醋酸杆菌属(Acetobacter)是大曲贮存期间的优势细菌,且不同贮存时间的细菌群落结构差异较大。由此可知,贮存过程是大曲最复杂、最关键的环节,大量微生物在自然环境变化下不断演替而趋于平衡,为后续酿酒的多边发酵提供重要的微生物基础[7]。然而,当前研究多集中于大曲贮存过程中的微生物群落结构组成,忽视了大曲贮存过程的功能演替,尤其是利用生物信息学对大曲贮存过程细菌群落的功能预测研究还鲜见报道。
当前关于大曲贮存过程细菌群落多样性及功能的研究还相对较少,导致大曲贮存过程的微生物生态学机制研究仍存在不足,尤其是在南方沿海地区生产的大曲。大部分研究者都是以我国比较著名的白酒生产所用大曲为研究对象,但是不同地域由于气候环境的不同,环境中微生物群落差异较大,而大曲通常是在相对干燥、通风良好,温湿度恒定的曲房进行贮藏,不可避免的会受到当地环境的影响。目前,针对沿海地区生产的大曲还尚未报道。本研究以浙江地区生产的中温大曲为研究对象,通过高通量测序技术,分析中温大曲贮存过程中细菌群落结构及多样性的演替规律,并基于多元统计分析揭示影响细菌群落结构组成变化的理化因素,同时,结合生物信息学对细菌功能进行预测,从微生物生态学视角全面深入理解大曲贮存过程的重要性,旨在为生产品质稳定的成熟中温大曲提供理论和数据参考。
1 材料与方法
1.1 材料与试剂
样品:取自浙江省某白酒大曲生产车间,成曲常温贮存90 d,每隔30 d取样。
邻苯二甲酸氢钾、酚酞、氢氧化钠(均为分析纯):国药集团化学试剂有限公司;DNeasy PowerSoil Pro Kit:德国QIAGEN公司;BMS1000-2核酸纯化磁珠:无锡百迈格生物科技有限公司;Taq脱氧核糖核酸(deoxyribo nucleic acid,DNA)聚合酶:上海生工生物工程股份有限公司;细菌16S rRNA V3-V4区扩增引物(343F:5′-TACGGRAGGCAGCAG-3′和806R:5′-GGACTACVVGGGTATCTAATC-3′):上海欧易生物医学科技有限公司;Nextera XT DNA Sample Preparation Kit:美国Illumina公司。
PowerSoil Pro Kit:德国QIAGEN公司;BMS1000-2核酸纯化磁珠:无锡百迈格生物科技有限公司;Taq脱氧核糖核酸(deoxyribo nucleic acid,DNA)聚合酶:上海生工生物工程股份有限公司;细菌16S rRNA V3-V4区扩增引物(343F:5′-TACGGRAGGCAGCAG-3′和806R:5′-GGACTACVVGGGTATCTAATC-3′):上海欧易生物医学科技有限公司;Nextera XT DNA Sample Preparation Kit:美国Illumina公司。
1.2 仪器与设备
AB-50型电子分析天平、FE28型pH计:瑞士Mettler-Toledo公司;GL-21M型高速冷冻离心机:长沙平凡仪器厂;VORTEX-3型旋涡混匀仪:德国IKA公司;NanoDrop 2000型紫外微量分光光度计:美国Thermo公司;DYCP-31DN型琼脂糖水平电泳仪电泳槽:北京六一仪器厂;SEDI G型聚合酶链式反应(polymerase chain reaction,PCR)仪、KETA G型全自动凝胶成像系统:美国Wealtec公司;Illumina PE250测序仪:美国Illumina公司。
1.3 试验方法
1.3.1 样品收集
为消除曲房空间差异和大曲个体差异,分别从曲房中间的曲堆上、中和下层取样,并粉碎混匀,将粉碎混匀的大曲分为两份,分别置于-20 ℃和-80 ℃保存,一份用于理化检测,一份用于DNA提取,每份样品设置三个平行样本。
1.3.2 理化特性分析
参考QB/T 4257—2011《酿酒大曲通用分析方法》[8]测定样品的pH、酸度和含水量。
1.3.3 DNA提取及高通量测序
根据DNeasy PowerSoil Pro Kit试剂盒说明书提取大曲样品中微生物的总DNA,使用NanoDrop 2000型紫外微量分光光度计测定其溶度,并检验是否受核糖核酸(ribonucleic acid,RNA)和蛋白质污染,随后采用1%琼脂糖凝胶电泳测定其纯度。采用细菌16S rRNA V3-V4区扩增引物以合格的DNA为模板进行PCR扩增。PCR扩增体系(20 μL):以16S rRNAV3-V4区扩增引物各0.1 μmol,模板10 ng,2×高保真DNA聚合酶混合物10 μL[9]。PCR扩增条件:94 ℃预变性2 min;94 ℃变性20 s,55 ℃退火30 s,72 ℃延伸1 min,共进行30个循环;最后72 ℃保持10 min[10]。扩增产物利用2%琼脂糖凝胶电泳进行目的片段纯化回收。将纯化的PCR扩增产物委托上海欧易生物医学科技有限公司完成建库和Illumina Miseq PE250高通量测序。
PowerSoil Pro Kit试剂盒说明书提取大曲样品中微生物的总DNA,使用NanoDrop 2000型紫外微量分光光度计测定其溶度,并检验是否受核糖核酸(ribonucleic acid,RNA)和蛋白质污染,随后采用1%琼脂糖凝胶电泳测定其纯度。采用细菌16S rRNA V3-V4区扩增引物以合格的DNA为模板进行PCR扩增。PCR扩增体系(20 μL):以16S rRNAV3-V4区扩增引物各0.1 μmol,模板10 ng,2×高保真DNA聚合酶混合物10 μL[9]。PCR扩增条件:94 ℃预变性2 min;94 ℃变性20 s,55 ℃退火30 s,72 ℃延伸1 min,共进行30个循环;最后72 ℃保持10 min[10]。扩增产物利用2%琼脂糖凝胶电泳进行目的片段纯化回收。将纯化的PCR扩增产物委托上海欧易生物医学科技有限公司完成建库和Illumina Miseq PE250高通量测序。
1.3.4 生物信息学分析
原始序列使用Qiime2 pipeline[11]进行处理,具体如下:利用Demux插件对原始序列进行解复用,使用Cutadapt插件去除测序引物,并过滤低质量碱基,随后采用DADA2插件合并序列并去除嵌合体,通过预训练SILVA 138数据库[12]对序列进行注释,得到非单扩增序列变异体(amplicon sequence variants,ASV),将叶绿体、线粒体和不能注释到门水平的ASV删除。以最少样本序列数为标准进行抽平处理,分析不同样品的多样性差异。利用R软件实现基于ASVs的柱状图、样本聚类分析、主坐标分析和冗余分析。
根据样品中各ASV代表序列及其丰度表,使用Phylogenetic Investigation of Communities by Reconstruction of Unobserved States 2(PICRUSt2)算法的标准集成基因组数据库推断16S rRNA基因的功能组成[13]。该方法是将样品的16S rRNA基因序列定位到京都基因与基因组百科全书(Kyoto Encyclopedia of Genes and Genomes,KEGG)数据库进行注释,并通过PICRUSt2算法预测群落的代谢途径丰度。
1.3.5 数据处理及统计分析
所有实验重复进行三次,数据结果以“平均值±标准偏差”表示。用SPSS 19.0软件进行方差分析,P<0.05表示差异显著。采用GraphPad Prism 8.0和Excel 2017绘制图表。
2 结果与分析
2.1 贮存期间大曲理化特征变化
大曲在自然环境变化胁迫下会发生复杂的理化反应,故从理化角度揭示大曲贮存过程中的各理化特征的差异。大曲贮存过程中pH、酸度和含水量的变化见图1。
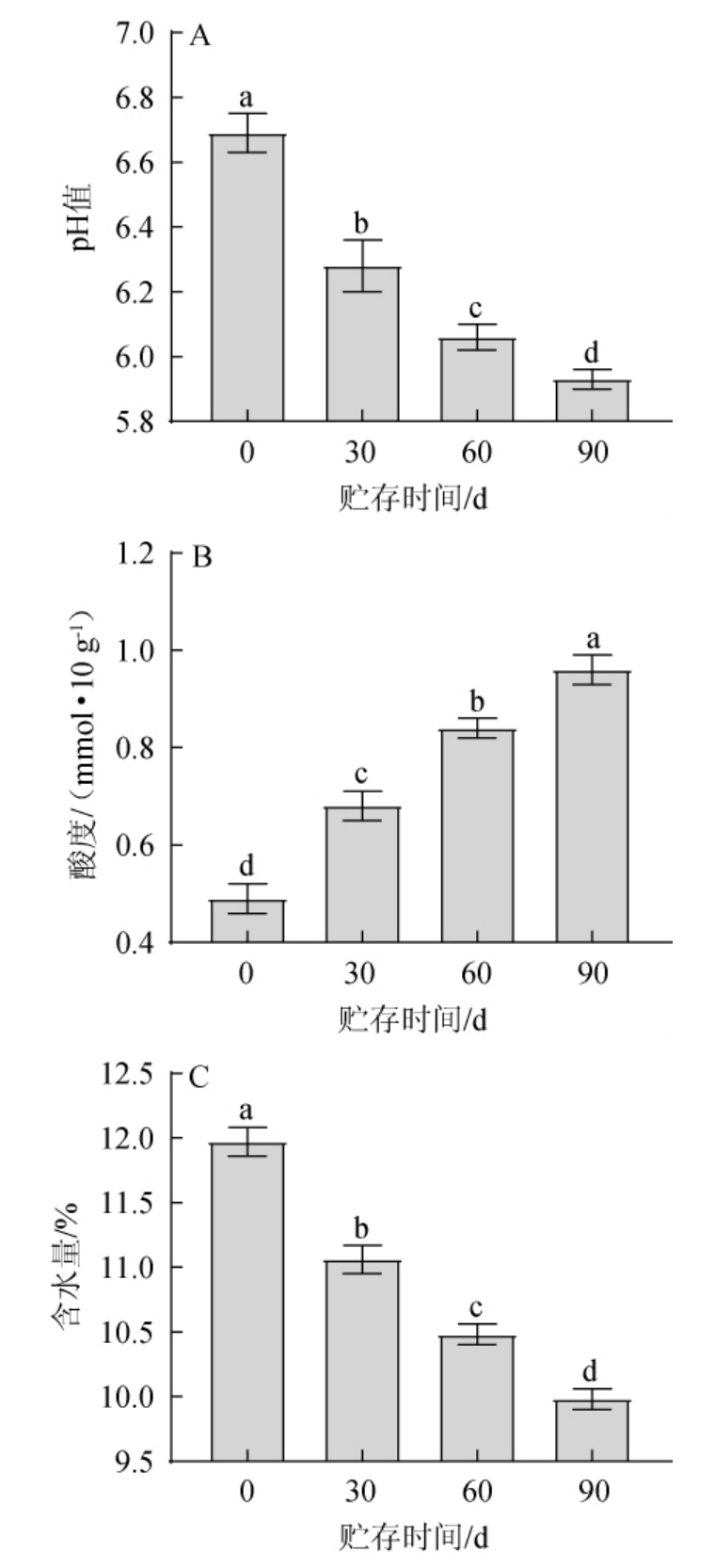
图1 大曲贮存期间pH(A)、酸度(B)和含水量(C)的动态变化
Fig.1 Dynamic changes of pH (A),acidity (B) and moisture (C)during Daqu storage
不同小写字母表示差异显著(P<0.05)。
由图1可知,在90 d贮存过程中,大曲样品的pH和含水量随贮存时间的不断延长而逐渐降低,而酸度则逐渐增加。成曲的pH和含水量分别为6.69和11.97%。贮存90 d后,大曲样品的pH和含水量分别下降至5.93和9.98%,显著低于成曲样品的pH和含水量(P<0.05)。此外,大曲的酸度在贮存90 d后可达0.96 mmol/10 g,比贮存前的成曲样品提高了95.92%,二者的酸度差异显著(P<0.05)。这一结果与GUAN T等[1]的研究结果相一致,其研究发现浓香型白酒大曲贮存6个月后会引起含水量和pH的降低以及酸度的升高。造成这一现象的原因可能是由于每块大曲砖块之间都留有一定的缝隙,而曲房是一个相对干燥、通风良好、温湿度恒定的空间[14]。pH的降低及酸度的增加与乳酸菌、醋酸菌等细菌的相对丰度持续较高有关[1]。故贮存会造成大曲的pH、酸度和含水量发生显著变化。
2.2 贮存期间大曲中细菌群落多样性变化
使用Observed指数、Shannon指数和Simpson指数评估大曲贮存过程中细菌群落的α-多样性变化,结果见表1。
表1 大曲贮存期间细菌群落序列数量和α-多样性分析结果
Table 1 Bacterial community sequence numbers and alpha-diversity analysis results during Daqu storage
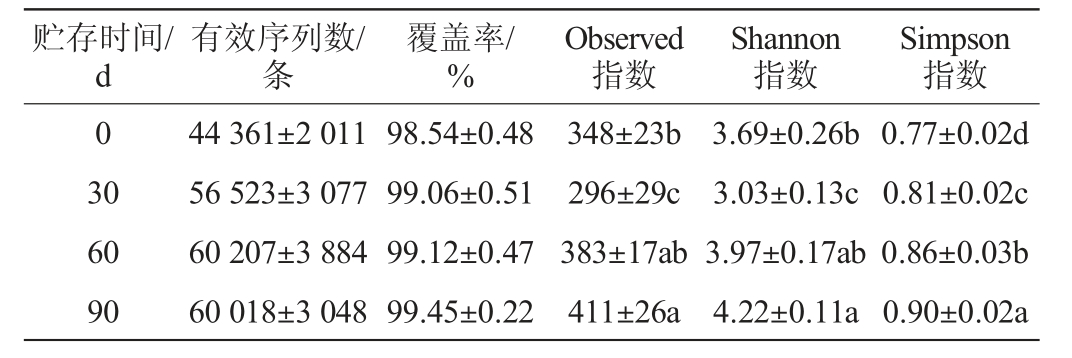
注:同列不同字母表示差异显著(P<0.05)。
由表1可知,12个大曲样品的Illumina高通量测序共得到663 332条有效序列,平均42 188~65 079条/大曲样品。各大曲样品的测序覆盖率均较高(>98%),说明这些有效测序序列可真实反映大曲中细菌群落的物种组成和多样性。成曲细菌群落的Observed指数、Shannon指数和Simpson指数分别为348、3.69和0.77。随着贮存时间的不断延长,大曲样品细菌群落的Observed指数和Shannon指数呈先下降后逐渐增加的变化趋势,而Simpson指数则呈逐渐增加的变化趋势。贮存90 d后,大曲细菌群落的Observed指数、Shannon指数和Simpson指数分别为411、4.22和0.90,与成曲细菌群落的α-多样性结果相比差异显著(P<0.05)。这与陈晓茹等[7]研究结果相一致。大曲样品细菌群落α-多样性的变化与大曲贮存环境有关,通常pH和酸度会显著影响微生物群落的α-多样性,表现为一些不耐酸的微生物不能适应该环境而消亡[15]。由于曲房具有良好的通风性,环境中耐酸的微生物可附着在大曲表面,利用高粱、小麦中的糖类物质进行生长繁殖,从而提高贮存后期大曲样品的α-多样性。
基于Bary-Curtis算法计算各大曲样品的距离,随后采用主坐标分析(principal coordinate analysis,PCoA)比较不同贮存时间大曲样品中细菌群落的差异,结果见图2。
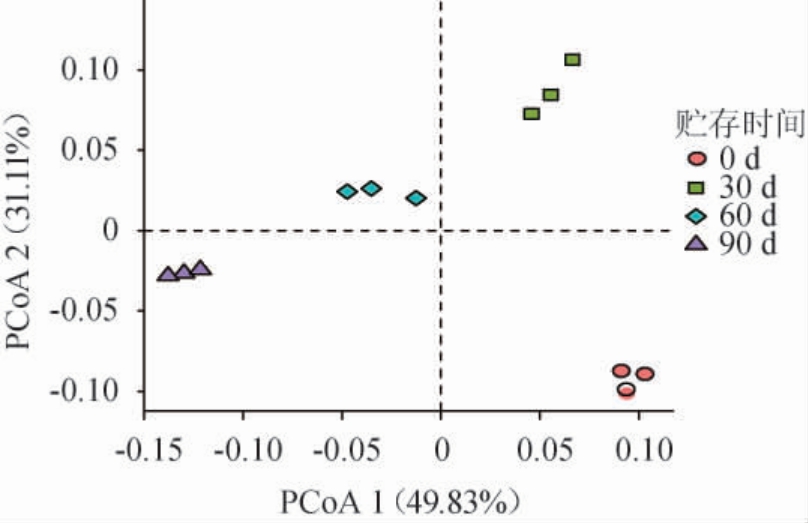
图2 大曲贮存期间细菌群落结构的主坐标分析结果
Fig.2 Principal coordinate analysis results of bacterial community structure during Daqu storage
由图2可知,主坐标第一轴方差贡献率为49.83%,而主坐标第二轴方差贡献率为31.11%,由此可知,物种差异的累计方差贡献率为80.94%,可利用前两个主成分来解释该模型的变量。不同贮存时间的大曲样品在主坐标前两轴的距离较远,说明各贮存时间的大曲样品中细菌群落结构差异显著,尤其是成曲样品和其他贮存时间的大曲样品差异最大。这一结果与陈晓茹等[7]研究结果相一致,表明大曲样品中细菌群落结构会随着贮存时间延长而不断变化。
2.3 贮存期间大曲中细菌群落结构组成演替
不同贮存时间大曲样品中细菌群落门水平的演替规律见图3。由图3可知,经物种分类注释后,12个大曲样品共检测到来自11个门水平的物种,其中厚壁菌门(Firmicutes)、变形菌门(Proteobacteria)和放线菌门(Actinobacteria)是大曲样品贮存期间相对丰度最高的三个门。厚壁菌门在大曲贮存期间占绝对优势地位,其相对丰度范围在86.83%~97.61%,表明贮存期间的大曲样品具有相对稳定的门水平细菌群落结构。这一结果与唐贤华等[16]研究结果相似,说明来自厚壁菌门的微生物普遍具有耐酸、分解高粱和小麦中淀粉和蛋白等特性。
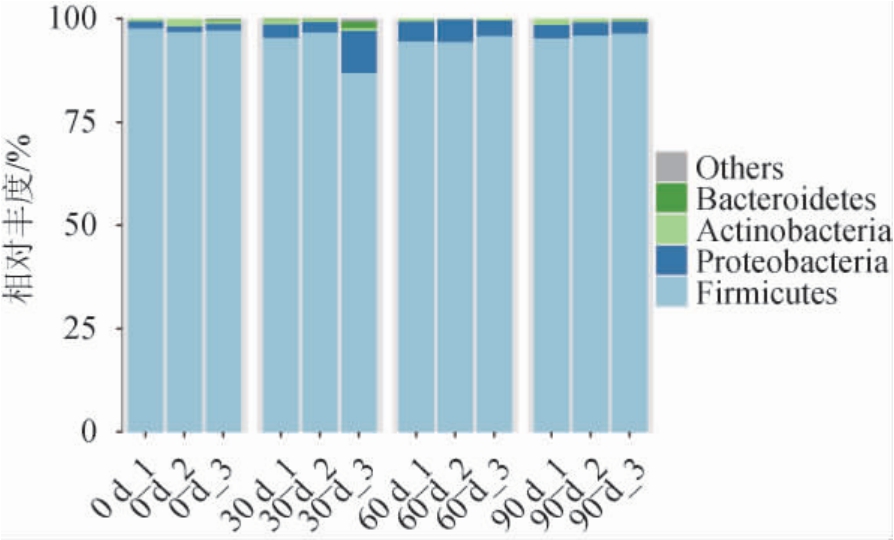
图3 大曲贮存期间细菌门的相对丰度动态变化
Fig.3 Dynamic changes of relative abundance of bacterial phyla during Daqu storage
Others:相对丰度<0.5%的所有细菌门。
为进一步更为准确的描述大曲样品在不同贮藏时间的细菌群落结构,从属水平揭示其组成差异。不同贮存时间大曲样品中细菌群落属水平的演替规律见图4。由图4可知,所有大曲样品共检测到79个属,其中乳酸杆菌属(Lactobacillus)、魏斯式菌属(Weissella)、片球菌属(Pediococcus)、明串珠菌属(Leuconostoc)、假单胞菌属(Pseudomonas)、芽孢杆菌属(Bacillus)、醋酸杆菌属(Acetobacter)、克罗彭斯特菌属(Kroppenstedtia)、不动杆菌属(Acinetobacter)、链霉菌属(Streptomyces)和乳球菌属(Lactococcus)等是大曲贮存期间的优势属。此外,在属水平的聚类分析也发现同一贮存时间的大曲样品被聚在一起,且整个贮存期间的大曲样品可主要分为两簇,成曲样品簇和其他贮存时间大曲样品簇,这一结果与图2的结果相一致,说明成曲和其他贮存时间的大曲样品在属水平组成上差异较大。成曲样品中乳酸杆菌属和魏斯式菌属的相对丰度最大,分别为47.56%和42.77%。随着贮存时间的不断延长,乳酸杆菌属的相对丰度逐渐升高,而魏斯式菌属的相对丰度则逐渐降低。贮存90 d后,大曲样品中乳酸杆菌属和魏斯式菌属的相对丰度分别为63.44%和25.16%。乳酸杆菌属是一类兼性厌氧的微生物,是谷物类产品发酵所必需的微生物之一,广泛存在于大曲及白酒酿造中,其可通过自身代谢系统将谷物中淀粉转化为乳酸[17-18]。这可能是促使大曲样品在贮存期间酸度增加的主要因素。魏斯式菌属也被确定为大曲中最丰富的细菌类群之一,其能够降解纤维二糖并在酸性发酵环境中生长代谢[17]。片球菌属和明串珠菌属的相对丰度在贮存30 d前逐渐升高,分别从2.71%和2.22%升高至5.57%和3.49%,之后随着贮存时间的增加逐渐降低,贮存90 d后,二者的相对丰度分别仅为1.89%和0.89%。这一结果与GUAN T等[1]研究结果相一致,表明片球菌属和明串珠菌属受其他微生物之间的竞争抑制,不能在贮存期间大量生长繁殖,从而达不到相对较高的相对丰度。据报道,从葡萄酒中分离出的片球菌具有酯化能力[19]。明串珠菌属通常附着在谷物原料表面,可将原料中糖类物质转化为甘露糖和葡聚糖,为其他乳酸菌的生长繁殖提供物质基础[20]。芽孢杆菌属和不动杆菌属在贮存期间的相对丰度范围分别为0.64%~2.07%和0~6.28%,这些少量的芽孢杆菌属具有产生蛋白水解酶和淀粉酶等特征,可提高大曲中有机酸和吡嗪类化合物的含量[21-23]。而不动杆菌属在大曲中的作用目前尚不清楚。此外,醋酸杆菌属和克罗彭斯特菌属的相对丰度则随贮存时间的不断延长而逐渐升高,二者分别从成曲样品的0.11%和0.04%升高至贮存90 d后大曲样品的2.04%和2.35%。醋酸杆菌属是一类好氧的微生物,可将谷物原料中糖类和酒精氧化成醋酸,从而提高大曲样品的酸度。克罗彭斯特菌属广泛存在各种不同香型的大曲中,可作为大曲成熟的生物标志微生物[24-25]。
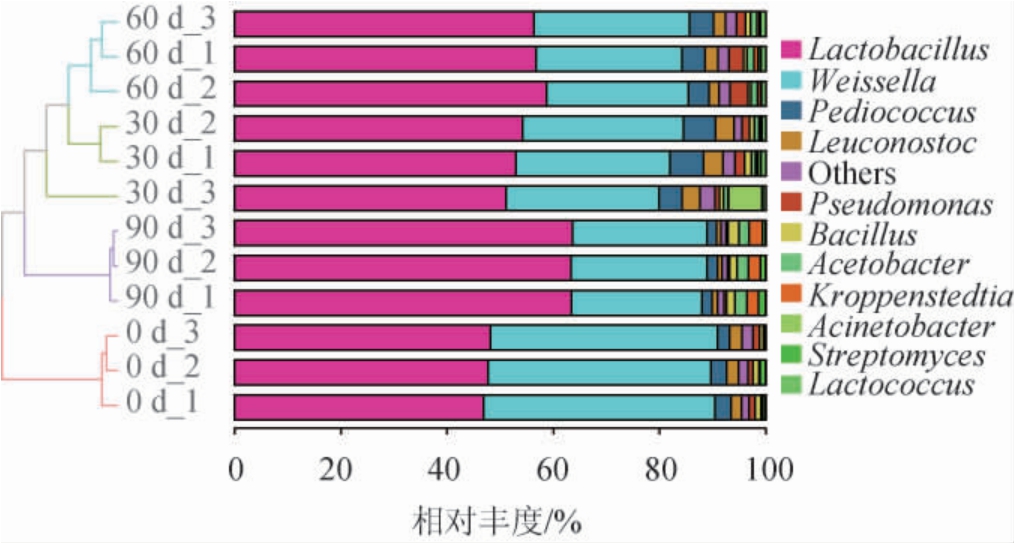
图4 大曲贮存期间细菌属的相对丰度动态变化
Fig.4 Dynamic changes of relative abundance of bacterial genera during Daqu storage
Others:相对丰度<0.5%的所有细菌属。
2.4 贮存期间大曲理化因子与细菌群落的冗余分析
冗余分析(redundancy analysis,RDA)是一种回归分析结合主成分分析的排序方法,可用于揭示响应变量和解释变量的线性关系。不同贮存时间大曲样品理化因子与优势细菌属的冗余分析见图5。
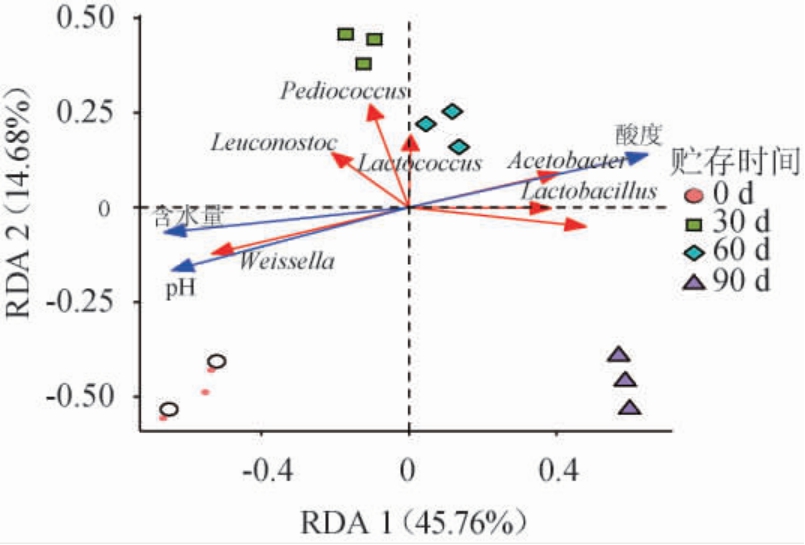
图5 大曲贮存期间理化因子与优势细菌属的冗余分析
Fig.5 Redundancy analysis of physicochemical factors and dominant bacterial genera during Daqu storage
蓝色和红线之间夹角为锐角表示正相关,钝角表示负相关。
由图5可知,贮存期间大曲理化因子对优势细群属的总解释度为60.44%,其中冗余分析第一轴的解释度为45.76%,可较好的区分不同贮存时间的大曲样品。魏斯式菌属相对丰度与含水量和pH呈正相关,与酸度呈负相关,而酸度则与乳酸杆菌属、醋酸杆菌属和克罗彭斯特菌属相对丰度呈正相关。此外,通过条件限制分析阐明每个理化因子对大曲贮存期间细菌群落结构的影响程度,发现pH、含水量和酸度对大曲细菌群落主要优势属演替的贡献度相差无几,说明多个理化因子共同调控贮存期间大曲样品细菌群落结构的演替变化。综上可知,贮存期间大曲样品细菌群落组成和理化因子变化显著,这些理化因子共同影响了大曲细菌群落多样性及结构稳定,从而促进了大曲的成熟。
2.5 贮存期间大曲细菌群落功能预测分析
利用PICRUSt2算法预测细菌的代谢功能,是宏基因组分析的一种代替方法,具有成本低,精度较高等特点。不同贮存时间大曲样品细菌群落的功能预测分析见图6。
由图6可知,贮存期间大曲样品共涉及6类一级功能层,包括代谢(L1)、遗传信息处理(L2)、环境信息处理(L3)、细胞过程(L4)、有机系统(L5)和人类疾病(L6),其中代谢、遗传信息处理和环境信息处理是主要功能,贮存期间的相对丰度分别为60.11%~61.82%、20.21%~21.05%和14.22%~14.84%。此外,这6类一级功能层的相对丰度在不同贮存时间的大曲样品中均无显著差异(P>0.05)。进一步对二级功能层进行分析,发现贮存期间大曲样品共涉及27个二级功能层,其中碳水化合物代谢(24.79%~26.85%),翻译(10.52%~11.02%)和膜转运(9.86%~10.21%)是最主要的三个二级功能层。通过聚类分析发现27个二级功能层可被分为两簇,其中运输和代谢、消化系统、碳水化合物代谢、氨基酸代谢、能力代谢等11个二级功能层的相对丰度在贮存90 d时相对高于其他贮存时间。而折叠、分选和降解、衰老、脂类代谢、萜类和聚酮类化合物代谢、翻译等9个二级功能层在贮存30 d时富集;膜转运、信号转导、核苷酸代谢、复制和修复及细胞运动5个二级功能层则在成曲样品中富集。
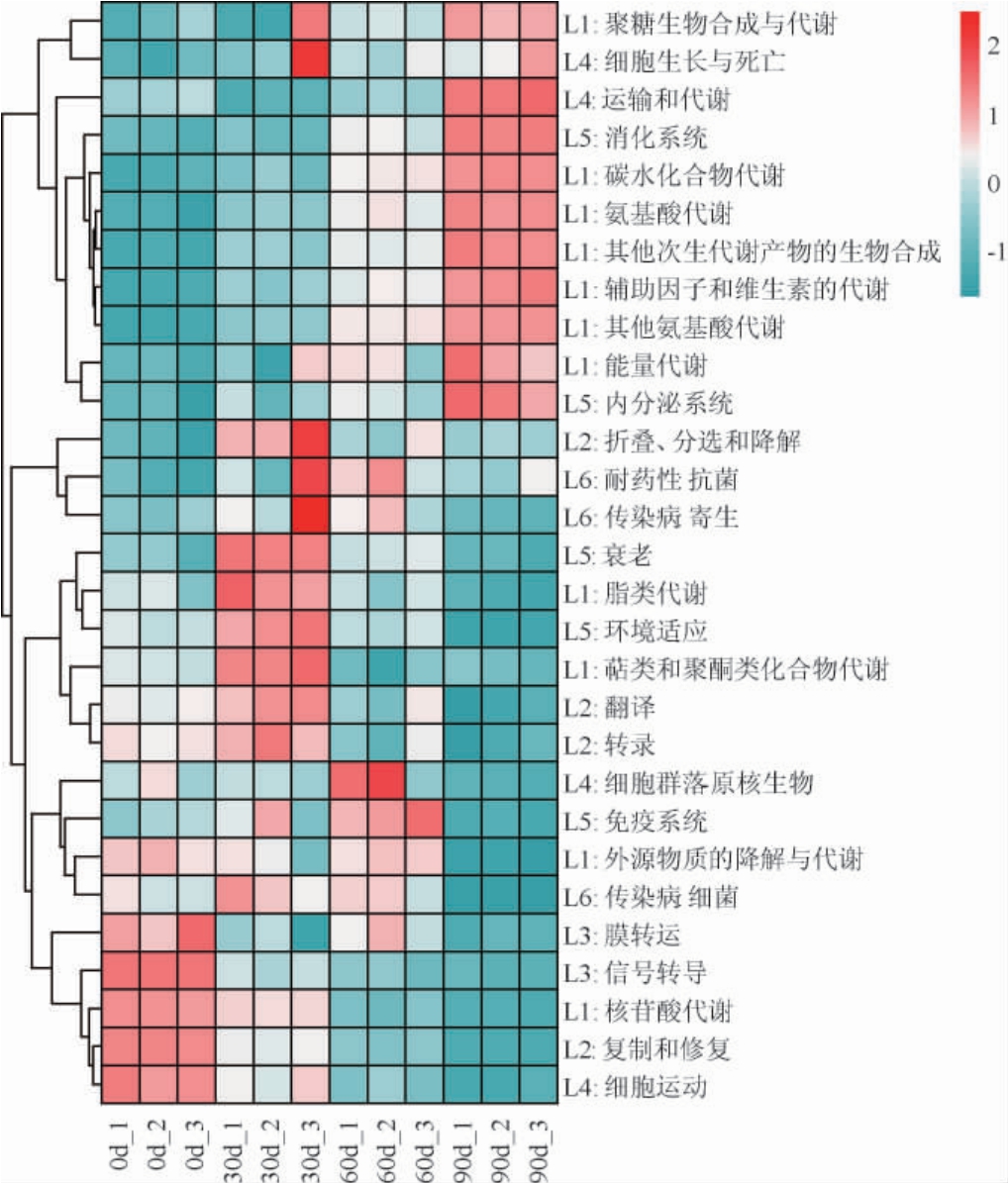
图6 大曲贮存期间细菌群落的PICRUSt2功能预测分析
Fig.6 PICRUSt2 functional predictive analysis of bacterial communities during Daqu storage
L1~L6表示一级功能层,L1:代谢、L2:遗传信息处理、L3:环境信息处理、L4:细胞过程、L5:有机系统、L6:人类疾病。
3 结论
本研究利用16S rRNA高通量测序技术探究浙江地区大曲贮存过程中理化特征和细菌群落结构的演替变化,并结合PICRUSt2预测贮存期间细菌群落功能差异。研究发现大曲的pH和含水量随着贮存时间的不断延长逐渐降低,而酸度呈相反的变化趋势。贮存90 d后,大曲样品的细菌群落结构和多样性差异显著(P<0.05)。门水平上,贮存期间的绝对优势菌门为厚壁菌门(Firmicutes),其相对丰度>85%。属水平上,乳酸杆菌属(Lactobacillus)和魏斯式菌属(Weissella)是大曲贮存期间的绝对优势属,其相对丰度范围分别为47.56%~63.44%和25.16%~42.77%。冗余分析结果表明魏斯式菌属相对丰度与含水量和pH呈正相关,而酸度与乳酸杆菌属、醋酸杆菌属和克罗彭斯特菌属相对丰度呈正相关。此外,PICRUSt2预测结果表明大曲贮存期间的基因功能主要与代谢、遗传信息处理和环境信息处理等功能相关。本研究旨在为生产品质稳定的成熟大曲提供理论和数据参考。
[1]GUAN T,YANG H,OU M,et al.Storage period affecting dynamic succession of microbiota and quality changes of strong-flavor Baijiu Daqu[J].LWT-Food Sci Technol,2021,139:110544.
[2]ZHU M,ZHENG J,XIE J,et al.Effects of environmental factors on the microbial community changes during medium-high temperature Daqu manufacturing[J].Food Res Int,2022:110955.
[3]HE G,HUANG J,ZHOU R,et al.Effect of fortified Daqu on the microbial community and flavor in Chinese strong-flavor liquor brewing process[J].Front Microbiol,2019,10:56-65.
[4]沈怡方.白酒生产技术全书[M].北京:中国轻工业出版社,2011:6-9.
[5]陶敏,明红梅,马世源,等.贮存时间对中高温大曲质量的影响[J].食品与发酵工业,2022,48(2):205-211.
[6]XU B,XU S,CAI J,et al.Analysis of the microbial community and the metabolic profile in medium-temperature Daqu after inoculation with Bacillus licheniformis and Bacillus velezensis[J].LWT-Food Sci Technol,2022,160:113214.
[7]陈晓茹,黄钧,周荣清,等.强化曲贮存过程中群落与代谢组分的变化规律[J].食品与发酵工业,2022,48(3):63-69.
[8]中华人民共和国工业和信息化部.QB/T 4257—2011 酿酒大曲通用分析方法[S].北京:中国标准出版社,2011.
[9]林钰宽,吴生文,游勇,等.不同培曲工艺对特香型大曲微生物群落结构的影响[J].食品与发酵工业,2022,48(6):188-194.
[10]宋凯.应用Illumina Miseq测序技术分析传统酸马奶细菌多样性[J].食品研究与开发,2021,42(16):175-182.
[11]BOLYEN E,RIDEOUT J R,DILLON M R,et al.Reproducible,interactive,scalable and extensible microbiome data science using QIIME 2[J].Nat Biotechnol,2019,37(8):852-857.
[12]QUAST C,PRUESSE E,YILMAZ P,et al.The SILVA ribosomal RNAgene database project:improved data processing and web-based tools[J].Nucleic Acid Res,2012,41(D1):D590-D596.
[13]DOUGLAS G M,MAFFEI V J,ZANEVELD J R,et al.PICRUSt2 for prediction of metagenome functions[J].Nat Biotechnol,2020,38(6):685-688.
[14]ZHANG C,AO Z,CHUI W,et al.Characterization of volatile compounds from Daqu-a traditional Chinese liquor fermentation starter[J].Int J Food Sci Technol,2011,46(8):1591-1599.
[15]LIU P,ZHANG L,DU X,et al.Dynamic analysis of physicochemical and biochemical indices and microbial communities of light-flavor daqu during storage[J].J Am Soc Brew Chem,2019,77(4):287-294.
[16]唐贤华,田伟,张崇军,等.中温大曲在发酵和贮存过程中微生物群落结构分析[J].中国酿造,2019,38(6):113-118.
[17]YANG Y,WANG S T,LU Z M,et al.Metagenomics unveils microbial roles involved in metabolic network of flavor development in mediumtemperature daqu starter[J].Food Res Int,2021,140:110037.
[18]TAN Y,ZHONG H,ZHAO D,et al.Succession rate of microbial community causes flavor difference in strong-aroma Baijiu making process[J].Int J Food Microbiol,2019,311:108350.
[19]MATTHEWS A,GRBIN P R,JIRANEK V.Biochemical characterization of the esterase activities of wine lactic acid bacteria[J].Appl Microbiol Biotechnol,2007,77(2):329-337.
[20]吴晓红,高生平,蒋彩云,等.榨菜发酵过程中原核微生物群落结构及其理化因子的动态演替[J].食品与发酵工业,2021,47(1):27-34.
[21]YI Z,JIN Y,XIAO Y,et al.Unraveling the contribution of high temperature stage to Jiang-flavor Daqu,a liquor starter for production of Chinese Jiang-flavor Baijiu,with special reference to metatranscriptomics[J].Front Microbiol,2019,10:472.
[22]梁慧珍,卢延想,刘正,等.高温大曲中高产吡嗪类物质芽孢杆菌的筛选与应用[J].中国酿造,2022,41(1):116-122.
[23]HE G,DONG Y,HUANG J,et al.Alteration of microbial community for improving flavor character of Daqu by inoculation with Bacillus velezensis and Bacillus subtilis[J].LWT-Food Sci Technol,2019,111:1-8.
[24]任爱容,黄永光.茅台镇不同主酿区域酱香型白酒酿造大曲中细菌菌群结构分析[J].食品科学,2021,42(4):167-174.
[25]刘婷,陈可丹,黄冰静,等.不同生产环境对特香型大曲的影响[J].中国酿造,2019,38(7):80-84.