豆腐乳,又称腐乳、霉豆腐,是以大豆为原料制成的发酵类食品,在国外有着东方“奶酪”的美誉,其制作方法最早可追溯到魏代,凭借其独特的风味和细腻的口感备受广大消费者的喜欢[1]。豆腐乳发酵是一个庞大微生物群落演替的过程,利用其微生物产生的多种酶系,促使豆腐乳中蛋白质分解为营养价值高的氨基酸和一些风味物质、脂肪分解为脂肪酸、淀粉转化为糖类等人体所必需且易吸收的小分子物质[2-3]。发酵后的豆腐乳大幅度提升了大豆蛋白的吸收率,同时富含有低聚糖、大豆异黄酮等功能性物质,对于抗氧化、提高人体免疫力、降血压等方面具有积极作用[4]。
我国地势辽阔,不同区域的气候差异性孕育了多种具有代表性的豆腐乳:如桂林豆腐乳、北京青豆腐乳、湖南永州茶油豆腐乳和四川特色豆腐乳等[5]。豆腐乳所具有的独特风味及营养价值与发酵期间微生物的群落结构密切相关,豆腐乳根据发酵类型分为自然发酵豆腐乳和纯种发酵豆腐乳,虽然市场销售已有工业化生产的纯种发酵豆腐乳,但自然发酵豆腐乳因制作成本低和操作简单等因素也出现在各大销售市场上。豆腐乳的风味往往由酶系的丰富程度所决定,自然发酵处于开放性环境中,其中包含有利于发酵的菌株同时也包括有害微生物,有害微生物在豆腐乳发酵过程中因其种类的不确定性和发酵不受控制,易造成产品质量的不稳定带来食品安全性等问题[6-7]。自然发酵是完全利用环境中的微生物协同发酵,分泌的多种酶系使口感和风味有所提升,但自然发酵不受人工控制,致病菌和腐败菌一但大量繁殖就会给产品安全性及消费者健康带来危害[8]。纯种发酵利用特定的菌种和先进工艺技术进行发酵,保证了产品的稳定和安全性,纯种发酵工艺随着越来越多的企业和研究人员的不断投入和研究,将带来更大的经济效应[9]。因此,研究豆腐乳传统自然发酵体系的微生物群落结构,为后续纯种的发酵豆腐乳的改善提供一定的理论基础。
变性梯度凝胶电泳(denaturinggradientgelelectrophoresis,DGGE)[10]、脱氧核糖核酸(deoxyribonucleic acid,DNA)克隆文库[11]和16S rRNA基因文库[12]等技术存在测序通量低、操作复杂、干扰较多和检测效率不高等问题,而高通量测序技术因检测通量更高、准确度高以及同时分析多个样品的特点[13],被广泛应用在豆瓣酱、豆豉、泡菜、食醋等发酵类食品中检测微生物的组成及其丰度的变化[14-15]。本研究基于高通量测序技术对四川乐山地区的两种发酵类型豆腐乳中的细菌和真菌群落多样性进行分析,以期了解其发酵优势菌属和可能导致腐败的菌株,为改善豆腐乳品质及优化发酵工艺提供一定理论基础。
1 材料与方法
1.1 材料与试剂
1.1.1 材料
纯种发酵豆腐乳(LA-1~LA-3):乐山阙剂食品有限公司;自然发酵豆腐乳(LB-1~LB-3);工厂自然发酵豆腐乳;自然发酵豆腐乳(LC-1~LC-3):市售;自然发酵豆腐乳(LD-1~LD-3):家庭自制。
1.1.2 试剂
DNA检测试剂盒:美国OMEGA公司;Qubit3.0 DNA检测试剂盒:美国Life公司;Taq DNA聚合酶和Hieff NGSTM DNA Selection Beads:上海翌圣生物科技股份有限公司。其他试剂均为国产分析纯。
1.2 仪器与设备
TP421型电子天平:上海舜宇恒平科学仪器有限公司;Pico-21台式离心机:美国Thermo Fisher公司;VORTEX 3旋涡混匀器:德国IKA公司;ETC 811聚合酶链式反应(poly merase chain reaction,PCR)仪:北京东胜创新生物科技有限公司;TND03-H-H混匀型干式恒温器:深圳拓能达科技有限公司;DYY-4C电泳仪:北京六一生物科技有限公司;Tanon-2500型凝胶成像仪:上海天能生物科技有限公司;1Sorvall Stratos冷冻高速离心机:美国赛默飞世尔科技公司。
1.3 方法
1.3.1 豆腐乳样品微生物基因组DNA的提取与检测
参考李荣源等[16]的方法,利用基因组DNA提取试剂盒对豆腐乳样品微生物的基因组DNA进行提取,使用1%琼脂糖凝胶电泳检测DNA的质量。
1.3.2 PCR扩增
参考苟萌等[17]的方法,对微生物总DNA精确定量,以其为模板,采用通用引物341F(5′-CCCTACACGACGCTCTTCCGATCTG-3′)和805R(5′-GACTGGAGTTCCGGCACCCGAGAATTCCA-3′)对细菌16S rDNA的V3-V4区基因序列进行PCR扩增。采用通用引物1737F(5-GGAAGTAAAAGTCGTAACAAGG-3′)和2043R(5′-GCTGCGTTCTTCATCGATCGATGC-3′)对真菌在ITS1区基因序列进行PCR扩增,PCR扩增条件:94 ℃预变性3 min;94 ℃变性30 s,55 ℃退火30 s,72 ℃延伸30 s,26个循环;最后72 ℃再延伸10 min。采用2%琼脂糖凝胶电泳检测PCR扩增产物,采用试剂盒回收纯化后的PCR扩增产物,并将样品送至生工生物工程(上海)股份有限公司进行Illumina MiSeq高通量测序。
1.3.3 数据处理及分析
经过高通量测序得到的序列通过拼接、精确匹配、去除无法拼接序列,得到最终序列。利用Usearch(3.0.1.1)软件将具有97%相似度的序列进行操作分类单元(operational taxonomic units,OTU)聚类,每个OTU对应一种代表序列;利用Uchime去除嵌合体序列。利用核糖体数据库项目(ribosomal database project,RDP)、Silva、Unite等数据库进行基因序列比对和注释。实验作图软件使用R软件和Origin 9.1。实验重复3次。
2 结果与分析
2.1 稀释性曲线
不同豆腐乳样品中微生物菌群稀释曲线见图1。由图1可知,豆腐乳样品中细菌和真菌菌群的OTU数均随序列数的增加呈先增加后趋于平坦的趋势,说明本次样品的取样量充足且测序数据合理。
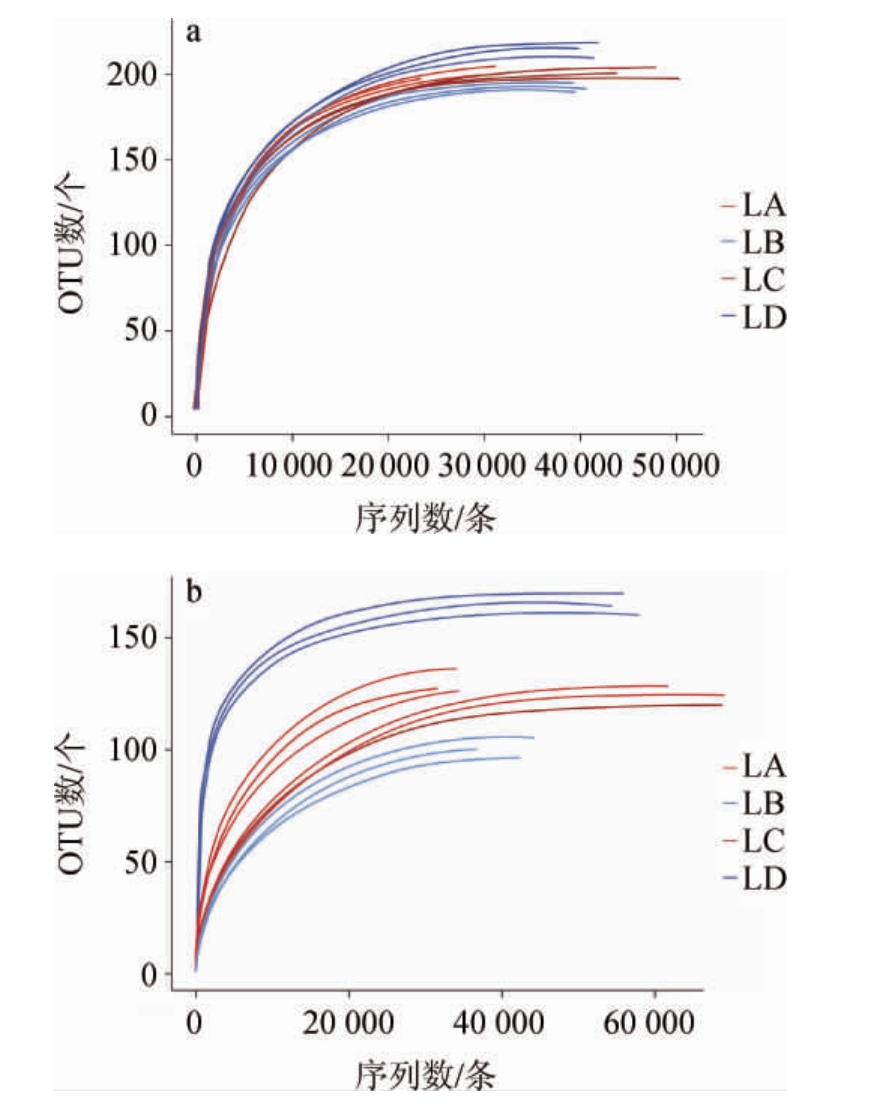
图1 不同豆腐乳样品细菌(a)和真菌(b)菌群的稀释性曲线
Fig.1 Dilution curves of bacterial (a) and fungal (b) flora of different sufu samples
2.2 数据统计与OTU分析
经过滤和双端拼接,细菌菌群的16S rDNA基因序列共获得160 858条有效序列,平均碱基长度为420 bp;真菌菌群的ITS1区基因序列共获得187 339条有效序列,平均碱基长度为310 bp。所获得的序列按照97%的相似度进行分类,细菌菌群得到838个OTU,真菌菌群得到509个OTU,采用R软件绘制韦恩(Venn)图,结果见图2。
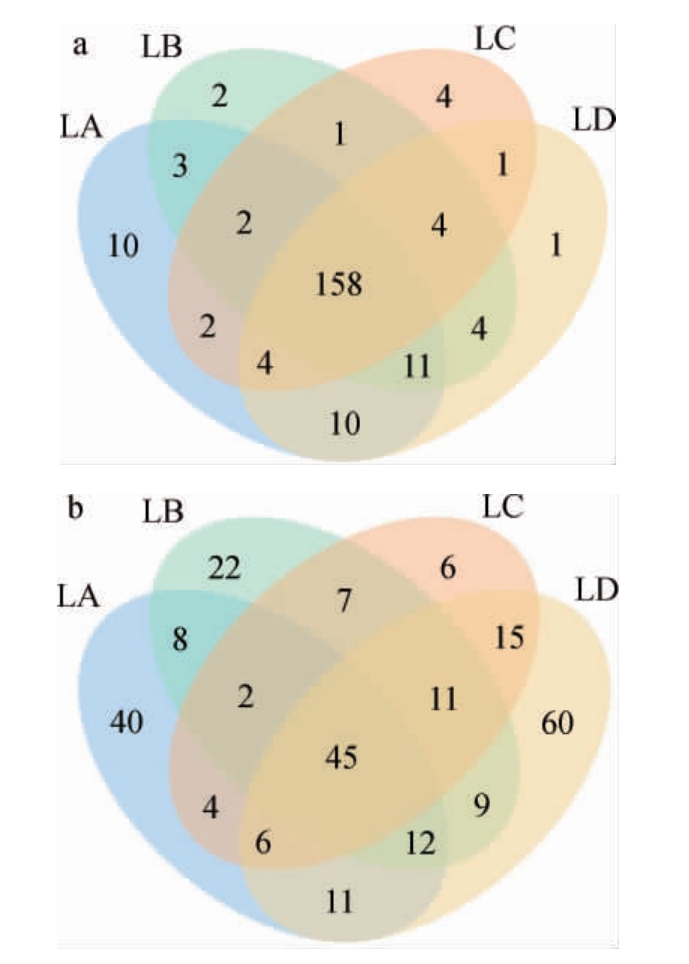
图2 不同豆腐乳样品细菌(a)和真菌(b)菌群OTU Venn图
Fig.2 OTU Venn diagram of bacterial (a) and fungal (b) flora of different sufu samples
由图2可知,4个豆腐乳样品共有的细菌OTU数为158个,真菌OTU数为45个,其中,样品LD的细菌和真菌OTU数均最高,分别为221个和169个,可以看出在家庭自制发酵豆腐乳中微生物的种类繁多不受人为环境控制,极易存在更高食品安全隐患。
2.3 α-多样性分析
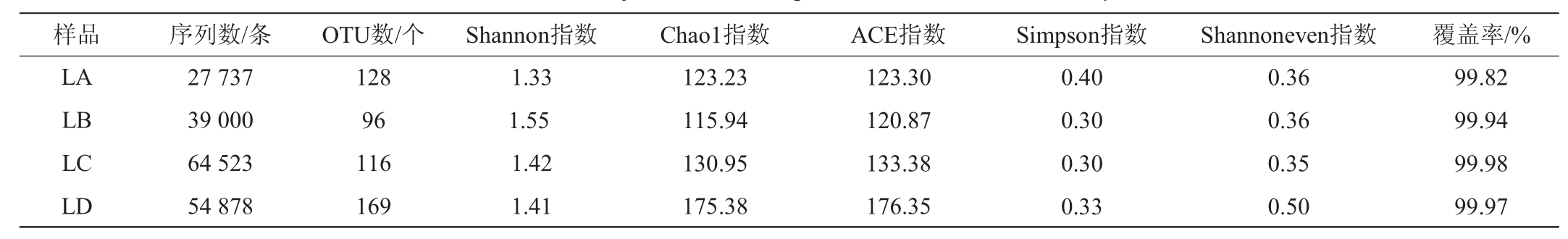
不同豆腐乳样品中细菌及真菌菌群的α-多样性分析结果分别见表1和表2。由表1可知,所有样品的覆盖率均>99%,说明测序结果代表了本次样品中微生物群落的真实情况。由OTU数、Chao1指数和ACE指数可以看出,无论是细菌菌群还是真菌菌群,样品LD的微生物菌群丰富度最高。由Simpson指数和Shannon指数可以看出,样品LA的多样性均大于其他3个样品,样品LA为纯种毛霉发酵而来,多样性指数偏高可能是因为在生产过程中由于操作不当造成微生物交替污染。虽然样品LA多样性较高,但OTU数却不是最多的;Shannon指数最低和Simpson指数最高的样品分别为LC和LD,但OTU数却不是最低的,因此,可以看出Shannon指数和Simpson指数的高低与样品中OTU数不成正相关。通过比较分析细菌与真菌菌群Shannon指数多样性可知,细菌菌群Shannon指数略高于真菌菌群,表明细菌菌群的多样性更为丰富,在豆腐乳发酵过程中同样起着重要的作用。细菌菌群在豆腐乳发酵期间的协同作用增添多种酶系,使豆腐乳的风味更加均浓厚,口感更加细腻,同样在豆腐乳发酵过程中一旦有害细菌过多或成为优势菌,会造成产品腐败变质[18]。
表1 不同豆腐乳样品细菌菌群的α-多样性指数
Table 1 α-diversity indexes of bacterial flora of different sufu samples
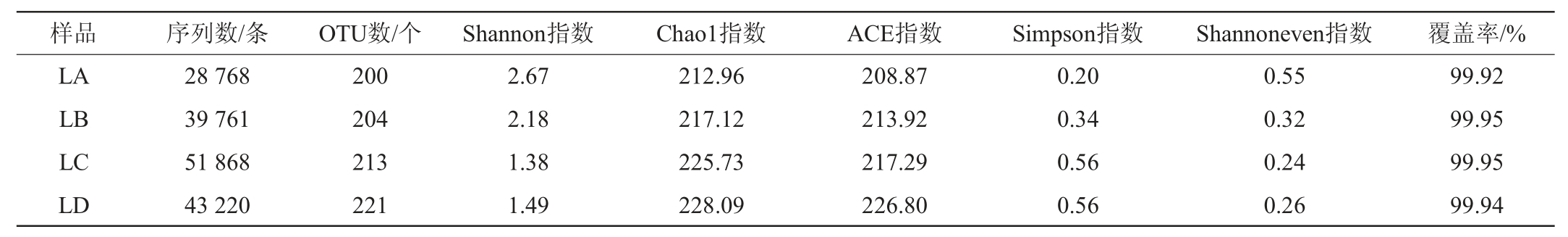
表2 不同豆腐乳样品真菌菌群的α-多样性指数
Table 2 α-diversity indexes of fungal flora of different sufu samples
2.4 微生物群落结构分析
2.4.1 基于门水平的微生物群落结构分析
基于门水平,不同豆腐乳样品中的微生物菌群结构见图3。由图3a可知,从4种豆腐乳样品中共得到3个优势细菌门(平均相对丰度>1%),分别为变形菌门(Proteobacteria)、厚壁菌门(Firmicutes)、拟杆菌门(Bacteroidetes),均为4种豆腐乳样品的优势细菌门。其中,变形菌门在样品LA中平均相对丰度最高,为59.83%;厚壁菌门在样品LB中平均相对丰度最高,为45%;拟杆菌门在样品LC中平均相对丰度最高,为34.43%。众多研究表明,变形菌门是细菌中最大的一类门,为革兰氏阴性菌且广泛存在于自然界中[19];厚壁菌门可以产生芽孢,在缺水及极端发酵环境中均发现其芽孢,这可能是厚壁菌门细菌在含盐量高、油渍产品中仍被发现的原因[20];拟杆菌门同样在发酵类食品(如奶制品、肉制品和酱油)中均有被发现,与杜瑞等[21]的研究结果一致。
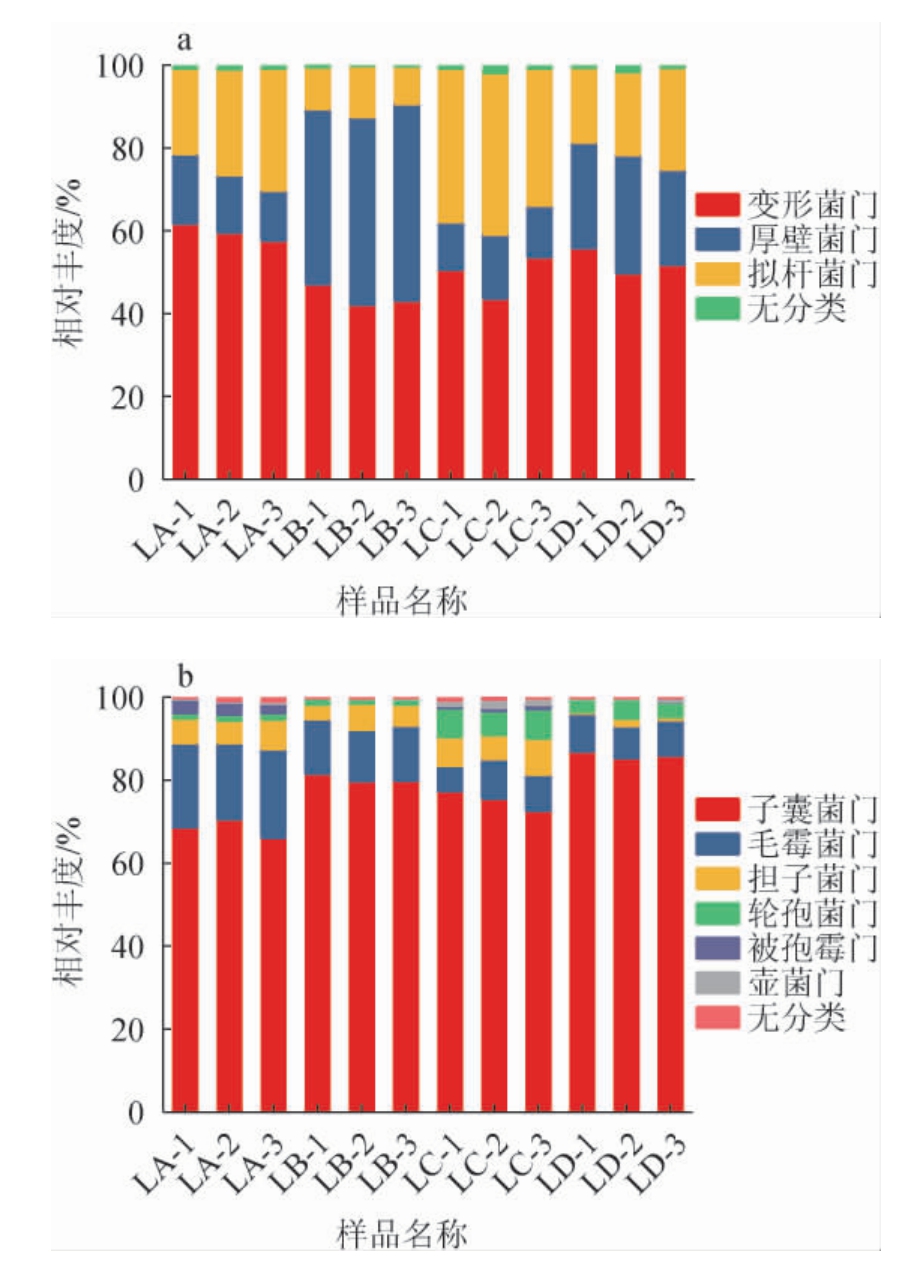
图3 基于门水平不同豆腐乳样品细菌(a)和真菌(b)菌群结构分析结果
Fig.3 Analysis results of bacterial (a) and fungal (b) flora structure of different sufu samples based on phylum level
由图3b可知,从4种豆腐乳样品中共得到6个优势真菌门(平均相对丰度>1%),分别为子囊菌门(Ascomycota)、毛霉菌门(Mucoromycota)、轮孢菌门(Rozellomycota)、胆子菌门(Basidiomycota)、被孢霉门(Mortierellomycota)和壶菌门(Chytridiomycota)。样品LA中的优势真菌门分别为子囊菌门、毛霉菌门和被孢霉门;样品LB中的优势真菌门分别为子囊菌门、毛霉菌门和担子菌门,样品LC中的优势真菌门分别为子囊菌门、毛霉菌门、轮孢菌门和壶菌门;样品LD中的优势真菌门分别为子囊菌门、毛霉菌门和担子菌门。4种豆腐乳共有的优势真菌门为子囊菌门、毛霉菌门和担子菌门,子囊菌门在样品LA、LB、LC和LD中的平均相对丰度分别为68.61%、80.55%、75.32%、86.23%;毛霉菌门在样品LA、LB、LC和LD中的平均相对丰度分别为20.31%、12.93%、8.11%和8.46%;担子菌门在样品LA、LB、LC和LD中的平均相对丰度分别为6.19%、5.11%、8.74%和1.75%。
2.4.2 基于属水平的微生物群落结构分析
基于属水平,不同豆腐乳样品中的微生物菌群结构见图4。
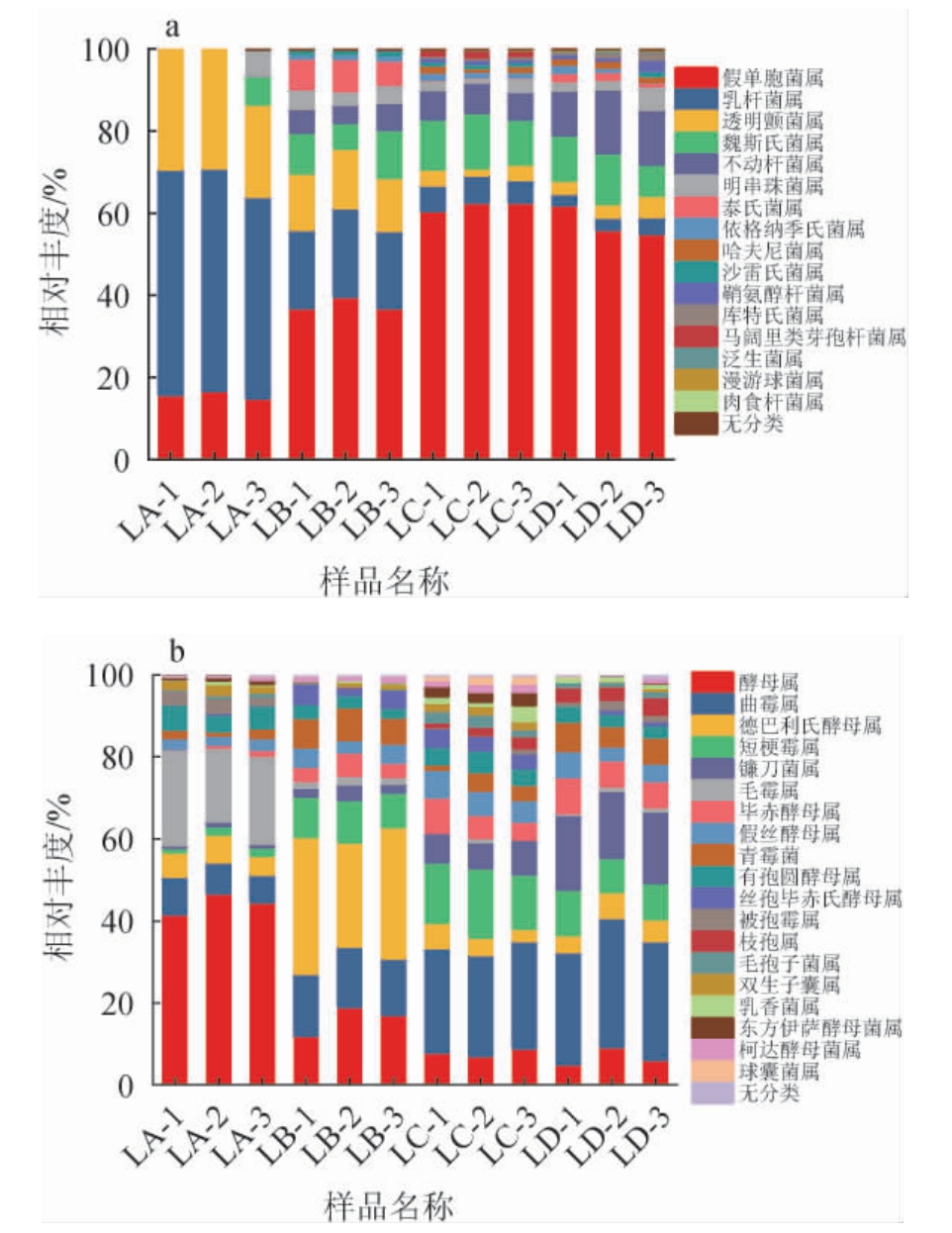
图4 基于属水平不同豆腐乳样品细菌(a)和真菌(b)菌群结构分析结果
Fig.4 Analysis results of bacterial (a) and fungal (b) flora structure of different sufu samples based on genus level
由图4a可知,从4种豆腐乳样品中共得到13个优势细菌属(平均相对丰度>1%),分别为假单胞菌属(Pseudomonas)、乳杆菌属(Lactobacillus)、透明颤菌属(Vitreoscilla)、魏斯氏菌属(Weissella)、不动杆菌属(Acinetobacter)、明串珠菌属(Leuconostoc)、泰氏菌属(Tissierella)、依格纳季氏菌属(Ignatzschineria)、哈夫尼菌属(Hafnia)、沙雷氏菌属(Serratia)、鞘氨醇杆菌属(Sphingobacterium)、库特氏菌属(Kurthia)、马阔里类芽孢杆菌属(Paenisporosarcina)。不同豆腐乳样品间的细菌菌群结构有所差异,在样品LA中的优势细菌属分别为乳杆菌属、透明颤菌属、假单胞菌属、魏斯氏菌属和明串珠菌属,平均相对丰度分别为52.77%、30.12%、15.92%、12.11%和6.24%;样品LB的优势细菌属分别为假单胞菌属、乳杆菌属、透明颤菌属、魏斯氏菌属、泰式菌属、不动杆菌和明串珠菌属,平均相对丰度分别为37.93%、19.87%、13.7%、9.31%、7.06%、5.64%和4.13%;样品LC的优势细菌属分别为假单胞菌属、魏斯氏菌属、不动杆菌属、乳杆菌属、透明颤菌属、明串珠菌属、依格纳季氏菌属、马阔里类芽孢杆菌属和沙雷氏菌属,平均相对丰度分别为62.05%、13.45%、7.53%、6.14%、3.08%、2.21%、1.55%、1.50%和1.04%;样品LD的优势细菌属分别为假单胞菌属、不动杆菌属、魏斯氏菌属、透明颤菌属、明串珠菌属、乳杆菌属、鞘氨醇杆菌属、哈夫尼菌属和库特氏菌属,平均相对丰度分别为59.12%、15.57%、12.45%、5.16%、4.27%、3.28%、1.91%、1.50%和1.03%。在4种豆腐乳样品中共有优势细菌属为假单胞菌属、乳杆菌属、透明颤菌属、魏斯氏菌属、不动杆菌属和明串珠菌属。其中假单胞菌属是微生物的重要类群,也是分布最广的微生物之一,为革兰氏阴性、无芽孢、需氧、直或稍弯曲杆状[22],其易分解蛋白质,在蛋白质含量较高的肉类、大豆和奶类制品常受到假单胞菌的污染,是造成食品腐败的常见菌属[23-24],且在市售及家庭小作坊自然发酵的豆腐乳中的相对丰度较高,表明易受该细菌的污染造成豆腐乳的腐败和变质。
由图4b可知,从4种豆腐乳样品中共得到19个优势真菌属,分别为酵母属(Saccharomyces)、曲霉属(Aspergillus)、德巴利氏酵母属(Debaryomyces)、短梗霉属(Aureobasidium)、镰刀菌属(Fusarium)、毛霉属(Mucor)、毕赤酵母属(Pichia)、假丝酵母属(Candida)、青霉菌(Penicillifer)、有孢圆酵母属(Torulaspora)、丝孢毕赤氏酵母属(Hyphopichia)、被孢霉属(Mortierella)、枝孢属(Cladophialophora)、毛孢子菌属(Trichosporon)、双生子囊属(Dipodascus)、乳香菌属(Talaromyces)、东方伊萨酵母菌属(Issatchenkia)、柯达酵母菌属(Kodamaea)、球囊菌属(Plectosphaerella)。在样品LA中的优势真菌属为酵母属、毛霉属、曲霉属、德巴利氏酵母属、有孢圆酵母属和被孢霉属,平均相对丰度分别为44.42%、20.87%、5.73%、7.86%、5.56%和5.22%;样品LB中的优势真菌属为德巴利氏酵母属、酵母属、曲霉属、短梗霉属、青霉属、毕赤酵母属、假丝酵母属、丝孢毕赤氏酵母属和有孢圆酵母属,平均相对丰度分别为31.23%、17.13%、14.92%、9.66%、7.14%、4.29%、4.12%、3.82%和2.93%;样品LC中的优势真菌属为曲霉属、短梗霉属、酵母属、镰刀菌属、毕赤酵母属、假丝酵母属、有孢圆酵母属、德巴利氏酵母属、丝孢毕赤氏酵母属、东方伊萨酵母菌属、双生子囊属、柯达酵母菌属和球囊菌属,平均相对丰度分别为25.43%、14.94%、8.09%、7.36%、6.22%、6.02%、4.52%、4.49%、4.02%、2.69%、1.97%、1.81和1.68;样品LD中的优势真菌属为曲霉属、镰刀菌属、短梗霉属、毕赤酵母属、酵母属、青霉属、德巴利氏酵母属、假丝酵母属、支孢属、有孢圆酵母属、被孢霉属、毛孢子菌属和乳香菌属,平均相对丰度分别为29.28%、17.41%、9.43%、7.18%、6.92%、6.16%、5.25%、4.69%、3.73%、3.31%、1.63%、1.27%和1.15%。在4种豆腐乳样品中共有优势真菌属为曲霉属、曲霉属、德巴利氏酵母属、短梗霉属、假丝酵母属和有孢圆酵母属。由此可以看出,豆腐乳真菌菌群以酵母菌为主。酵母菌是发酵食品中的优良发酵剂[25],不仅增加发酵食品的营养价值还赋予其独特的风味口感[26]。
2.5 基于属水平不同豆腐乳样品间微生物群落主成分分析结果
基于属水平,不同豆腐乳样品间的微生物群落主成分分析结果(principal component analysis,PCA)见图5。由图5a可知,样品LA与LD相距较远,说明这两种样品间的细菌菌群多样性差异较大,样品LA与LB主要集中在第三象限,表明这两种样品中的细菌菌落有一定的相似性,样品LC与其他3个样品相距较远,细菌菌群结构差异较大。由图5b可知,样品LA主要集中在第四象限,与其他3个样品分布距离较大,这可能是因为样品LA主要以纯种毛霉发酵而成。样品LC和LD主要集中在第一象限,表明这两个样品真菌菌群多样性差异较小,样品LB集中在第三象限,与其他3个样品相距较远。真菌菌群结构差异较大,这可能是因为生产环境中微生物种类复杂,温度及湿度等参数不固定导致样品间微生物种类及丰度有所差异[27]。综上,可以看出纯种发酵豆腐乳与其他自然发酵豆腐乳的微生物群落差异性较大。
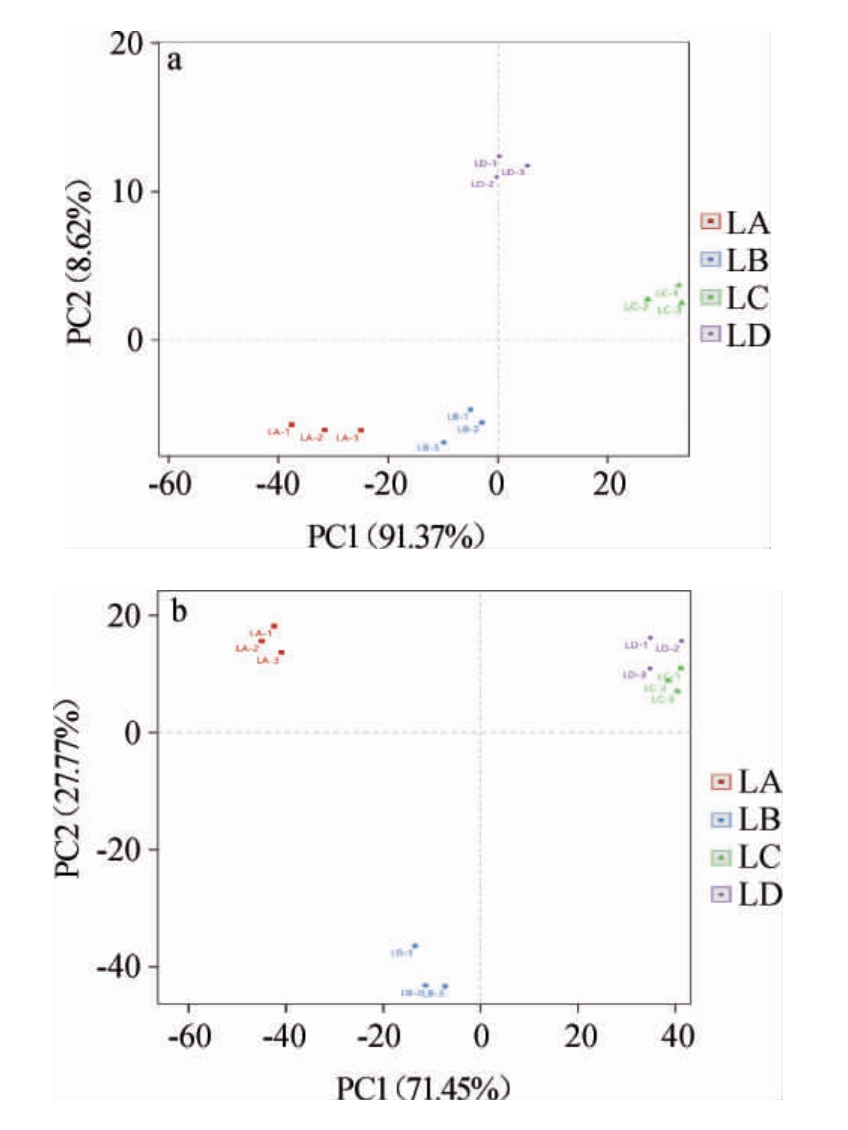
图5 基于细菌(a)和真菌(b)属水平不同豆腐乳样品的主成分分析结果
Fig.5 Principal component analysis results of different sufu samples based on bacteria (a) and fungi (b) at genus level
3 结论
本研究基于高通量测序技术对4种豆腐乳样品中微生物菌群多样性进行较为全面的分析。结果表明,4种豆腐乳样品中的微生物多样性及丰度存在一定的差异,在门水平上,共有优势细菌门(平均相对丰度>1%)为变形菌门、厚壁菌门、拟杆菌门;优势真菌门为子囊菌门、毛霉菌门和担子菌门;在属水平,优势细菌属为假单胞菌属、乳杆菌属、透明颤菌属、魏斯氏菌属、不动杆菌属和明串珠菌属;优势真菌属为曲霉属、曲霉属、德巴利氏酵母属、短梗霉属、假丝酵母属和有孢圆酵母属。这些共有优势菌影响着豆腐乳发酵体系风味物质的生成和产品品质。利用高通量测序技术分析纯种发酵和自然发酵豆腐乳中微生物组成及相对丰度,为发酵豆腐乳产品的微生物结构提供了一定的理论依据。
[1]张蒙冉,李淑英,高雅鑫,等.传统发酵豆制品研究进展[J].食品科技,2021,46(1):98-104.
[2]万红芳,赵勇,王正全,等.生产菌种及环境微生物与腐乳品质关系研究进展[J].食品发酵与工业,2019,45(6):255-261.
[3]LIU L,CHEN X,HAO L,et al.Traditional fermented soybean products:processing,flavor formation,nutritional and biological activities[J].Crit Rev Food Sci Nutr,2022,62(7):1971-1989.
[4]LI F J,YIN L J,LU X,et al.Changes In angiotensin I-converting enzyme inhibitory activities during the ripening of Douchi (a Chinese traditional soybean product)fermented by various starter cultures[J].Int J Food Prop,2010,13(3):512-524.
[5]周锦绣,林奇,唐卿燕.我国腐乳生产用菌的研究现状[J].食品科技,2007,32(11):20-23.
[6]HAN B Z,ROMBOUTS F M,NOUT M J.A Chinese fermented soybean food[J].Int J Food Microbiol,2001,65(1-2):1-10.
[7]姚粟,于学健,白飞荣,等.中国传统发酵食品用微生物菌种名单的研究[J].食品与发酵工业,2017,43(9):238-258.
[8]陶康,吴凌伟,金晓芳,等.基于高通量基因测序分析腐乳微生物多样性[J].食品科学,2021,42(8):143-149.
[9]ZHANG Q,WANG C Z,LI B K,et al.Research progress in tofu processing: from raw materials to processing conditions[J].Crit Rev Food Sci Nutr,2018,58(9):1448-1467.
[10]GREEN S J,LEIGH M B,NEUFELD J D.Denaturing gradient gel electrophoresis(DGGE)for microbial community analysis[M]//MCGENITY T,TIMMIS K,NOGALES B.Hydrocarbon and Lipid Microbiology Protocols.Heidelberg:Springer Protocols Handbooks,2015:77-99.
[11]QUAN J,TIAN J.Circular polymerase extension cloning for high-throughput cloning of complex and combinatorial DNA libraries[J].Nat Protoc,2011,2(6):242-251.
[12]JOHNSONJS,SPAKOWICZDJ,HONFBY,et al.Evaluation of16S rRNA gene sequencing for species and strain-level microbiome analysis[J].Nat Commun,2019,10(1):1-11.
[13]DI BELLA J M,BAO Y,GLOOR G B,et al.High throughput sequencing methods and analysis for microbiome research[J].J Microbiol Method,2013,95(3):401-414.
[14]关统伟,向慧平,王鹏昊,等.基于高通量测序的郫县豆瓣不同发酵期细菌群落结构及其动态演替[J].食品科学,2018,39(4):106-111.
[15]CHEN G,CHEN C C,LEI Z H.Meta-omics insights in the microbial community profiling and functional characterization of fermented foods[J].Trends Food Sci Tech,2017,65:23-31.
[16]李荣源,卢红梅,秦兴,等.高通量测序分析赤水晒醋各生产阶段微生物群落结构变化[J].食品科学,2020,41(24):78-86.
[17]苟萌,胡婕,张彤彤,等.Illumina MiSeq高通量测序技术研究原料乳冷藏过程中微生物群落演替规律[J].中国食品学报,2021,21(7):313-319.
[18]石黎琳,牟方婷,李安,等.基于高通量测序技术分析腐乳自然发酵过程微生物多样性[J].中国酿造,2021,40(2):144-149.
[19]HUANG X,YU S,HAN B,et al.Bacterial community succession and metabolite changes during sufu fermentation[J].LWT-Food Sci Technol,2018,97:537-545.
[20]HE B,LI H,HU Z,et al.Difference in microbial community and taste compounds between Mucor-type and Aspergillus-type Douchi during koji-making[J].Food Res Int,2019,121:136-143.
[21]杜瑞,王柏辉,罗玉龙,等.应用Illumina MiSeq 测序技术比较传统发酵乳、肉食品中细菌多样性[J].中国食品学报,2021,21(2):269-277.
[22]胡旺,陈梦娟,尹含靓,等.利用Illumina Miseq测序技术分析浏阳霉豆腐制作过程中微生物变化[J].食品与发酵工业,2021,47(24):265-270.
[23]MEI Y Z,YANG L,XU X T,et al.Complete genome sequence of a bacterium Pseudomonas fragi P121,a strain with degradation of toxic compounds[J].J Biotechnol,2016,224:68-69.
[24]BANSAL R,HULBERT S H,REESE J C,et al.Pyrosequencing reveals the predominance of Pseudomonadaceae in gut microbiome of a gall midge[J].Pathogens,2014,3(2):459-472.
[25]刘筱雪,袁文娟,丁涛,等.基于高通量测序对四川怀远特色发酵食品微生物群落结构分析[J].四川大学学报(自然科学版),2019,56(3):537-543.
[26]张鹏飞,乌日娜,武俊瑞.传统发酵大豆制品挥发性成分和微生物多样性的研究进展[J].中国酿造,2018,37(12):1-6.
[27]廖新浴,陈信贤,刘东红,等.不同种类腐乳产品中优势菌种的分离与鉴定[J].食品安全质量检测学报,2018,9(14):3755-3759.