传统发酵酸面团是由面粉或者其他谷物同水与环境中的微生物经自然发酵后保留一部分作为下次发面的菌种,也被称为“面头”、“酵面”、“酵子”和“面起子”[1-2],其是由酵母菌、乳酸菌及霉菌等多菌种组成的混合发酵体系[3],是一个微型的菌种资源库。
近年来,国内外越来越多的研究者针对传统酸面团中的微生物菌群进行研究。RANDAZZO C L等[4]使用聚合酶链式反应-变性梯度凝胶电泳(polymerase chain reactiondenaturing gradient gel electrophoresis,PCR-DGGE)技术和培养法对传统西西里小麦酸面团样品的细菌菌群结构进行研究,结果发现,优势菌种分别为旧金山乳杆菌(Lactobacillus sanfranciscensis)、发酵乳杆菌(Lactobacillus fermentium)、戊糖乳杆菌(Lactobacillus pentosus)和植物乳杆菌(Lactobacillus plantarum);MICHEL E等[5]分别通过培养法、实时定量PCR(real-time quantitative PCR,RT-qPCR)法和高通量测序技术对16个法国发酵面团样品中乳酸菌的相对丰度进行研究,发现13个样品的优势菌种均为旧金山乳杆菌;陶东娅等[6]利用变性梯度凝胶电泳技术和基因测序技术在粘豆包酸面团中检测到7种乳酸菌;张新杰[7]使用DGGE技术对不同配比原料制作的传统自然发酵粘豆包发酵面团进行研究,共检测出18种乳酸菌;乌日娜等[8]利用PCR-DGGE技术在酸汤子玉米面团中检测出4种细菌菌种,其中类肠膜魏斯氏菌(Weissella paramesenteroides)为优势菌种;杨健等[9]对黑龙江省肇源县粘豆包发酵面团中细菌菌群多样性进行检测,推测发酵乳杆菌、植物乳杆菌为优势菌种;吴斯日古冷[10]结合经典分离鉴定法和电泳技术研究内蒙古西部地区酸面团样品中生物多样性,发现优势乳酸菌种为旧金山乳杆菌、植物乳杆菌;张国华等[11]研究发现,山西省临汾市传统酸面团样品中参与发酵的微生物大约有210个种水平物种信息,且以乳杆菌属为优势菌属,以旧金山乳杆菌为优势菌种;李晓敏等[2]对采自山西、河北、青海、甘肃和河南5个地区酸面团样品中的细菌菌群结构进行研究,发现优势菌群为乳杆菌属、乳球菌属和魏斯氏菌属;高静等[12]采用高通量测序分析老面中的菌群多样性,发现在属水平上存在地区差异,河北、山西、山东样品中以片球菌属(Pediococcus)为主,而云南样品中以乳杆菌属(Lactobacillus)为主。传统发酵面团中微生物的多样性受多种因素影响,包括环境微生物种类、谷物种类及来源、和面用水、发酵工艺及其他成分等[13-14]。因此,研究不同地域来源酸面团中微生物菌群的组成,对菌种的筛选具有一定的指导意义。
本研究利用高通量测序技术对内蒙古不同地区传统酸面团的细菌群落多样性进行解析,并对酸面团中的细菌代谢途径进行预测和对比,以期为筛选优质特性的菌种资源提供参考。
1 材料与方法
1.1 材料与试剂
1.1.1 材料
传统酸面团样品:内蒙古西部地区(编号为nx1~nx5)、中部地区(编号为nz1~nz4)和东部地区(编号为nd1~nd5)的农户家中,共采集14个地市的样品。
1.1.2 试剂
MoBio PowerSoil脱氧核糖核酸(deoxyribonucleic acid,DNA)Isolation Kit:德国Qiagen公司;Agencourt AMPure
AMPure XP:美国Beckman Coulter公司;细菌16S rRNA V3-V4区PCR扩增引物338F(5'-AC-TCCTACGGGAGGCAGCA-3')和806R(5'-GGACTACHVG-GGTWTCTAAT-3'):北京奥维森基因科技有限公司。其他试剂均为国产分析纯。
XP:美国Beckman Coulter公司;细菌16S rRNA V3-V4区PCR扩增引物338F(5'-AC-TCCTACGGGAGGCAGCA-3')和806R(5'-GGACTACHVG-GGTWTCTAAT-3'):北京奥维森基因科技有限公司。其他试剂均为国产分析纯。
1.2 仪器与设备
Illumina MiSeq高通量测序平台:美国Illumina公司;9700 PCR仪、Step One Plus QPCR仪:美国ABI公司;5702 R台式冷冻离心机:德国Eppendorf公司。
1.3 方法
1.3.1 细菌菌群多样性分析
根据MoBio PowerSoil Isolation Kit操作手册提取酸面团样品中微生物的总DNA,以其为模板,采用338F和806R PCR扩增16S rDNA V3-V4区基因序列。将PCR扩增产物送至北京奥维森基因科技有限公司,采用Illumina MiSeq平台进行高通量测序。
1.3.2 数据处理及分析
利用Flash软件根据overlap原始数据进行拼接,采用Uchime软件[15]去除嵌合体,得到优质序列。采用Uclust软件在97%相似度下构建操作分类单元(operational taxonomic unit,OTU)。利用Mothur软件计算各样本的α多样性指数,基于Silva V128[16]、RDP v16和Greengenes v13.5数据库对OTU代表性序列进行注释;利用Qiime软件[17]对样品β多样性进行分析,OTU聚类分析通过R软件实现。采用PICRUSt软件[18]和京都基因百科全书(Kyoto Encyclopedia of Genes and Genomes,KEGG)数据库对样品中细菌的代谢功能进行预测。
2 结果与分析
2.1 传统酸面团样品细菌群落高通量测序结果及Alpha多样性分析结果
稀疏曲线可以判定样本的取样大小及测序深度是否合理[19]。3个地区酸面团样品的稀疏曲线见图1。
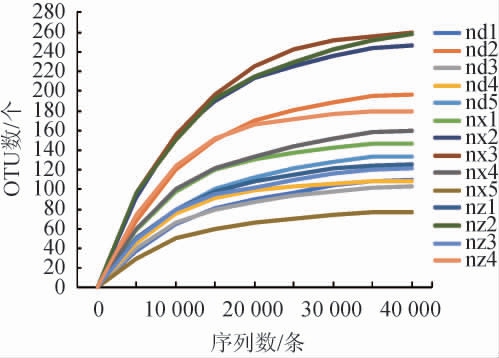
图1 传统酸面团样品中细菌菌群的稀疏曲线
Fig.1 Rarefaction curves of bacterial flora in traditional sourdough samples
由图1可知,随着序列数的增加,14个样品的OTU数量呈先增加后趋于平坦的趋势,说明此次测序数据合理,测序深度能反映样品中的物种信息。传统酸面团样品中细菌菌群的高通量测序及α多样性分析结果见表1。
表1 传统酸面团样品中细菌菌群高通量测序及Alpha多样性分析结果
Table 1 Results of high-throughput sequencing and Alpha diversity analysis of bacterial flora in traditional sourdough samples

由表1可知,14个酸面团样品共产生1 230 198条高质量序列,得到656个OTU,其Coverage为99.77%~99.94%,说明样品中序列被测出的概率较高。由表1亦可知,不同样品间的α多样性指数存在较大差异,其中样品nx3的OTU数(260)和Chao1指数(384.50)最大,而样品nd4的Shannon指数(4.15)最大,说明样品nx3中细菌菌群丰度最高,样品nd4中细菌菌群多样性最高。为进一步分析不同地区酸面团样品中细菌菌群的差异,对不同地区酸面团样品中细菌菌群的OTU绘制韦恩图,并基于OTU进行聚类分析,结果见图2。
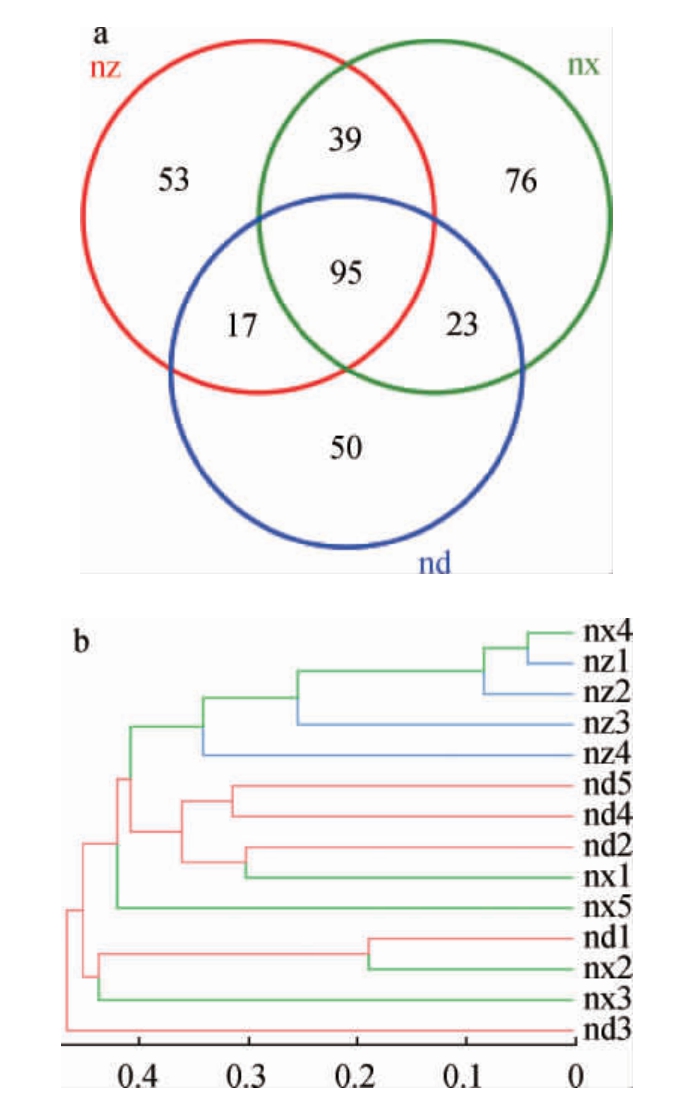
图2 细菌群落OTU水平的Venn图(a)及聚类分析结果(b)
Fig.2 Venn diagram (a) and cluster analysis result (b) of OTU level of bacterial community
由图2a可知,内蒙古东部地区酸面团样品的OTU数为185个,内蒙古中部地区酸面团样品的OTU数为204个,内蒙古西部地区酸面团样品的OTU数为233个,三个地区样品中共有的OTU数为95个。可见,来自不同地区的酸面团在细菌群落组成上存在相似性。内蒙古东部、中部和西部地区酸面团样品特有的OTU数分别为50个、53个和76个,分别占各地区总OTU数的27.03%、25.98%和32.62%,说明在三个地区酸面团样品中细菌组成存在一定的差异。
由图2b可知,3个地区14个酸面团样品可聚为3类,样品nd1、nx2、nx3聚为一类,相似性较高,样品nd3单独聚类,其他样品聚为一类,表明其细菌群落结构存在一定的相似性和差异性。
2.2 传统酸面团样品中细菌群落组成分析
2.2.1 基于门水平酸面团样品细菌菌群结构分析结果
为了解传统酸面团中细菌群落的组成,对不同地区酸面团样品的OTU进行分类学鉴定,在门分类学水平揭示不同地区间酸面团样品细菌群落的结构差异,结果见图3。
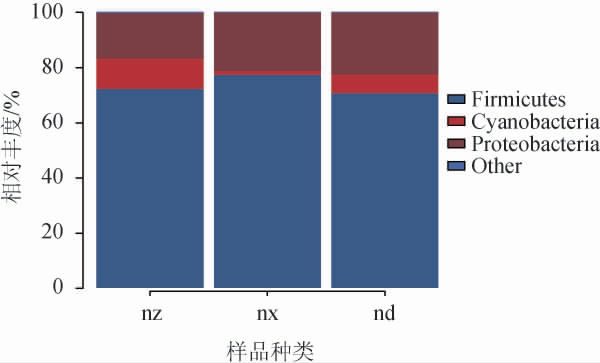
图3 基于门水平不同地区酸面团样品细菌群落组成分析结果
Fig.3 Analysis results of bacterial community composition of sourdough samples from different regions based on phylum level
Other:相对丰度<1%的细菌门。
由图3可知,在门水平上,内蒙古西部地区酸面团样品的优势细菌门(平均相对丰度>1%)为厚壁菌门(Firmicutes)、变形菌门(Proteobacteria),平均相对丰度分别为85.63%、13.78%;内蒙古中部地区酸面团样品的优势细菌门为厚壁菌门(Firmicutes)、蓝细菌门(Cyanobacteria)、变形菌门(Proteobacteria),平均相对丰度分别为83.33%、6.57%、6.31%;内蒙古东部地区酸面团样品的优势细菌门为厚壁菌门(Firmicutes)、变形菌门(Proteobacteria),平均相对丰度分别为83.33%、16.24%;3个地区14个酸面团样品中的细菌菌群主要归属于厚壁菌门(Firmicutes)、变形菌门(Proteobacteria)和蓝细菌门(Cyanobacteria),这与邢小龙等[20-21]的研究结果一致,与意大利成熟酸面团中的细菌门相同[22]。
2.2.2 基于属水平酸面团样品细菌菌群结构分析结果
在属分类学水平上探究不同地区酸面团样品细菌群落的组成,结果见图4。
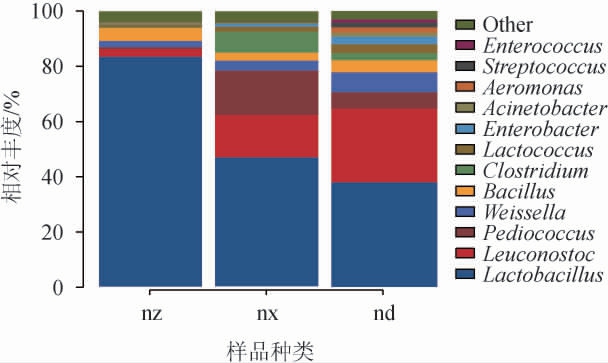
图4 基于属水平酸面团样品细菌群落组成分析结果
Fig.4 Analysis results of bacterial community composition of sourdough samples based on genus level
Other:相对丰度<1%的细菌属。
由图4可知,从3个地区14个酸面团样本中共检测出11个优势细菌属(平均相对丰度>1%),不同地区酸面团样品优势细菌属的种类及相对丰度差异明显,内蒙古中部地区酸面团样品中优势细菌属分别为乳杆菌属(Lactobacillus)、芽孢杆菌属(Bacillus)、明串珠菌属(Leuconostoc)和魏斯氏菌属(Weissella),平均相对丰度分别为75.52%、4.26%、2.52%、2.02%;内蒙古西部地区样品中优势细菌属分别为乳杆菌属(Lactobacillus)、片球菌属(Pediococcus)、明串珠菌属(Leuconostoc)、梭菌属(Clostridium)、魏斯氏菌属(Weissela)和芽孢杆菌属(Bacillus),平均相对丰度分别为41.92%、14.40%、13.73%、6.75%、3.20%、2.66%;内蒙古东部地区酸面团样品中优势细菌属分别为乳杆菌属(Lactobacillus)、明串珠菌属(Leuconostoc)、魏斯氏菌属(Weissella)、片球菌属(Pediococcus)、芽孢杆菌属(Bacillus)、乳球菌属(Lactococcus)和肠杆菌属(Enterobacter),平均相对丰度分别为27.42%、19.33%、5.19%、4.30%、3.14%、2.24%、2.04%。
乳杆菌属是乳酸菌中最重要的组成部分,多数样品以乳杆菌属为优势菌群[2,23-25],这可能是因为乳杆菌属具有独特的基因组特征,也可能是细胞内的碳水化合物活性酶存在特异性[26],能对碳水化合物高度适应并加以代谢,从而增强了其竞争力[27]。片球菌属能赋予食品柔和的酸味及香气,改善食品的营养和品质[28]。芽孢杆菌属能够分泌α-淀粉酶,有利于样品的糖化作用[29]。Clostridium由于产生丁酸和乙酸进而转化成丁醇、丙酮和乙醇,可能会提供酸面团面制品更丰富的风味物质[25]。不同地区酸面团样品中细菌组成存在一定差异,这可能是面粉基质和制作工艺不同的原因。也有研究表明,环境因素对细菌群落多样性和丰富度也有潜在影响[30]。
2.3 酸面团样品细菌菌群代谢功能预测分析结果
采用PICRUSt软件和KEGG数据库对样品中细菌菌群的代谢功能进行预测,结果见图5。
由图5a可知,内蒙古东部、中部、西部地区酸面团样品中细菌群落的功能(平均相对丰度>1%)均包括4类,分别为新陈代谢(metabolism)(79.87%、80.90%、79.24%)、遗传信息处理(genetic information processing)(12.95%、13.85%、14.21%)、环境信息处理(environmental information processing)(3.40%、2.60%、2.82%)、细胞加工(cellular processes)(3.122%、2.06%、2.83%)。
由图5b可知,内蒙古东部、中部、西部地区酸面团样品中细菌菌群的新陈代谢功能包括碳水化合物代谢(carbohydrate metabolism)(14.64%、13.25%、14.46%)、辅助因子和维生素代谢(metabolism of cofactors and vitamins)(111.56%、12.34%、11.47%)、氨基酸代谢(aminoacidmetabolism)(10.56%、10.33%、10.69%)、脂质代谢(lipid metabolism)(7.91%、10.00%、8.74%)、萜类化合物和聚酮化合物代谢(metabolism of terpenoids and polyketides)(9.02%、8.76%、7.75%)、能量代谢(energy metabolism)(5.39%、5.30%、4.88%)、外源生物降解和代谢(xenobiotics biodegradation and metabolism)(5.07%、5.11%、5.39%)、多聚糖的生物合成和代谢(glycan biosynthesis and metabolism)(3.03%、2.90%、2.83%)、核苷酸代谢(nucleotide metabolism)(2.23%、2.39%、2.44%)、其他次生代谢产物的生物合成(biosynthesis of other secondary metabo lites)(1.64%、1.37%、1.43%)。遗传信息处理功能包括复制与修复(replication and repair)(5.96%、6.37%、6.72%)、翻译(translation)(3.32%、3.64%、3.67%)、折叠分选与降解(folding,sorting and degradation)(2.99%、3.22%、3.16%)。细胞加工功能在内蒙古东部地区酸面团样品只涉及细胞生长和死亡(cell growth and death)(1.35%)一条功能通路,而中部、西部地区酸面团样品包括细胞生长和死亡(cell growth and death)(1.46%、1.48%)、细胞运动(cell growth and death)(1.01%、1.33%)两条通路。环境信息处理只涉及膜运输(membrane transport)(2.91%、2.29%、2.65%)一条功能通路。综上,3个地区酸面团样品中细菌群落的基因功能主要是碳水化合物代谢、辅助因子和维生素代谢、氨基酸代谢、脂质代谢、萜类化合物和聚酮化合物代谢5条KEGG途径。
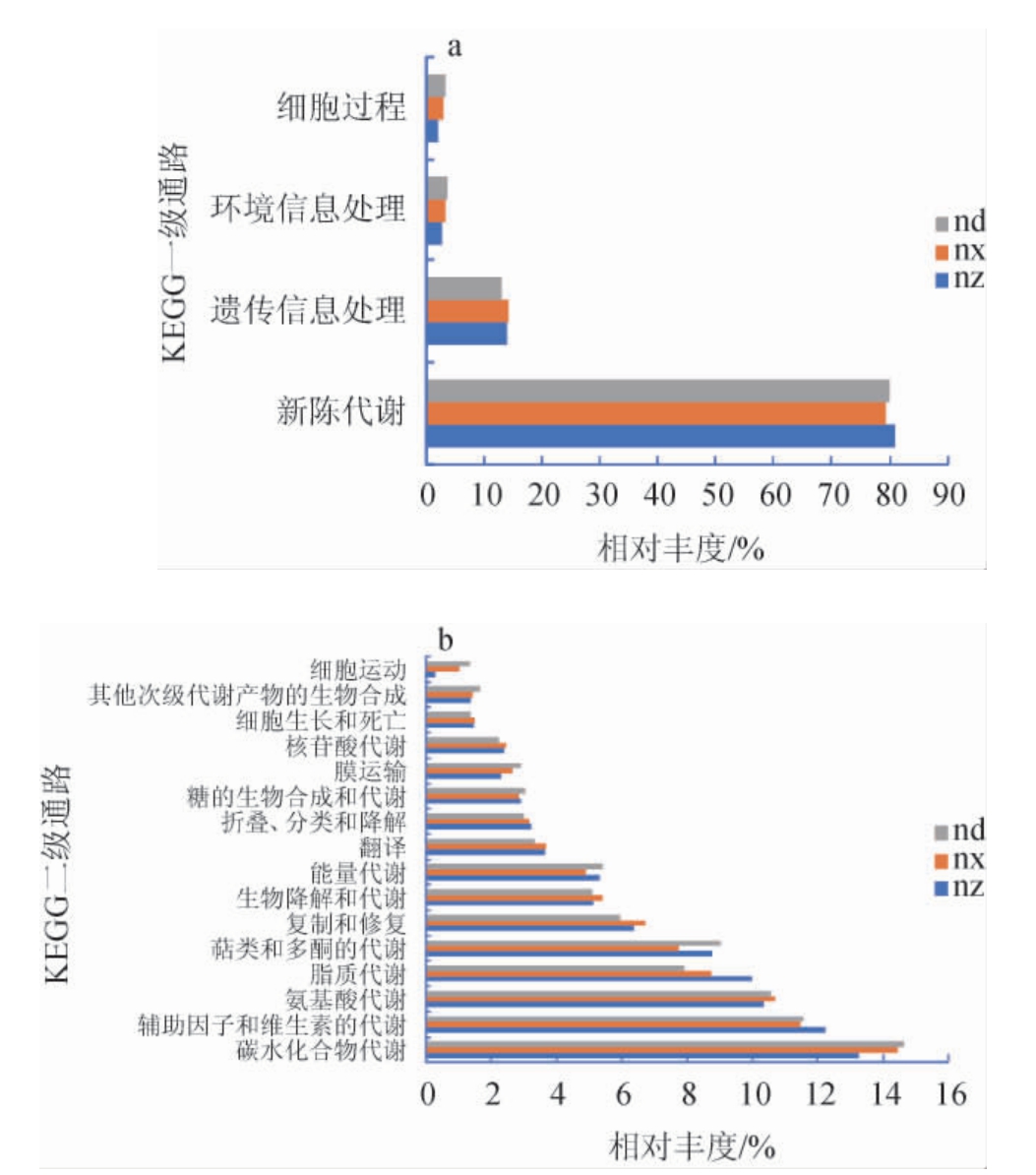
图5 传统酸面团中细菌群落所预测到KEGG一级(a)及二级(b)通路的相对丰度
Fig.5 Relative abundance of KEGG primary (a) and secondary (b)pathways predicted in bacterial community of traditional sourdough
3 结论
利用Illumina Miseq高通量测序技术对内蒙古地区传统酸面团样品中的细菌菌群多样性进行解析,并结合PICRUSt软件预测细菌功能的变化。结果发现,三个地区的样品共产生1 230 198条高质量序列,得到656个OTU,其覆盖率为99.77%~99.94%。3个地区酸面团样品检出3个优势细菌门和11个优势细菌属,共有优势细菌门为厚壁菌门和变形菌门,共有优势细菌属为乳杆菌属、明串珠菌属、魏斯氏菌属和芽孢杆菌属,不同地区样品间细菌菌群多样性和丰度存在明显差异。传统酸面团样品中细菌群落的基因功能主要富集在碳水化合物代谢、辅助因子和维生素代谢、氨基酸代谢、脂质代谢及萜类化合物和聚酮化合物代谢5条KEGG途径。本研究不仅可为乳酸菌发酵剂的筛选提供参考,还可为进一步研究不同地区面团发酵过程中起关键作用的菌种提供依据。
[1]刘娜.区域特色酸面团馒头及其优选乳酸菌发酵特性比较研究[D].无锡:江南大学,2014.
[2]李晓敏,韩伟,黎琪,等.传统酸面团菌群结构及其酵制馒头风味物质分析[J].食品科学,2021,42(10):162-170.
[3] ZHANG J,LIU W,SUN Z,et al.Diversity of lactic acid bacteria and yeasts in traditional sourdoughs collected from western region in Inner Mongolia of China[J].Food Control,2011,22(5):767-774.
[4] RANDAZZO C L,HEILIG H,RESTUCCIA C,et al.Bacterial population in traditional sourdough evaluated by molecular methods[J].J Appl Microbiol,2005,99(2):251-258.
[5]MICHEL E,MONFORT C,DEFFRASNES M,et al.Characterization of relative abundance of lactic acid bacteria species in French organic sourdough by cultural,qPCR and MiSeq high-throughput sequencing methods[J].Int J Food Microbiol,2016,239:35-43.
[6]陶东娅,金银旗.不同地区粘豆包酸面团中微生物多样性分析[J].食品研究与开发,2016,37(13):156-159.
[7]张新杰.东北地区传统粘豆包发酵面团的微生物多样性及其与理化性质的关系[D].哈尔滨:东北农业大学,2017.
[8]乌日娜,张颖,张红萧,等.酸汤子玉米面团中微生物多样性分析[J].食品科学,2017,38(12):21-26.
[9]杨健,孙大庆.东北粘豆包发酵面团中细菌多样性研究[J].黑龙江八一农垦大学学报,2015,27(5):83-86.
[10]吴斯日古冷.内蒙古西部地区酸面团中酵母菌和乳酸菌的分离鉴定及其生物多样性研究[D].呼和浩特:内蒙古农业大学,2011.
[11]张国华,王伟,涂建,等.基于宏转录组学技术解析传统酸面团中微生物代谢机理[J].中国粮油学报,2019,34(11):10-16.
[12]高静,刘敏,吴丹丹,等.基于高通量测序分析老面中菌群多样性及其对馒头品质的影响[J].中国粮油学报,2019,34(7):13-19.
[13]VUYST L D,KERREBROECK S V,LEROY F.Microbial ecology and process technology of sourdough fermentation[J].Adv Appl Microbiol,2017,100:49-160.
[14] ARORA K,AMEUR H,POLO A,et al.Thirty years of knowledge on sourdough fermentation:A systematic review[J].Trends Food Sci Tech,2021,108:71-83.
[15] EDGAR R C.Search and clustering orders of magnitude faster than BLAST[J].Bioinformatics,2010,26(19):2460-2461.
[16]CHRISTIAN Q,ELMAR P,PELIN Y,et al.The SILVA ribosomal RNA gene database project: improved data processing and web-based tools[J].Nucleic Acids Res,2013,41(D1):D590-596.
[17]CAPORASO J G,KUCZYNSKI J,STOMBAUGH J,et al.QIIME allows analysis of high-throughput community sequencing data[J]. Nat Meth,2010,7(5):335-336.
[18] LANGILLE M G I,ZANEVELD J,GREGORY CAPORASO J,et al.Predictive functional profiling of microbial communities using 16S rRNA marker gene sequences[J].Nat Biotechnol,2013,31(9):814-821.
[19]郝卓莉.酸菜发酵期间细菌群落结构动态变化分析[J].中国酿造,2020,39(7):56-61.
[20]邢小龙.河南地区老酵面团菌群结构及优势菌种复配研究[D].郑州:河南农业大学,2020.
[21]LIU X,ZHOU M,CHEN J,et al.Bacterial diversity in traditional sourdough from different regions in China[J].LWT-Food Sci Technol,2018:251-259.
[22] RIZZELLO C G,CAVOSKI I,TURK J,et al.Organic cultivation of Triticum turgidum subsp durum is reflected in the flour-sourdough fermentation-bread axis[J].Appl Environ Microbiol,2015,81(9):3192-3204.
[23]李梁,薛蓓,罗章,等.基于高通量测序分析冻干老面与新鲜老面细菌群落结构[J].中国食品添加剂,2018(10):126-131.
[24]张海燕,柳陈坚,刘祥祥,等.基于高通量测序分析老面细菌多样性[J].食品工业科技,2016,37(15):145-149.
[25]白雪.辽东地区酸汤子面团微生物多样性分析及其对面包品质的影响[D].沈阳:沈阳师范大学,2020.
[26] SWANSON K S,GIBSON G R,HUTKINS R,et al.The International Scientific Association for Probiotics and Prebiotics (ISAPP) consensus statement on the definition and scope of synbiotics[J].Nat Rev Gastroen Hepat,2020,17(11):687-701.
[27]VUYST L,KERREBROECK S,LEROY F.Microbial ecology and process technology of sourdough fermentation[J]. Adv Appl Microbiol,2017,100:46-160.
[28]曾雪峰.淡水鱼发酵对酸鱼品质影响的研究[D].无锡:江南大学,2013.
[29] LI Z,LING C,BAI Z,et al.Cultivable bacterial diversity and amylase production in three typical Daqus of Chinese spirits[J]. Int J Food Sci Tech,2014,49(3):776-786.
[30]FU W,RAO H,TIAN Y,et al.Bacterial composition in sourdoughs from different regions in China and the microbial potential to reduce wheat allergens[J].LWT-Food Sci Technol,2019,117:669-672.