1 基因组改组技术的基本概念及发展历史
基因组改组技术是在脱氧核糖核酸(deoxyribonucleic acid,DNA)重组基础上,结合传统育种技术,采用多亲本之间的DNA重组和全基因组片段交换,将多个优良表型重组在一起的过程。20世纪90年代中期,STEMMER W P[1]首次提出基因组改组的概念。2002年,ZHANG Y X等[2]在《Nature》杂志上发表了首篇应用基因组改组技术改良微生物菌株的报道。仅用1年的时间,通过2轮原生质体的递归融合,快速的增强了弗氏链霉菌(Streptomyces fradiae)合成泰乐菌素(Tylosin)的能力[2]。该基因组改组技术的改良成效,相当于诱变育种工作20年的效果[2]。
2 基因组改组技术的一般流程
基因组改组技术的一般流程见图1,包括亲本库的构建、原生质体的递归融合和目标表型的筛选3个部分。
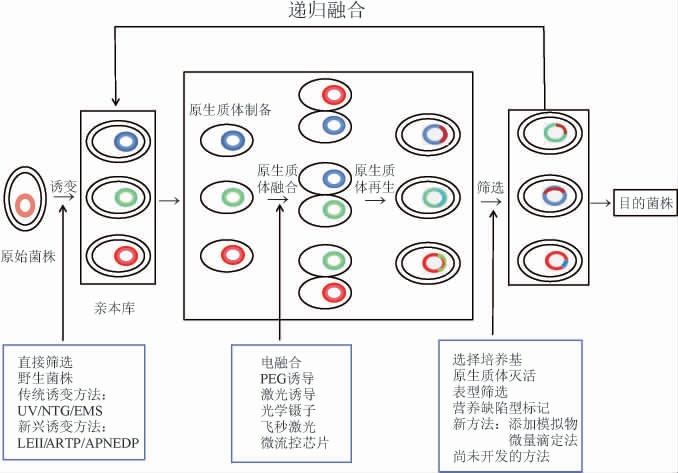
图1 基因组改组技术的一般流程
Fig.1 General process of genome shuffling technique
2.1 亲本库的构建
基因组改组的第一步是构建一个基因多样化的亲本库。首先要初步筛选具有良好的遗传多样性和表型变化明显的原始菌株,然后对原始菌株进行诱变处理,筛选出遗传性状优良的不同类型的突变株,作为后续原生质体融合的亲本库。目前,诱变仍是常见的亲本库的构建方法,通常用传统的物理诱变(紫外(ultraviolet,UV)诱变)或化学诱变(亚硝基胍(nitrosoguanidine,NTG)诱变、甲基磺酸乙酯(ethyl methyl sulfonate,EMS)诱变)处理,经过一轮或多轮诱变,从而获得丰富基因型的正突变株[3]。例如,王灏等[4]通过紫外诱变和化学诱变对酿酒酵母f4、f5、f6进行诱变,获得具有耐高温和耐乙醇能力的正突变株。但传统的诱变方法具有诱变时间长和诱变效率低的局限性,为了提高亲本菌株的突变效率,一些新兴的诱变方法得到了发展和应用,如常压室温等离子体(atmospheric room temperature plasma,ARTP)、常压非平衡放电等离子体(atmospheric nonequilibrium discharge plasma,APNEDP)和低能离子注入(low energy ion implantation,LEII)等方法[5-6]。NIU F X等[7]利用ARTP诱变,获得了9株莽草酸(shikimic acid,SA)滴度较高、16株生长速度较快、7株莽草酸滴度和生长速度均优于亲本的改良菌株。此外,根据随机组合的策略,为了更加合理地构建亲本库,如多重自动基因组工程(multiple automated genome engineering,MAGE)和真核生物多重自动基因组工程(eukaryotic multiplex genome engineering,eMAGE),可以引入到基因组改组中,筛选出具有更高多样性和更多功能的大型亲本库[7]。
2.2 原生质体的递归融合
原生质体的递归融合是基因组改组的关键步骤。来自亲本的原生质体经历混合、融合、再生等过程,将来自多个亲本菌株的遗传特征进行融合,使不同基因组的基因整合到一个全新的基因组中。传统融合方法主要使用化学试剂(聚乙二醇)或电脉冲诱导细胞融合,其融合效率较低[3];激光诱导、光学镊子、飞秒激光和微流控芯片等新兴融合方法显著提高了原生质体的融合速率,改善了传统融合方法效率较低的问题[8-10]。例如,GONG J等[11]利用拥有超高时空分辨率的飞秒激光诱导红发夫酵母(Phaffia rhodozyma)的原生质体融合,约20 min之后能够发现细胞融合,160 min之内两个细胞融合成一个较大的细胞,融合效率达到80%。
原生质体融合的效率受融合方法的影响,反过来融合方法又因菌株而异[12]。因此,在进行原生质体融合之前,首先需要对单个菌株的融合方法进行优化,以保证原生质体的高效融合和再生,从而获得所需的重组菌株[13]。除了优化使用方法外,还可以从技术方面提高原生质体的融合效率。目前,原核细胞和真核细胞已经成功地通过高效的原生质体制备和融合来快速筛选各种菌株[14]。总之,融合速度越快,获得目的表型的速度越快。
2.3 目的表型的筛选
基因组改组的最后一步是目的表型的筛选。筛选方法种类多样,筛选技术由于菌株改良的目标而有所不同。一般的筛选方法包括表型观察筛选、产物理化特性筛选、基质利用能力筛选、选择性压力筛选、高通量筛选(high throughputscreening,HTS)等[15]。例如,通过水解圈、透明圈和抑菌圈等筛选产量提高的优良菌株[16];通过生长状态和选择培养基筛选耐受能力较好的优良菌株[17];通过高通量筛选方法筛选所需的目的表型和底物利用率提高的菌株[18]。目前出现了一些新颖的筛选方法,例如,添加菌株产物类似物至培养基中,以便有效分离得到高产菌株[19];使用颜色或荧光筛选代谢物产量提高的菌株,通过识别和筛选色素或受荧光影响的产品来选择目的菌株的理想表型[20-21]。基于光谱装置,可以建立检测范围广泛的化学物质的高通量筛选方法,通过合适的高通量筛选方法可以迅速的分析目的菌株的多种性状,并且对目的菌株进行多种方式的快速分类[22]。总之,目的表型的筛选方法越高效,那么目的表型所需的筛选时间越短。
3 基因组改组技术在微生物育种方面的应用
基因组改组技术是一种快速改善细胞表型的新型全基因组工程技术,该技术已经被广泛用于种内、种间和属间许多重要菌株的表型改良[5,21,23]。
3.1 应用基因组改组技术提高代谢物产量
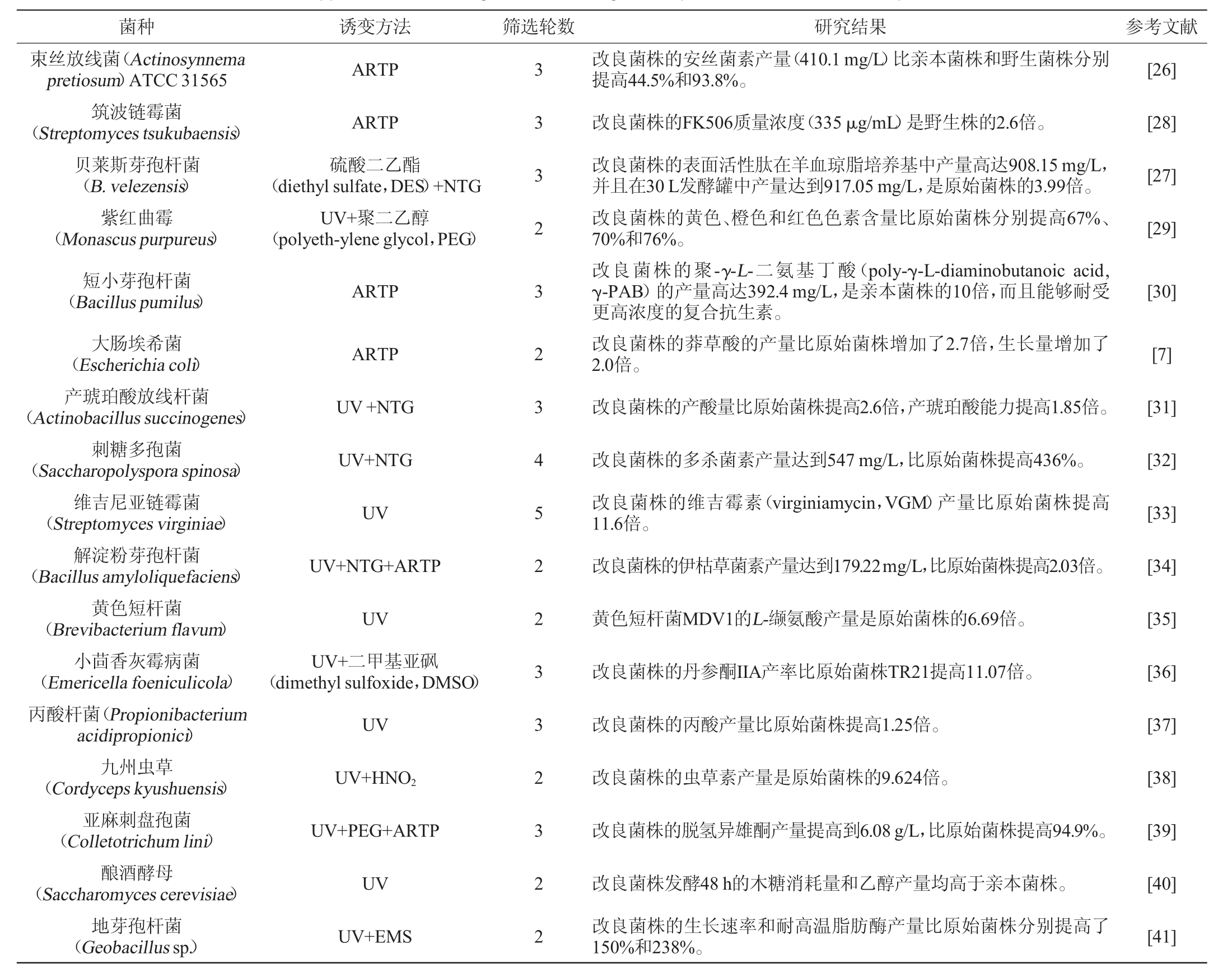
基因组改组最重要的应用就是提高代谢产物的产量(见表1)。代谢产物的高产性状在遗传水平上主要受多个基因的调控,利用传统的诱变育种方法都难以实现代谢物产量的提高[24]。基因组改组技术可以成功操纵复杂表型,快速获得所需表型的高产菌株。例如,安丝菌素具有较高的抗肿瘤活性和抗菌活性,能够抑制人肿瘤细胞的分裂,是一种很有前途的药物前体[25]。亲本菌株束丝放线菌(Actinosynnema pretiosum)ATCC 31565的安丝菌素产量较低,限制了其市场供应,LI J等[26]通过3轮基因组改组,使改良菌株G3-96的安丝菌素产量达到410.1 mg/L,比亲本菌株的安丝菌素产量提高44.5%,比野生菌株的安丝菌素产量提高93.8%。表面活性肽(surfactin)是一种很有前途的微生物脂肽,在食品、环境、农业和医药等领域有着广泛的应用,由于表面活性肽的生产效率较低,成本较高,其商业应用受到了很大限制。CHEN L等[27]采用基因组改组技术对贝莱斯芽孢杆菌(Bacillus velezensis)进行改良,经过3轮基因组改组,获得了高产、基因稳定的改良菌株F34,其表面活性肽产量从229.6 mg/L提高到908.15 mg/L,在30 L发酵罐中产量达到917.05 mg/L,是原始菌株的3.99倍。
表1 基因组改组技术提高代谢物产量的应用现状
Table 1 Application status of genome shuffling technique to increase metabolite production
3.2 应用基因组改组技术提高改良菌株耐受能力
微生物菌株的最重要性状之一就是对环境条件具有一定的耐受性,这些环境胁迫会严重影响微生物菌株的代谢活性和生产力[42]。许多耐受性的表型受基因组中分布的多基因调控,难以用传统育种、代谢工程或其他遗传操作方法进行修饰[43]。近年来,利用基因组改组技术提高微生物菌株对这些胁迫条件的耐受能力已经有了广泛的研究,如耐热性、耐糖性、耐酸性、耐盐性、乙醇耐受性和异丙醇耐受性(见表2)。例如:SHI D等[12]对酿酒酵母(S.cerevisiae)进行3轮基因组改组,获得的改良菌株SM-3能在55 ℃条件下的平板培养基上生长,提高了菌株SM-3的耐热性、乙醇耐受性和乙醇产量,缓解了温度对于酿酒酵母的发酵生产限制。YU L等[44]对鼠李糖乳杆菌(Lactobacillus rhamnosus)进行2轮基因组改组后,改组菌株的乳酸产量、细胞生长和葡萄糖消耗分别较野生型提高71.4%、44.9%和62.2%。基因组改组提高了菌株耐受葡萄糖能力的同时提高了L-乳酸的产量。TIAN K等[45]通过基因组改组技术提高了改良菌株G423耐酸性的同时也提高了Nisin的产量,使得菌株G423可以在pH3.7培养基上存活,且Nisin效价(4543 IU/mL)比原始菌株F44高59.9%。CAO X等[46]通过基因组改组技术提高了鲁氏酵母(Zygosaccharomyces rouxii)的耐盐性,筛选到的改良菌株S3-2不仅在高盐的培养基中生长状态良好,而且对氯化钾和氯化锂表现出更强的抗逆性,同时能够加速酱油的风味形成和提高酱油的品质。SNOEK T等[47]对酿酒酵母(S.cerevisiae)的亲本菌株进行了3轮基因组改组,淘汰了乙醇耐受性较低的改良菌株,得到了具有最大乙醇产量和乙醇耐受性提高7%的酿酒酵母改良菌株。DE GÉRANDO H M等[48]利用基因组改组技术对拜氏梭菌(Clostridium beijerinckii)进行改良,最终得到3株异丙醇耐受能力提高的改良菌株,其异丙醇耐受能力最高达50 g/L,比原始菌株提高42.85%。CHENG C等[49]通过基因组改组技术提高了酿酒酵母(S.cerevisiae)对木质纤维素水解物中存在的一种或多种抑制副产物的耐受能力,改组之后的酿酒酵母菌株对存在于木质纤维素生物质水解物中的抑制性副产物的耐受性增加。以上研究表明通过基因组改组技术不仅可以提高微生物菌株的单一性状,而且可以同时改善微生物菌株的多种性状,充分展示了该项技术的应用潜力。
表2 基因组改组技术提高菌株耐受能力的应用现状
Table 2 Application status of genome shuffling technique to improve strain tolerance

3.3 应用基因组改组技术提高底物利用效率
高效的底物利用能力是微生物菌株改良中所期望获得的目的表型之一,通过基因组改组的方法,可以获得底物利用率提高的菌株(见表3)。例如,JETTI K D等[51]利用酿酒酵母(S.cerevisiae)和树干毕赤酵母(Pichia stipitis)进行基因组改组,获得了一株底物的利用率明显高于亲本菌株的改良菌株SP2-18,该菌株能够利用木糖;JOHN R P等[23]使用德氏乳杆菌(Lactobacillus delbrueckii)和解淀粉芽孢杆菌(Bacillus amyloliquefaciens)作为亲本菌株,从淀粉废物中生产L-乳酸,3轮基因组改组之后,改良菌株F2从83 g/L木薯甘蔗渣(淀粉含量50%)中直接生产40 g/L乳酸,使淀粉转化率达到96%;赵晨等[58]以枯草芽孢杆菌(Bacillus subtilis)SFR-3A作为原始菌株,通过基因组改组技术筛选到具有高抗逆性D-核糖高产菌株SFRCP-100,该菌株在5 L发酵罐中的D-核糖产量达到38.2 g/L,其转化率达到35%。
表3 基因组改组技术提高菌株对底物转化率的应用现状
Table 3 Application status of genome shuffling technique to improve substrate conversion rate of strain

此外,通过基因组改组提高底物利用率也可以用于降解环境污染成分[59-62]。生物系统天生具有将复杂底物转化为简单分子的能力,使其能够对环境污染成分进行生物降解。由于从环境中分离出的野生菌株的降解速度较慢,因此,需要提高微生物降解高分子底物的能力。LEE B U等[59]通过基因组改组技术对具有降解2,4,6-三硝基甲苯(2,4,6-trinitrotoluene,TNT)能力的嗜麦芽寡养单胞菌(Stenotrophomonas maltophilia)OK-5进行4轮基因组改组后,改良菌株对TNT的降解作用比原始菌株提高6倍;YI L等[60]利用紫外诱变和亚硝基胍诱变结合基因组改组技术,提高了假单胞菌(Pseudomonas parafulva)YAB-1对于持久性有机污染物全氟辛酸(perfluorooctanoic acid,PFOA)的降解能力,改良菌株对PFOA的降解率达到58.6%,比原始菌株提高1.8倍[60]。
以上研究结果不仅表明了基因组改组技术在提高底物利用率方面的成功应用,而且证明了该项技术可以有效地促进微生物菌株对于有毒化合物的降解以及该项技术在改善环境微生物污染中的广阔的应用前景。
3.4 基因组改组技术在微生物菌株改良中其他方面的应用
研究发现通过基因克隆、突变或原生质体融合等技术可能会激活基因组中的沉默基因,从而产生新的活性代谢产物[63]。在这方面,一些研究人员已经证明了基因组改组在激活某些代谢方面也很有效(见表4),因此,基因组改组技术可以作为一项探索新基因和代谢产物的新方法。WANG M等[64]通过基因组改组得到的改良菌株G-444产生的18个新化合物,与原始菌株瘤座孢属(Tubercularia sp.)TF5的化合物在结构类型和代谢物上都有所不同,表明通过基因组改组之后一些沉默基因被激活;CAO X等[46]利用基因组改组技术筛选出的改良菌株S3-2在高盐液体发酵中,提升了酱油风味,而且减少了香气形成所需的总时间。此外,改良菌株S3-2具有较高的氨基酸氮含量,其中乙酸乙酯含量为对照的2.38倍,并且改良菌株S3-2使4-羟基-2-乙基-5-甲基-3-呋喃酮(4-hydroxy-2-ethyl-5-methyl-3-furanone)的生成率提高75%,同时产生了4-乙基愈创木酚(4-ethylguaiacol)这一重要的风味成分。
此外,通过基因组改组技术使微生物菌株的其他目的表型也得到了显著提高(见表4),如提高了植物乳杆菌(Lactobacillus plantarum)163的抑菌活性,提高了乳酸片球菌(Pediococcus acidilactici)对鸡沙门氏菌(Salmonella gallinarum)的抑菌活性,提高了植物乳杆菌(Lactobacillus plantarum)C88的粘附力,提高了链霉菌(Streptomyces bikiniensis)对黄瓜枯萎病菌(Cucumber fusarium)的生防活性[65-68]。充分说明了基因组改组技术是一种范围广泛、功能强大的微生物菌株改良技术。
表4 基因组改组技术在微生物育种其他方面的应用现状
Table 4 Application status of genome shuffling technique in other aspects of microbial breeding

4 结论与展望
基因组改组作为一种快速改良微生物菌株全基因组的技术,既可以对多个亲本的全基因组进行改良,又不需要十分了解改良菌株的遗传背景,成功地完成了许多微生物菌株的表型改良,其中包括药用微生物菌株,农业微生物菌株和工业微生物菌株的改良,具有极大的应用于实际生产的潜力,促进了微生物菌种选育工作的技术方法和路线上的变革,开创了菌种改良和改造工作的新局面[69]。但是基因组改组技术仍有较大的发展空间,首先,基因组改组技术可以与合理的基因工程相结合,可以提前将具有明确遗传背景的单个基因、代谢途径或代谢网络进行定向改造,然后用于进一步的基因组改组。而且,随着基因组学和生物信息学的发展,基因组改组作为连接其他技术的重要桥梁,将为阐明代谢网络和调控机制提供更大的机会。此外,根据自动化装置和快速检测方法的发展,高通量筛选方法也将随之更新。因此,建立普适的高通量筛选方法仍然存在较大困难。
相信随着多元策略创造遗传多样性、先进的原生质体诱导技术和高效的高通量筛选方法的应用,基因组改组技术将在未来的微生物菌株改良中发挥出更加重要的作用,创造出更大的社会价值和经济价值。
[1]STEMMER W P.DNA shuffling by random fragmentation and reassembly: in vitro recombination for molecular evolution[J].Proc Natl Acad Sci USA,1994,91(22):10747-10751.
[2] ZHANG Y X,PERRY K,VINCI V A,et al.Genome shuffling leads to rapid phenotypic improvement in bacteria[J].Nature,2002,415(6872):644-646.
[3]赵丽红,陈威,高艳娇,等.基因组重排技术在微生物育种中的应用研究进展[J].江苏农业科学,2018,46(18):1-5.
[4]王灏,王航,孟春,等.基因组改组技术选育耐高温、耐高乙醇酿酒酵母菌株的研究[J].微生物学通报,2007,34(4):705-708.
[5]GU C,WANG G,MAI S,et al.ARTP mutation and genome shuffling of ABE fermentation symbiotic system for improvement of butanol production[J].Appl Microbiol Biotechnol,2017,101(5):2189-2199.
[6]XU F,JIN H,LI H,et al.Genome shuffling of Trichoderma viride for enhanced cellulase production[J].Ann Microbiol,2012,62(2):509-515.
[7] NIU F X,HE X,HUANG Y B,et al.Biosensor-guided atmospheric and room-temperature plasma mutagenesis and shuffling for high-level production of shikimic acid from sucrose in Escherichia coli[J].J Agr Food Chem,2020,68(42):11765-11773.
[8] SKELLEY A M,KIRAK O,SUH H,et al.Microfluidic control of cell pairing and fusion[J].Nat Methods,2009,6(2):147-152.
[9] LIU P,WEN J,CHEN Y,et al.Femtosecond laser-based mutagenesis strategy for micronomicin production enhancement of Micromonospora sagamiensis ATCC 21826[J].World J Microbiol Biotechnol,2013,29(6):1121-1127.
[10]ZHONG M C,GONG L,ZHOU J H,et al.Optical trapping of red blood cells in living animals with a water immersion objective[J].Opt Lett,2013,38(23):5134.
[11]GONG J,ZHAO X,XING Q,et al.Femtosecond laser-induced cell fusion[J].Appl Phys Lett,2008,92(9):093901.
[12] SHI D,WANG C,WANG K.Genome shuffling to improve thermotolerance,ethanol tolerance and ethanol productivity of Saccharomyces cerevisiae[J].J Ind Microbiol Biotechnol,2009,36(1):139-147.
[13]SHI J,ZHANG M,ZHANG L,et al.Xylose-fermenting Pichia stipitis by genome shuffling for improved ethanol production[J].Microb Biotechnol,2014,7(2):90-99.
[14] GAO X,ZHAO H,ZHANG G,et al.Genome shuffling of Clostridium acetobutylicum CICC 8012 for improved production of Acetone-Butanol-Ethanol(ABE)[J].Curr Microbiol,2012,65(2):128-132.
[15]代兴华,蔡爱华,张厚瑞,等.基因组改组技术及其在工业微生物改良中的应用[J].食品与发酵工业,2011,37(7):142-147.
[16]申屠旭萍,姚佳忆,俞晓平.基因组重排技术在微生物菌种改良中的应用[J].中国计量大学学报,2018,29(2):204-210.
[17]GONG J,ZHENG H,WU Z,et al.Genome shuffling: progress and applications for phenotype improvement[J].Biotechnol Adv,2009,27(6):996-1005.
[18]MAGOCHA T A,ZABED H,YANG M,et al.Improvement of industrially important microbial strains by genome shuffling:current status and future prospects[J].Bioresour Technol,2018,257:281-289.
[19] HIDA H,YAMADA T,YAMADA Y.Genome shuffling of Streptomyces sp.U121 for improved production of hydroxycitric acid[J].Appl Microbiol Biotechnol,2007,73(6):1387-1393.
[20]LIU W,JIANG R.Combinatorial and high-throughput screening approaches for strain engineering[J].Appl Microbiol Biotechnol,2015,99(5):2093-2104.
[21]LI S,CHEN X,DONG C,et al.Combining genome shuffling and interspecific hybridization among Streptomyces improved ε-poly-l-lysine production[J].Appl Biochem Biotechnol,2013,169(1):338-350.
[22] ZENG W,GUO L,XU S,et al.High-throughput screening technology in industrial biotechnology[J].Trends Biotechnol,2020,38(8):888-906.
[23] JOHN R P,GANGADHARAN D,MADHAVAN NAMPOOTHIRI K.Genome shuffling of Lactobacillus delbrueckii mutant and Bacillus amyloliquefaciens through protoplasmic fusion for L-lactic acid production from starchy wastes[J].Bioresour Technol,2008,99(17):8008-8015.
[24]罗情情,王明兹,陈必链,等.基因组改组技术及其在微生物中的应用[J].农产品加工(学刊),2014(3):62-65,68.
[25] FAN Y,GAO Y,ZHOU J,et al.Process optimization with alternative carbon sources and modulation of secondary metabolism for enhanced ansamitocin P-3 production in Actinosynnema pretiosum[J].J Biotechnol,2014,192:1-10.
[26] LI J,GUO S,HUA Q,et al.Improved AP-3 production through combined ARTP mutagenesis,fermentation optimization,and subsequent genome shuffling[J].Biotechnol Lett,2021,43(6):1143-1154.
[27]CHEN L,CHONG X,ZHANG Y Y,et al.Genome shuffling of Bacillus velezensis for enhanced surfactin production and variation analysis[J].Curr Microbiol,2020,77(1):71-78.
[28] YE L,YE R,HU F,et al.Combination of atmospheric and room temperature plasma (ARTP) mutagenesis,genome shuffling and dimethyl sulfoxide(DMSO)feeding to improve FK506 production in Streptomyces tsukubaensis[J].Biotechnol Lett,2021,43(9):1809-1820.
[29] GHOSH S,DAM B.Genome shuffling improves pigment and other bioactive compound production in Monascus purpureus[J].Appl Microbiol Biotechnol,2020,104(24):10451-10463.
[30] LI S,WANG L,WANG N.Enhanced poly-γ-l-diaminobutanoic acid production in Bacillus pumilus by combining genome shuffling with multiple antibiotic-resistance[J].J Ind Microbiol Biotechnol,2020,47(12):1141-1154.
[31]HU S,YOU Y,XIA F,et al.Genome shuffling improved acid-tolerance and succinic acid production of Actinobacillus succinogenes[J].Food Sci Biotechnol,2019,28(3):817-822.
[32]TAO H,ZHANG Y,DENG Z,et al.Strategies for enhancing the yield of the potent insecticide spinosad in Actinomycetes[J].Biotechnol J,2019,14(1):1700769.
[33]TONG Q Q,ZHOU Y H,CHEN X S,et al.Genome shuffling and ribosome engineering of Streptomyces virginiae for improved virginiamycin production[J].Bioproc Biosyst Eng,2018,41(5):729-738.
[34] SHI J,ZHU X,LU Y,et al.Improving iturin a production of Bacillus amyloliquefaciens by genome shuffling and its inhibition against Saccharomyces cerevisiae in orange juice[J].Front Microbiol,2018,9:2683.
[35]HUANG Q G,ZENG B D,LIANG L,et al.Genome shuffling and highthroughput screening of Brevibacterium flavum MDV1 for enhanced L-valine production[J].World J Microbiol Biotechnol,2018,34(8):121.
[36]ZHANG P,LEE Y,WEI X,et al.Enhanced production of tanshinone iia in endophytic fungi Emericella foeniculicola by genome shuffling[J].Pharm Biol,2018,56(1):357-362.
[37] LUNA-FLORES C H,PALFREYMAN R W,KRÖMER J O,et al.Improved production of propionic acid using genome shuffling[J].Biotechnol J,2017,12(2):1600120.
[38]WANG Y,ZHANG G,ZHAO X,et al.Genome shuffling improved the nucleosides production in Cordyceps kyushuensis[J].J Biotechnol,2017,260:42-47.
[39] SUN J,LI H,NI Y,et al.Genome shuffling of Colletotrichum lini for improving 3β,7α,15α-trihydroxy-5-androsten-17-one production from dehydroepiandrosterone[J].J Ind Microbiol Biotechnol,2017,44(6):937-947.
[40]INOKUMA K,IWAMOTO R,BAMBA T,et al.Improvement of xylose fermentation ability under heat and acid Co-stress in Saccharomyces cerevisiae using genome shuffling technique[J].Front Bioeng Biotechnol,2017,5:81.
[41]CHALOPAGORN P,CHAROENPANICH J,CHOOWONGKOMON K.Retraction note to:genome shuffling enhances lipase production of thermophilic Geobacillus sp.[J].Appl Biochem Biotechnol,2017,183(4):1539-1539.
[42]ZHU Z,ZHANG J,JI X,et al.Evolutionary engineering of industrial microorganisms-strategies and applications[J].Appl Microbiol Biotechnol,2018,102(11):4615-4627.
[43]CHEN L,XIN Q H,MA L M,et al.Applications and research advance of genome shuffling for industrial microbial strains improvement[J].World J Microbiol Biotechnol,2020,36(10):158.
[44] YU L,PEI X,LEI T,et al.Genome shuffling enhanced L-lactic acid production by improving glucose tolerance of Lactobacillus rhamnosus[J].J Biotechnol,2008,134(1-2):154-159.
[45]TIAN K,LI Y,WANG B,et al.The genome and transcriptome of Lactococcus lactis ssp. lactis f44 and g423: insights into adaptation to the acidic environment[J].J Dairy Sci,2019,102(2):1044-1058.
[46]CAO X,HOU L,LU M,et al.Genome shuffling of Zygosaccharomyces rouxii to accelerate and enhance the flavour formation of soy sauce[J].J Sci Food Agr,2010,90(2):281-285.
[47]SNOEK T,PICCA NICOLINO M,VAN DEN BREMT S,et al.Largescale robot-assisted genome shuffling yields industrial Saccharomyces cerevisiae yeasts with increased ethanol tolerance[J].Biotechnol Biofuels,2015,8(1):32.
[48] DE GÉRANDO H M,FAYOLLE-GUICHARD F,RUDANT L,et al.Improving isopropanol tolerance and production of Clostridium beijerinckii DSM 6423 by random mutagenesis and genome shuffling[J].Appl Microbiol Biotechnol,2016,100(12):5427-5436.
[49] CHENG C,ALMARIO M P,KAO K C.Genome shuffling to generate recombinant yeasts for tolerance to inhibitors present in lignocellulosic hydrolysates[J].Biotechnol Lett,2015,37(11):2193-2200.
[50]WANG W,WU B,QIN H,et al.Genome shuffling enhances stress tolerance of Zymomonas mobilis to two inhibitors[J].Biotechnol Biofuels,2019,12(1):288.
[51]JETTI K D,GNS R R,GARLAPATI D,et al.Improved ethanol productivity and ethanol tolerance through genome shuffling of Saccharomyces cerevisiae and Pichia stipitis[J].Int Microbiol,2019,22(2):247-254.
[52]LI S B,QIAN Y,LIANG Z W,et al.Enhanced butanol production from cassava with Clostridium acetobutylicum by genome shuffling[J].WorldJ Microbiol Biotechnol,2016,32(4):53.
[53]ZHENG P,LIU M,LIU X,et al.Genome shuffling improves thermotolerance and glutamic acid production of Corynebacteria glutamicum[J].World J Microbiol Biotechnol,2012,28(3):1035-1043.
[54]HOU L.Novel methods of genome shuffling in Saccharomyces cerevisiae[J].Biotechnol Lett,2009,31(5):671-677.
[55] WEI P,LI Z,HE P,et al.Genome shuffling in the ethanologenic yeast Candida krusei to improve acetic acid tolerance[J].Biotechnol Appl Biochem,2008,49(2):113-120.
[56]WANG Y,LI Y,PEI X,et al.Genome-shuffling improved acid tolerance and L-lactic acid volumetric productivity in Lactobacillus rhamnosus[J].J Biotechnol,2007,129(3):510-515.
[57] PATNAIK R,LOUIE S,GAVRILOVIC V,et al.Genome shuffling of Lactobacillus for improved acid tolerance[J].Nat Biotechnol,2002,20(7):707-712.
[58]赵晨,赵祥颖,张家祥,等.基于基因组重排技术选育D-核糖高产菌株[J].中国酿造,2019,38(2):159-163.
[59]LEE B U,CHO Y S,PARK S C,et al.Enhanced degradation of TNT by genome-shuffled Stenotrophomonas maltophilia OK-5[J].Curr Microbiol,2009,59(3):346-351.
[60]YI L,PENG Q,LIU D,et al.Enhanced degradation of perfluorooctanoic acid by a genome shuffling-modified Pseudomonas parafulva YAB-1[J].Environ Technol,2019,40(24):3153-3161.
[61]LEEBU,CHOIMS,KIMDM,etal.GenomeshufflingofStenotrophomonas maltophilia OK-5 for improving the degradation of explosive RDX(hexahydro-1,3,5-trinitro-1,3,5-triazine)[J].Curr Microbiol,2017,74(2):268-276.
[62] DAI M,COPLEY S D.Genome shuffling improves degradation of the anthropogenic pesticide pentachlorophenol by Sphingobium chlorophenolicum ATCC 39723[J].Appl Environ Microbiol,2004,70(4):2391-2397.
[63]HOPWOOD D A,CHATER K F.Fresh approaches to antibiotic production[J].Philos Trans R Soc Lond B Biol Sci,1980,290(1040):313-328.
[64]WANG M,LIU S,LI Y,et al.Protoplast mutation and genome shuffling induce the endophytic fungus Tubercularia sp.TF5 to produce new compounds[J].Curr Microbiol,2010,61(4):254-260.
[65]SUN J,LIU H,DANG L,et al.Genome shuffling of Lactobacillus plantarum 163 enhanced antibacterial activity and usefulness in preserving orange juice[J].Lett Appl Microbiol,2021,73(6):741-749.
[66]HAN G G,SONG A A,KIM E B,et al.Improved antimicrobial activity of Pediococcus acidilactici against Salmonella gallinarum by UV mutagenesis and genome shuffling[J].Appl Microbiol Biotechnol,2017,101(13):5353-5363.
[67]ZHAO Y,DUAN C,GAO L,et al.Genome shuffling of Lactobacillus plantarum C88 improves adhesion[J].Biosci Biotechnol Biochem,2017,81(1):184-193.
[68] ZHAO S,TIAN C Y,DU C M.Production of hybrids of Streptomyces bikiniensis strain HD-087 by genome shuffling and enhancement of its biocontrol activity against Fusarium oxysporum F.sp.cucumerinum[J].J Horticul Sci Biotechnol,2014,89(2):147-152.
[69]李义勇,张亚雄.基因重组技术在工业微生物菌种选育中应用的研究进展[J].中国酿造,2009,28(1):11-14.