嗜热链球菌(Streptococcus thermophilus)是发酵乳中常用的益生菌种之一,虽然属于链球菌属,但其是链球菌属内唯一对人体健康有益的乳酸菌[1]。嗜热链球菌在美国具有“公认安全性(generally recognized as safe,GRAS)”,并且具有非常悠久的使用历史[2]。在乳品发酵工业中是仅次于乳酸乳球菌(Lactococcus lactis)的一种非常重要的乳酸菌。含有嗜热链球菌的乳制品全球每年消费市场约400亿美元[2]。嗜热链球菌已经被列入国食药监注[2005]第202号发布的《可用于保健食品的益生菌名单》中,同时已经被国家卫生部列入《可用于食品中的菌种名单》(卫办监督发[2010]65号),说明该菌在具有保健性质的益生效果的同时具有食品方面的益生作用。研究表明,嗜热链球菌在发酵过程中可以分泌多种功能活性物质,如双乙酰、胞外多糖、细菌素和维生素等[3-6];可以通过有机酸代谢等方式降低体内的胆固醇[7-9]。此外,其可以与保加利亚乳杆菌(Lactobacillus bulgaricus)进行协同发酵,并且其分泌物能够刺激嗜酸乳杆菌(Lactobacillus acidophilus)的生长,因此在发酵乳发酵过程中其经常被用作引子与保加利亚乳杆菌和瑞士乳杆菌(Lactobacillus helveticus)混合发酵风味发酵乳[10-13]。
乳酸菌菌种的选择决定了发酵乳的风味和益生效果,准确的使用优质的乳酸菌是获得优质发酵乳的关键技术[14-15],因此,对乳酸菌菌种的鉴定具有重要的意义。乳酸菌菌株相似性较高,所以对乳酸菌的鉴定也是研究的难点。目前常用的鉴定方法是菌落形态观察、镜检及生理生化试验方法,该方法实验步骤繁琐,稳定性差,对检验员的技术和经验要求较高。因此,建立一种快速稳定可靠的鉴定方法在以后的食品检测和微生物分类的研究中是非常必要的。实时荧光定量聚合酶链式反应(real-time fluorescent quantitative polymerase chain reaction,RT-fqPCR)法是目前技术成熟、结果稳定可靠的微生物菌种鉴定的方法,目前,已有大量研究采用该方法对发酵乳杆菌(Lactobacillus fermentium)、副干酪乳杆菌(Lactobacillus paracei)、鼠李糖乳杆菌(Lactobacillus rhamnosus)、干酪乳杆菌(Lactobacillus casei)等[16-20]进行鉴定和定量研究。以16S rRNA基因为基础的生物信息学及菌种鉴定是目前研究最多的分子生物学菌种鉴定方法,董银苹等[21]使用16S rRNA基因对乳酸杆菌和嗜热链球菌进行鉴定并取得了可靠的结果;STACHELSKA M A等[22]根据16S rRNA基因设计引物对德氏乳杆菌(Lactobacillus delbrueckii)和嗜热链球菌进行定量分析,取得了与平板计数方法相似的结果。因此,建立一种RT-fqPCR方法用于鉴定嗜热链球菌是可行且非常必要的。
有文献[23]报道,16S rRNA基因属内亲缘关系较近的种间相似度通常会>99%,因此,在进行菌种水平的鉴定时会很难进行区分。本研究通过基因发育树分析确定目标基因,建立一种实时荧光定量PCR法对发酵乳中嗜热链球菌进行鉴定,并通过特异性实验、灵敏度实验和抗干扰实验验证该方法的稳定性和准确性,以期为发酵乳样品中嗜热链球菌的检测和菌种鉴定提供技术支撑。
1 材料与方法
1.1 材料与试剂
1.1.1 材料
60份发酵乳样品:市售,所有样品标签标示含有嗜热链球菌。
1.1.2 菌株
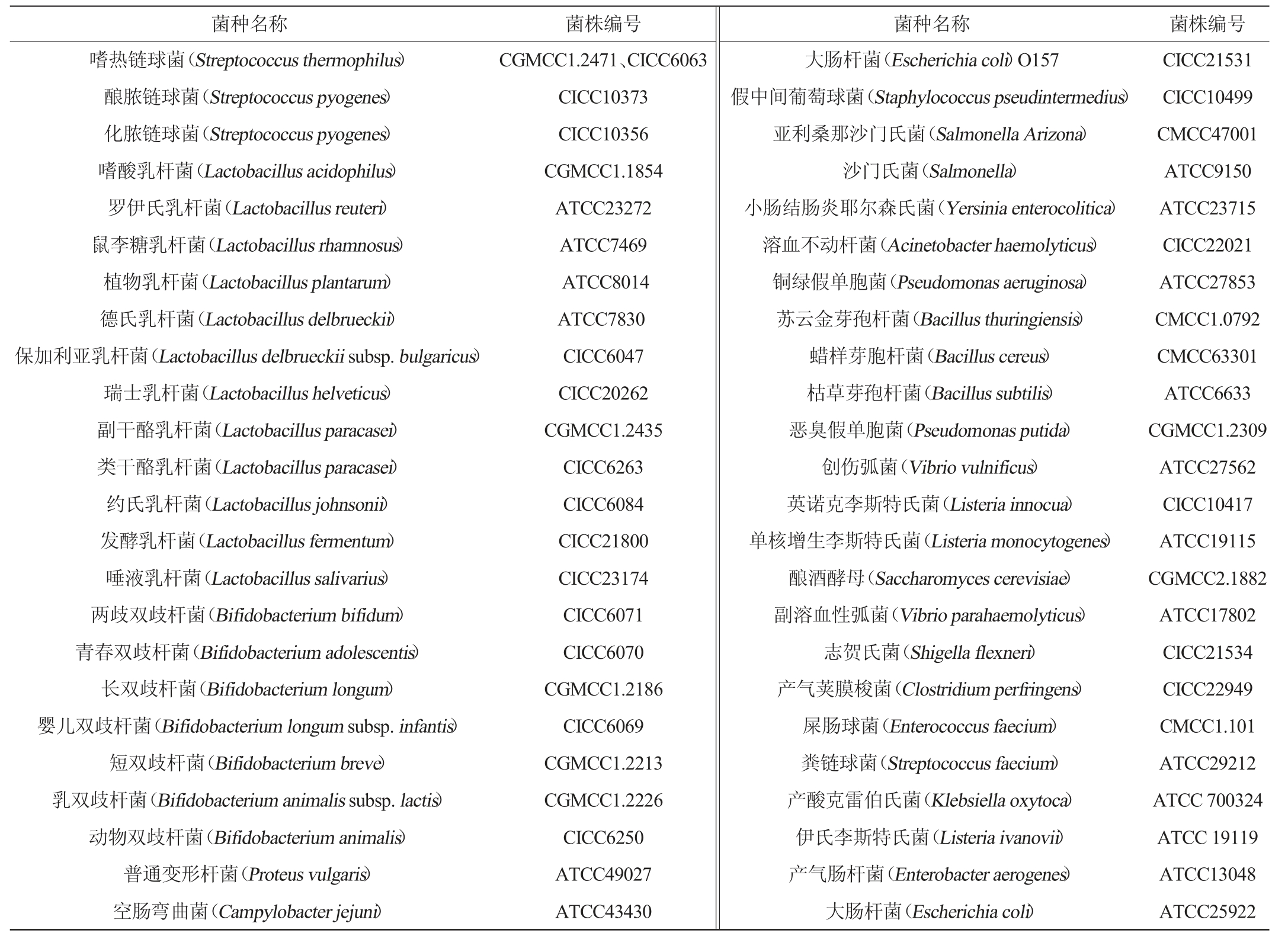
本实验所用菌株均购自中国工业微生物菌种保藏管理中心(China center of industrial culture collection,CICC),菌株信息见表1。
表1 本研究所用菌株信息
Table 1 Information of strains used in the study
1.1.3 试剂
MC琼脂培养基:北京路桥技术有限公司;细菌基因组脱氧核糖核酸(deoxyribonucleic acid,DNA)提取试剂盒、蛋白酶K(20 mg/mL)、溶菌酶(50 mg/mL):天根生化科技有限公司;Taq man master mix:美国ABI公司。
1.2 仪器与设备
ABIQuantStudio 7实时荧光定量PCR仪:美国ABI公司;VITEK Compac微生物分析系统:法国生物梅里埃公司。
Compac微生物分析系统:法国生物梅里埃公司。
1.3 方法
1.3.1 目的基因的确定
根据文献[24-27]确定选取16S rRNA、16S-23S rRNA、hsp60和recA等基因作为候选目标基因,在美国国立生物技术信息中心(national center for biotechnology information,NCBI)网站下载候选目标基因序列,采用DNAMAN软件进行序列比对。选取在链球菌属间具有很好的种间特异性的基因作为目的基因,采用MEGAX生物学软件构建系统发育树。
1.3.2 引物和探针的设计
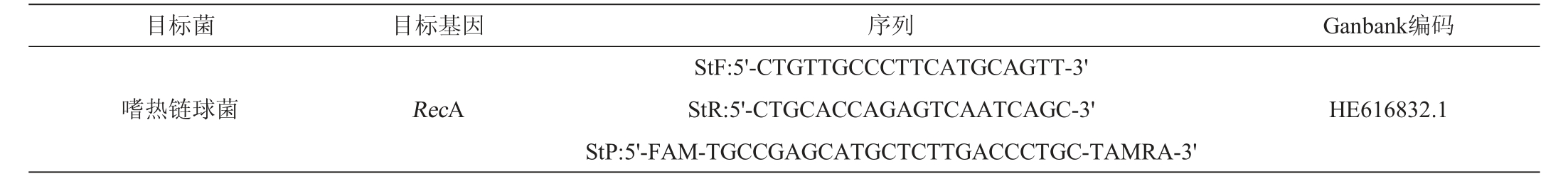
使用Primer Express V3.0设计目的基因的引物和探针,设计得到的引物探针在NCBI网站进行primerBlast,选出特异性较好的引物、探针委托上海生物工程有限公司合成。引物及探针序列见表2。
表2 实时荧光PCR引物和探针序列
Table 2 Sequences of primers and probes of real-time fluorescence PCR
1.3.3 菌株活化
将冷冻干燥的2株嗜热链球菌标准菌株首先转接于MC琼脂培养基中,37 ℃需氧培养(48±2)h,转接两代回复活力,用于后续实验。
1.3.4 DNA提取
按照细菌基因组DNA提取试剂盒说明书提取表1中细菌的基因组DNA。
1.3.5 实时荧光定量PCR
实时荧光定量PCR扩增体系:2×master mix 12.5 μL、引物对StF和StR(10 μmol/L)各1 μL、探针StP(10 μmol/L)0.5 μL、模板DNA 1 μL、双蒸水(ddH2O)9 μL。
实时荧光定量PCR扩增参数:50℃、2min,95℃、10 min,95 ℃、5 s,60 ℃、40 s;同时收集羧基荧光素荧光,进行40个循环,4 ℃保存反应产物。
实时荧光定量PCR扩增曲线指数期明显,Ct值<35可直接判定为阳性,Ct值为35~40判定为可疑,需要进行重复实验,Ct值>40判定为阴性。
1.3.6 实时荧光定量PCR扩增特异性实验
以2株嗜热链球菌及表1中其他细菌DNA为模板进行实时荧光定量PCR扩增。
1.3.7 实时荧光定量PCR扩增灵敏性实验
灵敏度实验包括绝对灵敏度和相对灵敏度实验[28]。绝对灵敏度实验:将提取得到的嗜热链球菌CICC6063 DNA溶液稀释至10 ng/μL、1 ng/μL、0.1 ng/μL、0.01 ng/μL、0.001 ng/μL、0.000 5 ng/μL、0.000 1 ng/μL。所有稀释液在同一条件下进行实时荧光定量PCR扩增。相对灵敏度实验:将嗜热链球菌CICC6063添加到巴氏灭菌的发酵乳中,添加菌落数分别为107 CFU/mL、106 CFU/mL、105 CFU/mL、104 CFU/mL、103 CFU/mL、102 CFU/mL、10 CFU/mL,使用细菌基因组提取试剂盒提取DNA后进行实时荧光定量PCR扩增。灵敏度实验每个反应设置5个平行,重复实验4次,记录阳性扩增次数。
1.3.8 实时荧光定量PCR抗干扰实验
对本次建立方法的抗干扰实验设计了基因组水平的抗干扰实验和培养物水平的抗干扰实验。
基因组水平的抗干扰实验:将嗜热链球菌CICC6063的DNA分别稀释至10 ng/μL、1 ng/μL、0.1 ng/μL、0.01 ng/μL,分别按照1∶1的体积比添加10 ng/μL的大肠杆菌ATCC25922 DNA或TE缓冲液,以混合后的DNA作为模板进行实时荧光定量PCR扩增,阴性对照以10 ng/μL大肠杆菌ATCC25922基因组作为模板,阳性对照以10 ng/μL嗜热链球菌CICC6063基因组作为模板,空白对照以无菌水作为模板。记录干扰DNA对实验结果的影响。
培养物水平的抗干扰实验:将嗜热链球菌CICC6063培养液的菌体浓度稀释到107 CFU/mL、106 CFU/mL、105 CFU/mL、104 CFU/mL,分别按照1∶1的体积比添加107 CFU/mL的大肠杆菌ATCC25922或无菌水,充分混匀后,按照细菌DNA提取试剂盒的说明书提取DNA,然后进行实时荧光定量PCR扩增,阴性对照以107 CFU/mL大肠杆菌ATCC25922作为模板,阳性对照以107 CFU/mL嗜热链球菌CICC6063作为模板,空白对照以无菌水作为模板记录非特异性菌对实验结果的影响。
1.3.9 市售样品的检测
将购买得到的发酵乳样品按照国标GB 4789.35—2016《食品安全国家标准食品微生物学检验乳酸菌检验》的方法进行培养,从每个样品的MC平板上挑取10个菌落,使用细菌DNA提取试剂盒提取DNA,将该DNA作为模板进行实时荧光定量PCR扩增,记录阳性扩增样品数。
2 结果与分析
2.1 系统发育分析结果
通过分析发现recA基因在链球菌属间具有很好种间特异性,基于recA基因构建嗜热链球菌、前庭链球菌、唾液链球菌、乳杆菌属等菌株的系统发育树,结果见图1。由图1可知,嗜热链球菌与前庭链球菌、唾液链球菌和乳杆菌等菌株具有比较大的差异,其种间差异率>10%。说明recA基因能够很好的用于嗜热链球菌和乳杆菌属等菌株的菌种鉴定和区分,较之16S rRNA具有更高的分辨率[23,29]。
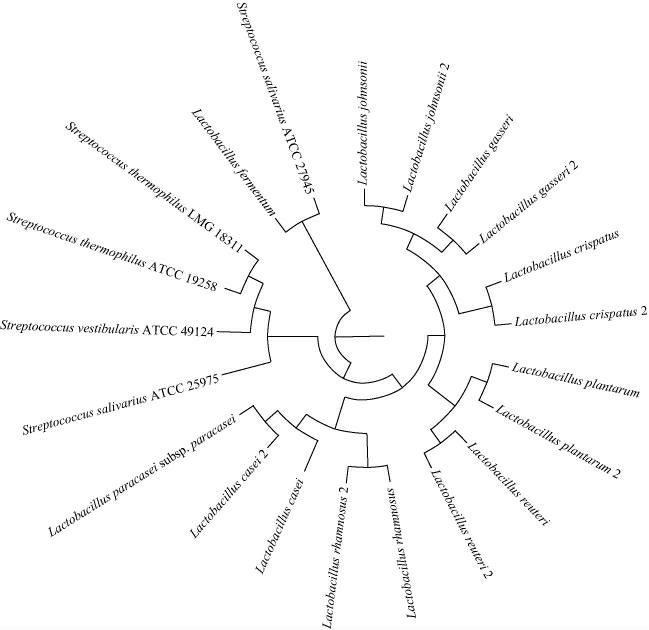
图1 基于recA基因序列21株菌株的系统发育树
Fig.1 Phylogenetic tree of 21 strains based on recA gene sequence
2.2 实时荧光定量PCR扩增法鉴定嗜热链球菌的特异性验证结果
实时荧光定量PCR扩增法鉴定嗜热链球菌的特异性验证结果见图2。
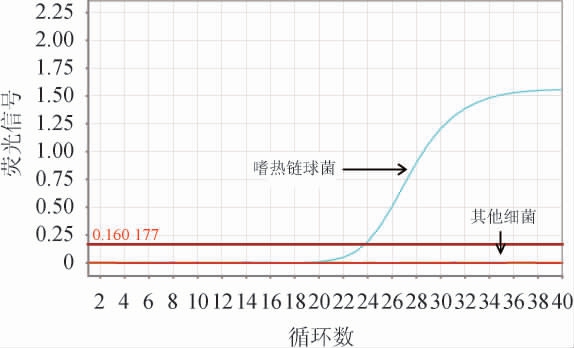
图2 实时荧光定量PCR扩增法鉴定嗜热链球菌的特异性实验结果
Fig.2 Specificity test results for Streptococcus thermophilus identification by real-time fluorescence quantitative PCR amplification method
由图2可知,以嗜热链球菌DNA为模板的实时荧光定量PCR具有明显的扩增曲线,以其亲缘关系较近的其他乳酸菌和食品中常见的细菌DNA为模板的实时荧光定量PCR均无荧光扩增信号。由此可以判断,本研究建立的实时荧光定量PCR方法能够特异性的扩增嗜热链球菌的recA基因序列。
2.3 实时荧光定量PCR扩增法鉴定嗜热链球菌的灵敏性验证结果
2.3.1 绝对灵敏度验证
实时荧光定量PCR扩增法鉴定嗜热链球菌的绝对灵敏度实验结果见表3。
表3 实时荧光定量PCR扩增法鉴定嗜热链球菌的绝对灵敏度实验结果
Table 3 Absolute sensitivity test results for Streptococcus thermophilus identification by real-time fluorescence quantitative PCR amplification method
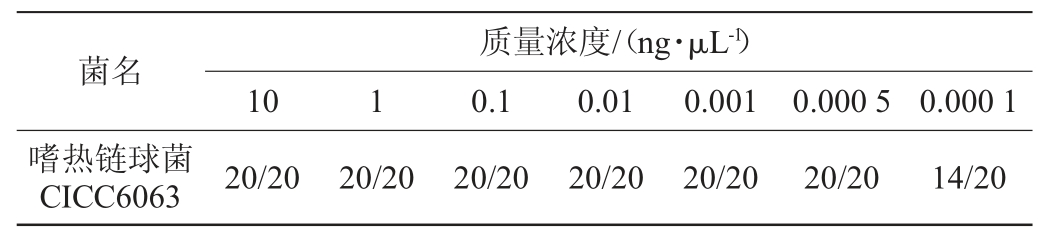
注:表中数据表示“检出次数/检测次数”。下同。
由表3可知,嗜热链球菌CICC6063可稳定检出的质量浓度为0.000 5 ng/μL,表示检测下限可以达到1 pg/μL。结果表明,本研究建立的实时荧光定量PCR方法的绝对灵敏度为1 pg/μL。
2.3.2 相对灵敏度验证
实时荧光定量PCR扩增法鉴定嗜热链球菌的相对灵敏度实验结果见表4。
表4 实时荧光定量PCR扩增法鉴定嗜热链球菌的相对灵敏度实验结果
Table 4 Relative sensitivity test results for Streptococcus thermophilus identification by real-time fluorescence quantitative PCR amplification method

由表4可知,可以稳定检出的最低菌落浓度为103CFU/mL。结果表明,本次建立的实时荧光定量PCR方法的相对灵敏度为103 CFU/mL。
2.4 实时荧光定量PCR扩增法鉴定嗜热链球菌的抗干扰能力验证结果
2.4.1 基因组水平的抗干扰实验结果
基因组水平的抗干扰实验结果见表5。由表5可知,在干扰基因组浓度是目标基因组浓度1 000倍的情况下,Ct值无显著影响(P>0.05),因此可以看出,本研究建立的方法具有很好的抗干扰能力。
表5 基因组水平实时荧光定量PCR扩增法鉴定嗜热链球菌的抗干扰实验结果
Table 5 Anti-disturbance test results for Streptococcus thermophilus identification by real-time fluorescence quantitative PCR amplification method at the level of the genome
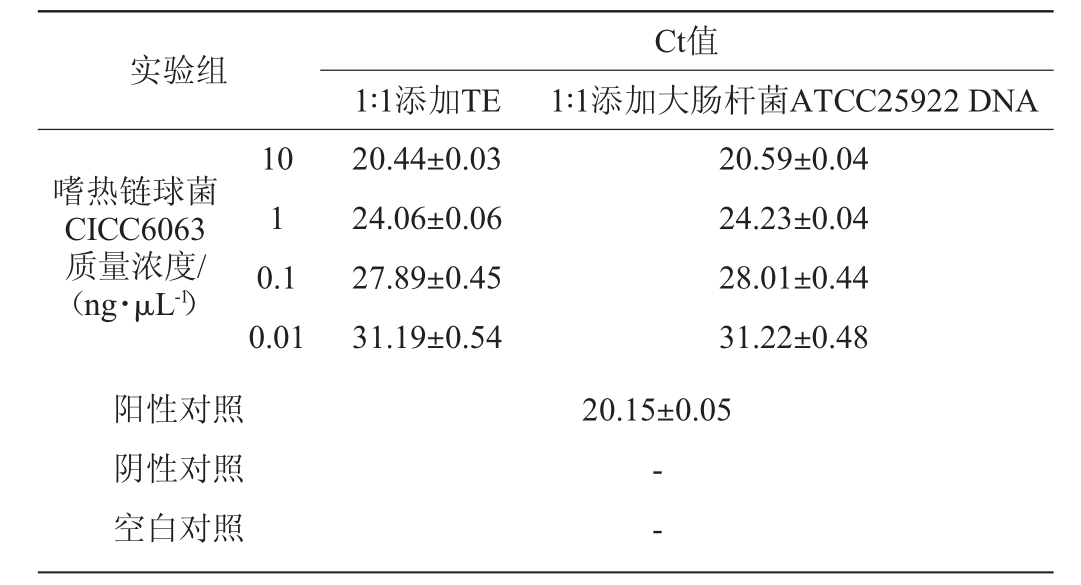
注:“-”表示Ct值>40。下同。
2.4.2 培养物水平的抗干扰实验结果
培养物水平的抗干扰实验结果见表6。由表6可知,在干扰菌浓度达到目标菌浓度1 000倍的情况下,Ct值无显著影响,均不影响嗜热链球菌的检出,由此可以看出,本研究建立的实时荧光定量PCR方法在培养物水平上具有很好的抗干扰能力。
表6 培养物水平实时荧光定量PCR扩增法鉴定嗜热链球菌的抗干扰实验结果
Table 6 Anti-disturbance test results for Streptococcus thermophilus identification by real-time fluorescence quantitative PCR amplification method at the culture level
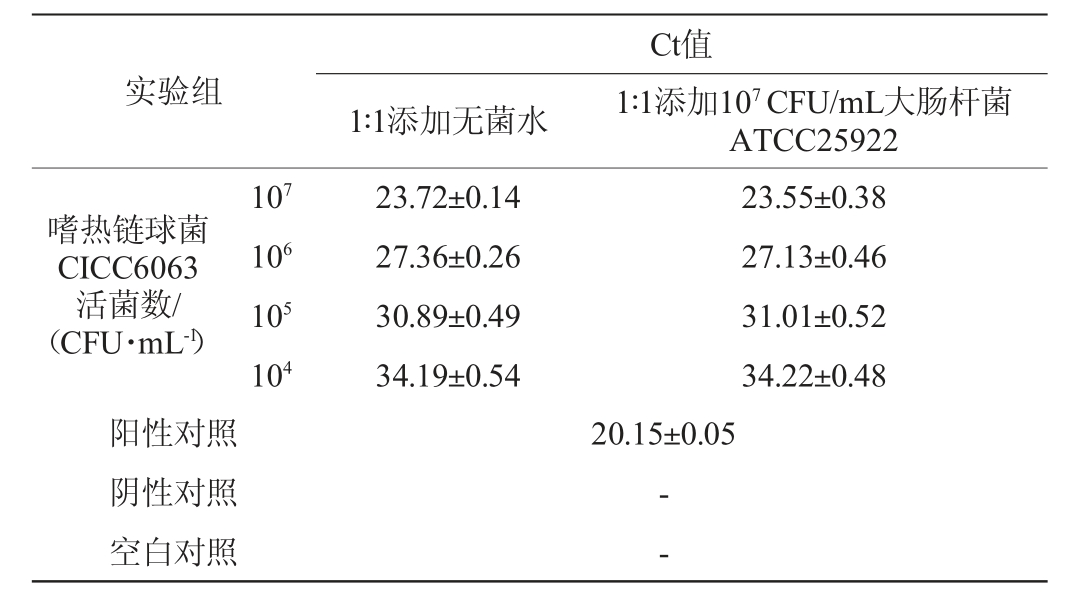
2.5 市售样品中嗜热链球菌的检测结果
使用本研究建立的实时荧光定量PCR扩增方法对市售的60份表示含有嗜热链球菌的发酵乳样品进行检测,检测结果显示均检出嗜热链球菌。
3 结论
本研究基于recA基因序列对嗜热链球菌、前庭链球菌、唾液链球菌、乳杆菌属等菌株构建系统发育树发现,种间差异率>10%,说明该基因可以用于嗜热链球菌的特异性鉴定。使用该基因设计引物和探针,建立了一种可用于发酵乳中嗜热链球菌检测的实时荧光定量PCR扩增方法,通过特异性、灵敏性及抗干扰实验验证该方法能够特异性的对发酵乳中的嗜热链球菌进行检测,并且具有很好的重复性和稳定性;绝对灵敏度可达到1 pg/μL,相对灵敏度可以达到103CFU/mL;在菌种水平和DNA水平都具有非常好的抗干扰能力。对市售的发酵乳样品的检测结果表明,60份标示添加嗜热链球菌的发酵乳样品均能检测到嗜热链球菌成分,说明该方法具有很好的实用性。本研究建立的方法可以为以后发酵乳样品中嗜热链球菌的检测提供技术支撑,recA基因具有很好的菌种特异性,为以后乳杆菌或双歧杆菌的鉴定提供了一定的参考数据。
[1]YONG J G,GOIN C,O'FLAHERTY S,et al.Specialized adaptation of a lactic acid bacterium to the milk environment:the comparative genomics of Streptococcus thermophilus LMD-9[J].Microbial Cell Factories,2011,10(Suppl 1):S22.
[2]DELOME C.Safety assessment of dairy microorganisms: Streptococcus thermophilus[J].Int J Food Microbiol,2008,126(3):274-277.
[3]VANIN GELGEM F,ZAMFIR M,MOZZI F,et al.Biodiversity of exopolysaccharides produced by Streptococcus thermophilus strains is reflected in their production and their molecular and functional characteristics[J].Appl Environ Microbiol,2004,70(2):900-912.
[4]PASCAL H,H FRÉDÉRIC,LAETITIA F,et al.New insights in the molecular biology and physiology of Streptococcus thermophilus revealed by comparative genomics[J].Fems Microbiol Rev,2005(3):435-463.
[5]ZOTTA T,RICCIARDI A,CIOCIA F,et al.Diversity of stress responses in dairy thermophilic streptococci[J].Int J Food Microbiol,2008,124(1):34-42.
[6]BOLOTIN A,QUINQUIS B,RENAULT P,et al.Complete sequence and comparative genome analysis of the dairy bacterium Streptococcus thermophilus[J].Nat Biotechnol,2004,22(12):1554.
[7]官雪芳,郑怡,徐庆贤,等.一株酸奶发酵嗜热链球菌的筛选及特性研究[J].西北农业学报,2018,27(1):124-130.
[8]HA C H,SWEARINGIN B,JEON Y K,et al.Effects of combined exercise on HOMA-IR,HOMA β-cell and atherogenic index in Korean obese female[J].Sport Sci Health,2015,11(1):49-55.
[9]ROSSI F,MARZOTTO M,CREMONESE S,et al.Diversity of Streptococcus thermophilus in bacteriocin production;inhibitory spectrum and occurrence of thermophilin genes[J].Food Microbiol,2013,35(1):27-33.
[10]ZIELIN′ SKA D,RZEPKOWSKA A,RADAWSKA A,et al. In vitro screening of selected probiotic properties of Lactobacillus strains isolated from traditional fermented cabbage and cucumber[J]. Curr Microbiol,2015,70:183-194.
[11]许本发.酸奶和乳酸菌饮料加工[M].北京:中国轻工业出版社,1994:134-135.
[12]赵瑞香,孙俊良,于涛,等.嗜酸乳杆菌与嗜热链球菌共发酵互生机理的研究[J].生物技术,2005,15(3):28-31.
[13]GERRIT S,SMIT B A,ENGELS W J M.Flavour formation by lactic acid bacteria and biochemical flavour profiling of cheese products[J].Fems Microbiol Rev,2005,29(3):591-610.
[14]贾庆超,梁艳美.模糊数学感官评价结合响应面法优化黑枸杞-刺梨风味发酵乳发酵工艺[J].中国酿造,2021,40(12):125-132.
[15]孙昕萌,袁惠萍,赵钜阳.发酵乳风味及其分析技术研究进展[J].食品安全质量检测学报,2021,12(15):6111-6117.
[16]MONNET C,CORREIA K,SARTHOU A S,et al.Quantitative detection of Corynebacterium casei in cheese by real-time PCR[J].Appl Environ Microbiol,2006,72(11):6972-6979.
[17]POSTOLLEC F,FALENTIN H,PAVAN S,et al.Recent advances in quantitative PCR (qPCR) applications in food microbiology[J]. Food Microbiol,2011,28(5):848-861.
[18]SINGH S,GOSWAMI P,SINGH R,et al.Application of molecular identification tools for Lactobacillus,with a focus on discrimination between closely related species:A review[J].LWT-Food Sci Technol,2009,42(2):448-457.
[19]ZAGOM,BONVINIB,CARMINATI D,et al.Detection and quantification ofEnterococcusgilvusin cheese byreal-time PCR[J].Syst Appl Microbiol,2009,32(7):514-521.
[20]FURET J P,QUÉNÉE P,TAILLIEZ P.Molecular quantification of lactic acid bacteria in fermented milk products using real-time quantitative PCR[J].Int J Food Microbiol,2004,97(2):197-207.
[21]董银苹,崔生辉,李凤琴,等.乳酸杆菌及嗜热链球菌的种水平鉴定——16SrRNA基因PCR扩增及序列分析[J].卫生研究,2010,39(4):454-458.
[22]STACHELSKA M A.Identification of Lactobacillus delbrueckii and Streptococcus thermophilus strains present in artisanal raw cow milkcheese using real-time PCR and classic plate count methods[J]. Pol J Microbiol,2017,66(4):491-499.
[23]MOHKAM M,NEZAFAT N,BERENJIAN A,et al.Identification of Bacillus probiotics isolated from soil rhizosphere using 16S rRNA,recA,rpoB gene sequencing and RAPD-PCR[J].Prob Antimicrob Proteins,2016,8(1):8-18.
[24]YOU I,KIM E B.Genome-based species-specific primers for rapid identification of six species of Lactobacillus acidophilus group using multiplex PCR[J].PLoS ONE,2020,15(3):e0230550.
[25]LU W,KONG W,YANG P,et al.A one-step PCR-based method for specific identification of 10 common lactic acid bacteria and Bifidobacterium in fermented milk[J].Int Dairy J,2015,41:7-12.
[26]DIOS J D,CL MARTÍNEZ,TATO M,et al.94 Comparison between MALDI-TOF and recA gene sequencing for the identification of Burkholderiacepacia complex species isolated in a cystic fibrosis unit[J].J Cyst Fibros,2016,15:S75.
[27]JENA R,CHOUDHURY P K,PUNIYA A K,et al.Isolation and species delineation of genus Bifidobacterium using PCR-RFLP of partial hsp60 gene fragment[J].LWT-Food Sci Technol,2017,80:286-293.
[28]肖其胜,杨捷琳,杨惠琴,等.实时荧光PCR法鉴定食品中双歧杆菌[J].食品科学,2016,37(20):6.
[29]多拉娜,宋宇琴,李伟程,等.干酪乳杆菌的近缘种及亚种部分看家基因的系统发育分析[J].微生物学通报,2018,45(12):11.