凤头姜是湖北省恩施土家族苗族自治州来凤县的一种特色农作物,来凤凤头姜亦是我国国家地理标志产品之一,其因外形形似古代神话中凤凰的头而得名[1]。凤头姜具有品质优良、风味独特、富含硒元素和营养丰富等优点,是姜中独具特色的佳品[2]。凤头姜的加工制品包括脱水姜片、红生姜、姜油、姜汁和泡姜等[3],其中泡姜是将凤头姜去皮切碎后与红辣椒、大蒜、花椒、白酒和盐等佐料混合后入坛密封发酵而成,具有质地脆嫩和口感酸鲜的特点[3]。
泡凤头姜发酵过程中微生物对产品的营养价值和安全性有着不可忽视的影响,因而对泡姜中微生物群系进行全面解析具有极为必要的意义。作为不依赖于纯培养技术的技术方法,克隆文库、磷脂脂肪酸(phospholipid fatty acid,PLFA)和变性梯度凝胶电泳(denatured gradient gel electrophoresis,DGGE)技术在传统发酵食品微生物群落结构研究中有着广泛的应用[4],但具有通量低和无法实现大样本间群落结构平行分析的缺点[5]。近年来兴起的以MiSeq高通量测序为代表的第二代测序技术[6],具有测序结果可信度高,且能够全面和客观分析样本中微生物群落结构的特点[7],在发酵食品微生物多样性研究[8]和植物内生菌多样性解析[9]方面有着广泛地应用。因而,将MiSeq高通量测序技术运用于来凤泡凤头姜盐水细菌多样性的研究中具有一定的可行性。
本研究采用MiSeq高通量测序技术对湖北恩施土家族苗族自治州来凤地区泡凤头姜盐水中的细菌多样性进行解析,在明确其细菌构成的基础上,进一步对核心菌群进行甄别,并采用PICRUSt(phylogenetic investigation of communities by reconstruction of unobserved states)软件对细菌类群的基因功能进行预测,以期为掌握来凤地区泡凤头姜盐水中细菌类群及多样性提供一定的参考依据。
1 材料与方法
1.1 材料与试剂
1.1.1 材料
6份泡凤头姜盐水样品(编号为PJ1、PJ2、PJ3、PJ4、PJ5和PJ6):采集自湖北省恩施州来凤县城南农贸大市场(E109°18′~109°27′,N29°25′~29°36′),均为当地农户家中自制。其制作方法为在干燥清洁的土(陶)坛中放入洗净的凤头姜,加入当地的辣椒、料酒、食盐和花椒等辅料,加入适量陈泡菜水后混匀,盖上坛盖并用清水封住坛口以隔绝空气,将土(陶)坛放在18~20 ℃的条件下自然发酵30 d即可。采集的样品发酵时间均在30~40 d之间。
1.1.2 试剂
QIAGENDNeasymericonFoodKit:德国QIAGEN公司;10×聚合酶链式反应(polymerase chain reaction,PCR)缓冲液、脱氧核糖核苷三磷酸、脱氧核糖核酸(deoxyribonucleic acid,DNA)聚合酶:北京全式金生物技术有限公司;正反向引物338F/806R:武汉天一辉远生物科技有限公司合成。
1.2 仪器与设备
Veriti FAST梯度PCR仪:美国ABI公司;164-5050基础电源电泳仪:美国BIO-RAD公司;UVPCDS8000凝胶成像分析系统:美国Protein Simple公司;MiSeq PE300高通量测序平台/PE300高通量测序平台:美国Illumina公司;R920机架式服务器:美国DELL公司;ND-2000C微量紫外分光光度计:美国Nano Drop公司。
1.3 实验方法
1.3.1 泡凤头姜盐水样品DNA的提取
根据DNA提取试剂盒说明书提取泡凤头姜盐水样品的宏基因组DNA,并使用1.0%的琼脂糖凝胶电泳和Nano Drop对DNA质量及浓度进行检测,以期获得高质量和高浓度的DNA片段。
1.3.2 细菌16S rRNA V3~V4区PCR扩增及测序
以提取的基因组DNA为模板,采用正向引物338F(5'-ACTCCTACGGGAGGCAGCAG-3')和反向引物806R(5'-GGACTACHVGGGTWTCTAAT-3')对泡凤头姜盐水中微生物的16S rRNA V3~V4区基因序列进行PCR扩增[10-11],将PCR扩增合格的产物清洁后委托上海美吉生物医药科技有限公司,使用MiSeq高通量测序平台进行测序。
1.3.3 生物信息学分析
将测序结果进行质控[12],使用QIIME(v1.70)平台完成生物信息学处理[13]。具体方法如下:构建操作分类单元(operational taxonomic units,OTU)[14]、去除嵌合体[15]、使用核糖体数据库项目(ribosomal database project,RDP)(ribosomal database project,RDP)11.5数据库进行同源性比对确定微生物学分类地位[16],同时使用PICRUSt软件对泡凤头姜盐水中细菌微生物的基因功能进行预测[17],并使用热图对结果进行可视化。
1.3.4 数据处理
采用Origin 8.5(OriginLab Corp,MA,USA)绘制序列分布长度、细菌门和属相对含量以及OTU出现次数柱状图;采用皮尔森相关性分析对核心优势OTU的相关性进行计算,根据其相关系数的大小使用R语言软件(v4.1.0)绘制热图;使用R语言软件(v4.1.0)绘制瀑布图。
2 结果与分析
2.1 测序序列长度分布
纳入本研究的6份泡凤头姜盐水样品共产生123 508条序列,经质量控制后得到123 471条有效序列,去除引物序列和barcode标签序列后所有样品的有效序列的碱基长度的分布情况见图1。
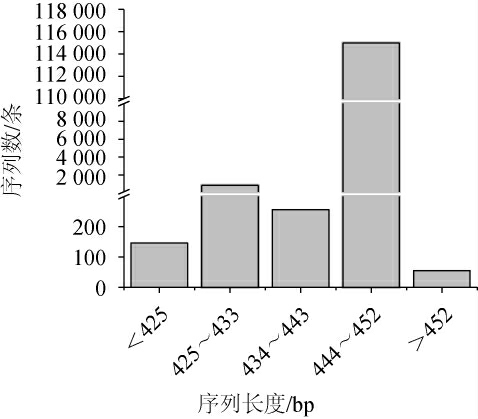
图1 泡凤头姜盐水样品中细菌菌群有效序列的碱基长度分布情况
Fig.1 Distribution of base length of effective sequences of bacterial community in fermented Fengtou ginger brine samples
由图1可知,泡凤头姜盐水样品中细菌菌群有效序列的碱基长度主要集中分布在444~452 bp,序列数占总序列数的93.10%,有6.53%的基因序列碱基长度集中在425~433 bp,另外仅有0.37%的基因序列碱基长度集中在0~433 bp。有研究表明,细菌16S rRNA的V3~V4区基因序列的碱基长度为338~806 bp[18],结果表明,本研究测序所得到的序列基本可以覆盖16S rRNA的V3~V4区,可用于后续研究。
2.2 泡凤头姜盐水的细菌菌群结构分析
2.2.1 基于门水平泡凤头姜盐水的细菌菌群结构
本研究将有效序列按照97%的相似度划分共得到10 513个OTU,去除36个嵌合体OTU后,余下10 477个OTU,挑选代表性序列在数据库中进行比对后,将其鉴定到19个门、46个纲、96个目、163个科和393个属,其中分别有0.05%和7.45%的序列无法鉴定到细菌门和属水平。本研究将平均相对含量大于1.0%的细菌门和属分别定义为优势细菌门和优势细菌属,泡凤头姜盐水样品中优势细菌门的相对含量及分布情况见图2。
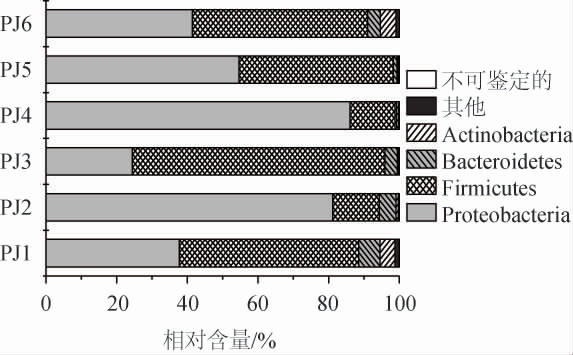
图2 泡凤头姜盐水样品中优势细菌门相对含量的分析结果
Fig.2 Analysis results of relative content of dominant bacterial phyla in fermented Fengtou ginger brine samples
由图2可知,6个泡凤头姜盐水中的优势细菌门分别为变形菌门(Proteobacteria)、厚壁菌门(Firmicutes)、拟杆菌门(Bacteroidetes)和放线菌门(Actinobacteria),其平均相对含量分别为54.25%、40.26%、3.24%和1.76%。值得注意的是,Proteobacteria在样品PJ4中的相对含量高达86.11%,而其在样品PJ3中的相对含量仅为24.50%,Firmicutes在样品PJ3和PJ4中亦存在与Proteobacteria同样的差异。由此可见,尽管优势细菌门在所有样品中均存在,但不同细菌门在各个样品中的含量差异较大。
2.2.2 基于属水平泡凤头姜盐水的细菌菌群结构
泡凤头姜盐水样品中优势细菌属的相对含量及分布情况见图3。
由图3可知,6个泡凤头姜盐水样品中的优势细菌属主要为隶属于Firmicutes的乳杆菌属(Lactobacillus)、芽孢杆菌属(Bacillus)和乳球菌属(Lactococcus),其平均相对含量分别为27.73%、7.67%和3.31%;隶属于Proteobacteria的假单胞菌属(Pseudomonas)、不动杆菌属(Acinetobacter)、罗尔斯通菌属(Ralstonia)、气单胞菌属(Aeromonas)、拉恩氏菌属(Rahnella)、弧菌属(Vibrio)、肠杆菌属(Enterobacter)、克吕沃氏菌属(Kluyvera)、果胶杆菌属(Pectobacterium)和欧文氏菌属(Erwinia),其平均相对含量分别为5.99%、5.67%、5.34%、3.91%、2.60%、2.03%、1.88%、1.78%、1.32%和1.02%;隶属于Bacteroidetes的黄杆菌属(Flavobacterium),平均相对含量分别为1.06%。由此可见,Lactobacillus是泡凤头姜盐水中主要的细菌类群。值得注意的是,其中属于乳酸菌类群的有Lactobacillus和Lactococcus两个属,累计平均相对含量为31.04%。XIONG T等[19]对泡菜发酵过程中乳酸菌菌群的动态变化进行了跟踪,结果发现明串珠菌属(Leuconostoc)和Enterococcus出现在发酵的最早期,其他乳酸菌种类在发酵前期开始陆续出现并增殖,到了发酵中期部分乳酸菌开始消亡,而Lactobacillus一直存在至发酵终期。与此同时,XIONG T等[20]还研究了盐浓度对泡菜发酵的影响,结果发现高浓度盐会延缓泡菜的发酵进程,同时减少乳酸菌含量。因而,本研究泡凤头姜盐水中乳酸菌种类少且含量较低这一现象可能是由于纳入的部分样品已处于发酵后期阶段,亦或是当地人制作的泡凤头姜盐浓度较高。后期可结合泡凤头姜的感官和理化指标展开研究,以期探究泡凤头姜盐水与其他泡菜盐水中菌群存在差异的具体原因。
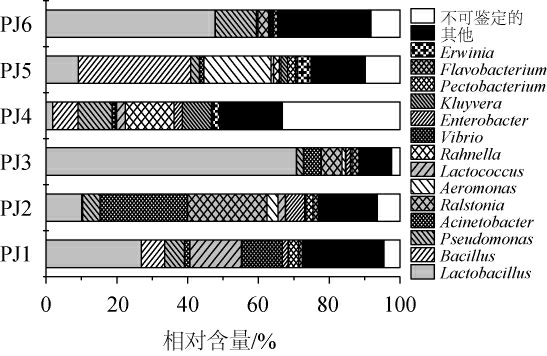
图3 泡凤头姜盐水样品中优势细菌属相对含量的分析结果
Fig.3 Analysis results of relative content of dominant bacterial genera in fermented Fengtou ginger brine samples
除此之外,由图3还可发现,泡凤头姜盐水样品中存在着一定的条件致病菌,如Pseudomonas[21]、Acinetobacter[22]和Enterobacter[23]等,目前已有大量研究证明这些菌属中的部分菌种具有一定的感染性和导致炎症的可能性。究其原因,可能是与制作环境开放以及原料和器具上有害微生物进入了发酵坛中有关,从而导致泡凤头姜盐水中微生物类群被污染。因此,解析泡凤头姜盐水的细菌群落结构对泡凤头姜的产业化生产和降低其中存在的安全隐患具有重要作用。
2.2.3 基于OTU水平泡凤头姜盐水的细菌菌群结构分析
在对泡凤头姜盐水细菌类群进行揭示的基础上,本研究进一步对每个样品的OTU数量和序列相对含量进行了统计分析,进而对6个泡凤头姜盐水样品特有和共有细菌类群的含量进行解析,结果见图4。
由图4可知,去除嵌合体后余下的10 477个OTU中,有7 841个OTU仅出现在一个样品中,占OTU总数的74.85%,但其包含的序列数仅占总序列数的7.35%;而在所有样品中均出现的OTU有105个,仅占OTU总数的1.00%,但其包含的序列数却占总序列数的41.01%。本研究将在所有样品中均出现的OTU定义为核心OTU。核心OTU中分别有22和12个OTU隶属于Pseudomonas和Lactobacillus,包含的序列数分别占总序列数的2.98%和21.26%。由此可见,尽管每个样品各自都拥有着较多的独特OTU数量,但他们之间仍存在着大量以Lactobacillus为主的核心菌群。
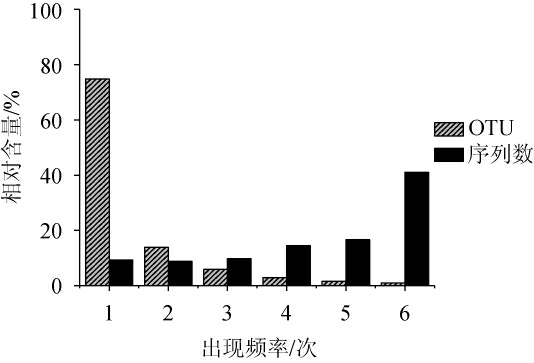
图4 样品中OTU出现次数与其包含序列的相对含量
Fig.4 Occurrences frequency and relative content of the contained sequences of OTU
本研究将在每个样品中均存在且平均相对含量大于1.0%的OTU定义为核心优势OTU,泡凤头姜盐水中的核心优势OTU及其平均相对含量见图5。
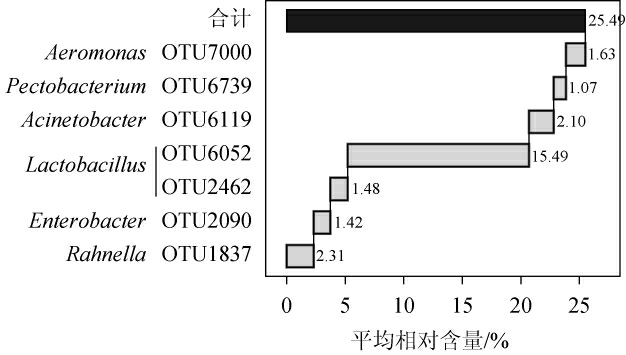
图5 泡凤头姜盐水样品中核心优势OTU瀑布图
Fig.5 Waterfall plot of core dominant OTU in fermented Fengtou ginger brine samples
由图5可知,泡凤头姜盐水样品中的核心优势OTU共有7个,分别为OTU7000、OTU6739、OTU6119、OTU6052、OTU2462、OTU2090和OTU1837,平均相对含量分别为1.63%、1.07%、2.10%、15.49%、1.48%、1.42%和2.31%,累计平均相对含量为25.49%。经序列比对发现,OTU6052和OTU2462中代表性序列被鉴定为隶属于Lactobacillus,累计平均相对含量为16.97%,由此可见,Lactobacillus是泡凤头姜盐水中的核心细菌属,这与图2的结果相一致。本研究进一步对核心优势OTU间的相关性进行分析,结果见图6。
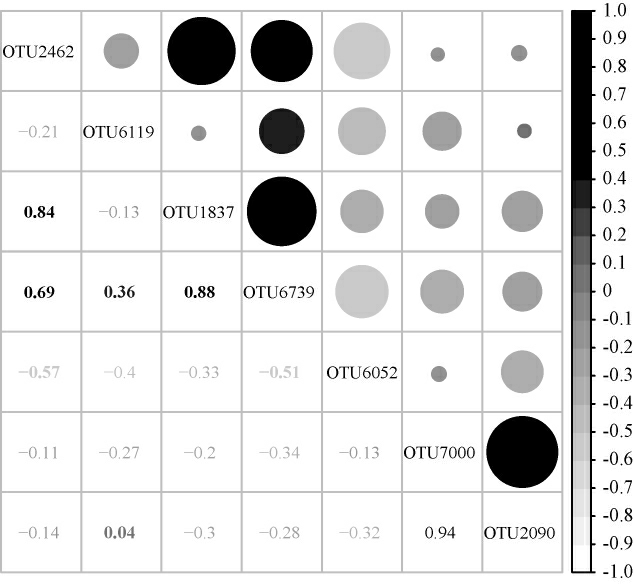
图6 核心优势OTU的相关性热图
Fig.6 Heat map of correlation analysis of core dominant OTU
由图6可知,OTU1837(Rahnella)与OTU2462(Lactobacillu)和OTU6739(Pectobacterium)均呈现显著正相关(P<0.05),OTU7000(Aeromonas)与OTU2090(Enterobacter)呈现极显著正相关关系(P<0.01)。由此可见,泡凤头姜盐水样品中的部分核心细菌类群可能存在共生关系。
2.3 PICRUSt基因功能预测
使用PICRUSt软件对泡凤头姜盐水样品中的细菌菌群基因功能进行预测,根据蛋白质直系同源簇数据库(clusters of orthologous groups of proteins,COG)进行功能注释[24]。本研究共注释到3 567个COGs,所有的COGs分别隶属于20个功能大类,其结果见图7。
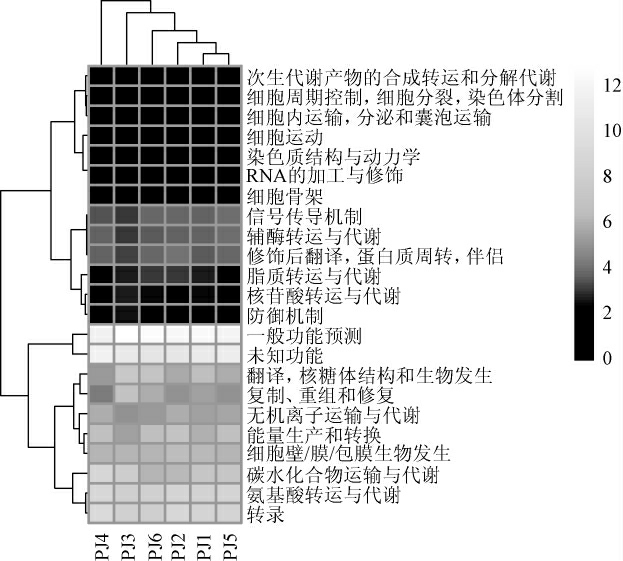
图7 泡凤头姜盐水样品中细菌基因功能的预测结果
Fig.7 Prediction results of bacterial gene function in fermented Fengtou ginger brine samples
由图7可知,泡凤头姜盐水样品中细菌的基因主要富集在氨基酸转运与代谢、碳水化合物运输和代谢以及能量生产和转换功能上,而在核糖核酸(ribonucleic acid,RNA)加工与修饰以及细胞运动相关的基因功能上富集量较低。乳酸菌作为泡菜发酵过程中的主要微生物,在发酵过程中会产生大量有机酸和氨基酸等代谢物质[25],这些代谢物质的产生可能是泡凤头姜盐水中细菌类群在氨基酸转运与代谢功能上有着较高表达的原因。
3 结论
本研究利用MiSeq高通量测序技术对来凤地区泡凤头姜盐水样品细菌类群进行分析发现,泡凤头姜盐水样品中的细菌菌群归属于19个门、46个纲、96个目、163个科和393个属;优势细菌门为变形菌门(Proteobacteria)、厚壁菌门(Firmicutes)、拟杆菌门(Bacteroidetes)和放线菌门(Actinobacteria),其平均相对含量分别为54.25%、40.26%、3.24%和1.76%;优势细菌属为乳杆菌属(Lactobacillus)、芽孢杆菌属(Bacillus)、假单胞菌属(Pseudomonas)、不动杆菌属(Acinetobacter)、罗尔斯通菌属(Ralstonia)、气单胞菌属(Aeromonas)、乳球菌属(Lactococcus)、拉恩氏菌属(Rahnella)、弧菌属(Vibrio)、肠杆菌属(Enterobacter)、克吕沃氏菌属(Kluyvera)、果胶杆菌属(Pectobacterium)、黄杆菌属(Flavobacterium)和欧文氏菌属(Erwinia),其平均相对含量分别为27.73%、7.67%、5.99%、5.67%、5.34%、3.91%、3.31%、2.60%、2.03%、1.88%、1.78%、1.32%、1.06%和1.02%;Lactobacillus为泡凤头姜盐水样品中主要的核心细菌类群,累计相对含量达到16.97%;经PICRUSt基因功能预测发现,泡凤头姜盐水样品中细菌的基因主要富集在氨基酸转运与代谢、碳水化合物运输和代谢以及能量生产和转换功能上。由此可见,来凤地区泡凤头姜盐水中细菌类群多样性较高,且富含Lactobacillus。
[1]胡洪涛,朱志刚,杨靖钟,等.不同处理对高山凤头姜姜瘟病的防效及土壤细菌群落结构和功能的影响[J].微生物学通报,2020,47(6):1763-1775.
[2]李伟,程超,莫开菊.凤头姜醇溶和水溶黄酮的抗氧化作用[J].食品科学,2017,38(19):137-142.
[3]田程飘,朱伟伟,宋雅玲,等.生姜与醋泡姜抗氧化、抑菌和抗肿瘤活性比较研究[J].食品工业科技,2019,40(14):18-23.
[4]郭壮,赵慧君,雷敏,等.白酒窖泥微生物多样性研究方法及进展[J].食品研究与开发,2018,39(22):200-207.
[5]LIU M,TANG Y,ZHAO K,et al.Determination of the fungal community of pit mud in fermentation cellars for Chinese strong-flavor liquor,using DGGE and Illumina MiSeq sequencing[J]. Food Res Int,2017,91(1):80-87.
[6]WEI Y J,WU Y,YAN Y Z,et al.High-throughput sequencing of microbial community diversity in soil,grapes,leaves,grape juice and wine of grapevine from China[J].Plos One,2018,13(3):e0193097.
[7]于国萍,陈媛,姚宇秀,等.利用Illumina MiSeq高通量测序技术分析原料乳的菌群分布[J].食品科学,2018,39(16):186-191.
[8]BOYNTON P J,GREIG D.Fungal diversity and ecosystem function data from wine fermentation vats and microcosms[J].Data Brief,2016,8(12):225-229.
[9]ZEBIN C,TIYUAN X,DINGKANG W,et al.Diversity analysis of endophytic fungi in mentha using Illumina MiSeq high-throughput sequencing technology[J].Med Plant,2016,7(Z1):46-50.
[10]夏梦雷,杨帆,陆锴,等.高通量测序技术在传统发酵食品微生物群落中的应用研究[J].中国酿造,2021,40(5):1-7.
[11]XU N,TAN G,WANG H,et al.Effect of biochar additions to soil on nitrogen leaching,microbial biomass and bacterial community structure[J].Eur J Soil Biol,2016,74(3):1-8.
[12]郭壮,王玉荣,葛东颖,等.腊肠发酵过程中细菌多样性评价及其对风味的影响[J].食品科学,2021,42(6):186-192.
[13]CAPORASO J G,KUCZYNSKI J,STOMBAUGH J,et al.QIIME allows analysis of high-throughput community sequencing data[J]. Nat Method,2010,7(5):335-336.
[14]王玉荣,沈馨,董蕴,等.鲊广椒细菌多样性评价及其对风味的影响[J].食品与机械,2018,34(4):25-30.
[15]PUENTES-TÉLLEZ P E,SALLES J F.Dynamics of abundant and rare bacteria during degradation of lignocellulose from sugarcane biomass[J].Microbial Ecol,2020,79(2):312-325.
[16]COLE J R,WANG Q,FISH J A,et al.Ribosomal database project:data and tools for high throughput rRNA analysis[J]. Nucleic Acids Res,2014,42(D1):D633-D642.
[17]WILKINSON T J,HUWS S A,EDWARDS J E,et al.CowPI:a rumen microbiome focussed version of the PICRUSt functional inference software[J].Front Microbiol,2018,9:1095.
[18]王玉荣,杨成聪,葛东颖,等.扩增区域对鲊广椒细菌MiSeq测序的影响[J].食品科学,2019,40(10):134-140.
[19]XIONG T,GUAN Q,SONG S,et al.Dynamic changes of lactic acid bacteria flora during Chinese sauerkraut fermentation[J].Food Control,2012,26(1):178-181.
[20]XIONG T,LI J,LIANG F,et al.Effects of salt concentration on Chinese sauerkraut fermentation[J].LWT-Food Sci Technol,2016,69(6):169-174.
[21]ERGONUL O,TOKCA G,KESKE S,,et al.Elimination of healthcareassociated Acinetobacter baumannii infection in a highly endemic region[J].Int J Infect Dis,2022,114(1):11-14.
[22]AZAM M W,KHAN A U.Updates on the pathogenicity status of Pseudomonas aeruginosa[J].Drug Discov Today,2019,24(1):350-359.
[23]ANNAVAJHALA M K,GOMEZ-SIMMONDS A,UHLEMANN A C.Multidrug-resistant Enterobacter cloacae complex emerging as a global,diversifying threat[J].Front Microbiol,2019,10(1):44.
[24]GALPERIN M Y,WOLF Y I,MAKAROVA K S,et al.COG database update:focus on microbial diversity,model organisms,and widespread pathogens[J].Nucleic Acids Res,2021,49(D1):D274-D281.
[25]XIONG T,CHEN J,HUANG T,et al.Fast evaluation by quantitative PCR of microbial diversity and safety of Chinese Paocai inoculated with Lactobacillus plantarum NCU116 as the culture starter[J]. LWT Food Sci Tcchnol,2019,101(3):201-206.