益生菌是能够为人类提供健康益处的活微生物的统称,主要是能够利用可发酵碳水化合物产生大量乳酸的细菌,其能够改善消化系统健康、预防感染性腹泻、肠应激综合征和炎症性肠病等[1-3]。但益生菌的健康益处具有菌种或菌株特异性,并非所有的乳酸菌都可以被认为是益生菌[4]。双歧杆菌(Bifidobacterium)是益生菌的重要成员之一,已发现双歧杆菌物种参与了多种促进健康的活性化合物的生物合成,包括短链脂肪酸(short chain fatty acid,SCFA)、维生素和有机酸[5],其对宿主具有抗癌[6]和降低胆固醇水平等作用[7]。有研究表明,人类肠道中的双歧杆菌可为人体提供能量、营养、调节免疫系统和调整宿主的肠道生理[8-11]。
双歧杆菌属于革兰氏阳性、非运动性和过氧化氢酶阴性的厌氧杆菌,其可以在人类肠道中存活。目前,在人类肠道中能够存活和被发现的双歧杆菌主要有长双歧杆菌(Bifidobacterium longum)、两歧双歧杆菌(Bifidobacterium bifidum)、短双歧杆菌(Bifidobacterium breve)、青春双歧杆菌(Bifidobacterium adolescentis)、假长双歧杆菌(Bifidobacterium pseudomonas)和嗜热双歧杆菌(Bifidobacterium thermophilum)等[12-14]。双歧杆菌属内不同的亚种或菌株具有不同的益生效果,如动物双歧杆菌乳亚种(Bifidobacterium animalis subsp.lactis),又称乳双歧杆菌,其具有较强的抗炎作用和抑制病原菌的作用,可以改善人体免疫系统[15],已经被用作各种功能性食品中的活性成分[16-17]。然而,动物双歧杆菌动物亚种却不能够在牛奶中生长[18];长双歧杆菌不同亚种能分泌不同的糖酵解酶[19]。因此,区分双歧杆菌亚种是非常必要的。此外,在向消费者提供的益生菌产品标签中标注明确到亚种或株的物种信息和其声称的产品益生效果也是至关重要的[20-21]。近些年,国外有研究报告显示,商业益生菌产品存在标签不准确的问题,比如缺少标注中明确的某些物种、标注物种分类信息不准确以及甚至出现了未申报物种被添加的情况[19,22]。然而,目前对于双歧杆菌的检测主要还是依靠传统的生理生化方法,该方法已被证明不能对双歧杆菌的不同种和亚种进行区分。因此,目前还没有可靠的检测方法来区分双歧杆菌的不同种和亚种。
基于聚合酶链式反应(polymerase chain reaction,PCR)的方法已经被广泛用于益生菌、乳制品、肉制品和海鲜制品中细菌菌株的鉴定[23-25]。特别是16S rRNA基因已被用作细菌鉴定的常用靶基因,该基因在属间鉴定具有非常好的分辨率,然而在亲缘关系非常近的物种间具有非常高的相似性(平均值>95%),所以具有非常低的分辨率[26-27]。近些年,国外也有研究使用新的管家基因(如tuf基因[28]、recA基因[28]、hsp60基因[29]等)替代16S rRNA基因用于区分双歧杆菌。这些基因与16S rRNA基因相比具有更高的分辨率,但是仍然无法区分相似性更高的物种和亚种。因此,这些基因只能应用于有限的物种区分[30]。
基于此,本研究通过生物信息学分析并建立系统发育树,选择区分度更高的基因建立一种能够在亚种层面对动物双歧杆菌乳亚种进行鉴定的实时荧光定量聚合酶链式反应(real-time fluorescence quantitative polymerase chain reaction,RT-fqPCR)方法。通过特异性、灵敏性和抗干扰实验对该方法进行验证,并对市售的64份标识含有乳双歧杆菌的婴幼儿配方乳粉样品进行检测,以期为双歧杆菌检测标准的修订提供参考,为动物双歧杆菌杆菌乳亚种的鉴定提供技术支撑。
1 材料与方法
1.1 材料与试剂
1.1.1 材料
64份标识含有动物双歧杆菌乳亚种的婴幼儿配方乳粉样品:北京市各大超市或销售门店。
1.1.2 试剂及菌种
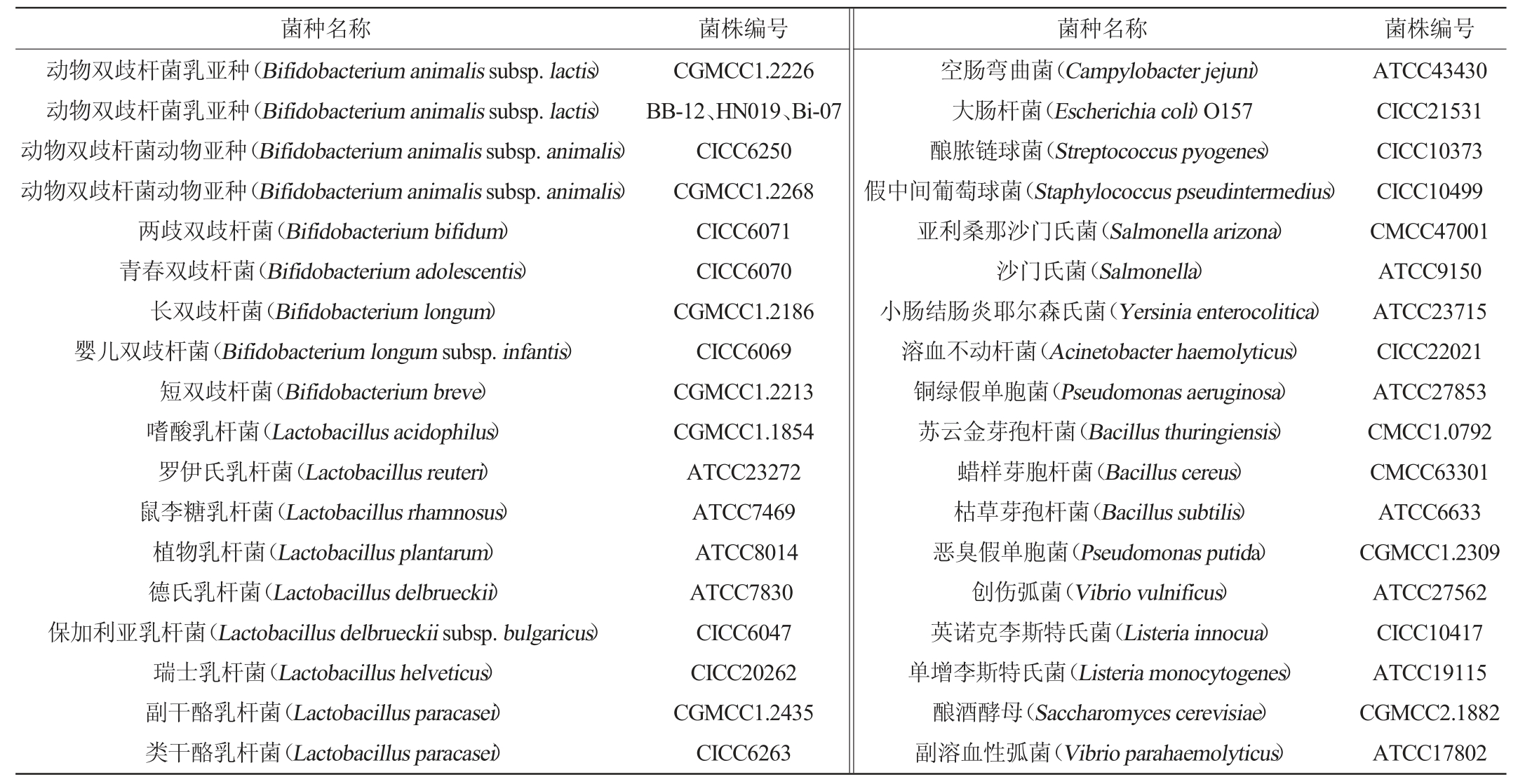
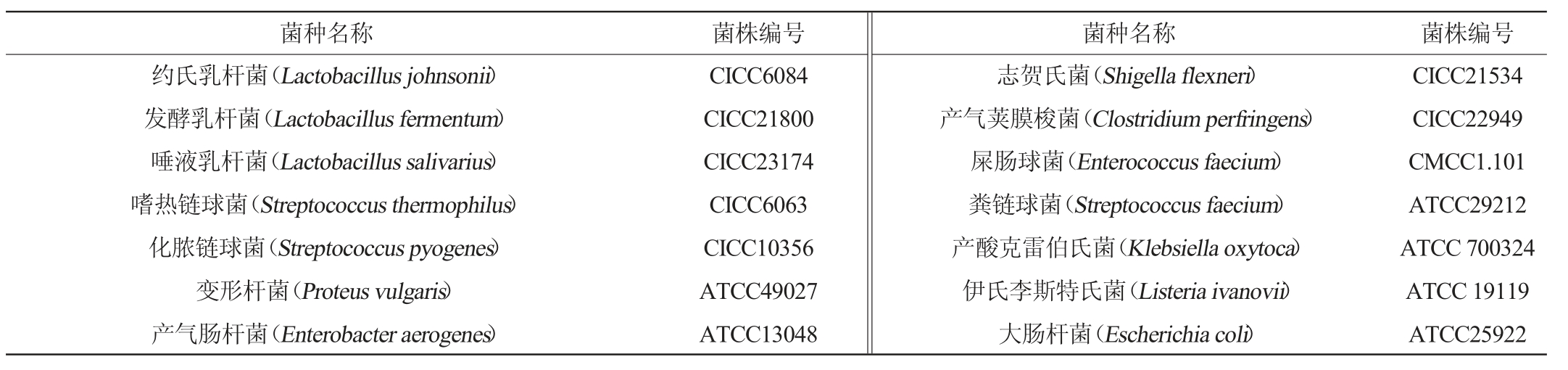
MRS琼脂培养基、MC培养基:北京陆桥技术有限公司;细菌基因组脱氧核糖核酸(deoxyribonucleic acid,DNA)提取试剂盒、蛋白酶K(20 mg/mL)、溶菌酶(50 mg/mL):天根生化科技有限公司;实验所用菌株(表1):中国工业微生物菌种保藏管理中心(China center of industrial culture collection,CICC)。
表1 实验所用菌株信息
Table 1 Information of strains used in the experiment
续表
1.2 仪器及设备
Taqman master mix、ABI QuantStudio 7实时荧光PCR仪:美国ABI公司;VITEK Compact微生物分析系统:法国生物梅里埃公司。
Compact微生物分析系统:法国生物梅里埃公司。
1.3 方法
1.3.1 菌株活化及处理
将冷冻干燥保藏的动物双歧杆菌乳亚种标准菌株转接于MRS琼脂培养基,37 ℃厌氧培养(48±2)h,转接两代回复活力,用于后续实验。
1.3.2 DNA提取
按照细菌基因组DNA提取试剂盒说明书提取DNA。
1.3.3 引物探针设计
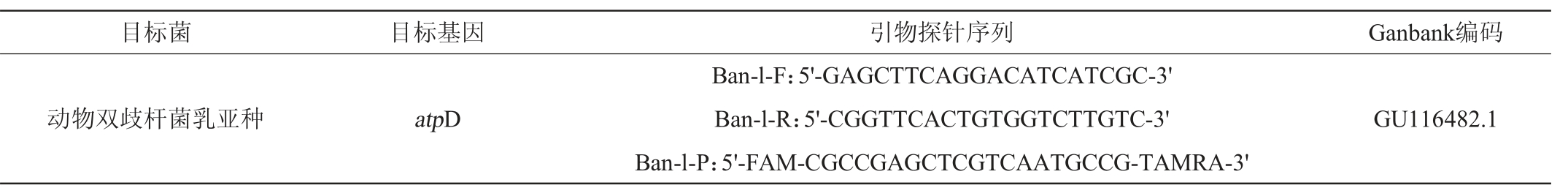
根据已发表文献[26-30]确定选取16S-23S rRNA、Tuf、hsp60、recA、atpD等基因作为候选目标基因,在美国国立生物技术信息中心(national center for biotechnology information,NCBI)网站下载动物双歧杆菌乳亚种及亲缘关系较近的其他双歧杆菌和乳杆菌的候选目标基因序列,使用DNAMAN V9.0软件进行序列比对,以MEGA-X生物信息学软件中的邻接(neighbor-joining,NJ)法构建系统发育树,分析该基因在动物双歧杆菌乳亚种水平的区分度,根据区分度确定目标基因。选择目标基因的一段突变区序列作为引物和探针设计的目标片段。使用Primer Express V3.0设计引物和探针,设计得到的引物探针在NCBI网站进行primer Blast,选出特异性较好的引物探针由上海生物工程有限公司合成。引物探针序列见表2。
表2 实时荧光PCR引物和探针序列
Table 2 Sequence of primers and probes of real-time fluorescence PCR
1.3.4 实时荧光定量PCR
实时荧光定量PCR扩增体系(25 μL):2×master mix 12.5μL、引物对(10μmol/L)各1 μL、探针(10μmol/L)0.5μL、模板DNA 1 μL、双蒸水(ddH2O)9 μL。
实时荧光PCR扩增条件:50 ℃、2 min;95 ℃、10 min;95 ℃、5 s,64 ℃、40 s,40个循环;同时收集FAM荧光。
对照设置:阳性对照以动物双歧杆菌乳亚种CGMCC 1.2226 DNA为模板,阴性对照以大肠杆菌(Escherichia coli)ATCC25922 DNA为模板;空白对照以双蒸水为模板。
实时荧光定量PCR扩增曲线指数期明显,Ct<35可直接判定为阳性;Ct值为35~40判定为可疑,需要进行重复实验;Ct>40判定为阴性。
1.3.5 特异性及灵敏性试验
特异性实验以动物双歧杆菌乳亚种及表1中的菌株DNA作为扩增模板,按照1.3.4中的方法进行实时荧光定量PCR扩增。
灵敏度实验包括绝对灵敏度和相对灵敏度试验[31]。绝对灵敏度试验:测定动物双歧杆菌乳亚种CGMCC1.2226 DNA溶液的质量浓度,根据质量浓度梯度稀释至10 ng/μL、1 ng/μL、0.1 ng/μL、0.01 ng/μL、0.001 ng/μL、0.000 5 ng/μL、0.000 1 ng/μL。所有稀释液在同一反应条件下进行实时荧光定量PCR扩增。相对灵敏度试验:将稀释后不同菌体浓度的动物双歧杆菌乳亚种菌悬液添加到无菌婴幼儿配方乳粉中,添加菌落数分别为107 CFU/mL、106 CFU/mL、105 CFU/mL、104 CFU/mL、103 CFU/mL、102 CFU/mL、101 CFU/mL,使用细菌基因组DNA提取试剂盒提取DNA后进行实时荧光定量PCR扩增。灵敏度实验每个反应设置5个平行,重复实验4次共20次实验,记录阳性扩增次数。
1.3.6 抗干扰试验
为了验证方法的抗干扰能力,设计基因组水平的抗干扰试验和物种水平的抗干扰试验。
基因组水平的抗干扰试验:将提取得到的动物双歧杆菌乳亚种CGMCC1.2226的DNA稀释至10 ng/μL、1 ng/μL、0.1 ng/μL、0.01 ng/μL,按照1∶1的体积比分别添加10 ng/μL的大肠杆菌ATCC25922基因组DNA或TE缓冲液,混合后的DNA作为模板按照1.3.4的方法进行实时荧光定量PCR扩增,记录干扰DNA对试验结果的影响。
培养物水平的抗干扰试验:将动物双歧杆菌乳亚种CGMCC1.2226培养物稀释到107 CFU/mL、106 CFU/mL、105 CFU/mL、104 CFU/mL,按照1∶1的体积比分别添加107 CFU/mL的大肠杆菌ATCC25922或无菌水,充分混匀后取1 mL,按照细菌基因组DNA提取试剂盒的说明书提取DNA后进行实时荧光定量PCR扩增,记录非特异性菌对实验结果的影响。
1.3.7 市售婴幼儿配方乳粉样品的检测
将购买得到的婴幼儿配方乳粉样品按照国标GB 4789.34—2016《食品安全国家标准食品微生物学检验双歧杆菌检验》的方法进行培养。从每个样品MRS平板上挑取10个单菌落,使用细菌基因组DNA提取试剂盒提取DNA,将该DNA作为模板进行实时荧光定量PCR扩增,记录阳性扩增样品数。
2 结果与分析
2.1 系统发育树分析结果
基于NCBI网站下载的动物双歧杆菌乳亚种及亲缘关系较近的其他双歧杆菌和乳杆菌的候选目标基因16S-23S rRNA、Tuf、hsp60、recA、atpD等的序列构建系统发育树,结果发现,atpD基因具有较好的区分度,其系统发育树见图1。
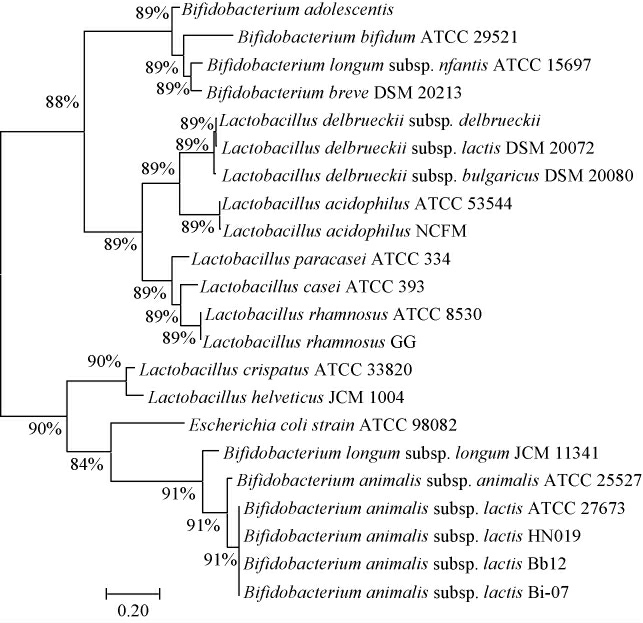
图1 基于atpD基因菌株的系统发育树
Fig.1 Phylogenetic tree of strains based on atpD gene
由图1可知,动物双歧杆菌乳亚种和动物双歧杆菌动物亚种虽然在同一个大的分支,但其差异率接近10%,与目前常用的菌种鉴定基因16S rRNA相比具有更大的分辨率[32]。atpD基因与乳杆菌和其他双歧杆菌间的差异更为明显,差异率>10%。结果表明,atpD基因可以用于动物双歧杆菌亚种间的鉴定和区分。
2.2 特异性验证结果
实时荧光定量PCR扩增法鉴定动物双歧杆菌乳亚种的特异性验证结果见图2。
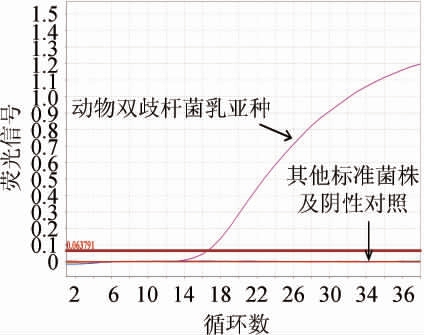
图2 实时荧光定量PCR法鉴定动物双歧杆菌乳亚种的特异性实验结果
Fig.2 Specificity test results of identification of Bifidobacterium animalis subsp. lactis by real-time fluorescence quantitative PCR method
由图2可知,以动物双歧杆菌乳亚种的DNA为模板的实时荧光定量PCR具有明显的荧光扩增曲线,以亲缘关系较近的其他乳酸菌和食品中常见的细菌DNA为模板的实时荧光定量PCR均无荧光扩增信号。由此可以判断,该引物探针可以特异性的扩增动物双歧杆菌乳亚种atpD基因,并能够与亲缘关系较近的其他双歧杆菌进行很好的区分。
2.3 灵敏性验证实验结果
2.3.1 绝对灵敏度验证
实时荧光定量PCR扩增法鉴定动物双歧杆菌乳亚种的绝对灵敏度实验结果见表3。
表3 实时荧光定量PCR法鉴定动物双歧杆菌乳亚种的绝对灵敏度实验结果
Table 3 Absolute sensitivity test results of identification of Bifidobacterium animalis subsp. lactis by real-time fluorescence quantitative PCR method
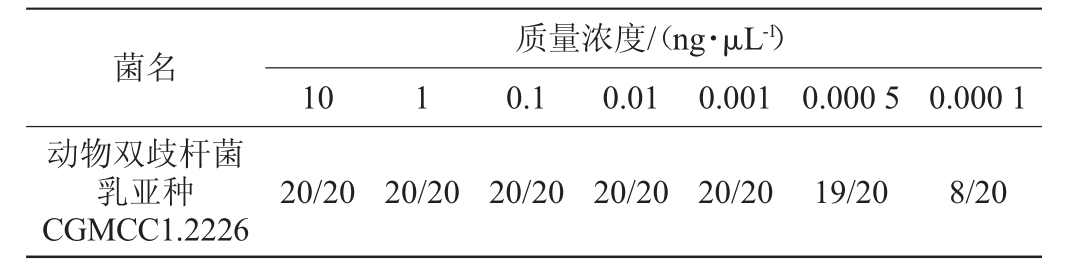
注:表中数据表示“阳性检出次数/总检测次数”。下同。
由表3可知,当动物双歧杆菌乳亚种CGMCC1.2226的DNA质量浓度为0.001 ng/μL时,20组试验结果均为阳性;当DNA质量浓度为0.000 5 ng/μL时,出现阴性结果的情况,说明该方法在基因组水平的可稳定检出的质量浓度为0.001 ng/μL,表示检测下限可以达到1 pg/μL。因此,确定本研究建立的实时荧光定量PCR方法的绝对灵敏度为1pg/μL。
2.2.2 相对灵敏度验证
实时荧光定量PCR扩增法鉴定动物双歧杆菌乳亚种的相对灵敏度实验结果见表4。
表4 实时荧光PCR法鉴定动物双歧杆菌乳亚种的相对灵敏度实验结果
Table 4 Relative sensitivity test results of identification of Bifidobacterium animalis subsp. lactis by real-time fluorescence PCR method

由表4可知,当动物双歧杆菌乳亚种CGMCC1.2226的菌体浓度为103 CFU/mL时,20组试验结果均为阳性;当菌体浓度为102CFU/mL时,出现阴性结果的情况,说明本研究所建立的实时荧光定量PCR法从婴幼儿配方乳粉中可稳定检出的动物双歧杆菌乳亚种最低菌体浓度为103 CFU/mL。因此,确定本研究建立的实时荧光定量PCR法的相对灵敏度可以达到103 CFU/mL。
2.3 抗干扰能力验证实验结果
2.3.1 基因组水平的抗干扰能力验证
根据检测过程的真实情况,往往检测对象是多种杂菌与目标菌株共同存在的混合菌,导致提取得到的DNA不只是目标菌种一种基因组,因此,本实验设计了基因组水平的抗干扰实验,结果见表5。
表5 基因组水平实时荧光定量PCR扩增法鉴定动物双歧杆菌乳亚种的抗干扰实验结果
Table 5 Anti-disturbance test results of identification of Bifidobacterium animalis subsp. lactis by real-time fluorescence quantitative PCR method at genome level
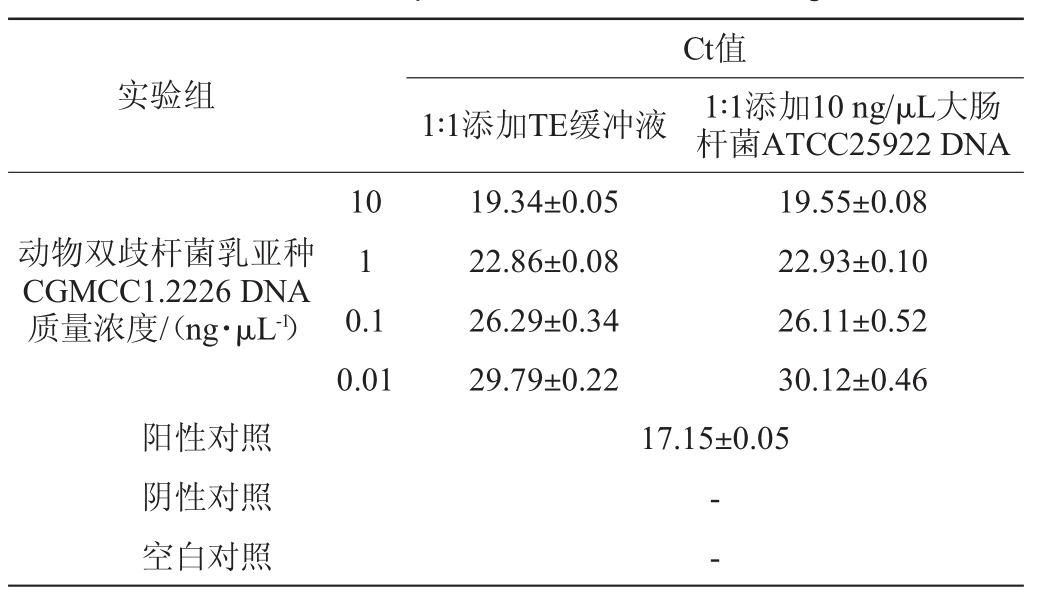
注:“-”表示Ct值>40。下同。
由表5可知,当动物双歧杆菌乳亚种CGMCC1.2226的DNA质量浓度分别为10ng/μL、1ng/μL、0.1ng/μL、0.01ng/μL时,Ct值均<35,结果均为阳性,说明在干扰基因组DNA质量浓度是目标基因组质量浓度1 000倍的情况下,Ct值无显著影响(P>0.05)。结果表明,本研究建立的方法在基因组水平上具有非常好的抗干扰能力。
2.3.2 培养物水平的抗干扰实验结果
培养物水平实时荧光定量PCR扩增法鉴定动物双歧杆菌乳亚种的抗干扰实验结果见表6。
表6 培养物水平实时荧光定量PCR扩增法鉴定动物双歧杆菌乳亚种的抗干扰实验结果
Table 6 Anti-disturbance test results of identification of Bifidobacterium animalis subsp. lactis by real-time fluorescence quantitative PCR method at culture level
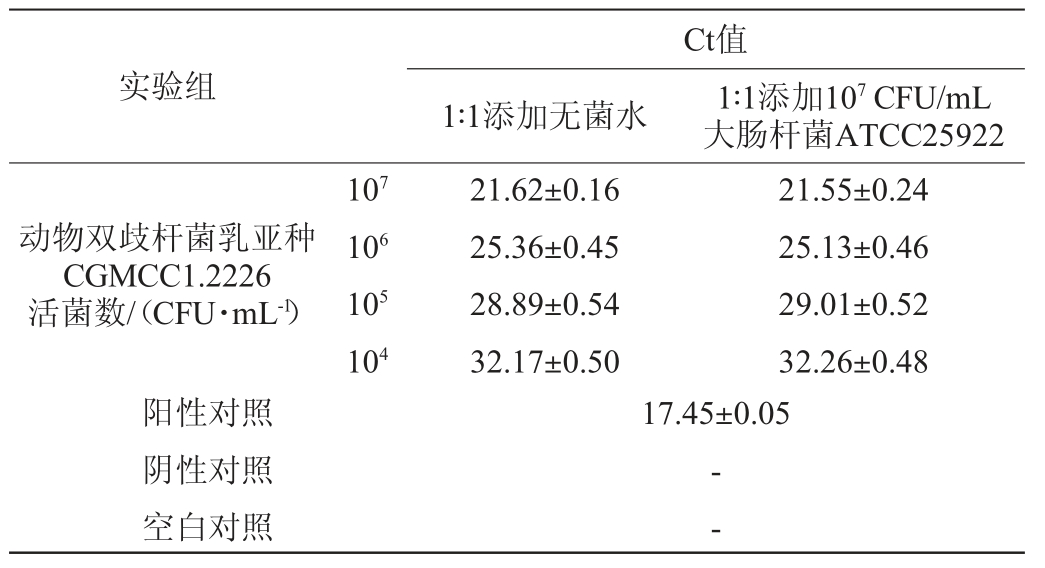
由表6可知,当动物双歧杆菌乳亚种CGMCC1.2226的菌体浓度分别为107 CFU/mL、106 CFU/mL、105 CFU/mL、104 CFU/mL时,Ct值均<35,结果均为阳性,说明在干扰菌体浓度是目标菌体浓度1 000倍的情况下,Ct值未有显著影响(P>0.05),均不影响动物双歧杆菌乳亚种的检出,结果表明,本研究建立的实时荧光定量PCR方法在培养物水平上具有很好的抗干扰能力。
2.4 市售婴幼儿配方乳粉样品的检测结果
使用本研究建立的实时荧光定量PCR方法对市售的64份标明含有动物双歧杆菌乳亚种的婴幼儿配方乳粉进行检测,检测结果显示均检出动物双歧杆菌乳亚种。从该结果可以反映出国内销售的婴幼儿配方乳粉添加动物双歧杆菌乳亚种(乳双歧杆菌)的添加种类及浓度均满足国家标准GB10765—2010《食品安全国家标准婴儿配方食品》[33]的要求。
3 讨论
根据目前文献报道,动物双歧杆菌动物亚种和动物双歧杆菌乳亚种在表型和基因型方面都具有非常高的相似性,但是它们又具有不同的功能,如它们在牛奶中的生长情况或表达酶的能力[18-19]。为了区分这些物种或亚种,目前认为可信度更高的方法是全基因组测序和实时荧光定量PCR方法。全基因组测序的方法虽然比较成熟,能够更好的区分不同的种或亚种,但其鉴定成本过高,导致其在日常检测中很难得到广泛的应用。实时荧光定量PCR方法虽然是一种技术成熟、操作简便、检测成本较低的技术方法,但是建立一种能够在种或亚种进行鉴定的实时荧光定量PCR方法也存在比较大的技术难度。根据目前的文献报道,在双歧杆菌属或种水平的鉴定中选择的目标基因主要是16S rRNA和Tuf基因,由于这两个基因在亚种间具有非常高的相似度,因此很难用于亚种间的鉴定[34-35]。也有文献报道[36],通过比较基因组学分析的方法获得功能基因,选用功能基因作为靶基因设计实时荧光定量PCR方法对长双歧杆菌长亚种和长双歧杆菌婴儿亚种进行鉴定。但是该种方法具有一定的局限性,比如,长双歧杆菌婴儿亚种的特异性唾液酸酶基因仅在一些长双歧杆菌婴儿亚种中存在,并不是所有长双歧杆菌婴儿亚种中都存在,该基因也被发现存在于一些两歧双歧杆菌中。此外,长双歧杆菌长亚种特异性激酶基因,被证实在青春双歧杆菌中也存在。因此,选择功能基因作为目标基因建立的实时荧光定量PCR方法具有一定的局限性。为了能够改善先前研究中存在分辨率低和局限性问题,通过大量的基因序列比对和发育树的构建与分析,选取atpD基因作为目标基因设计引物探针。通过发育树结果可以看出,动物双歧杆菌乳亚种与动物双歧杆菌动物亚种之间基因差异接近10%,与其他双歧杆菌和乳杆菌差异率均>10%,可以用于实时荧光定量PCR引物探针的设计。在此基础上建立的实时荧光定量PCR方法具有非常好的特异性、灵敏性和抗干扰能力。
4 结论
本研究基于动物双歧杆菌乳亚种、动物双歧杆菌动物亚种、长双歧杆菌、短双歧杆菌、两歧双歧感觉、婴儿双歧杆菌和部分乳杆菌等22株菌的atpD基因建立系统发育树,结果发现,该基因在双歧杆菌乳亚种间具有较高的种间特异性,种间差异率>10%,因此,利用该基因设计引物探针,利用实时荧光定量PCR法对动物双歧杆菌乳亚种进行鉴定。结果发现,该方法能够特异性的检测动物双歧杆菌乳亚种;绝对灵敏度可以达到1 pg/μL,相对灵敏度可以达到103 CFU/mL;基因水平和培养物水平抗干扰能力良好。采用该方法从64份标识含有动物双歧杆菌乳亚种的乳粉样品均能检测出动物双歧杆菌乳亚种,表明基于atpD基因建立的RT-fqPCR方法能够快速准确的对动物双歧杆菌乳亚种进行检测。本研究建立的实时荧光定量PCR法可为婴幼儿配方乳粉中乳双歧杆菌的检测提供技术支撑,为其他双歧杆菌的鉴定方法的建立提供了一定的数据参考。
[1]HIDALGO-CANTABRANA C,DELGADO S,RUIZ L,et al.Bifidobacteria and their health-promoting effects[J].Microbiol Spectrum,2017,5(3):DOI:https://doi.org/10.1128/microbiolspec.BAD-0010-2016.
[2]AGAF M H F M.The role of prebiotics and probiotics in gastrointestinal disease[J].Gastroenterol Clin N,2018,47(1):179-191.
[3]SHEHATA H R,RAGUPATHY S,SHANMUGHANANDHAN D,et al.Guidelines for validation of qualitative real-time PCR methods for molecular diagnostic identification of probiotics[J].J AOAC Int,2019,102(6):1774-1778.
[4]PINTO-SANCHEZ M I,HALL G B,GHAJAR K,et al.Probiotic Bifidobacterium longum NCC3001 reduces depression scores and alters brain activity:A pilot study in patients with irritable bowel syndrome[J].Gastroenterology,2017,153(2):448.
[5]SARKAR A,MANDAL S. Bifidobacteria-insight into clinical outcomes and mechanisms of its probiotic action[J].Microbiol Res,2016,192:159-171.
[6]INOUE Y,IWABUCHI N,XIAO J Z,et al.Suppressive effects of Bifidobacterium breve strain M-16V on T-helper type 2 immune responses in a murine model[J].Biol Pharm Bull,2009,32(4):760-763.
[7]ZHANG R,HE L,LING Z,et al.Screening of cholesterol-lowering Bifidobacterium from Guizhou Xiang Pigs,and evaluation of its tolerance to oxygen,acid,and bile [J].Korean J Food Sci Anim Resour,2016,36(1):37-43.
[8]CHIARA F,ROSARIO S,LUCA C,et al.How to feed the mammalian gut microbiota:bacterial and metabolic modulation by dietary fibers[J].Front Microbiol,2017,8:1749.
[9]MILANI C,DURANTI S,BOTTACINI F,et al.The first microbial colonizers of the human gut:Composition,activities,and health implications of the infant gut microbiota[J].Microbiol Mol Biol Rev Mmbr,2017,81(4):00036-17.
[10]LEBLANC J G,MILANI C,GIORI G,et al.Bacteria as vitamin suppliers to their host:A gut microbiota perspective[J].Curr Opin Biotech,2012,24(2):160-168.
[11]VENTURA M,O'TOOLE P W,VOS W D,et al.Selected aspects of the human gut microbiota[J].Cell Mol Life Sci,2018,75(1):81-82.
[12]JUNICK J,BLAUT M.Quantification of human fecal Bifidobacterium species by use of quantitative real-time PCR analysis targeting the groEL gene[J].Appl Environ Microbiol,2012,78(8):2613.
[13]AH U V,MOZZETTI V,LACROIX C,et al.Classification of a moderately oxygen-tolerant isolate from baby faeces as Bifidobacterium thermophilum[J].Bmc Microbiol,2007,7(1):79-79.
[14]TURRONI F,FORONI E,PIZZETTI P,et al.Exploring the diversity of the bifidobacterial population in the human intestinal tract[J].Appl Environ Microbiol,2009,75(6):1534-15345.
[15]WEIZMAN Z,ASLI G,ALSHEIKH A.Effect of a probiotic infant formula on infections in child care centers:comparison of two probiotic agents[J].Pediatrics,2005,115(1):5-9.
[16]DAN L W,QUILICI F A,MICHZPUTEN S,et al.The effect of probiotic fermented milk that includes Bifidobacterium lactis CNCM I-2494 on the reduction of gastrointestinal discomfort and symptoms in adults:a narrative review[J].Nutr Hosp,2015,32(2):501-509.
[17]AYOUNG L,JU L Y,JIN Y H,et al.Consumption of dairy yogurt containing Lactobacillus paracasei ssp.paracasei,Bifidobacterium animalis ssp. lactis and heat-treated Lactobacillus plantarum improves immune function including natural killer cell activity[J].Nutrients,2017,9(6):558.
[18]MASCO L.Polyphasic taxonomic analysis of Bifidobacterium animalis and Bifidobacterium lactis reveals relatedness at the subspecies level:reclassification of Bifidobacterium animalis as Bifidobacterium animalis subsp.animalis subsp.nov.and Bifidobacterium lacti[J].Int J Syst Evol Micr,2004,54(4):1137.
[19]LEWIS Z T,SHANI G,MASARWEH C,et al.Validating bifidobacterial species and subspecies identity in commercial probiotic products[J].Pediatr Res,2016,79(3):445-452.
[20]SHEHATA H R,RAGUPATHY S,SHANMUGHANANDHAN D,et al.Guidelines for validation of qualitative real-time PCR methods for molecular diagnostic identification of probiotics[J].J AOAC Int,2019,102(6):1774-1778.
[21]卫生部关于公布可用于婴幼儿食品的菌种名单的公告[J].中国食品添加剂,2011(6):223.
[22]WESLEY M,HIBBERD A A,BRYAN Z,et al.Genotyping by PCR and high-throughput sequencing of commercial probiotic products reveals composition biases[J].Front Microbiol,2016,7:1747.
[23]BINETTI A G,CAPRA M L,ALVAREZ M A,et al.PCR method for detection and identification of Lactobacillus casei/paracasei bacteriophages in dairy products[J].Int J Food Microbiol,2008,124(2):147-153.
[24]C CAMMÀ,MD DOMENICO,MONACO F.Development and validation of fast real-time PCR assays for species identification in raw and cooked meat mixtures[J].Food Control,2012,23(2):400-404.
[25]KIM E,KIM H J,YANG S M,et al.Rapid identification of Staphylococcus species isolated from food samples by matrix-assisted laser desorption/ionization time-of-flight mass spectrometry[J].J Microbiol Biotechn,2019,29(4):548-557.
[26]VENTURA M,CANCHAYA C,CASALE AD,et al.Analysis of bifidobacterial evolution using a multilocus approach[J].Int J Syst Evol Micr,2006,56(12):2783-2792.
[27]JUNICK J,BLAUT M.Quantification of human fecal Bifidobacterium species by use of quantitative real-time PCR analysis targeting the groELgene[J].Appl Environ Microbiol,2012,78(8):2613.
[28]VENTURA M,ZINK R.Comparative sequence analysis of the tuf and recA genes and restriction fragment length polymorphism of the internal transcribed spacer region sequences supply additional tools for discriminating Bifidobacterium lactis from Bifidobacterium animalis[J].Appl Environ Microbiol,2003,69(12):7517-7522.
[29]LIN Z,WEI L,DONG X.Species identification of genus Bifidobacterium based on partial HSP60 gene sequences and proposal of Bifidobacterium thermacidophilum subsp.porcinum subsp.nov[J].Int J Syst Evol Micr,2003,53(Pt 5):1619-1623.
[30]DIOS J D,CL MART NEZ,TATO M,et al.94 Comparison between MALDI-TOFandrecAgenesequencingfortheidentification of Burkholderia cepacia complex species isolated in a cystic fibrosis unit[J].J Cyst Fibr,2016,15:S75-S75.
NEZ,TATO M,et al.94 Comparison between MALDI-TOFandrecAgenesequencingfortheidentification of Burkholderia cepacia complex species isolated in a cystic fibrosis unit[J].J Cyst Fibr,2016,15:S75-S75.
[31]肖其胜,杨捷琳,杨惠琴,等.实时荧光PCR法鉴定食品中双歧杆菌[J].食品科学,2016,37(20):177-182.
[32]多拉娜,宋宇琴,李伟程,等.干酪乳杆菌的近缘种及亚种部分看家基因的系统发育分析[J].微生物学通报,2018,45(12):11.
[33]中华人民共和国卫生部.GB 10765—2010食品安全国家标准婴幼儿配方食品[S].北京:中国标准出版社,2010.
[34]TANNOCK G W,LAWLEY B,MUNRO K,et al.Comparison of the compositions of the stool microbiotas of infants fed goat milk formula,cow milk-based formula,or breast milk[J].Appl Environ Microbiol,2013,79(9):3040-3048.
[35]KURAKAWA T,OGATA K,TSUJI H,et al.Establishment of a sensitive system for analysis of human vaginal microbiota on the basis of rRNAtargeted reverse transcription-quantitative PCR[J].J Microbiol Methods,2015,111:93-104.
[36]BLAIR,LAWLEY,KAREN,et al.Differentiation of Bifidobacterium longum subspecies longum and infantis by quantitative PCR using functional gene targets[J].Peer J,2017,5:e3375.