发酵豆制品是以各种豆类为原料,在开放环境中经自然发酵而形成特色风味的发酵成品[1]。酱豆作为我国主要的传统发酵豆制品之一,在发酵的过程中受微生物作用产生独特的风味,发酵后的豆制品营养丰富,容易被人体消化吸收[2-4]。在我国,酱豆的食用历史比较悠久,深受广大消费者的喜爱,其作为发酵豆制品具有抑制肿瘤生长、降血压、降血脂、延缓衰老和抗氧化等功效[4-6]。由于酱豆相对开放的发酵环境,导致其在发酵过程中的微生物菌群组成丰富且较为复杂,由于酱豆中所含的微生物对产品的风味造成影响,因此对酱豆中的微生物进行解析显得尤为重要。JUNG J Y等[7]利用焦磷酸测序技术研究韩国发酵豆酱中酱醅的微生物动态变化,结果发现,发酵豆酱中酱醅的优势细菌和真菌分别为芽孢杆菌(Bacillus)和毛霉(Mucor);张颖等[8]利用聚合酶链式反应-变性梯度凝胶电泳(polymerase chain reaction-denaturing gradient gel electrophoresis,PCR-DGGE)结合高通量测序技术对东北传统自然发酵豆酱发酵过程中的细菌多样性进行研究,结果发现,不同发酵阶段豆酱的优势细菌属为乳杆菌属(Lactobacillus)、明串珠菌属(Leukonostoc)、四联球菌属(Tetragenococcus)和肠球菌属(Enterococcus);安飞宇等[9]对发酵豆酱的微生物多样性进行了解析,结果发现豆酱中的优势细菌属为乳杆菌属和四联球菌属,优势真菌属为青霉菌属(Penicillium)和异常威克汉姆酵母属(Wickerhamomyces)。
高通量测序技术是分析食品中微生物多样性的方法之一,其通量高,测序结果准确,能够产生大量的有用信息[10-12]。目前,高通量测序技术已经广泛应用于医学[13-14]、植物[15-16]以及食品[17-19]等诸多领域,其中在分析食品中微生物的多样性研究中应用尤为广泛。高世南等[20]采用Illumina MiSeq高通量测序技术对新疆新源县酸马奶的细菌多样性进行了解析,结果显示酸马奶中的优势细菌门主要为厚壁菌门(Firmicutes),优势细菌属主要为乳杆菌属(Lactobacillus)。
本研究以湖北省恩施土家族苗族自治州利川市的酱豆为研究对象,利用Illumina MiSeq高通量测序技术对其中的真菌多样性和群落结构进行解析,以期为后续酱豆的工业化生产提供数据和理论支持。
1 材料与方法
1.1 材料与试剂
1.1.1 材料
采集自湖北省恩施土家族苗族自治州利川市集市上10个不同摊位的酱豆样品,样品编号依次为LCJM1、LCJM2、LCJM3、LCJM4、LCJM5、LCJM6、LCJM7、LCJM8、LCJM9和LCJM10。
1.1.2 试剂
QIAGEN DNeasy maricon Food Kit脱氧核糖核酸(deoxyribonucleic acid,DNA)基因组提取试剂盒:德国QIAGEN公司;FastPfuFlyDNAPolymerase、5×TransStartTM、脱氧核糖核苷三磷酸(deoxy-ribonucleoside triphosphate,dNTPs)Mix:北京全式金生物技术有限公司;111860琼脂糖:香港基因有限公司;其他试剂均为国产分析纯。
1.2 仪器与设备
2100芯片生物分析仪:美国Agilent公司;Vetiri梯度基因扩增仪:美国AB公司;5810R台式高速冷冻离心机:德国Eppendorf公司;UVPCDS8000凝胶成像分析系统:美国BIO-RAD公司;ND-2000C微量紫外分光光度计:美国Nano Drop公司;DYY-12电泳仪:北京六一仪器厂;MiSeq高通量测序平台:美国Illumina公司。
1.3 试验方法
1.3.1 利川酱豆样品基因组的提取
使用QIAGEN DNeasy maricon Food Kit DNA基因组提取试剂盒提取利川酱豆样品的基因组DNA,并用1%琼脂糖凝胶电泳进行检测,将符合要求的DNA条带置于-20 ℃保存备用。
1.3.2 PCR扩增及Illumina MiSeq高通量测序
以基因组DNA为模板,参考王丹丹等[21]的方法进行真菌18S核糖体核糖核酸(ribosomal ribonucleic acid,rRNA)PCR扩增及Illumina MiSeq高通量测序。PCR扩增体系:5×PCR缓冲液4 μL,dNTP Mix 2 μL,正向引物SSU0817F(5'-TTAGCATGGAATAATRRAATAGGA-3',加入7个核苷酸通用标签)0.8 μL,反向引物SSU1196R(5'-TCTGGACCTGGTGAGTTTCC-3')0.8 μL,rTaq酶0.4 μL,DNA模板10 ng,双蒸水(ddH2O)补至20 μL。PCR扩增程序:95 ℃预变性3min;95 ℃变性30 s,55 ℃退火30 s,72 ℃延伸45 s,共30次循环;72 ℃再延伸10 min。PCR扩增结束后用2%琼脂糖凝胶电泳检测PCR扩增产物,检测合格后委托上海美吉生物医药科技有限公司进行Illumina MiSeq高通量测序。
1.3.3 利川酱豆样品生物信息学分析
将测序后的序列拼接后,进一步按照WANG Y Y等[22]的方法进行质控后筛选出优化序列。使用QIIME(v1.7.0)对样品进行生物物种分析[23];使用软件PyNAST[24]对齐序列后,用软件UCLUST[25]以97%相似性划分操作分类单元(operational taxonomic units,OTU);使用SILVA数据库[26]在每个OTU中挑选一个代表性序列进行样品的真菌分类信息分析。
1.3.4 多元统计学分析
使用R(v4.0.3)软件绘制瀑布图及相关性热图;使用软件Origin 8.5绘制OTU出现次数统计图、稀释曲线图、香农指数曲线图和柱状堆积图;使用Excel 2019绘制饼图。
2 结果与分析
2.1 利川酱豆样品真菌菌群多样性分析
2.1.1 利川酱豆样品真菌菌群测序结果分析
对利川酱豆样品进行高通量测序后,由测序结果可知,10个利川酱豆样品经序列比对后成功得到365 351条序列,其中样品LCJM1、LCJM2、LCJM3、LCJM4、LCJM5、LCJM6、LCJM7、LCJM8、LCJM9和LCJM10的序列总数分别为33 171条、40 474条、38 331条、44 969条、37 239条、33 051条、34 578条、36 619条、30 287条和36 632条。为评估样品的测序量是否足够,绘制利川酱豆样品的稀释曲线和香农指数曲线,结果见图1。
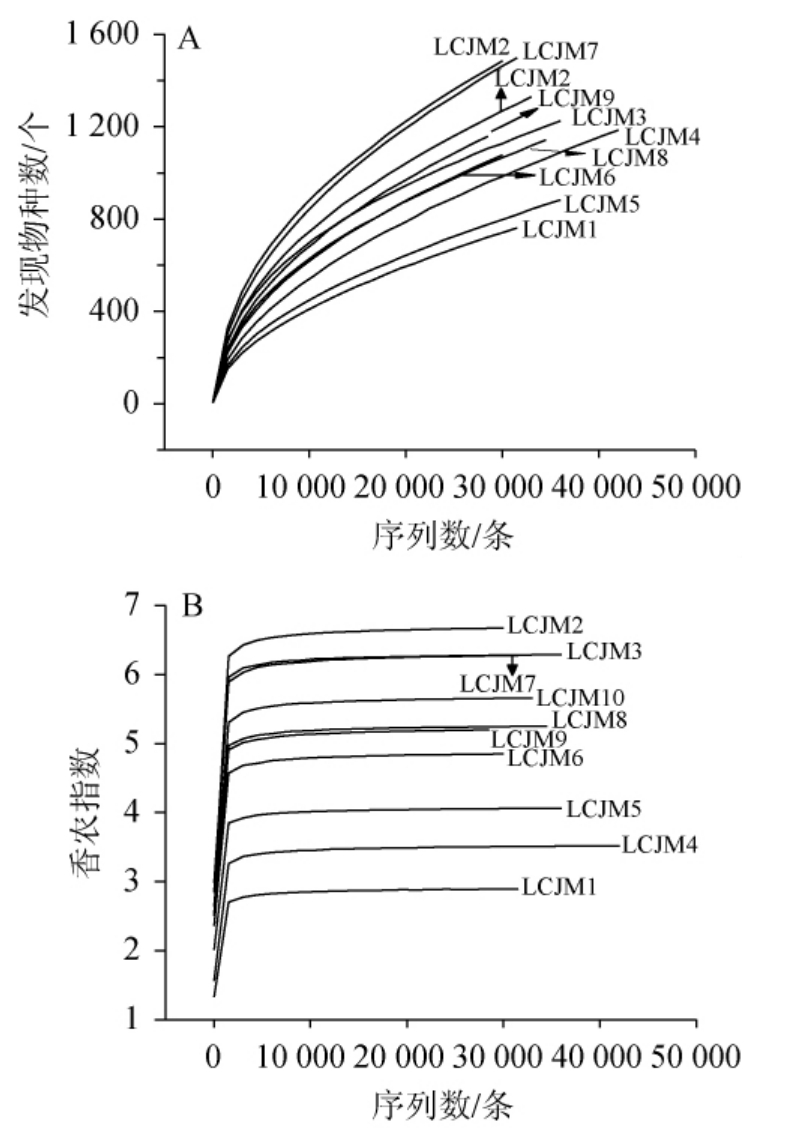
图1 利川酱豆样品的稀释曲线(A)和香农指数曲线(B)
Fig.1 Dilution curves (A) and Shannon index curves (B) of Lichuan Jiangdou samples
由图1A可知,10个利川酱豆样品中发现物种数随着序列数的增加而增加,说明新的种系型可能会随着序列数的增加而被发现。由图1B可知,10个利川酱豆样品的香农指数随着序列数的增加逐渐趋于平缓,说明随着序列数的增加样品中的多样性不会再有变化,样品的测序深度已足够。由此可知,样品的测序量能够满足要求,可进一步进行生物信息学分析。
2.1.2 利川酱豆样品真菌OTU分析
将酱豆样品中相对含量≥1%的OTU定义为核心OTU。OTU数量可反应出样品中微生物的丰度[27],本研究对样品OTU水平上的真菌微生物进行多样性分析,结果见图2。
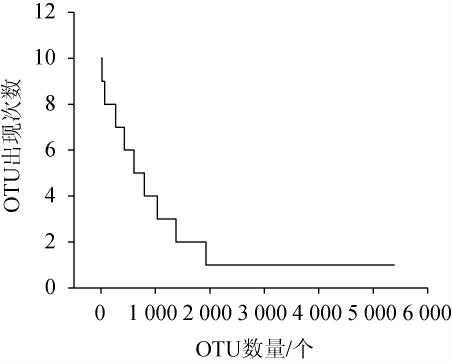
图2 利川酱豆样品中OTU出现次数统计结果
Fig.2 Statistical result of OTU occurrence times in Lichuan Jiangdou samples
由图2可知,10个利川酱豆样品中共产生5 391个真菌OTU,其中有18个OTU在10个样品中均出现,占OTU总数的0.33%,但这些OTU中所包含的序列条数为210 186条,占测序序列总数的57.53%;而利川酱豆样品中仅出现1次的OTU有3 466个,占OTU总数的64.29%,但其包含的序列数为4 238条,仅占序列总数的1.16%。由此说明不同的利川酱豆样品中有大量的共有真菌群落,尽管不同的酱豆样品拥有大量的独有菌群,但这部分微生物所占比例很低。为研究利川酱豆样品中核心真菌群落组成及各自所占比例,本研究进一步对核心OTU的构成和相对含量进行了解析,其分析结果见图3。
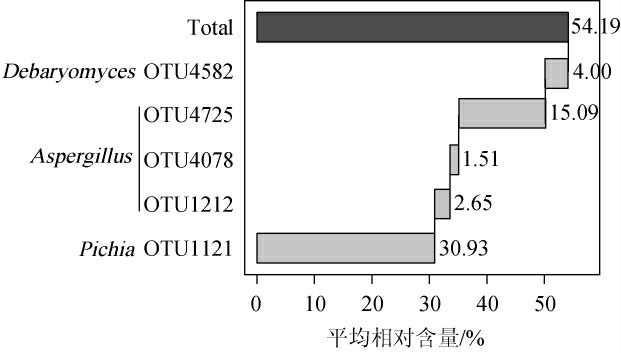
图3 核心OTU的构成及相对含量分析结果
Fig.3 Result of composition and relative content analysis of core OTU
由图3可知,在10个利川酱豆样品中的核心OTU共有5个,这些OTU占OTU总数的0.09%,但其所包含的序列数占总序列数的54.19%,分别为OTU4582(4.00%)、OTU4725(15.09%)、OTU4078(1.51%)、OTU1212(2.65%)和OTU1121(30.93%)。其中OTU1121隶属于毕赤酵母属(Pichia),OTU4582隶属于德巴利氏酵母属(Debaryomyces),OTU4078、OTU4725和OTU1212隶属于曲霉属(Aspergillus)。由此可见,Pichia、Debaryomyces和Aspergillus为利川酱豆中的优势真菌。
2.2 利川酱豆样品真菌菌群结构分析
由利川酱豆样品的物种注释结果分析发现,所有样品中的真菌被鉴定为3个门、15个纲、27个目、34个科、45个属。本研究将样品中相对含量<1.0%的细菌门或属归为其他类(others),平均相对含量≥1.0%的细菌门或属定义为核心细菌门或属。基于门水平对利川酱豆样品中的真菌菌群结构进行分析,结果见图4。
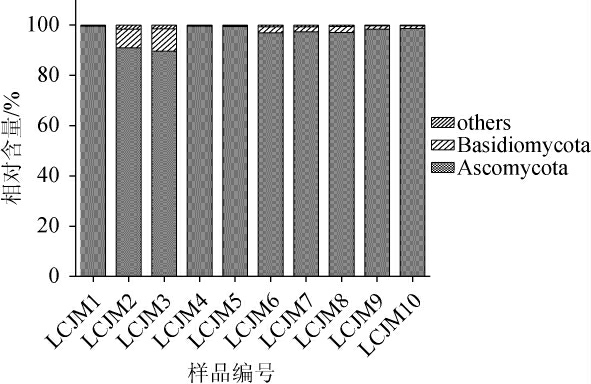
图4 基于门水平利川酱豆样品真菌菌群结构分析结果
Fig.4 Analysis results of fungal flora structure of Lichuan Jiangdou samples based on phylum level
由图4可知,10个利川酱豆样品中被鉴定出的真菌门有子囊菌门(Ascomycota)、担子菌门(Basidiomycota)和毛霉亚门(Mucoromycotina),其平均相对含量分别为96.79%、2.63%和0.02%,其中Ascomycota和Basidiomycota为核心真菌门(平均相对含量≥1%),毛霉亚门(Mucoromycotina)只在样品LCJM1、LCJM3、LCJM5和LCJM6中存在,且平均相对含量低,分别为0.006%、0.02%、0.16%和0.16%。进一步基于属水平对利川酱豆样品中的真菌菌群结构进行分析,结果见图5。
由图5可知,10个利川酱豆样品中的核心真菌属为毕赤酵母属(Pichia)、曲霉属(Aspergillus)、德巴利氏酵母属(Debaryomyces)、假丝酵母属(Candida)、Starmerella、节担菌属(Wallemia)和镰刀菌属(Fusarium),其平均相对含量分别为37.46%、29.48%、3.92%、2.86%、2.62%、2.19%和1.03%。通过进一步分析核心真菌属在不同样品中的分布,发现Starmerella未在样品LCMJ4中鉴定出,Wallemia未在样品LCMJ5中鉴定出,而Pichia、Aspergillus、Debaryomyces、Candida和Fusarium在10个利川酱豆样品中均存在。除此之外,还有15.72%的真菌序列未鉴定到属水平,因此,利川酱豆样品中的真菌资源仍有待发掘。
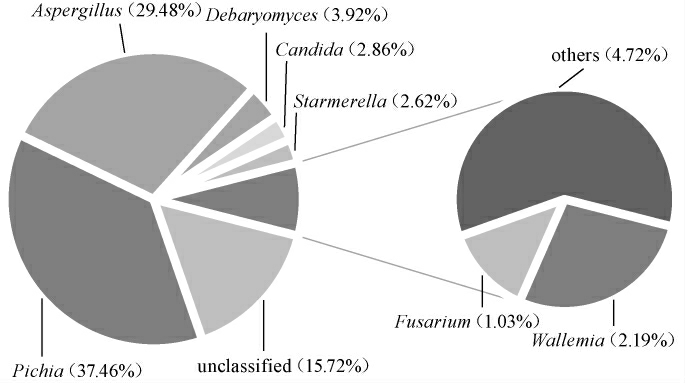
图5 基于属水平利川酱豆样品真菌菌群结构分析结果
Fig.5 Analysis results of fungal flora structure of Lichuan Jiangdou samples based on genus level
毕赤酵母属(Pichia)作为利川酱豆样品中的优势真菌属,研究发现,其为非酿酒酵母且在自然界中分布较为广泛,因其独特的生理特性,可作为一种拮抗微生物而应用到生物防治中[28];德巴利氏酵母属(Debaryomyces)能够通过代谢乳糖、蛋白质和乳酸盐而产生香味,同时也会产生一些有助于人体消化的小分子物质[29];曲霉属(Aspergillus)作为利川酱豆的优势真菌属,是一类丝状真菌,在自然界中分布广泛,其代谢的产物(多糖类、蒽醌类和生物碱类等)具有抗病毒、抗菌、抗氧化和抗癌等功能,且其广泛应用于发酵食品中,如酱油、醋等[30-31];由于利川酱豆发酵的开放环境,导致了酱豆在发酵过程中微生物的多样性,因此在发酵过程中也会产生一些不利于人体健康的真菌属,如镰刀菌属(Fusarium),该菌属可侵染植物造成植物的腐烂从而导致减产,有些镰刀菌属(Fusarium)可产生真菌毒素,食用后会造成食物中毒[32]。
2.3 核心真菌群落间的相关性分析
微生物彼此间相关关系的解析对于酱豆制品后续的标准化生产和品质控制必不可少,为进一步确定利川酱豆样品中核心真菌群落之间的相关性,本研究对酱豆样品的核心真菌属间及核心OTU间的相关性进行了分析,结果见图6。
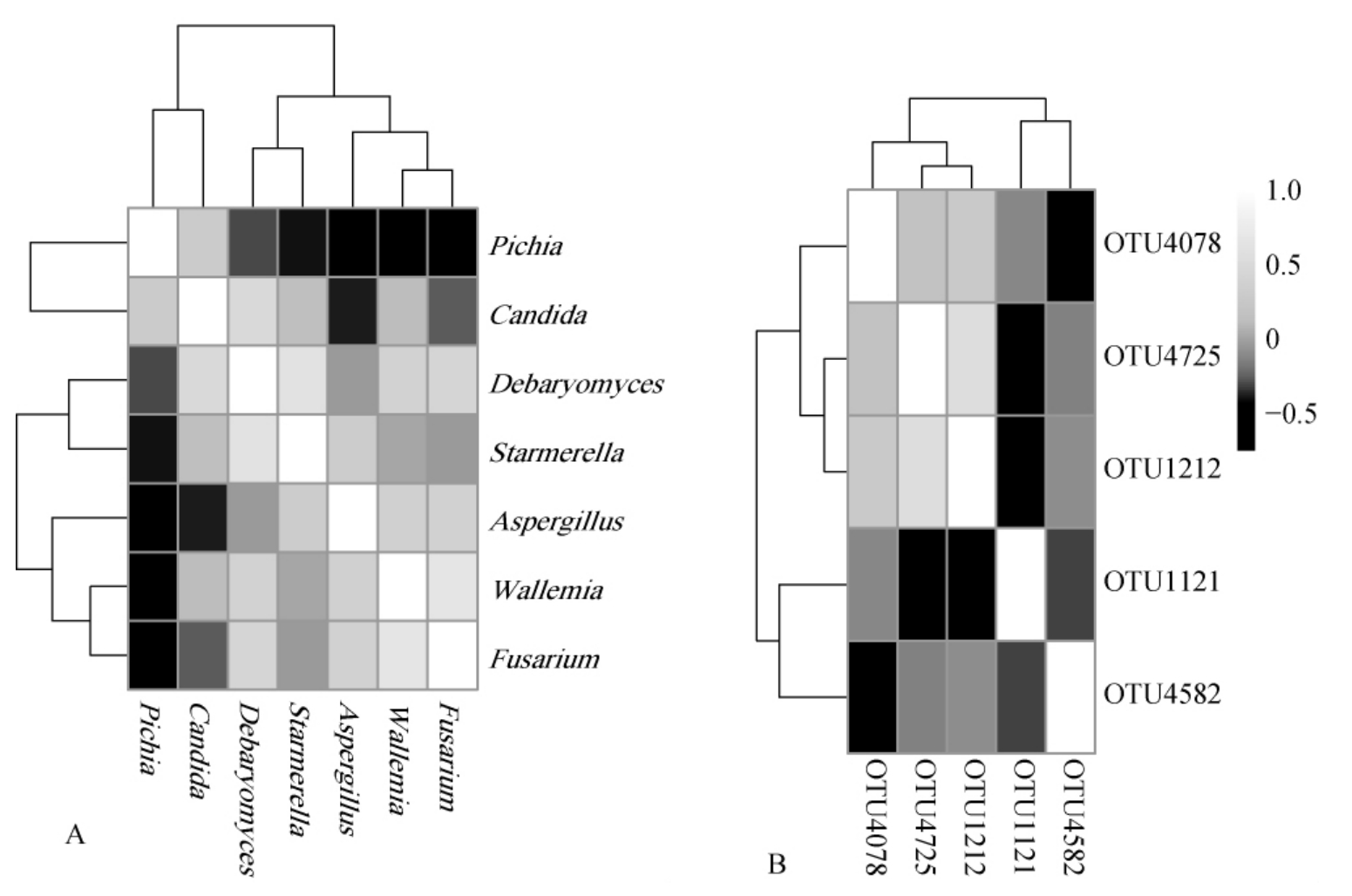
图6 核真菌属间(A)和核心OTU间(B)相关性分析结果
Fig.6 Results of correlation analysis between core fungi (A) and core OTUs (B)
由图6可知,在利川酱豆样品的核心真菌属之间,其相关性均不显著(P>0.05);核心OTU之间的相关性也均不显著(P>0.05)。由此可知,核心真菌群落间相关性不显著(P>0.05)。
3 结论
采用Illumina MiSeq高通量测序对利川酱豆样品真菌多样性进行解析,结果显示,利川酱豆样品中的真菌菌群隶属于3个门、15个纲、27个目、34个科、45个属,核心真菌门为子囊菌门(Ascomycota)(96.79%)和担子菌门(Basidiomycota)(2.63%);核心真菌属为毕赤酵母属(Pichia)(37.46%)、曲霉属(Aspergillus)(29.48%)、德巴利氏酵母属(Debaryomyces)(3.92%)、假丝酵母属(Candida)(2.86%)、Starmerella(2.62%)、节担菌属(Wallemia)(2.19%)和镰刀菌属(Fusarium)(1.03%),且核心真菌之间的相关性均不显著(P>0.05)。
[1]谭悦,阚建全,陈光静,等.发酵豆制品潜在风险因子分析[J].中国食品学报,2020,20(3):233-243.
[2]贾璠,郭霞,何晨,等.传统发酵豆制品营养功能成分研究进展[J].中国酿造,2019,38(4):1-6.
[3]张蒙冉,李淑英,高雅鑫,等.传统发酵豆制品研究进展[J].食品科技,2021,46(1):98-104.
[4]刘琪,张佩娜,陈静,等.发酵豆制品中安全风险因子的研究现状及对策[J].中国酿造,2018,37(2):1-5.
[5]李雪,陈雪,吴长庆,等.发酵豆制品的功能性及其机理研究现状[J].食品与发酵工业,2015,41(9):247-252.
[6]ZHAO X,JU J,PARK K Y.Comparisons of properties, in vitro anti-mutagenicity,and anti-cancer effects of short-term fermented soybean foods(Chungkukjang,Shuidouchi,and Natto)[J].J Korean Soc Food Sci Nutr,2017,46(10):1253-1257.
[7]JUNG J Y,LEE S H,JEON C O.Microbial community dynamics during fermentation of doenjang-meju,traditional Korean fermented soybean[J].Int J Food Microbiol,2014,185(1):112-120.
[8]张颖,乌日娜,孙慧君,等.豆酱不同发酵阶段细菌群落多样性及动态变化分析[J].食品科学,2017,38(14):30-35.
[9]安飞宇,姜静,武俊瑞,等.自然发酵豆酱的滋味特性与微生物多样性分析[J].中国食品学报,2020,20(7):207-215.
[10]HONG S W,CHOI Y,LEE H,et al.Microbial community structure of Korean cabbage kimchi and ingredients with denaturing gradient gel electrophoresis[J].J Microbiol Biotechn,2016,26(6):1057-1062.
[11]DI BELLA J M,BAO Y,GLOOR G B,et al.High throughput sequencing methods and analysis for microbiome research[J].J Microbiol Meth,2013,95(3):401-414.
[12]郝卓莉.遂宁榨菜发酵过程中细菌群落多样性和基因功能预测分析[J].中国酿造,2021,40(5):59-64.
[13]李奕萱,姜春明.高通量测序分析妊娠期糖尿病对早产儿肠道菌群的影响[J].中国微生态学杂志,2021,33(5):542-547.
[14]杨德仁,王卓,马蓉.应用高通量测序技术检测甲状腺乳头状癌相关基因变异[J].临床检验杂志,2020,38(12):891-893,913.
[15]陈菲菲,丛欣,向极钎,等.应用Illumina MiSeq高通量测序技术分析堇叶碎米荠根际土壤微生物多样性[J].湖北农业科学,2020,59(17):58-62,96.
[16]邱艳红,田文,徐秀兰.高通量测序技术在植物病毒检疫中的应用与发展[J].植物检疫,2020,34(3):1-6.
[17]赵馨馨,崔梦君,董蕴,等.应用Illumina MiSeq高通量测序技术分析巴东地区豆瓣酱中微生物多样性[J].现代食品科技,2019,35(9):297-303.
[18]李桂澜,匡华.高通量测序技术在食品微生物检测中的应用[J].食品安全质量检测学报,2019,10(15):5091-5097.
[19]肖剑.分子生物学技术在食品微生物检测中的应用[J].广西轻工业,2011,27(3):10-12.
[20]高世南,徐志华,李杨,等.新疆新源县酸马奶细菌菌群分析[J].中国酿造,2021,40(5):65-69.
[21]王丹丹,沈馨,董蕴,等.孝感凤窝酒曲真菌多样性评价[J].中国酿造,2017,36(11):38-42.
[22]WANG Y Y,SHE M N,GUO Z,et al.Determination of fungal diversity of acidic gruel by using culture-dependent and independent methods[J].Food Sci Nutr,2020,8(11):5832-5840.
[23]CAPORASO J,KUCZYNASKI J,STOMBAUGH J,et al.QIIME allows integration and analysis of high-throughput community sequencing data[J].Nat Meth,2010,7(5):335-336.
[24]CAPORASO J,BITTINGER K,BUSHMAN F D,et al.PyNAST:a flexible tool for aligning sequences to a template alignment[J].Bioinformatics,2010,26(2):266-267.
[25]EDGAR R C.Search and clustering orders of magnitude faster than BLAST[J].Bioinformatics,2010,26(19):2460-2461.
[26]FERNANDEZ D,SANTOS P,AGOSTINI C,et al.Coffee(Coffea arabica L.) genes early expressed during infection by the rust fungus (Hemileia vastatrix)[J].Mol Plant Pathol,2010,5(6):527-536.
[27]QIU Z,LI N,LU X,et al.Characterization of microbial community structure and metabolic potential using Illumina MiSeq platform during the black garlic processing[J].Food Res Int,2018,106(1):428-438.
[28]马小莉,王晓东.响应面法优化异常毕赤酵母(Pichia anomala)0732-1抑菌产物发酵条件[J].中国酿造,2018,37(3):57-60.
[29]王新惠,潘攀,孙劲松,等.基于氮阻遏效应分析汉逊德巴利酵母组胺调控[J].食品科学,2022,43(2):93-99.
[30]胡倩.两株曲霉属对虾肠道真菌次级代谢产物的研究[D].杭州:浙江大学,2017.
[31]江北,吕梦霞,蒋冬花.曲霉属真菌活性代谢产物及在农业生产中的应用研究进展[J].微生物学杂志,2019,39(2):103-110.
[32]张道宏,李培武,张奇,等.污染粮油食品的主要真菌毒素及胶体金免疫层析技术在快速检测中的应用[J].中国油料作物学报,2010,32(4):577.