中国白酒酿造具有悠久历史,山西主产清香型白酒,其白酒产业已成为山西省酿造业的支柱产业[1-2]。据酿造产业协会报道,仅山西年产白酒近700万t,按生产1 t的白酒产生3倍质量的酒糟[3],副产物酒糟达2 000万t以上。酒糟中含有蛋白质、氨基酸、脂肪、纤维素、钙、磷、维生素等营养物质[4],其中粗蛋白17.15%、粗纤维素37.91%、淀粉12.35%[5-7]。过去利用酒糟主要作为动物饲料辅料[8-10],利用价值较低,随着节约型、生态型经济发展,副产物酒糟高值化利用已迫在眉睫[11-12]。
目前,国内外对酒糟的研究多集中在探讨酒糟生产蛋白饲料的微生物发酵工艺方面[13-16],如白酒酒糟培养基中营养成分的组成及配比研究、发酵菌种及菌种组合筛选研究等,而对白酒酒糟本身的微生物菌群结构分析研究甚少。Illumina MiSeq高通量测序技术分析微生物多样性具有速度快、通量高、更具全面性的优势[17-20],已被广泛应用于土壤、水体及食品等微生物菌群多样性研究[21-22]。黎江华等[23]采用高通量测序技术分析五倍子发酵用酒曲与酒糟微生物多样性发现,浓香型白酒酒糟中共检测出56个细菌属和28个真菌属;袁帅[24]以酱香型白酒酒糟为研究对象,检测出酒糟样本中细菌主要分属11个门。但是目前针对清香型白酒酒糟中微生物菌群结构解析的研究鲜见报道。
因此,本研究通过Illumina MiSeq高通量测序技术对清香型白酒酒糟中微生物菌群结构进行解析,以期为后续建立清香型白酒酒糟的微生物体系指标以及进一步优化其生产蛋白饲料等奠定基础,为目前广泛应用于白酒酒糟转化利用方面的微生物发酵技术提供理论和技术依据,为深入了解清香型白酒发酵过程中的微生物菌群结构奠定基础,对促进清香型白酒产业循环经济的发展具有重要意义。
1 材料与方法
1.1 材料与试剂
清香型堡子酒酒糟:山西堡子酒业有限公司;Qubit3.0脱氧核糖核酸(deoxyribonucleic acid,DNA)检测试剂盒:美国Life公司;E.Z.N.ATMMag-Bind Soil DNA Kit:美国OMEGA公司![]() 聚合酶链式反应(polymerase chain reaction,PCR)MasterMix、HieffNGSTMDNASelectionBeads:翌圣生物科技(上海)股份有限公司;其他试剂均为国产分析纯。
聚合酶链式反应(polymerase chain reaction,PCR)MasterMix、HieffNGSTMDNASelectionBeads:翌圣生物科技(上海)股份有限公司;其他试剂均为国产分析纯。
1.2 仪器与设备
ETC 811 PCR仪:北京东胜创新生物科技有限公司;![]() 荧光计:美国英杰生命技术有限公司;DYY-6C型电泳仪:北京市六一仪器厂;FR-1000凝胶成像系统:上海复日科技有限公司。
荧光计:美国英杰生命技术有限公司;DYY-6C型电泳仪:北京市六一仪器厂;FR-1000凝胶成像系统:上海复日科技有限公司。
1.3 方法
1.3.1 样品预处理
取清香型堡子酒酒糟堆(堆积2~3 d)上部、中部、下部新鲜酒糟并混合,参考沈才萍等[25]的方法用磷酸盐缓冲液(phosphate buffer saline,PBS)对酒糟样品进行预处理。
1.3.2 基因组DNA提取
采用E.Z.N.ATM Mag-Bind Soil DNA Kit提取酒糟中微生物的基因组DNA。
1.3.3 PCR扩增
以基因组DNA为模板,采用引物341F(5'-CCTACGGGNGGCWGCAG-3')/805R(5'-GACTACHVGGGTATCTAATCC-3')和ITS1F(5'CTTGGTCATTTAGAGGAAGTAA-3')/ITS2R(5'-GCTGCGTTCTTCATCGATGC-3')分别对细菌16S rDNA V3-V4区和真菌ITS1-ITS2区基因序列进行两轮PCR扩增[26]。
1.3.4 Illumina MiSeq高通量测序
PCR扩增结束后,用2%琼脂糖凝胶电泳检测文库,使用Qubit 3.0荧光定量仪进行文库浓度测定,然后将PCR扩增产物委托生工生物工程(上海)股份有限公司进行Illumina MiSeq高通量测序。
1.3.5 Illumina MiSeq高通量测序数据处理
高通量测序得到的原始序列是双端序列数据,且序列中含有起始加入的引物和接头序列以及barcode标签序列。使用cutadapt软件、PEAR软件、PRINSEQ软件等处理数据。首先需要对引物的接头序列进行去除,然后根据双端序列之间的相互重复关系,将所有成对的单个序列拼接成一条序列,再根据barcode标签序列区分酒糟样品中真菌及细菌的各序列数据,之后对各序列数据进行质量控制,最后得到各序列的有效数据。
1.3.6 Illumina MiSeq高通量测序数据分析
运用Usearch软件对非重复序列进行操作分类单元(operational taxonomic units,OTU)聚类分析,并对应相应的数据库进行分类学分析。对聚类分析结果进行Alpha多样性分析,使用R软件绘制清香型堡子酒酒糟中微生物的稀释性曲线、Rank abundance曲线、香农指数曲线等。
2 结果与分析
2.1 Illumina Miseq高通量测序数据统计结果与质量分析
采用Illumina Miseq高通量测序平台得到的清香型堡子酒酒糟样本原始序列与其碱基长度见表1。
表1 酒糟样本的高通量测序数据统计结果
Table 1 Statistics results of high-throughput sequence data of distiller's grains samples
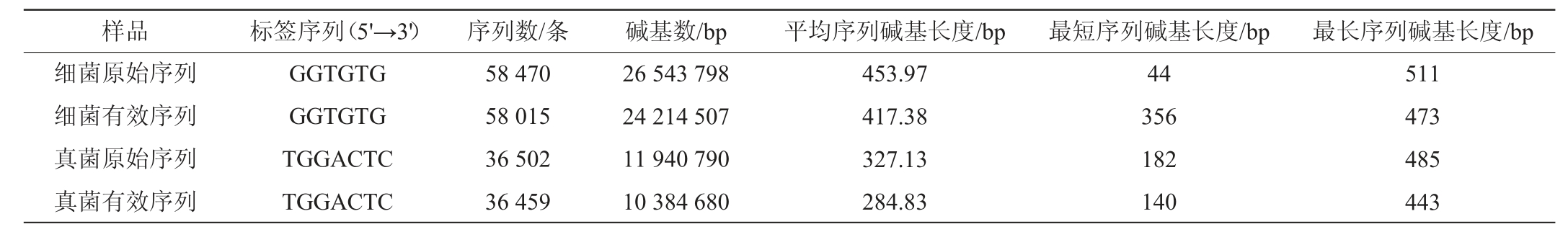
由表1可知,清香型堡子酒酒糟样品中得到的细菌原始序列数为58 470条,平均序列碱基长度为453.97 bp;经拼接、质控过滤后,细菌有效序列数为58 015条,平均序列碱基长度为417.38 bp。酒糟样品中得到的真菌原始序列数为36 502条,平均序列碱基长度为327.13 bp;经拼接、质控过滤后,真菌有效序列数为36 459条,平均序列碱基长度为284.83 bp。细菌与真菌的有效序列占比均达到99%以上,这表明本次高通量测序得到的有效序列可以满足后续对清香型堡子酒酒糟样品中微生物菌群结构的研究。
随机抽取一定数量的序列,与他们所代表的物种数目构建稀释性曲线,结果见图1。
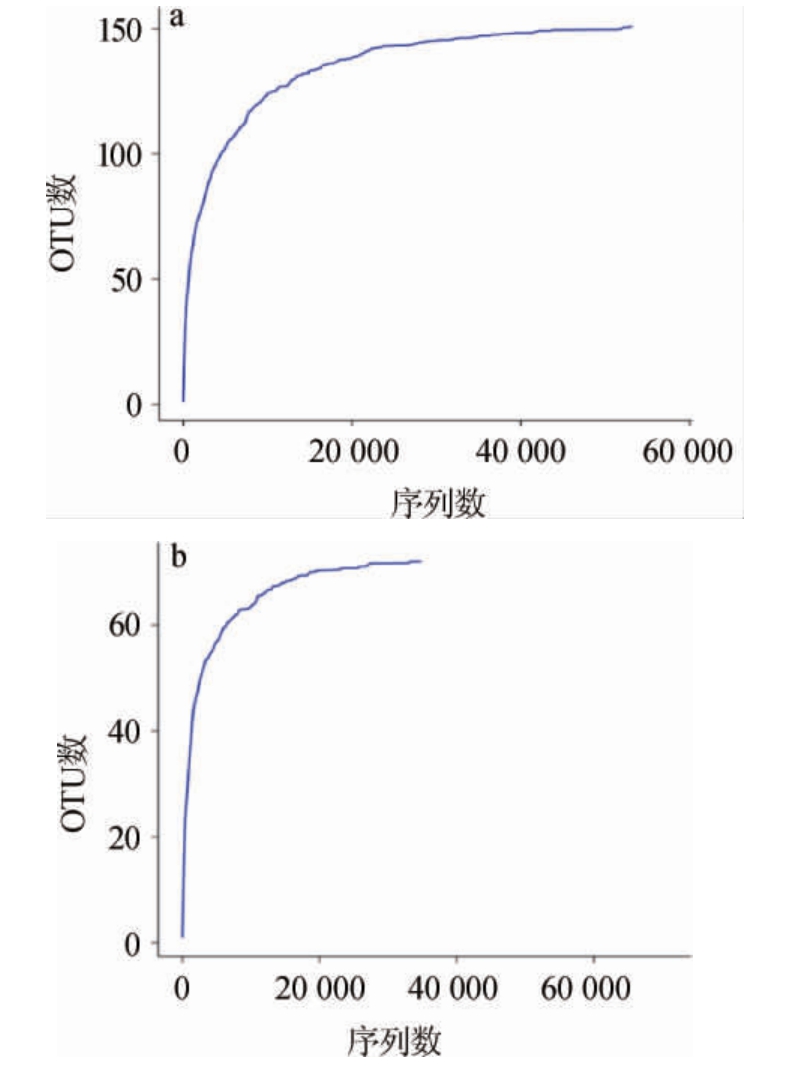
图1 酒糟样品中细菌菌群(a)及真菌菌群(b)的稀释性曲线
Fig.1 Rarefaction curves of bacteria flora (a) and fungi flora (b) in distiller's grains samples
由图1 可知,当测序序列数>20 000时,曲线趋向平坦,说明即使增加测序数据,OTU数也不会再增加,说明本次的测序量合理,能够覆盖酒糟样本中的绝大部分物种,数据量合理且足够。
Rank-abundance曲线是分析多样性的一种方式,用于反应样品多样性的两个方面,即样品所含物种的丰富程度和均匀程度[27]。酒糟样品中细菌菌群及真菌菌群的Rankabundance曲线见图2。由图2可知,酒糟样品中细菌和真菌菌群的丰富程度和均匀程度存在差异,其中细菌菌群的Rank-abundance曲线相对于真菌菌群较平缓,水平跨度较大,所以细菌菌群的物种组成较丰富,均匀程度较高;真菌菌群的Rank-abundance曲线陡峭,水平跨度小,微生物丰度较低,某一类或几类微生物占了大部分比例,具有数量优势。
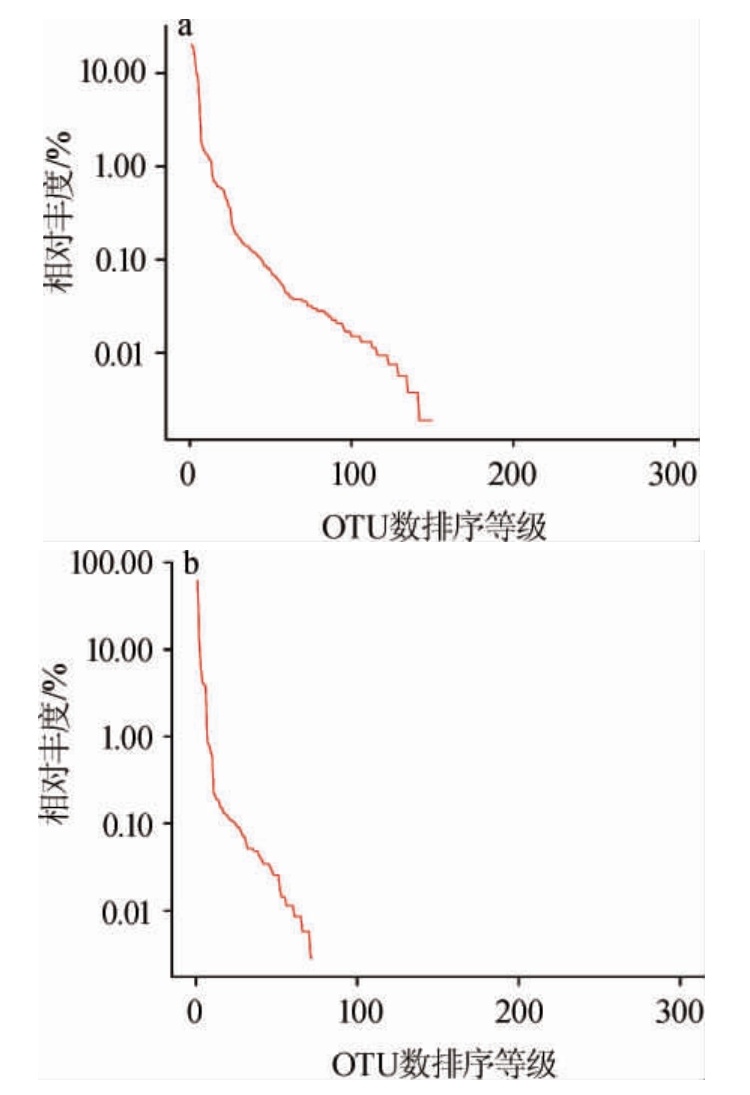
图2 酒糟样品中细菌(a)及真菌(b)的Rank-abundance曲线
Fig.2 Rank-abundance curves of bacteria (a) and fungi (b) in distiller's grains samples
2.2 酒糟样品微生物菌群的Alpha多样性分析
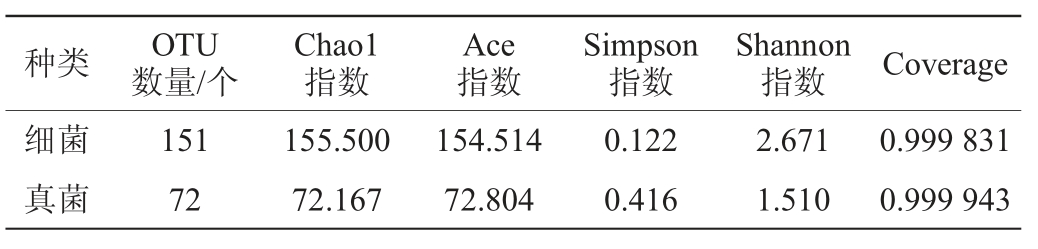
Alpha多样性反映微生物群落的丰富度和多样性[24],酒糟样品微生物菌群的α多样性分析结果见表2。由表2可知,细菌菌群的超1(Chao1)指数、Ace指数均大于真菌菌群,说明酒糟样本内,细菌菌群的物种丰富度远高于真菌菌群[28]。酒糟样品中细菌菌群的香农(Shannon)指数略大于真菌菌群,而细菌菌群的辛普森(Simpson)指数小于真菌菌群,从而表明清香型堡子酒酒糟中的细菌菌群多样性略大于真菌菌群多样性。另外,两者的Coverage指数都接近1,说明对清香型堡子酒酒糟样品文库的覆盖率很高[29],进一步表明本实验测序结果能很真实有效地反映清香型堡子酒酒糟的微生物多样性,本研究有一定研究意义和研究价值。
表2 酒糟样品中微生物群落的α-多样性分析结果
Table 2 Alpha diversity analysis results of microbial community in distiller's grains samples
2.3 酒糟样品OTU物种统计
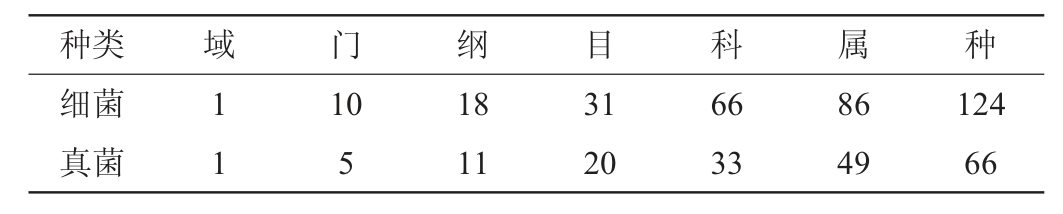
对酒糟样品微生物在各分类水平上的菌群结构进行分析,结果见表3。由表3可知,清香型堡子酒酒糟样品中的细菌菌群分布在10个门、18个纲、31个目、66个科、86个属、124种上;真菌菌群分布在5个门、11个纲、20个目、33个科、49个属、66个种上。细菌菌群在各分类水平的数量分布均高于真菌菌群,进一步说明清香型堡子酒酒糟中细菌菌群的多样性高于真菌菌群。另外,未测出酒糟样品中细菌菌群在种水平上的分类,分析原因可能是因为16S rDNA有很高的保守性,对亲缘关系较近的物种种属不容易分辨[30]。
表3 酒糟样品中微生物在各分类水平上的菌群结构
Table 3 Community structure of microbe in distiller's grains at different classification levels
2.4 酒糟样品中微生物菌群结构分析
2.4.1 基于门水平酒糟样品中细菌菌群结构分析
基于门水平清香型堡子酒酒糟样品中的细菌菌群结构见图3。由图3可知,清香型堡子酒酒糟样品中有4个优势细菌门(相对丰度≥1%),分别为变形菌门(Proteobacteria)(66.51%)、放线菌门(Actinobacteria)(26.21%)、厚壁菌门(Firmicutes)(4.40%)、拟杆菌门(Bacteroidetes)(1.67%)。
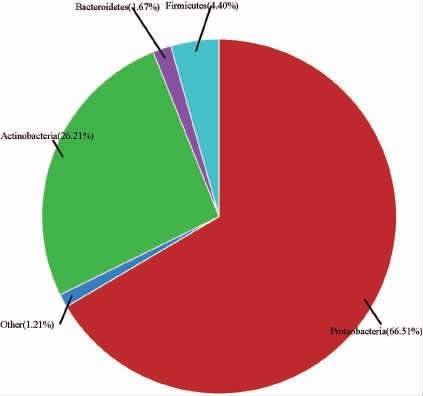
图3 基于门水平酒糟样品中的细菌菌群结构分析结果
Fig.3 Bacterial community structure analysis results of distiller's grain samples based on phylum level
Other表示相对丰度<1%的所有菌门。下同。
2.4.2 基于门水平酒糟样品中真菌菌群结构分析
基于门水平清香型堡子酒酒糟样品中的真菌菌群结构见图4。由图4可知,清香型堡子酒酒糟样品中有3个优势真菌门,分别为子囊菌门(Ascomycota)(85.04%)、毛霉门(Mucoromycota)(11.45%)及担子菌门(Basidiomycota)(2.77%),其中子囊菌门(Ascomycota)具有绝对优势。
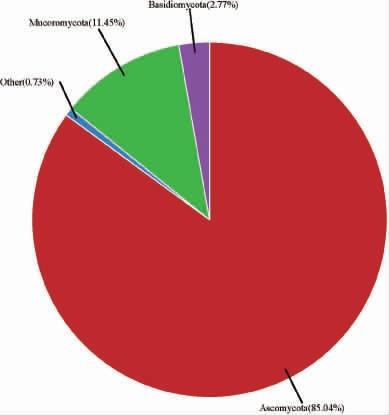
图4 基于门水平酒糟样品中的真菌菌群结构分析结果
Fig.4 Fungal community structure analysis results of distiller's grain samples based on phylum level
综上,在门水平上,清香型堡子酒酒糟中细菌菌群较真菌菌群更为丰富,细菌群落中各菌门的相对丰度差异较小,真菌群落中相对丰度差异较大,主要集中在子囊菌门内。
2.4.3 基于属水平酒糟样品中细菌菌群结构分析
基于属水平清香型堡子酒酒糟样品中的细菌菌群结构见图5。由图5可知,清香型堡子酒酒糟样品中有12个优势细菌属,分别为根瘤菌属(Rhizobium)(20.43%)、拉乌尔菌属(Raoultella)(18.96%)、谷氨酸杆菌属(Glutamicibacter)(15.00%)、红球菌属(Rhodococcus)(9.07%)、短波单胞菌属(Brevundimonas)(4.42%)、不动杆菌属(Acinetobacter)(2.52%)、假单孢菌属(Pseudomonas)(2.44%)、嗜冷杆菌属(Psychrobacter)(1.58%)、Devosia(1.57%)、嗜盐单胞菌属(Halomonas)(1.19%)、未分类-鼠杆菌属(unclassified-Murib-aculaceae)(1.15%)、乳杆菌属(Lactobacillus)(1.14%)。

图5 基于属水平酒糟样品中的细菌菌群结构分析结果
Fig.5 Bacterial community structure analysis results of distiller's grain samples based on genera level
Other表示相对丰度<1%的所有菌属。下同。
2.4.4 基于属水平酒糟样品中真菌菌群结构分析
基于属水平清香型堡子酒酒糟样品中的真菌菌群结构见图6。由图6可知,清香型堡子酒酒糟样品中有6个优势真菌属,分别为孢圆酵母属(Torulaspora)(62.59%)、伊萨酵母属(Issatchenkia)(12.79%)、横梗霉属(Lichtheimia)(7.08%)、根霉属(Rhizopus)(4.29%)、曲霉属(Aspergillus)(4.06%)、酵母属(Saccharomyces)(3.78%)。
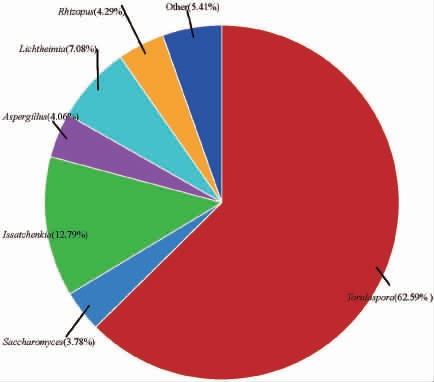
图6 基于属水平酒糟样品中的真菌菌群结构分析结果
Fig.6 Fungal community structure analysis results of distiller's grain samples based on genera level
黎江华等[23]对浓香型酒糟微生物多样性进行了研究,检测出浓香型白酒糟菌群优势菌属主要包括魏斯氏菌属(Weissella)(11%)、未定义蓝细菌(unidentified Cyanobacteria)(8%)等17个,而优势真菌属主要包括丝孢酵母属(Trichosporon)(34%)、伊萨酵母属(Issatchenkia)(9%)等11个,与本文中检测的细菌属、真菌属在丰度以及属别上有较大差异,这可能与两种酒型在发酵用曲、酿造工艺等不同有关。
3 结论
本研究采用Illumina MiSeq高通量测序技术对清香型堡子酒酒糟微生物菌群多样性进行分析,得出酒糟中的细菌丰富度和多样性均高于真菌,细菌菌群归属于10个门、18个纲、31个目、66个科、86个属、124个种,真菌菌群归属于5个门、11个纲、20个目、33个科、49个属、66个种。优势细菌门(相对丰度≥1.0%)为变形菌门(Proteobacteria)(66.51%)、放线菌门(Actinobacteria)(26.21%)、厚壁菌门(Firmicutes)(4.40%)、拟杆菌门(Bacteroidetes)(1.67%),优势细菌属为根瘤菌属(Rhizobium)(20.43%)、拉乌尔菌属(Raoultella)(18.96%)、谷氨酸杆菌属(Glutamicibacter)(15.00%)、红球菌属(Rhodococcus)(9.07%)、短波单胞菌属(Brevundimonas)(4.42%)、不动杆菌属(Acinetobacter)(2.52%)、假单孢菌属(Pseudomonas)(2.44%)、嗜冷杆菌属(Psychrobacter)(1.58%)、Devosia(1.57%)、嗜盐单胞菌属(Halomonas)(1.19%)、未分类-鼠杆菌属(unclassified-Muribaculaceae)(1.15%)、乳杆菌属(Lactobacillus)(1.14%);优势真菌门为子囊菌门(Ascomycota)(85.04%)、毛霉门(Mucoromycota)(11.45%)及担子菌门(Basidiomycota)(2.77%),优势真菌属为孢圆酵母属(Torulaspora)(62.59%)、伊萨酵母属(Issatchenkia)(12.79%)、横梗霉属(Lichtheimia)(7.08%)、根霉属(Rhizopus)(4.29%)、曲霉属(Aspergillus)(4.06%)、酵母属(Saccharomyces)(3.78%)。本研究为继续深入研究酒糟的发酵转化肥料及为动物饲料开发等提供了理论参考,同时,也为白酒酿造过程中菌群结构变化规律解析提供了理论依据。
[1]杨帆,周兴鹏,张敬铖,等.混蒸混烧生产清香型白酒工艺[J].食品研究与开发,2018,39(9):76-81.
[2]木喜叶勒.白酒功能菌对清香型白酒品质的影响研究[D].呼和浩特:内蒙古农业大学,2020.
[3]高铭坤,温广宇,钱芳.微生物对白酒酒糟的转化利用研究进展[J].食品与机械,2018,34(2):191-194.
[4]周小兵.白酒酒糟发酵生产丁二酸[D].无锡:江南大学,2013.
[5]聂永增.对于酒糟综合利用现状及创新应用的思考[J].轻工科技,2021,37(9):11-12.
[6]程海青.汾酒糟的现状与可持续发展浅探[J].现代畜牧科技,2021(9):38-39.
[7]张丹丹,程景,靳光,等.酒糟和醋糟营养成分差异及其瘤胃降解特性分析[J].中国饲料,2021(9):118-123.
[8]池永宽,熊康宁,宋淑珍,等.西南喀斯特地区白酒糟饲料化开发利用关键技术研究[J].中国饲料,2018(1):77-82.
[9]魏来.发酵白酒糟工艺优化及其对育肥猪生长性能的影响[D].武汉:武汉轻工大学,2015.
[10]赖玉城.产多酶体系混菌固态发酵酒糟生产蛋白饲料[D].福州:福建师范大学,2014.
[11]张丽华,王小媛,李昌文,等.酒糟再利用的研究进展[J].食品与发酵工业,2017,43(11):250-256.
[12]左上春,杨海泉,邹伟.白酒酒糟资源化利用研究进展[J].食品工业,2016,37(1):246-249.
[13]刘瑞娟.利用白酒酒糟发酵饲料的工艺优化[J].产业与科技论坛,2021,20(14):57-58.
[14]胡志强,李存福,张国顺,等.复合菌种协同发酵酒糟生产饲料蛋白分析[J].生物化工,2019,5(4):64-67.
[15]杨丽华,张婷婷,孟建宇.混菌固态发酵白酒酒糟制蛋白饲料的研究[J].中国饲料,2021(13):135-138.
[16]刘华南,汤超宏,梁士伟,等.微生物发酵白酒糟饲料及其在猪生产上的应用研究进展[J].湖南饲料,2021(1):43-48.
[17] SHOKRALLA S,SPALL J L,GIBSON J F,et al.Next-generation sequencing technologies for environmental DNA research[J].Mol Ecol,2012,21(8):1794-1805.
[18]王兴春,杨致荣,王敏,等.高通量测序技术及其应用[J].中国生物工程杂志,2012,32(1):109-114.
[19]杜贞娜,程斐,单之初,等.高通量测序技术及其在黄酒微生物多样性研究中的应用[J].中国酿造,2021,40(8):14-19.
[20]刘婷,肖仲久,李小霞,等.高通量测序技术分析茅台酿酒高粱根际细菌群落特征[J].中国酿造,2021,40(7):54-57.
[21]张宝宝.陕北石油污染土壤微生物多样性及降解菌特性研究[D].延安:延安大学,2021.
[22]万甜,何梦夏,任杰辉,等.渭河流域水体细菌群落的环境响应及生态功能预测[J].环境科学,2019,40(8):3588-3595.
[23]黎江华,刘超兰,冯兴,等.高通量测序分析五倍子发酵用酒曲与酒糟微生物多样性[J].中成药,2020,42(8):2113-2118.
[24]袁帅.用16S rDNA V4区高通量测序分析两种酒糟细菌多样性[D].贵阳:贵州师范大学,2014.
[25]沈才萍,李德林,沈才洪,等.酒醅微生物DNA提取预处理方法研究[J].四川理工学院学报(自然科学版),2013,26(3):16-20.
[26]张双虹,刘孟涛,陆婉瑶,等.Illumina Miseq高通量测序方法对袋装番茄酱微生物多样性分析[J].食品工业科技,2021,42(18):119-125.
[27]宋晓秋,徐亚杰,肖瀛,等.肉桂精油微胶囊对小鼠抗氧化活性与肠道菌群的影响[J].食品科学,2021,42(17):143-152.
[28]李斌,胡俊杰,张兰兰,等.基于高通量测序浓香型和芝麻香型白酒酒曲真菌群落结构的分析[J].中国酿造,2019,38(10):96-100.
[29]姜明慧,许玲,于文娟,等.高通量测序技术分析酱香型白酒下沙轮次堆积过程的微生物多样性[J].酿酒,2020,47(4):51-55,59.
[30]刘筱雪,袁文娟,丁涛,等.基于高通量测序对四川怀远特色发酵食品