四川盆地拥有适宜酿造的地域性环境,盛产各型宜人风格的浓香型白酒[1]。白酒的主要成分为乙醇和水,含量占据整个酒体的98%,余下1%~2%的微量成分,包含呈香呈味的酸类、酯类、醇醛等小分子物质,是决定白酒独特风格与品质的骨架成分[2]。浓香型白酒以窖池为其独特的发酵容器,窖池的每轮次发酵周期在90 d左右。长发酵期使窖池形成密闭的厌氧环境,其中栖息的微生物尤其是细菌类微生物对白酒骨架香味成分的积累、白酒的风格或质量的形成具有明显的决定作用[3]。俗话说“好窖出好酒”,优质窖泥中的微生物,经历了酿造工艺的长期筛选与驯化,形成了有益于酿造的独特群落体系,从而塑造了浓香型白酒以芳香浓郁、绵柔甘洌为主的典型风格。窖池微生态中微生物群落结构及丰度的差异,是不同窖池所酿白酒具有质量差异的主要因素[4]。以现代化微生物可培养或非培养[5]的各类技术实现窖泥酿造微生态体系的定向改良,使浓香型白酒发酵质量可控,是传统白酒产业走向现代化、机械化、自动化的核心问题,前提条件是解剖并认识成熟窖泥的微生物群落结构,辨识其中的酿造关键微生物并掌握其培养方式[5]。本研究基于高通量测序技术,比较了新窖泥及成熟窖泥中的细菌群落结构多样性,分析了细菌群落结构与酿造质量的相关性,为浓香型白酒酿造质量的提升提供了参考依据。
1 材料与方法
1.1 材料与试剂
供试窖泥样品(标记为LCX):来自于宜宾六尺巷酒业正常发酵4年且生产的原酒主体香不突出、诸香欠缺协调的新窖池;市内其他三家酒厂中正常发酵15年且生产的原酒相对风格典型、诸味协调的成熟老窖池,所选窖池执行同一套生产工艺,窖期均为90 d,取窖底泥作为样品,并依次标记为YRF、XJF、CXF;以无菌袋装样,4 ℃冷冻密封转运至实验室后,立即开展总脱氧核糖核酸(deoxyribonucleic acid,DNA)的提取。
AxyPrepDNA凝胶回收试剂盒:美国AXYGEN公司;DNA Marker D2000:北京天根生化科技有限公司;带识别条码(barcode)的细菌16S rRNA基因V3-V4区扩展引物338F(5'-ACTCCTACGGGAGGCAGCAG-3')和806R(5'-GGACTACHVGGTWTCTAAT-3'):北京诺禾致源生物信息科技有限公司。
1.2 仪器与设备
JXFSTPRP-32全自动样品快速研磨仪:上海净信实业发展有限公司;Velocity 18R冷冻高速离心机:Dynamica Scientific Ltd.;Tarzan 96梯度聚合酶链式反应(polymerase chain reaction,PCR)仪:莫纳生物科技有限公司;UV 8000紫外可见分光光度计:上海元析仪器有限公司;TanonEPS300凝胶电泳仪、Tanon 2500B凝胶成像系统:上海天能科技有限公司;AC2-6S1生物安全操作柜:新加坡艺思高科技有限公司。
1.3 方法
1.3.1 窖泥总DNA提取及聚合酶链式反应扩增
采用十六烷基三甲基溴化铵(hexadecyltrimethylammonium bromide,CTAB)溶解细胞膜的方法[6]提取窖泥总DNA。PCR扩增体系:Taq Mix 10 μL、模版1μL、正反向引物各0.5 μL,使用双蒸水(ddH2O)补足20 μL;PCR扩增条件:94 ℃预变性10 min;每个循环包含94 ℃变性40 s,58 ℃退火30 s,72 ℃延伸30 s,一共循环30次;循环结束后,72 ℃延伸10 min。使用2%琼脂糖凝胶对PCR产物进行检测。选择大小在500 bp 左右,符合16S rRNA V3-V4区扩增产物要求的清晰单一条带,切胶回收。对符合OD260nm值/OD280nm值=1.8~2.0、OD230nm值/OD260nm值>2.0条件的高浓度、高纯度胶条,送往擎科生物科技有限公司,借助Illumina Miseq平台及相关资源完成高通量测序,并返回原始序列(rawdata)。
1.3.2 生物信息学分析
为了确保分析数据的准确性,利用Mothur软件[7]对原始序列进行筛选,去掉低质量的序列,识别和消除嵌合体,分类并去除序列首尾两端的barcode,识别并剔除单次出现序列(singleton),得到过滤后的高质量拼接序列(clean tags)。利用USEARCH软件[8]依据序列相似性>97%的分类要求,将clean tags聚类成为可操作分类单元(operational taxonomic units,OTUs),并在聚类过程中去除一些测序错误的序列,提高分析的准确性。随后选取每个OTU中出现频数最高的完整序列作为该OTU的代表性序列,用于后续物种注释。利用QIIME2软件[9]中的assign_taxonomy.py脚本将每个OTU的代表序列与16S rRNA基因扩增子注释数据库Silva比对,获得OTUs的物种注释信息。再使用核糖体数据库项目(ribosomal database project,RDP)Classifier软件[10],设置置信度为0.5以上,校对并确认物种注释信息的分类学结果。分类学结果包括界、门、纲、目、科、属、种7个层级。将可注释的OTUs应用于物种多样性分析,以主成分分析(principal component analysis,PCA)法[11]将样品的群落差异最大化,找出对差异的形成具有主要奉献度且相对丰度>0.01%的优势OTUs。分析过程使用R语言完成作图[11]。根据共培养法和非培养法的微生物群落结构中同属的物种在代谢特征上具有相似性的发现[4],使用基本局部搜索工具(basic local alignment tool,BLAST)(https://blast.ncbi.nlm.nih.gov/Blast.cgi)检索与优势OTUs一致性(identity)高且正式发表的参考物种(reference species),用于系统发育多样性分析。
2 结果与分析
2.1 测序合理性
高通量测序后,各份样品获得的高质量clean tags,经聚类及与Silva数据库比对后,获取到能够注释的OTUs总数见表1。以稀释性曲线评估不同样本的测序合理程度,结果如图1所示。由表1、图1可知,当clean tags总数在10 000以上时,所有样本的曲线末端趋向平坦,表明各样本的测序数据量足够且合理,测序深度足以覆盖样品中存在的大多数微生物。同时,稀释性曲线表明,样品按物种丰富度从大到小排序依次是XJF、YRF、CXF、LCX。
表1 窖泥微生物群落多样性比较
Table 1 Comparison of the diversity of microbial community in pit mud
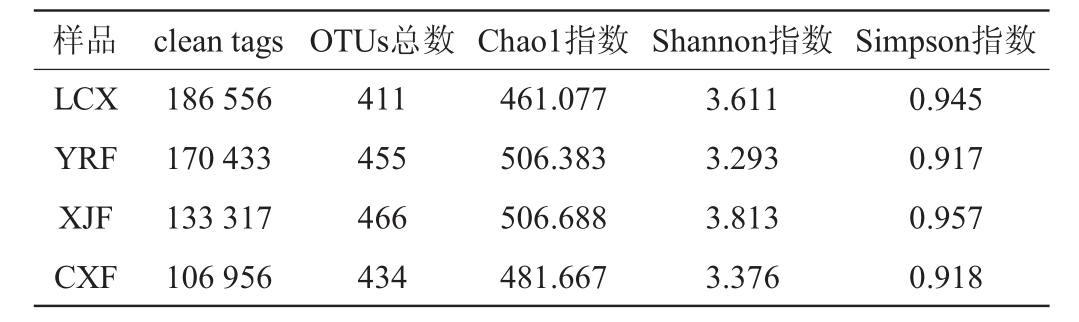
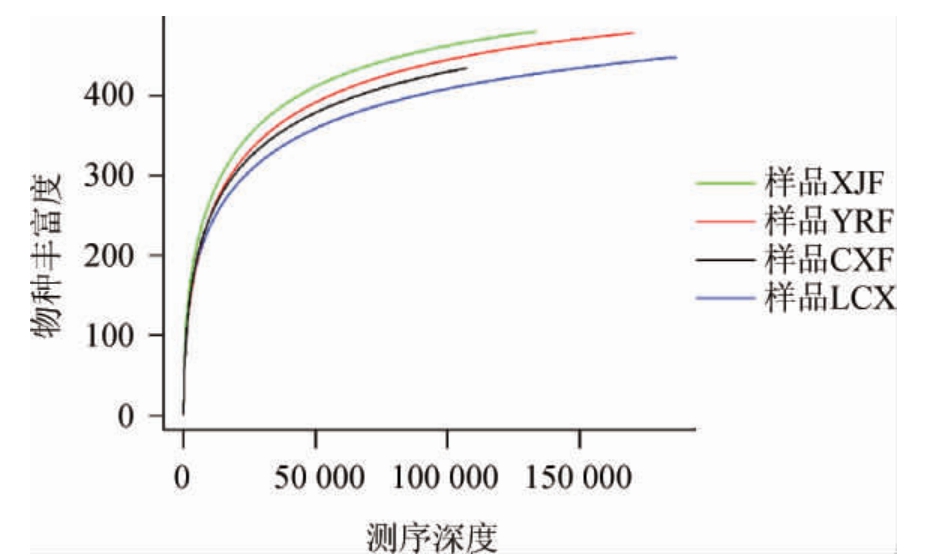
图1 窑泥样品稀释性曲线
Fig.1 Dilution curves of pit mud samples
以等级丰度曲线评估样品的多样性,结果如图2所示。
由图2可知,样品XJF的曲线在横轴上的跨度最长最平坦,表明样品XJF的物种含量相对最丰富,物种组成相对最均匀。以alpha多样性指数评估OTUs的多样性(表1),样品XJF与YRF的Chao1指数最大,反映了这两份样品拥有比其他样品更丰富的物种,Chao1指数结论与稀释性曲线结论一致;样品XJF的Shannon指数、Simpson指数均最大,反映了该份样品中物种的复杂度、物种的均匀性与丰度相对其他样品高,Simpson指数的结论与等极丰度曲线结论一致。而4份样品的Chao1、Shannon、Simpson指数均十分理想,表明高通量测序结果良好,可用于后续物种多样性分析。
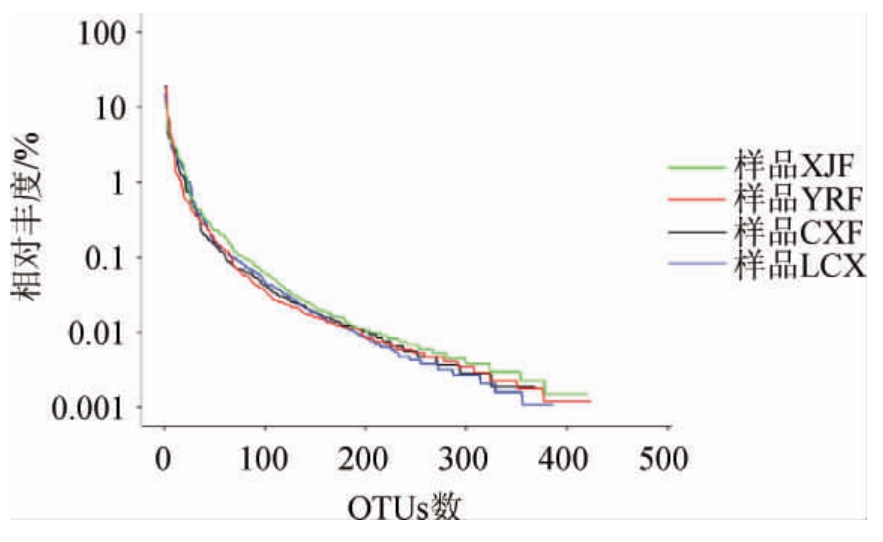
图2 窑泥样品的等级丰度曲线
Fig.2 Grade abundance curves of pit mud samples
2.2 优势微生物
以交集方式对4份样品的OTUs开展相关性分析,结果如图3所示。
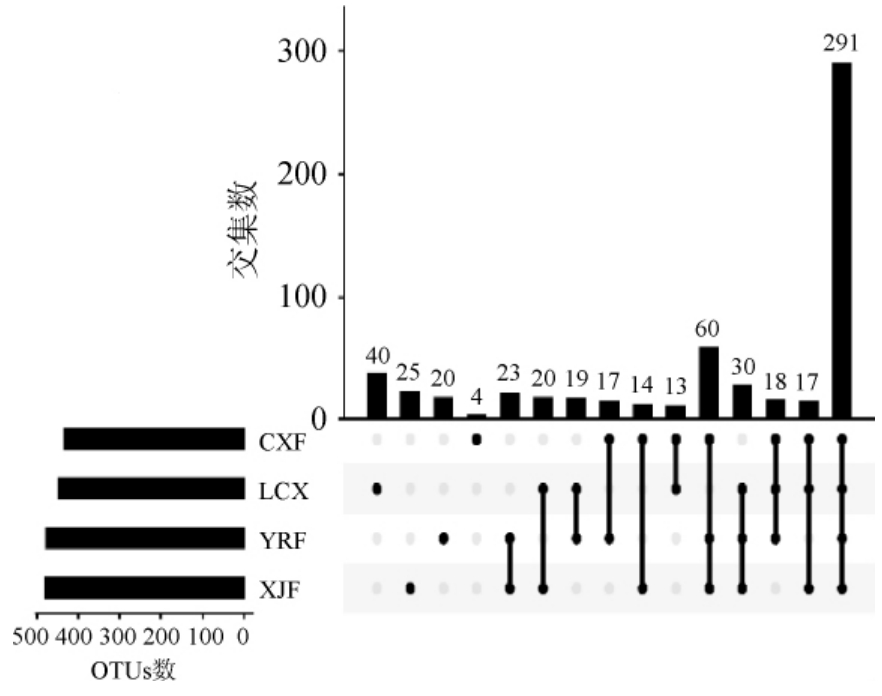
图3 窖泥样品的分类操作单元交集
Fig.3 Count of intersection of operational taxonomic units from pit mud samples
由图3可知,4份样品的共有OTUs数目为291个,相对丰度达到98.764 3%。任意3份样品的OTUs相对丰度的总和为0.731 4%。各份样品独有OTUs数量少,总丰度只有0.139 1%。经过RDP classifier检校OTUs的分类地位,发现4份样品中的共有OTUs主要来自于放线菌门(Actinobacteria)、拟杆菌门(Bacter-oidetes)、厚壁菌门(Firmicutes)、变形菌门(Proteobacteria)、螺旋菌门(Spirochaetae)、柔壁菌门(Tenericutes),表明同地域的不同浓香型白酒酿造厂的窖泥细菌群落类型具有高度的相似性。
基于属水平的群落结构丰度图(图4)直观地横向比对出各份样品细菌群落在属上具有明显差异,同时也发现Bacteroidetes、Spirochaetae、Firmicutes门类微生物在各样品中的相对丰度占绝对优势。
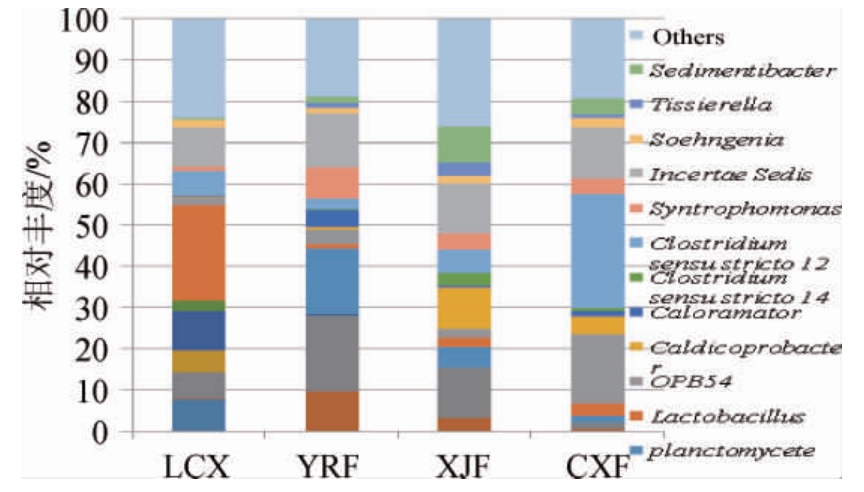
图4 窖泥细菌群落在属水平的结构分布
Fig.4 Structure distribution of bacterial communities in pit mud at genus level
对样品进行PCA,发现30个OTUs对窖泥样品间细菌群落结构的差异形成具有重要影响(图5),且全部来源于样品共有的OTUs中,属于优势OTUs。
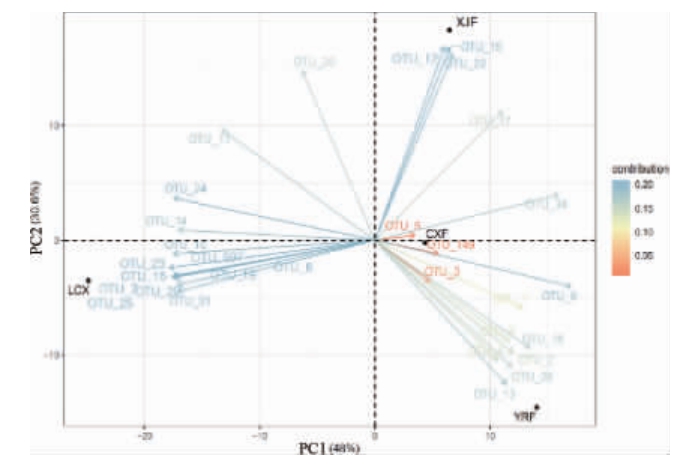
图5 对样品差异具有重要影响的操作分类单元矩阵的主成分分析
Fig.5 Principal component analysis of matrix of operational taxonomic units with important effects on sample difference
2.3 窖泥细菌系统发育多样性分析
经过BLAST检索,优势OTUs及其参考物种形成见表2。参考物种主要来源于各种各样的厌氧环境[3,12-36],在系统分类上全部归属于Bacteroidetes,Spirochaetae,Firmicutes,对优势OTUs具有较高的序列相似性(91%~100%)。将参考物种的属作为酿造的最小功能单元,并代表优势OTUs解剖窖泥样品的细菌系统发育多样性,可分析新窖池与成熟窖池形成酿造质量差异的原因。
Bacteroidetes微生物在新窖泥样品LCX中的相对丰度为31.59%,属于绝对优势类群,而在成熟窖泥样品中相对丰度相对较低,分别为28.33%(YRF)、15.53%(XJF)、2.09%(CXF)。Bacteroidetes的参考物种来源于5个属(表2),均具备代谢糖类物质产生小分子酸的能力[12-17],对寡聚糖、多糖、蛋白质等生物大分子具有良好的底物特异性,其代谢活动在维持自身繁殖的同时,也为下游微生物的生长提供基础性的能源、碳源、氮源等小分子物质[13-16],是窖泥酿造微生态中主导碳循环的重要微生物。
表2 优势操作分类单元矩阵的参考物种
Table 2 Reference species of matrix of dominant operational taxonomic units
Spirochaetes门微生物在新窖泥样品LCX中的相对丰度显著低,只有0.07%,在成熟窖泥样品中的相对丰度分别为15.92%(YRF)、4.78%(XJF)、1.43%(CXF),只包含planctomycete一个属。planctomycete属的微生物以CO2作为唯一碳源,在厌氧代谢过程将环境体系中有机物矿化、硝酸盐的异养还原作用而产生的NH4+氧化成N2、NO2-或NO3-,并从中获取繁殖所需的能量[18],对窖泥酿造微生态的氮循环有促进作用。
Firmicutes门微生物被认为是窖泥酿造微生态中产酸产香的关键微生物,其丰度常被用来评价窖泥的质量[4]。Firmicutes门微生物在窖泥样品中的多样性相对最丰富,主要包含芽孢杆菌纲(Bacilli)与梭菌纲(Clostridia),及相关的11个属(表2)。窖泥样品中的Bacilli微生物,主要包含乳杆菌属(Lactobacillus),是代谢合成乳酸的主要微生物[19-21],其相对丰度在新窖泥LCX中为23.20%,显著高于成熟窖泥的0.97%(YRF)、2.41%(XJF)、3.25%(CXF)。窖泥样品中的Clostridia微生物,特别是其中的梭菌属(Clostridium),是窖池中合成己酸的酿造关键微生物[22-36],其相对丰度在成熟窖泥中分别为36.01%(YRF)、51.26%(XJF)、73.95%(CXF),属于绝对优势微生物。Clostridia微生物在新窖泥LCX中的相对丰度为21.05%,相对低于Bacilli微生物的相对丰度。已有研究表明[1,4],窖池产己酸能力与Clostridia相对丰度为正相关,与Bacilli的相对丰度呈现负相关。Clostridia与Bacilli相对丰度在新窖泥LCX中为0.907 3%,显著低于成熟窖泥的相对丰度37.041 6%(YRF)、21.287 9(XJF)、22.786 8(CXF)。可认为Clostridia微生物在新窖泥LCX中相对失调的丰度是导致新窖池所酿原酒主体香不突出,酒质比成熟窖池差的主要原因。
3 结论
对新窖泥与成熟窖泥的细菌系统发育分析发现,螺旋菌门(Spirochaetae)、厚壁菌门(Firmicutes)、拟杆菌门(Bacteroidetes)是新窖泥与成熟窖泥中的共有优势类群,且Firmicutes微生物的多样性相对最丰富;新窖泥中Bacilli微生物的相对丰度显著高于成熟窖泥,Clostridia微生物与Bacilli微生物的丰度比显著低于成熟窖泥。可见浓香型白酒成熟窖泥中“好窖出好酒”的典型细菌群落结构,是Clostridia占绝对优势。以16S rRNA作为分子标记研究窖泥环境细菌群落,能有效揭示细菌系统发育的多样性,但要充分了解窖泥微生物的代谢特征与酿造质量的关系,还需借助微生物的可培养手段,并构建窖泥微生物的miRNA表达库,在后续再深入研究。
[1] ZOU W,YE G B,ZHANG K Z.Diversity,function,and application of Clostridium in Chinese strong flavor Baijiu ecosystem:A review[J].J Food Sci,2018,83(5):1193-1199.
[2]王忠彦.白酒色谱骨架成分的含量及比例关系对香型和质量的影响[J].酿酒科技,2000(6):93-96.
[3]WANG Q,WANG C D,LI C H,et al.Clostridium luticellarii sp.nov.,isolated from a mud cellar used for producing strong aromaticliquors[J]. Int J Syst Evol Micr,2015,65(12):4730-4733.
[4]LIANG H P,LI W F,LUO Q C,et al.Analysis of the bacterial community in agedand aging pit mud of Chinese Luzhou-flavour liquor by combined PCR-DGGE and quantitative PCR assay[J].J Sci Food Agr,2015,95(13):2729-2735.
[5]MU D S,LIANG Q Y,WANG X M,et al.Metatranscriptomic and comparative genomic insights into resuscitation mechanisms during enrichment culturing[J].Microbiome,2018,6(1):230.
[6]潘玲玲,王媚,罗明有,等.浓香型白酒黄水、窖泥中微生物总DNA 提取方法比较[J].中国酿造,2016,35(4):42-46.
[7] SCHLOSS P D,WESTCOTT S L,RYABIN T,et al.Introducing mothur:Open-source,platform-independent,community-supported software for describing and comparing microbial communities[J]. Appl Environ Microb,2009,75(23):7537-7541.
[8] EDGAR R C.UPARSE:Highly accurate OTU sequences from microbial amplicon reads[J].Nat Methods,2013,10:996-998.
[9]BOLYEN E,RIDEOUT J R,DILLON M R,et al.Reproducible,interactive,scalable and extensible microbiome data science using QIIME 2[J]. Nat Biotechnol,2019,37(9):1091.
[10] WANG Q,GARRITY G M,TIEDJE J M,et al.Naive bayesian classifier for rapid assignment of rRNA sequences into the new bacterial taxonomy[J].Appl Environ Microb,2007,73(16):5261-5267.
[11]PARADIS E,CLAUDE J,STRIMMER K.APE:Analyses of phylogenetics and evolution in R language[J].Bioinformatics,2004,20(2):289-290.
[12]TAKADA K,HAYASHI K,SATO Y,et al.Prevotella dentasini sp.nov.,a black-pigmented species isolated from the oral cavity of donkeys[J].Int J Syst Evol Micr,2009,60(7):1637-1639.
[13]HAHNKE S,LANGER T,KOECK D E,et al.Description of Proteiniphilum saccharofermentans sp.nov.,Petrimonas mucosa sp.nov.and Fermentimonas caenicola gen.nov.,sp.nov.,isolated from mesophilic laboratory-scale biogas reactors,and emended description of the genus Proteiniphilum[J].Int J Syst Evol Micr,2016,66(3):1466-1475.
[14]GRABOWSKI A,TINDALL B J,BARDIN V,et al.Petrimonas sulfuriphila gen.nov.,sp.nov.,a mesophilic fermentative bacterium isolated from a biodegraded oil reservoir[J]. Int J Syst Evol Micr,2005,55(3):1113-1121.
[15] KITAHARA M,SAKAMOTO M,TSUCHIDA S,et al. Parabacteroides chinchillae sp.nov.,isolated from chinchilla(Chincilla lanigera)faeces[J].Int J Syst Evol Micr,2013,63(9):3470-3474.
[16] TAN H Q,LI T T,ZHU C. Parabacteroides chartae sp.nov.,an obligatelyanaerobic species from wastewater of a paper mill[J].Int J Syst Evol Microbiol,2012,62(11):2613-2617.
[17] UEKI A,ABE K,OHTAKI Y,et al. Bacteroides paurosaccharolyticus sp.nov.,isolated from a methanogenic reactor treating waste from cattle farms[J].Int J Syst Evol Microbiol,2011,61(2):448-453.
[18]BOLLMANN J,ENGELBRECHT S,MARTIENSSEN M.Autofluorescent characteristics of Candidatus Brocadia fulgida and the consequencesfor FISH and microscopic detection[J].Syst Appl Microbiol,2019,42(2):135-144.
[19] ZHENG J S,WITTOUCK S,SALVETTI E,et al.A taxonomic note on the genus Lactobacillus:Description of 23 novel genera,emended description of the genus Lactobacillus Beijerinck 1901,and union of Lactobacillaceae and Leuconostocaceae[J].Int J Syst Evol Microbiol,2020,70(4):2782-2858.
[20] ENTAN E,MASA H,SUZUKI K I. Lactobacillus acetotolerans,a new species from fermented vinegar broth[J].Int J Syst Bacteriol,1986,36(4):544-549.
[21] VOGEL R F,BÖCKER G,STOLZ P.Identification of Lactobacilli from Sourdough and description of Lactobacillus pontis sp.nov[J]. Int J Syst Bacteriol,1994,44(2):223-229.
[22]SIZOVA M V,IZQUIERDO J A,PANIKOV N S,et al.Cellulose-and xylan-degrading Thermophilic anaerobic bacteria from biocompost[J]. Appl Environ Microb,2011,77(7):2282-2291.
[23]LIU L,JIAO J Y,SALAM N.Rhabdanaerobiumthermarum gen.nov.,sp.nov.,a novelanaerobic bacterium isolated from a hot spring[J].Int J Syst Evol Microbiol,2017,67(11):4584-4588.
[24]LABRADOR C R,BAENA S,CARDENAS D C,et al.Caloramator uimbayensis sp.nov.,an anaerobic,moderately thermophilic bacterium isolated from aterrestrial hot spring[J].Int J Syst Evol Microbiol,2013,63(4):1396-1402.
[25] YIN Q,TAO Y,ZHU X Y,et al. Clostridium liquoris sp.nov.,isolated from a fermentation pit used for the production of Chinese strong-flavoured liquor[J].Int J Syst Evol Microbiol,2016,66(2):749-754.
[26] HU X L,DU H,XU Y.Identification and quantification of the caproic acid-producing bacterium Clostridium kluyveri in the fermentation of pit mud used for Chinese strong-aroma type liquor production[J].Int J Food Microbiol,2015,214:116-122.
[27] ABRINI J,NAVEAU H,NYNS E J. Clostridium autoethanogenum,sp.nov.,an anaerobic bacterium that produces ethanol from carbon monoxide[J].Arch Microbiol,1994,161(4):345-351.
[28]WU C G,DONG X Z,LIU X L.Syntrophomonas wolfei subsp.methylbutyratica subsp.nov.,and assignment of Syntrophomonas wolfei subsp.saponavida to Syntrophomonas saponavida sp.nov.comb.nov.[J]. Syst Appl Microbiol,2007,30(5):376-380.
[29]MOORE W E C,JOHNSON J L,HOLDEMAN L V.Emendation of Bacteroidaceae and Butyrivibrio and descriptions of desulfornonas gen.nov.and ten new species in the genera Desulfomonas, Butyrivibrio, Eubacterium,Clostridium,and Ruminococcus[J]. Int J Syst Bacteriol,1976,26(2):238-252.
[30] KIM B C,JEON B S,KIM S,et al. Caproiciproducens galactitolivorans gen.nov.,sp.nov.,a bacterium capable of producing caproic acid from galactitol,isolated from a wastewater treatment plant[J]. Int J Syst Evol Microbiol,2015,65(12):4902-4908.
[31]JEON B S,et al.Production of hexanoic acid from D-galactitol by a newly isolated Clostridium sp.BS-1[J].Appl Microbiol Biotechnol,2010,88(5):1161-1167.
[32]MITSUOSAKAMOTO,TAKAOLINO,MORIYAOHKUMA.Faecalimonas umbilicata gen.nov.,sp.nov.,isolated fromhuman faeces,and reclassifica-tion of Eubacterium contortum, Eubacterium fissicatena and Clostridium oroticum as Faecalicatena contorta gen.nov.,comb.nov., Faecalicatena fissicatena comb.nov.and Faecalicatena orotica comb.nov.[J].Int J Syst Evol Microbiol,2017,67(5):1219-1227.
[33] PARSHINA S N,et al. Soehngenia saccharolytica gen.nov.,sp.nov.and Clostridium amygdalinum sp.nov.,two novelanaerobic,benzaldehydeconverting bacteria[J].Int J Syst Evol Microbiol,2003,53(6):1791-1799.
[34] FARROW J A E,LAWSON P A,HIPPE H,et al.Phylogenetic evidence that the gram-negative nonsporulating bacterium Tissierella (Bacteroides)praeacuta is a member of the Clostridium subphylum of the gram-positive bacteria and description of Tissierella creatinini sp.nov.[J]. Int J Syst Bacteriol,1995,45(3):436-440.
[35] IMACHI H,SAKAI S,KUBOTA T,et al. Sedimentibacter a cidaminivorans sp.nov.,an anaerobic,amino-acid-utilizing bacterium isolated from marine subsurface sediment[J]. Int J Syst Evol Microbiol,2016,66(3):1293-1300.
[36] BREITENSTEIN A,WIEGEL J,HAERTIG C,et al.Reclassification of Clostridium hydroxybenzoicum as Sedimentibacter hydroxybenzoicus gen.nov.,comb.nov.,and description of Sedimentibacter saalensis sp.nov.[J].Int J Syst Evol Microbiol,2002,52(3):801-807.