发酵是世界上一种最古老的食品保存技术之一。腐乳作为传统发酵食品,以其独特的风味特色在我国饮食文化中占据了一席重要地位。在腐乳发酵过程中,微生物分泌产生的蛋白酶、脂肪酶和糖化酶等多种酶能够将大豆中的蛋白质、脂肪和淀粉等大分子物质分解为游离氨基酸、脂肪酸、单糖和低聚糖等这些人体所必需且更易被吸收的小分子物质[1],因此与未发酵的大豆相比,腐乳大大提高了大豆蛋白的生物效价,且含有丰富的维生素B12、核黄素、大豆异黄酮等生物活性物质[2]。
腐乳按其色泽、风味可分为红腐乳、白腐乳、青腐乳、酱腐乳等;根据菌种类型可分为毛霉腐乳、根霉腐乳、细菌腐乳及无菌腐乳等。尽管目前腐乳已有工业化的生产方式,但仍有很多地方的商户采取自然发酵的方式生产腐乳来售卖。这种家庭式作坊生产的腐乳在开放的环境中进行自然发酵,其发酵过程不受控制,导致了产品的多样性和质量的不稳定性,值得引起重视。
目前大部分的研究都是基于改善腐乳的加工工艺或对风味的影响[3-4],对腐乳微生物多样性尤其是在发酵过程中微生物群落变化的研究较少。如今高通量测序技术已发展成为研究微生物多样性的有力工具,许多研究都利用该技术来进行微生物多样性的分析,如豆豉[5]、豆酱[6]、酸汤[7]、腐乳[8-9]等,因此利用该技术对自然发酵过程中的腐乳进行微生物群落变化的研究是很有必要的,可以使我们更全面的了解腐乳中细菌和真菌群落在不同发酵阶段的变化,了解其在各个发酵阶段的优势菌属,为改善腐乳的发酵工艺来保障其产品质量提供可靠的科学依据,对指导腐乳的生产具有实际意义。
1 材料与方法
1.1 材料与试剂
1.1.1 样品
腐乳样品:采购于重庆市北碚区菜市场,根据当地采用的发酵工艺结合腐乳发酵的变化选择在三个阶段进行取样。采样第一阶段:豆腐开始有长菌丝的趋势时;采样第二阶段:豆腐表面生长菌丝完全时;采样第三阶段:菌丝倒完的成品腐乳;采样过程中均使用无菌采样袋进行采样。样品的第一阶段编号为A1,第二阶段编号为A2,第三阶段编号为A3。
1.1.2 试剂
脱氧核糖核酸(deoxyribonucleic acid,DNA)提取试剂盒:美国MP Biomedicals生物医学公司;琼脂糖凝胶:西班牙BIOWEST公司;凝胶回收试剂盒:美国AXYGEN公司。其他试剂均为国产分析纯。
1.2 仪器与设备
ABI GeneAmp 9700型聚合酶链式反应(polymerase chain reaction,PCR)仪:美国ABI公司;QuantiFluorTM-ST蓝色荧光定量系统:美国Promega公司;MiSeq高通量测序平台:美国Illumina公司。
9700型聚合酶链式反应(polymerase chain reaction,PCR)仪:美国ABI公司;QuantiFluorTM-ST蓝色荧光定量系统:美国Promega公司;MiSeq高通量测序平台:美国Illumina公司。
1.3 方法
1.3.1 基因组DNA抽提
采用DNA提取试剂盒对腐乳样品中微生物的基因组DNA进行抽提,利用1%琼脂糖凝胶电泳检测抽提的基因组DNA。
1.3.2 PCR扩增
按指定测序区域,合成带有barcode的特异性引物515F(5'-GTGCCAGCMGCCGCGG-3')、907R(5'-CCGTCAATTCMTTTRAGTTT-3')、ITS1F(5'-CTTGGTCATTTAGAGGAAGTAA-3')和ITS2R(5'-GCTGCGTTCTTCATCGATGC-3')。为保证后续数据分析的准确性及可靠性,需满足两个条件:(1)尽可能使用低循环数扩增;(2)保证每个样本扩增的循环数一致。随机选取具有代表性的样本进行预实验,确保在最低循环数中使绝大多数样本能够扩增出浓度合适的产物。每个样本3个重复,将同一样本的PCR扩增产物混合后用2%琼脂糖凝胶电泳检测,使用AxyPrepDNA凝胶回收试剂盒切胶回收PCR扩增产物,Tris-HCl洗脱,2%琼脂糖电泳检测。
PCR扩增体系:5×FastPfu Buffer 5 μL,2.5 mmol/L脱氧核糖核苷三磷酸(deoxy-ribonucleoside triphosphates,dNTPs)2.5 μL,5 μmol/L正向引物1 μL,5 μmol/L反向引物1 μL,FastPfu DNA聚合酶0.5 μL,牛血清白蛋白(bovine serum albumin,BSA)0.25 μL,DNA模板10 ng,补充双蒸水(ddH2O)至20 μL。
细菌PCR扩增条件:95 ℃预变性3 min,27次循环(95 ℃变性30 s,55 ℃退火30 s,72 ℃延伸45 s),最后72 ℃延伸5 min。
真菌PCR扩增条件:95 ℃预变性3 min,32次循环(95 ℃变性30 s,55 ℃退火30 s,72 ℃延伸45 s),最后72 ℃延伸5 min。
1.3.3 荧光定量
参照电泳初步定量结果,将PCR扩增产物用QuantiFluorTM-ST蓝色荧光定量系统进行检测定量,之后按照每个样本的测序量要求,进行相应比例的混合。
1.3.4 Illumina PE250文库构建
连接“Y”字形接头;使用磁珠筛选去除接头自连片段;利用PCR扩增进行文库模板的富集;氢氧化钠变性,产生单链DNA片段。
1.3.5 Illumina PE250测序
DNA片段的一端与引物碱基互补,固定在芯片上;另一端随机与附近的另外一个引物互补,也被固定住,形成“桥(bridge)”;PCR扩增,产生DNA簇;DNA扩增子线性化成为单链;加入改造过的DNA聚合酶和带有4种荧光标记的dNTP,每次循环只合成一个碱基;用激光扫描反应板表面,读取每条模板序列第一轮反应所聚合上去的核苷酸种类;将“荧光基团”和“终止基团”化学切割,恢复3'端粘性,继续聚合第二个核苷酸;统计每轮收集到的荧光信号结果,获知模板DNA片段的序列。
1.3.6 测序数据分析
Illumina PE250测序得到的PE reads首先根据Overlap关系进行拼接,同时对序列质量进行质控和过滤,根据PE reads之间的Overlap关系,将成对的Reads拼接成一条序列;最后按照Barcode和引物序列拆分得到每个样本的优质序列,并在过程中根据正反Barcode和引物方向校正序列方向以及去除嵌合体,用Usearch软件和gold数据库,采用Donovo和Reference结合的方式去除嵌合体。区分样本后进行操作分类单元(operational taxonomic unit,OTU)聚类分析和物种分类学分析,细菌16S rDNA基因序列基于Silva[10]数据库,真菌5.8 ITS基因序列基于Unite[11]数据库,用Usearch对OTU进行聚类分析,基于OTU聚类分析结果,对OTU进行Alpha多样性指数分析,以及对测序深度的检测,用Mothur[12]做Rarefaction分析,利用R语言工具作稀释性曲线图;基于分类学信息,在各个分类水平上进行群落结构的统计分析并用R语言工具作图。
2 结果与分析
2.1 稀释性曲线
稀释性曲线[13]采用对样本序列进行随机抽样的方法,以抽到的序列数与它们所能代表OTU的数目构建稀释性曲线,它可以用来比较测序数据量不同的样本中物种的丰富度,也可以用来说明样本的测序数据量是否合理。当曲线趋向平坦时,说明测序数据量合理,更多的数据量只会产生少量新的OTU,反之则表明继续测序还可能产生较多新的OTU。腐乳样品细菌及真菌稀释性曲线见图1。由图1可知,曲线最终的趋势都趋于平坦,说明此次测序数据合理,测序深度能反映样品中的物种信息。
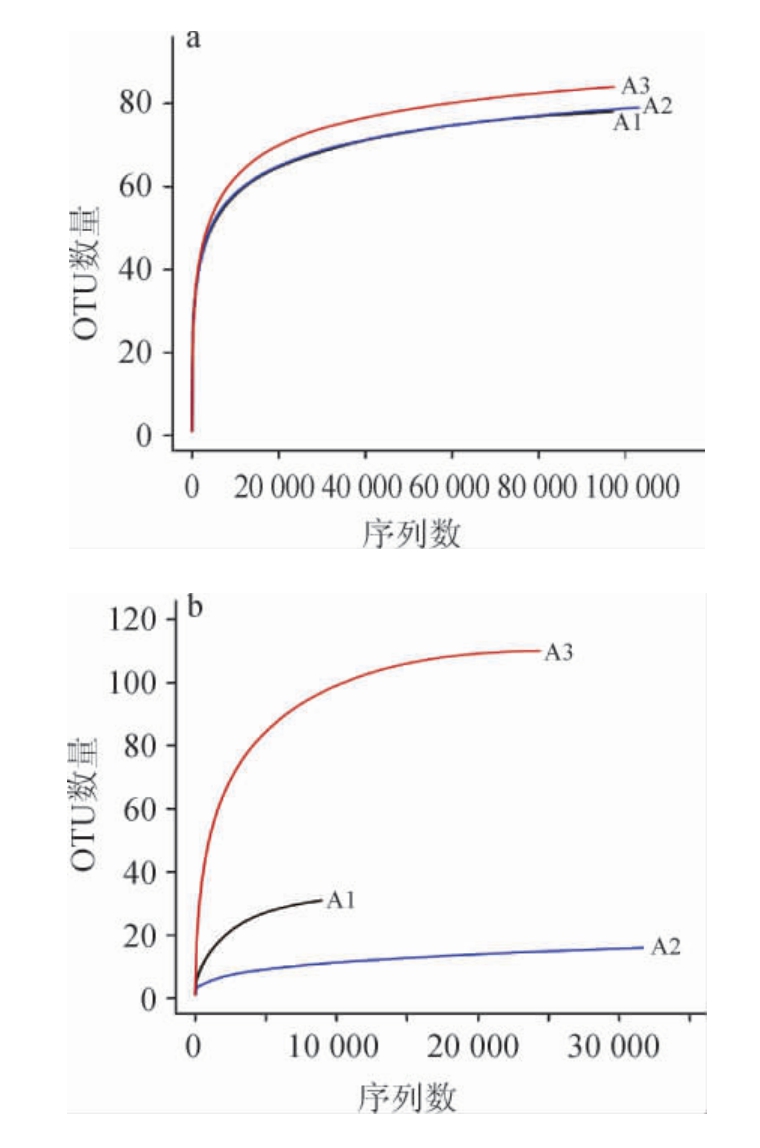
图1 细菌(a)及真菌(b)稀释性曲线
Fig.1 Dilution curve of bacteria (a) and fungi (b)
2.2 Alpha多样性分析
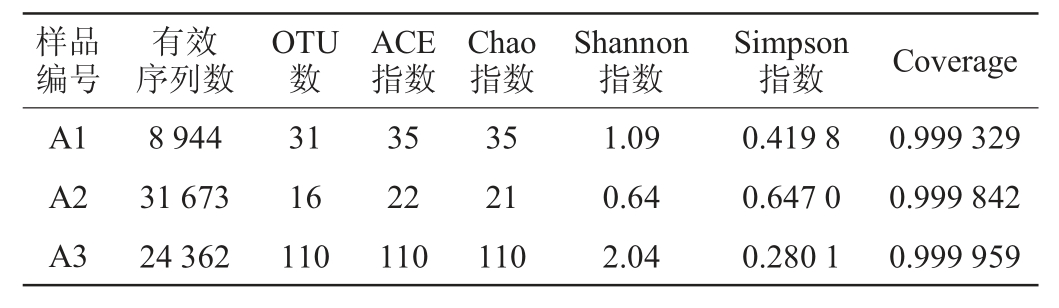
通过样本的多样性分析(Alpha多样性)可以反映微生物群落的丰度和多样性,Chao指数是用Chao1算法估计样本中所含OTU数目的指数,Chao指数和ACE指数可以用来代表菌群的丰度。辛普森(Simpson)指数与香农(Shannon)指数都是用来估算样本中微生物多样性的指数,Simpson指数值越大,说明群落多样性越低,Shannon指数值越大,说明群落多样性越高。Coverage是指各样本文库的覆盖率,其数值越高,则样本中序列被测出的概率越高,该指数反映测序结果是否代表了样本中微生物的真实情况。腐乳样品细菌Alpha多样性指数见表1,真菌Alpha多样性指数见表2。由表1及表2可知,所有样本的测序Coverage指数均>0.999,说明腐乳样品中序列基本都被测出,未被测到的概率较低,本次测序结果可以反映样品真实情况。由Chao指数和ACE指数可以看出无论是细菌还是真菌,在发酵的第三阶段菌群丰度是最高的,其中随着发酵时间的推移,细菌的菌群丰度变化不大,而真菌的菌群丰度有较明显的变化。由Simpson指数和Shannon指数可以看出,在发酵的第二阶段,细菌和真菌的群落多样性是最低的,而细菌群落多样性最高是在发酵的第一阶段,真菌群落多样性最高是在发酵的第三阶段。
表1 腐乳样品中细菌Alpha多样性指数
Table 1 Alpha diversity index of bacteria in sufu samples
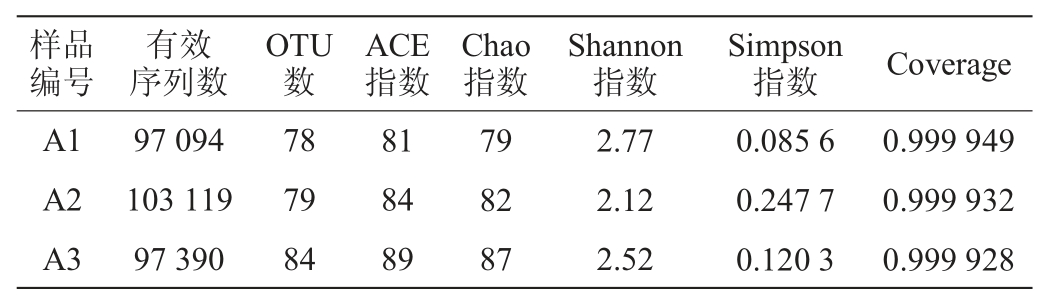
表2 腐乳样品中真菌Alpha多样性指数
Table 2 Alpha diversity index of fungal in sufu samples
2.3 微生物群落的OTU分布
用韦恩(Venn)图来表示样本中所共有和独有的OTU数目,可以比较直观的表现样本的OTU数目组成相似性及重叠情况,腐乳样品中细菌及真菌群落的OUT分布Venn图见图2。由图2可知,第一阶段的细菌OTU数是78个,第二阶段的细菌OTU数是79个,第三阶段的细菌OTU数是84个,三个样本中共有的OTU数是56个,说明在三个发酵阶段中细菌组成大部分相似。由图2亦可知,第一阶段的真菌OTU数是31个,第二阶段的真菌OTU数是11个,第三阶段的真菌OTU数是96个,三个样本中共有的OTU数是6个,说明在三个发酵阶段中真菌组成相似性不高。
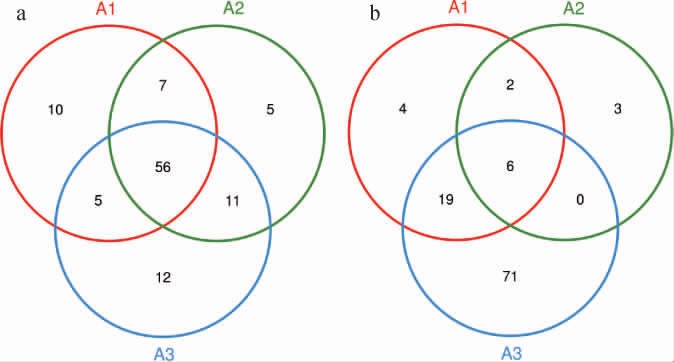
图2 细菌(a)及真菌(b)群落OTU分布Venn图
Fig.2 Venn diagram of OTU distribution of bacterial (a) and fungal (b)community
2.4 微生物群落结构分布
使用统计学的分析方法分析测序结果,并用柱状图的形式展现出来,可以用来观测样本在不同分类水平上的群落结构分布[14],并将多个样本的群落结构进行对比,可以更直观的感受到腐乳在发酵过程中微生物群落结构的变化。
2.4.1 细菌群落多样性
基于门和属水平,各个发酵阶段腐乳样品中细菌群落结构的分布情况见图3,发酵完成后,腐乳样品中细菌在域、门、纲、目、科、属等分类学水平上的物种比例和分布见图4。

图3 基于门(a)及属(b)水平细菌群落结构分布
Fig.3 Distribution of bacteria community structure at the phylum (a)and genus (b) level
由图3可知,在细菌门水平上,样本中主要存在的是变形菌门(Proteobacteria)、厚壁菌门(Firmicutes)和拟杆菌门(Bacteroidetes)这三类,在发酵的第一阶段,样品的细菌群落组成以变形菌门(61%)为主,第二阶段拟杆菌门(49%)和变形菌门(43%)分别占有较大比例,而第三阶段占比较多的则是厚壁菌门(50%)和变形菌门(45%)。由图3及图4可知,在样本中共鉴定出16个属和其他未被鉴定出的肠杆菌科,在发酵的第一阶段相对丰度含量较高的依次是不动杆菌属(Acinetobacter)、乳球菌属(Lactococcus)、假单胞菌属(Pseudomonas)、类香味菌属(Myroides)和丛毛单胞菌属(Comamonas)。第二阶段占比较大的菌属是类香味菌属(Myroides),其次是丛毛单胞菌属(Comamonas)和假单胞菌属(Pseudomonas)。第三阶段的占比较大的菌属则有乳球菌属(Lactococcus)、果胶杆菌属(Pectobacterium)、明串珠菌属(Leuconostoc)、漫游球菌属(Vagococcus)。可以看到随着发酵时间的推移不动杆菌属的数量越来越少;类香味菌属在发酵中期占很大比例,而在发酵完成后的含量却很低;假单胞菌属在发酵完成后含量显著低于前两个阶段;乳球菌属、未被鉴定出的肠杆菌科和果胶杆菌属都是呈现先减少后增加的趋势;丛毛单胞菌属则呈现出先增加后减少的趋势。
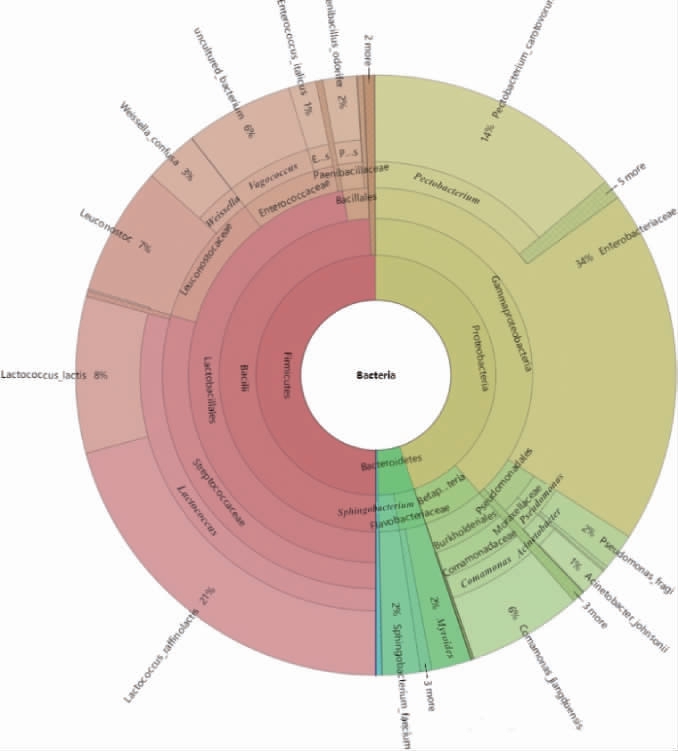
图4 腐乳发酵第三阶段细菌多级物种组成图
Fig.4 Bacterial multistage species composition at the third
不动杆菌属[15]广泛分布于自然界、水体和土壤中,医院环境与健康人的皮肤、呼吸道、消化道也会存在,目前对不动杆菌属的研究大部分集中在鲍曼不动杆菌耐药性方面。GUO X等[16]从豆豉这类发酵大豆食品中分离出了可以降解亚硝酸盐的不动杆菌属类细菌,刘亚栋[17]用16S rDNA基因序列分析的方法鉴定腐乳的微生物多样性时发现了不动杆菌属。类香味菌属[18]不分解糖类,脲酶阳性,可以还原亚硝酸盐,临床上类香味菌属细菌感染非常少见,大部分菌株对青霉素、头孢菌素、氨基糖苷类等药物耐药,其在发酵食品中出现的作用机理目前尚不明确。假单胞菌属可以分解蛋白质,所以在富含蛋白质的肉类、豆类等食品中常以假单胞菌属这类腐败菌为优势菌属,因此假单胞菌属大量存在于发酵前两个阶段中来分解豆制品中的蛋白质[19]。乳球菌属主要从乳制品、植物产品中分离出,发酵完成后的样品中存在的乳球菌属是棉子糖乳球菌(Lactococcus raffinolactis)和乳酸乳球菌(Lactococcus lactis),棉子糖乳球菌具有产γ-氨基丁酸的能力[20],γ-氨基丁酸是一种生理活性物质,这赋予了腐乳较好的生理活性功能,并且曾承露等[21]还初步证实了棉子糖乳球菌具有抑制金黄色葡萄球菌这类致病菌生成的作用,这对腐乳的卫生安全也是有益的。乳酸乳球菌是同型发酵细菌,广泛应用于发酵乳制品的制作,能够将牛乳中的蛋白质或脂肪降解成特定的风味物质或前体。肠杆菌科因其分布范围广,寄主范围大,因此与人类关系密切,肠杆菌科里的很多菌属都是食物中常见的并能够引起细菌性感染的致病菌。果胶杆菌属里存在的菌种是胡萝卜软腐果胶杆菌巴西亚种(Pectobacterium carotovorum subsp.brasiliense),冯志琴等[22]研究发现,这种细菌能够侵染十字花科、茄科、菠菜、芹菜和大多数葫芦科作物,引起其软腐病,所以推测这种细菌在发酵完成后的样品中增加可能是因为商家将发酵完成的腐乳包在白菜里而侵染了这种细菌。丛毛单胞菌属目前多用于生物降解的研究,在降解环境污染物方面有较好作用[23],刘亚栋[17]也在腐乳中发现过丛毛单胞菌属。明串珠菌属[24]是泡菜、干酪等发酵食品中的优势菌群,研究发现明串珠菌属的某些细菌有抑菌作用,对一些常见致病菌如金黄色葡萄球菌、伤寒沙门菌等有较好的拮抗作用,在腐乳、豆酱、泡菜中都分离出了明串珠菌属,明串珠菌属还应用到了酵素食品的加工中[25]。漫游球菌属[26]是一类可感染动物的细菌,关于漫游球菌属在发酵食品中的报道也很少见,其中河流漫游球菌可作为益生菌应用,而水獭漫游球菌可对人类健康产生影响。
2.4.2 真菌群落多样性
基于门和属水平,各个发酵阶段腐乳样品中真菌群落结构的分布情况见图5,发酵完成后,腐乳样品中真菌在域、门、纲、目、科、属等分类学水平上的物种比例和分布见图6。
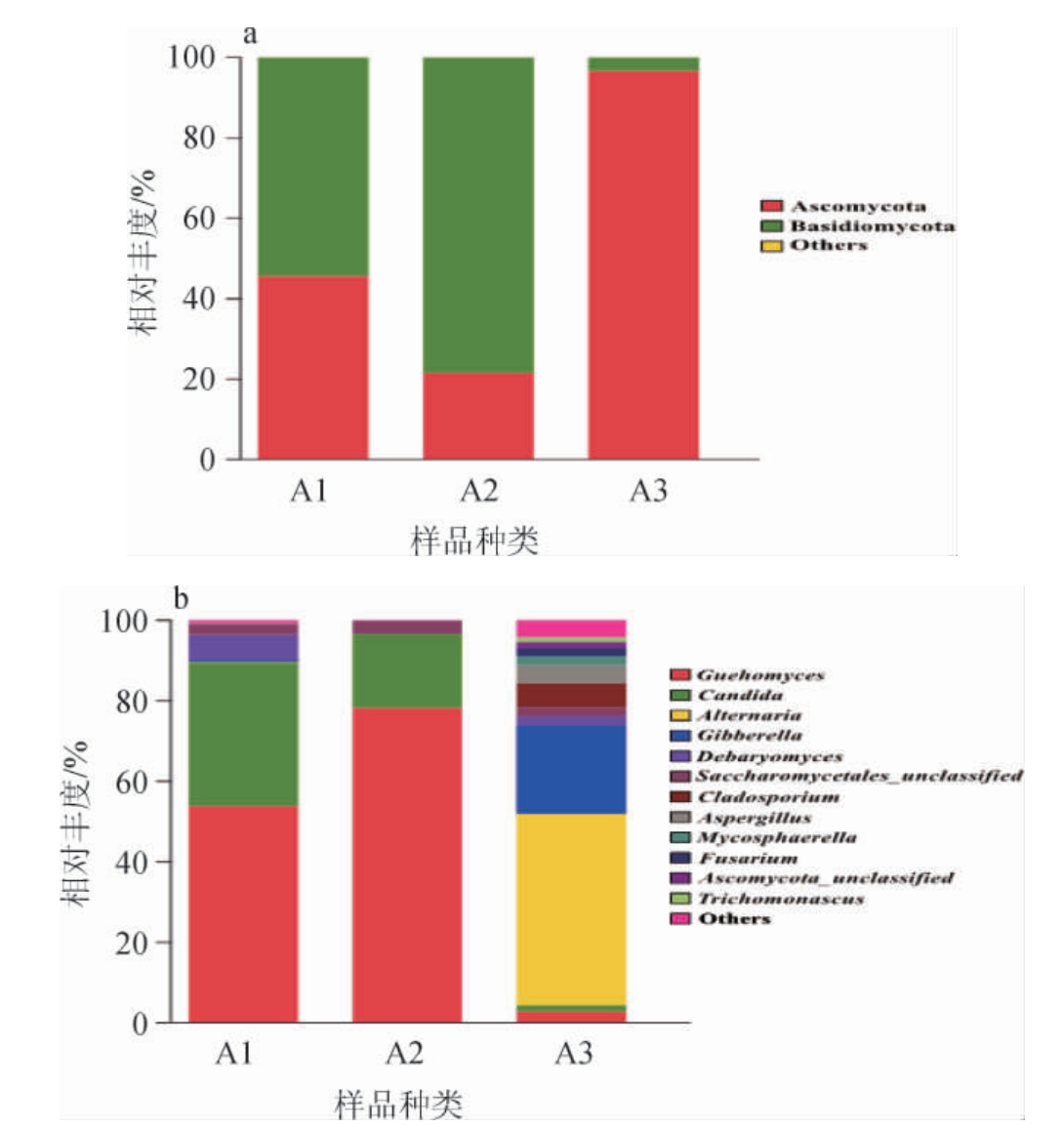
图5 基于门(a)及属(b)水平真菌群落结构分布
Fig.5 Distribution of fungi community structure at the phylum (a) and genus (b) level
由图5可知,在整个发酵过程中,真菌群落的组成相对细菌来说较简单,在发酵第三阶段的样本中,真菌群落的种类相对前两个阶段显著增加。在真菌门水平上,主要存在的是子囊菌门(Ascomycota)和担子菌门(Basidiomycota),发酵完成后子囊菌门占绝大优势。
由图5及图6可知,在样本中共鉴定出12个真菌属,其中大部分都是在第三阶段才出现在样本中。在发酵的第一阶段和第二阶段,占比较大的真菌菌属为久浩酵母属(Guehomyces)和假丝酵母属(Candida),而在发酵的第三阶段这两种菌属都降至很低,说明在发酵前期起主导作用的是酵母菌,酵母菌不仅对形成产品的风味物质有贡献,还可以抑制有害菌的生长,有的酵母菌的代谢产物还有利于乳酸菌的生长[27]。此外,链格孢属(Alternaria)、赤霉菌属(Gibberella)、枝孢属(Cladosporium)、曲霉属(Aspergillus)在发酵前期几乎不存在,发酵完成后大量出现,其中链格孢属和赤霉菌属占比最大,说明从发酵开始到发酵结束,真菌群落结构分布有着显著的变化。久浩酵母属在鲊广椒、酸汤子面团等发酵食品中都有被检测到[28-29],应该是一类参与食品发酵的重要菌属,但在腐乳中没有被报道过。假丝酵母属[30]有的菌种具有酒精发酵能力,可通过发酵作用增加产品的醇香,有的菌种能利用农副产品或碳氢化合物生产蛋白质,有的菌种则会引起感染而致病,假丝酵母属因其优良的生物特性常被用于食品工业、制药业和化妆品业[31]。链格孢属[32]在自然界分布广泛,一些链格孢属产生的真菌毒素能够引发多种农作物的病害,在一些瓜果蔬菜、酒水饮品、食用油里均检测到过链格孢毒素。赤霉菌属主要寄生于植物体内,是一类在农业生产上具有破坏性的植物病原真菌,推测其出现可能是由于在发酵完成后接触了其他物质而导致。王晖等[33]也曾在腐乳中检测到过枝孢属和曲霉属。
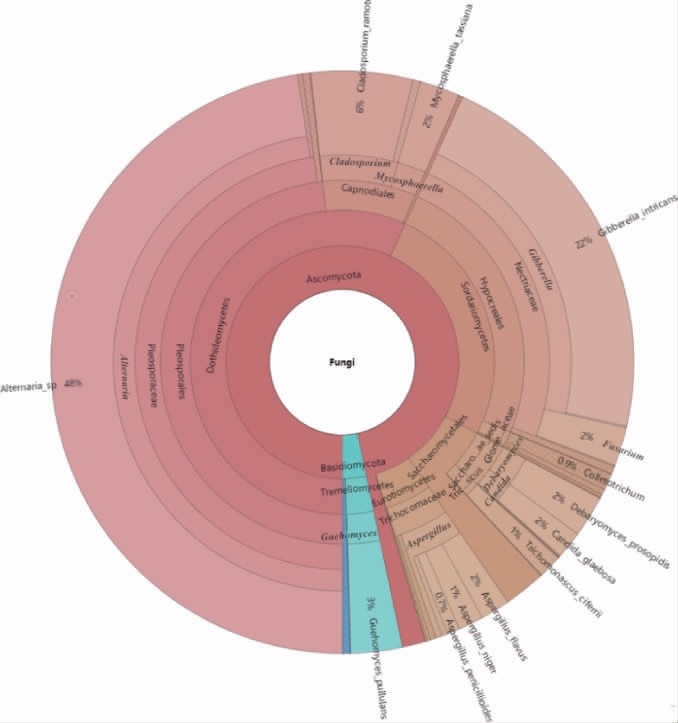
图6 腐乳发酵第三阶段真菌多级物种组成图
Fig.6 Fungi multistage species composition at the third stage of sufu fermentationstage of sufu fermentation
3 结论
腐乳的自然发酵与微生物的作用密切相关,本研究利用高通量测序技术,对腐乳自然发酵过程中的微生物多样性进行分析,结果表明,发酵的第一阶段,占比较大的细菌门为变形菌门,真菌门为担子菌门,细菌属为不动杆菌属、乳球菌属、假单胞菌属,真菌属为久浩酵母属、假丝酵母属;发酵的第二阶段,占比较大的细菌门为拟杆菌门,真菌门为担子菌门,细菌属为类香味菌属、丛毛单胞菌属、假单胞菌属,真菌属为久浩酵母属、假丝酵母属;发酵的第三阶段,占比较大的细菌门为厚壁菌门,真菌门为子囊菌门,细菌属为乳球菌属、果胶杆菌属,真菌属为链格孢属、赤霉菌属。不同阶段中主要存在的菌属不同,在腐乳整个发酵过程中既出现了有益于发酵的菌属,也有会影响产品质量的菌属。
[1]ZHAO C J,SCHIEBER A,GANZLE M G.Formation of taste-active amino acids,amino acid derivatives and peptides in food fermentations-A review[J].Food Res Int,2016,89(1):39-47.
[2]刘亚,杨光影,吴悦,等.腐乳研究进展[J].农产品加工学刊,2013(23):64-66.
[3]卢靖,刘平,张丽珠,等.腐乳发酵过程挥发性风味成分的变化[J].食品科学,2014,35(16):175-179.
[4]哈尔乐哈西·布勒斯别克.改善腐乳品质的加工工艺技术研究[D].杭州:浙江大学,2015.
[5]YANG L,YANG H L,TU Z C,et al.High-throughput sequencing of microbial community diversity and dynamics during douchi fermentation[J].Plos One,2016,11(12):e0168166.
[6]JUN R W,JIA C Z,PU S,et al.Bacterial community involved in traditional fermented soybean paste dajiang made in northeast China[J].Ann Microbiol,2013,63(4):1417-1421.
[7]王琪琪,田界先,潘宗东,等.基于Illumina MiSeq 分析贵州凯里酸汤独特风味的优势菌群[J].食品与发酵工业,2020,46(14):40-47.
[8]徐琼,刘洋,曲勤凤,等.高通量测序分析不同地区红腐乳细菌多样性[J].食品科学,2020,41(10):110-116.
[9]XU D D,WANG P,ZHANG X,et al.High-throughput sequencing approach to characterize dynamic changes of the fungal and bacterial communities during the production of sufu,a traditional Chinese fermented soybean food[J].Food Microbiol,2020,86:103340.
[10]QUAST C,PRUESSE E,YILMAZ P,et al.The SILVA ribosomal RNA gene database project:Improved data processing and webbased tools[J].Nucleic Acids Res,2013,41(1):590-596.
[11]ABARENKOV K,NILSSON R H,LARSSON K,et al.The UNITE database for molecular identification of fungi-recent updates and future perspectives[J].New Phytol,2010,186(2):281-285.
[12]SCHLOSS P D,GEVVERS D,WESTCOTT S L,et al.Reducing the effects of PCR amplification and sequencing artifacts on 16S rRNA-based studies[J].Plos One,2011,6(12):e27310.
[13]AMATO K R,YEOMAN C J,KENT A,et al.Habitat degradation impacts black howler monkey(Alouatta pigra)gastrointestinal microbiomes[J].Isme J,2013,7(7):1344-1353.
[14]OBERAUNER L,ZACHOW C,LACKNER S,et al.The ignored diversity:complex bacterial communities in intensive care units revealed by 16S pyrosequencing[J].Sci Rep,2013,3(1):1413-1413.
[15]安娜.临床医学微生物检验基础与诊断技术[M].北京:科学技术文献出版社,2014:76.
[16]GUO X,LIU B F,GAO L N,et al.Isolation of nitrite-degrading strains from Douchi and their application to degrade high nitrite in Jiangshui[J].J Sci Food Agr,2019,99(1):219-225.
[17]刘亚栋.利用16S rDNA 测序的方法鉴定腐乳中微生物的种类多样性[D].济南:山东师范大学,2017.
[18]JAMES H J,MICHAEL A P.临床微生物学手册:第1 卷第11 版[M].北京:中华医学电子音像出版社,2017:1051.
[19]黄文,江美都,肖作兵,等.食品添加剂[M].北京:中国计量出版社,2013:114.
[20]黄德娜,李锋,曾承露.Lactococcus raffinolactis Y-12 产γ-氨基丁酸发酵条件优化[J].中国食品添加剂,2017(9):95-101.
[21]曾承露,李锋,黄德娜.棉籽糖乳球菌Y-12 产细菌素培养条件优化[J].食品工业科技,2017,38(18):123-128.
[22]冯志琴,MENG X L,CHAI A L,等.胡萝卜软腐果胶杆菌巴西亚种(Pectobacterium carotovorum subsp.brasiliense)引起的黄瓜细菌性软腐病在中国发生[J].中国植保导刊,2017,37(7):58-62.
[23]赵燕,薛林贵,李琳,等.丛毛单胞菌在环境污染物降解方面的研究进展[J].微生物学通报,2012,39(10):1471-1478.
[24]岳媛媛,豆酱中明串珠菌的分离筛选[D].沈阳:沈阳农业大学,2017.
[25]沈燕飞,聂小华,孟祥河,等.酵素食品加工微生物与功能特性研究进展[J].浙江农业科学,2019,60(1):112-114.
[26]郑勤琴,段纲,项勋,等.漫游球菌研究进展[J].中兽医医药杂志,2017,36(5):27-30.
[27]崔玉琦,王雅炜,武瑞霞,等.酵母菌在发酵乳制品中的应用研究[J].食品科技,2014,39(10):53-56.
[28]尚雪娇,雷炎,代凯文,等.当阳地区鲊广椒品质的评价[J].食品研究与开发,2018,39(24):33-38.
[29]乌日娜,张颖,张红萧,等.酸汤子玉米面图中微生物多样性分析[J].食品科学,2017,38(12):21-26.
[30]纪铁鹏,崔雨荣.乳品微生物学[M].北京:中国轻工业出版社,2007:98.
[31]KIELISZEK M,KOT A M,ANNA B W,et al.Biotechnological use of Candida yeasts in the food industry:A review[J].Fungal Biol Rev,2017,31(4):185-198.
[32]王婧,翟伟卜,高环,等.链格孢引起的病害严重危害农作物生产并危及农产品安全[J].植物保护,2017,43(4):9-15.
[33]王晖,钟冠山.青腐乳发酵的初步探讨[J].中国酿造,2003,21(3):13-20.