酸鱼是侗族和苗族等少数民族制作的一种特色食品,因其口感美味和营养丰富而广受欢迎[1]。酸鱼是在密封环境下自然发酵而成的,主要原料为新鲜的淡水鱼,包括青鱼、草鱼和鲫鱼等[2],制作过程大致为清洗原料鱼、放置腌料(腌料的主要成分为胡椒、辣椒、小米、盐、糖和酒等)和装坛发酵,样品需要经过至少半个月的时间进行发酵[3]。民间传统的发酵方法不仅需要较长的发酵周期,且发酵标准和产品品质无法得到保障,这制约了传统发酵食品的现代化和工业化发展。为改善这种情况,BAO R等[4]通过研究发现接种乳酸乳球菌(Lactococcus lactis)M10和食窦魏斯氏菌(Weissella cibaria)M3可在降低发酵周期的同时保持发酵鱼的品质,且二者还能增强发酵鱼风味的形成;ZANG J H等[5]对酸鱼发酵过程中微生物群落的多样性进行了研究,发现酸鱼在发酵过程中细菌和真菌的结构是随着发酵时间而变化的,且在相同的发酵环境中,真菌的多样性大于细菌的多样性;夏秀东等[2]发现添加辣椒可抑制酸鱼发酵前期的菌落数量,并减少发酵过程中挥发性盐基氮的含量,但是具体的影响机制还仍待讨论。目前有关酸鱼的研究仍相对较少,并且对成品酸鱼的群落细菌多样性及功能预测分析还尚未开展。
近几年来,随着高通量测序技术的快速发展,以Illumina MiSeq为代表的第二代高通量测序技术因具有测序成本低、操作简单和结果准确等特点[6-7]而被广泛关注和使用,目前在酸奶[8]、腊鱼[9]、风干猪肉[10]、泡菜[11]和辣椒酱[12]等方面均有所应用。随着人们对16S rRNA序列认识的深入和高通量测序技术的发展,研究者发现基于16S扩增子分析所获得的信息相对有限,因此研究者开始探讨如何将分类单元操作(operational taxonomic unit,OTU)与细菌基因组功能建立联系,于是PICRUSt(Phylogenetic Investigation of Communities by Reconstruction of Unobserved States)技术应运而生,自此为16S扩增子的相关研究提供了研究方向,近几年来已被广泛的用于土壤[13]和食品[14]微生物类群揭示及其功能预测分析。
本研究从湖南省张家界市采集7 个酸鱼样品,采用Illumina MiSeq测序技术对其细菌多样性进行了解析,用PICRUSt技术对其微生物基因功能进行预测,以期为后续酸鱼生产用微生物菌株筛选提供理论依据。
1 材料与方法
1.1 材料与试剂
酸鱼:采集自湖南省张家界市慈利县,样品编号分别为SY1、SY2、SY3、SY4、SY5、SY6和SY7。
脱氧核糖核苷三磷酸、10×聚合酶链式反应(polymerase chain reaction,PCR)缓冲液、脱氧核糖核酸(deoxyribonucleic acid,DNA)酶:北京全式金生物技术有限公司;338F/806R正反向引物:武汉天一辉远生物科技有限公司;微生物基因组提取试剂盒:德国QIAGEN公司。
1.2 仪器与设备
Vetiri梯度基因扩增仪:美国AB公司;R930机架式服务器:美国DELL公司;Illumina MiSeq PE300高通量测序平台:美国Illumina公司;ND-2000C微量紫外分光光度计:美国Nano Drop公司;FluorChem FC3化学发光凝胶成像系统:美国ProteinSimple公司;CR21N型高速离心机:日本日立金属株式会社。
1.3 试验方法
1.3.1 样品采集及细菌基因组DNA提取
样品采集自湖南省张家界市慈利县慈利大市场、城西农贸市场和生活家菜市场,选取以草鱼和红辣椒为原料、发酵20~30 d左右且采用土陶坛发酵的酸鱼,装入采样袋后置于装有冰袋的样品箱。7个酸鱼样品由不同农户手工制作,样品运送回实验室后去除鱼头和鱼刺,鱼肉使用组织搅拌机打碎后,使用基因组提取试剂盒进行微生物宏基因组提取。
1.3.2 细菌16S rRNA扩增及高通量测序
以基因组为模板,使用带有7 个核苷酸序列的338F(5'-ACTCCTACGGGAGGCAGCAG-3')和806R(5'-GGACTACHVGGGTWTCTAAT-3')引物,参照王玉荣等[15]的扩增体系对细菌16S rRNA序列V4-V5区进行PCR扩增,结束后进行检测和清洁。使用Illumina MiSeq PE300平台进行高通量测序。参照ZHANG W等[16]的方法将下机序列进行拼接,并按照王玉荣等[17]的方法进行序列质量控制,进而得到质量控制合格的有效序列。
1.3.3 生物信息学分析
将质控合格的有效序列上传至QIIME分析平台进行生物信息学分析。其主要分析流程如下:(1)使用PyNAST软件对序列进行校准与对齐处理[18]。(2)使用UCLUST算法分别按照100%和97%对序列进行OTU的建立[19]。(3)使用ChimeraSlayer去除含有嵌合体序列的OTU。(4)将每个OTU中第一条序列分别与Greengenes、RDP和SILVA数据库[20]进行比对分析,以便在门、纲、目、科和属水平上对酸鱼样品的细菌群落进行分析,进而计算超1(Chao1)和香农(Shannon)指数。
1.3.4 PICRUSt基因功能预测
将合格的高质量序列与Greengenes数据库进行比对,用PICRUSt软件[20]对酸鱼的基因功能进行预测,然后基于蛋白质直系同源簇数据库[21](Clusters of Orthologous Groups of proteins,COGs)进行功能注释。
1.3.5 数据分析
用SAS软件进行相关性分析,用R软件进行UpSet图、相关性图和热图的绘制,其他图形都是采用Origin8.6软件绘制。
2 结果与分析
2.1 样品序列多样性及丰富度分析
本研究采用Illumina MiSeq高通量测序技术对7个酸鱼样品的细菌群落结构进行了解析。序列经过质控后,7个酸鱼样品共产生了274 102条高质量序列,平均每个样品39 157条。经与16S rRNA标准数据库比对得到274 098条序列,其中有4 条序列比对失败,因而共有274 098条序列参加分类单元操作划分。7个酸鱼样品的测序结果以及分类学地位如表1所示。
表1 酸鱼样品测序结果及各分类地位数量
Table 1 Sequencing results of sour fish and number of classified positions
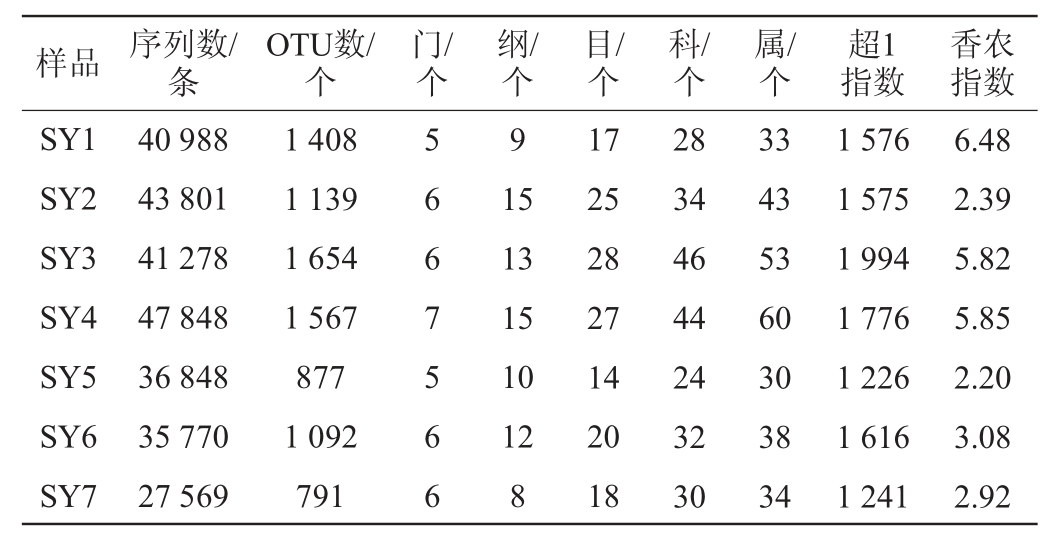
注:计算每个样品的超1指数和香农指数时,样品的测序量都是26 510条序列。
由表1可知,样品SY3的超1指数最高,而样品SY1的香农指数最高,这说明样品SY3中细菌的丰富度最大,而样品SY1中细菌的多样性最高。由表1亦可知,按97%相似性进行分类单元操作划分共得到3 612个OTU,平均每个样品1 218 个,在分类单元操作划分的基础上,将所有序列划分为7个门、19个纲、64个目、94个科和148个属。
2.2 基于各分类学地位酸鱼细菌相对含量的构成分析
本研究将酸鱼样品中平均相对含量<1.0%的细菌归为其他,基于门水平细菌构成情况如图1所示。
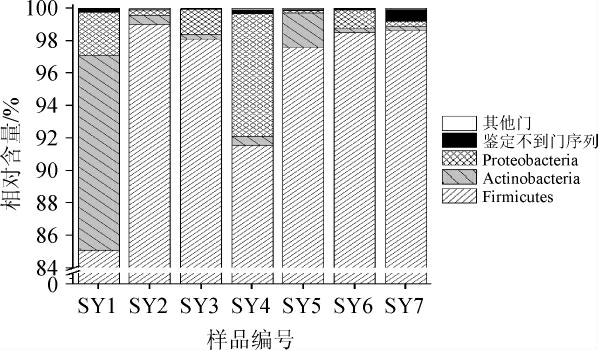
图1 酸鱼样品中平均含量高于1.0%的细菌门
Fig.1 Bacteria phylum with average content higher than 1.0% in sour fish samples
由图1可知,平均相对含量高于1.0%的有3 个门,分别为厚壁菌门(Firmicutes)、放线菌门(Actinobacteria)和变形菌门(Proteobacteria),其平均相对含量分别为95.49%、2.29%和1.96%,其中有0.2%鉴定不到门序列。酸鱼样品中平均相对含量>1.0%的细菌属构成如图2所示。
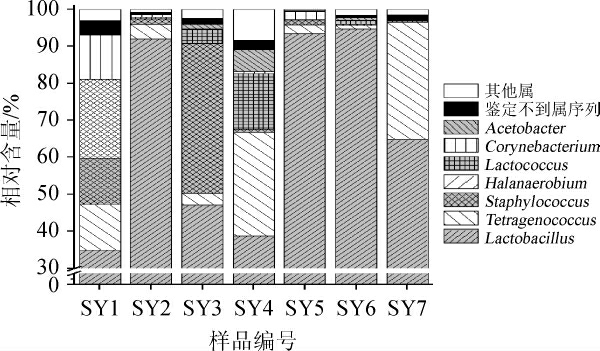
图2 酸鱼样品中平均含量高于1.0%的细菌属
Fig.2 Bacteria genera with average content higher than 1.0% in sour fish samples
由图2可知,酸鱼中平均相对含量大于1.0%的细菌属主要有隶属于Firmicutes的乳酸杆菌属(Lactobacillus)、四联球菌属(Tetragenococcus)、葡萄球菌属(Staphylococcus)、盐厌氧菌属(Halanaerobium)和乳球菌属(Lactococcus),其平均相对含量分别为66.53%、11.77%、8.10%、3.07%和2.99%;隶属于Actinobacteria的棒状杆菌属(Corynebacterium),其平均相对含量为2.20%;隶属于Proteobacteria的醋酸杆菌属(Acetobacter),其平均相对含量分别为1.15%。Lactobacillus、Tetragenococcus和Lactococcus均为乳酸菌,三者的累计平均相对含量为81.29%,由此可见,酸鱼样品中蕴含了丰富的乳酸菌。单玉鑫等[22]通过对贵州和湖南地区酸鱼微生物群落结构与品质的联系研究发现,酸鱼产品的主要优势细菌为Lactobacillus和Tetragenococcus,研究结论与本研究相同。Halanaerobium是一种嗜盐且严格厌氧的细菌,其可利用碳水化合物和果胶等物质来产酸和产氢,李春生等[23]研究鱼露发酵过程中细菌的演替以及品质变化,发现Halanaerobium会通过抑制乙酸乙酯的生成进而促进三甲胺的释放,从而可能会导致发酵产品产生鱼腥味,降低品质。
游刚等[24]研究表明使用复合乳酸菌发酵剂可以增加六齿金线鱼肉中醇、醛和酮类物质的含量,降低胺类物质的产生,进而改善了产品的风味品质;郑心如等[25]亦评价了复合发酵剂对发酵鱼肉香肠品质的影响,研究发现使用复合发酵剂可显著降低总挥发性盐基氮(total volatile base nitrogen,TVB-N)含量和硫代巴比妥酸(thiobarbituric acid,TBA)值,同时使得氨基酸态氮(amino acid nitrogen,AAN)值显著升高,进而使得产品品质提高。由此可见,为了缩短发酵时间、提高产品品质和提升发酵鱼产品的安全性,在对酸鱼样品中优势细菌属进行分析的基础上,对其中的优势类群进行分离,同时对优良发酵特性菌株进行筛选并确定其最佳的发酵复配方案,进而实现乳酸菌发酵剂在酸鱼生产中的应用是一个比较可行的研究思路。
本研究进一步对酸鱼样品中OTU的含量及分布情况进行分析,结果如图3所示。
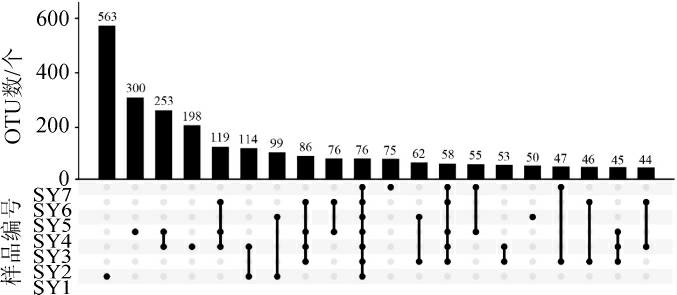
图3 基于OTU水平的UpSet图
Fig.3 UpSet diagram based on OTU level
由图3可知,有76 个OTU存在于所有的样品中,共包含188 289条序列,占序列总数的68.69%;同时分别有563、300、198、75和50个OTU仅存在于SY1、SY4、SY3、SY7和SY5样品中,分别包含了5 270、1 136、678、269和159条序列,分别占总序列数的1.92%、0.41%、0.25%、0.10%和0.06%。由此可见,7 个酸鱼样品中存在大量的核心细菌类群,虽然部分样品可能含有特殊的细菌类群,但其相对含量较少。将在所有样品中均存在的OTU定义为核心OTU,进一步分析发现其中8 个核心OTU的相对含量大于1.0%,其结果如图4所示。
由图4可知,8 个核心OTU被注释为3 类细菌属,其分别为Lactobacillus、Tetragenococcus和Staphylococcus。OTU11996注释为Tetragenococcus,其平均含量为9.89%,OTU4509、OTU5083、OTU3571、OTU7361、OTU1144和OTU11972均注释为Lactobacillus,其平均含量分别为1.11%、1.18%、1.42%、1.45%、1.95和43.27%,OTU6717注释为Staphylococcus,且其平均含量为5.50%。值的一提的是,注释为乳酸菌的7 个核心OTU平均含量累计达60.27%,由此可知,乳酸菌在酸鱼中为主要的优势菌,这和图2的结论是一致的。进一步对核心OTU之间的相关性进行研究,结果如图5所示。
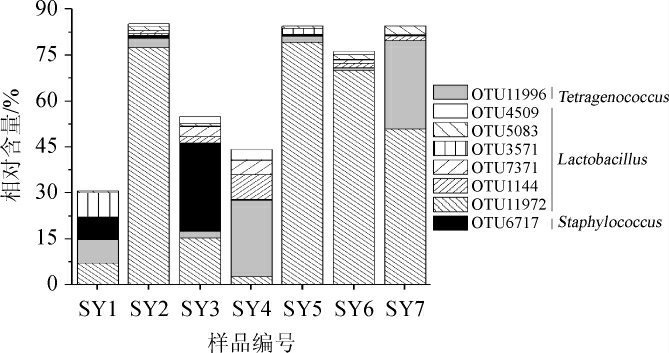
图4 平均相对含量大于1.0%的核心OTU
Fig.4 Core OTU with average relative content higher than 1.0%
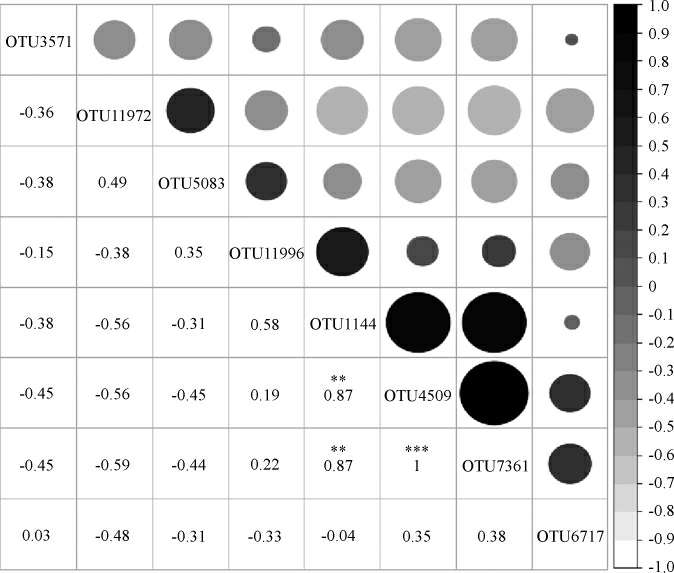
图5 核心OTU之间的相关性
Fig.5 Correlation between core OTUs
“**”为相关性非常显著(P<0.01),“***”为相关性极显著(P<0.001);圆圈颜色越深且直径越大,则表明其相关系数越大。
由图5可知,OTU7361(鉴定为Lactobacillus)与OTU4509(鉴定为Lactobacillus)的相关系数为1.00,相关性极显著(P<0.001);OTU7361(鉴定为Lactobacillus)与OTU1144(鉴定为Lactobacillus)的相关系数为0.87,相关性非常显著(P<0.01);OTU4509(鉴定为Lactobacillus)与OTU1144(鉴定为Lactobacillus)的相关系数亦为0.87,相关性亦非常显著(P<0.01)。由此可见,酸鱼样品中部分Lactobacillus类群可能具有一定的共生关系。
2.3 PICRUSt功能预测
本研究使用PICRUSt软件对酸鱼中细菌微生物的基因功能进行预测,同时参考蛋白质直系同源簇数据库(COGs)对功能类别进行了注释,其结果如图6所示。
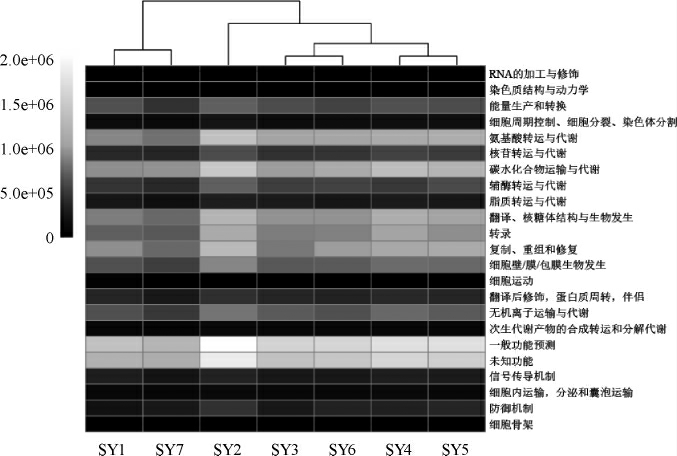
图6 酸鱼样品细菌的功能预测
Fig.6 Functional prediction of bacteria in sour fish samples
由图6可知,从纳入本研究的7个酸鱼样品中分析得到4 048个COG,且其隶属于21个功能类别,其中“氨基酸转运与代谢”和“碳水化合物运输和代谢”等功能在酸肉细菌类群中表达较高,而“RNA的加工与修饰”、“染色质结构与动力学”、“细胞周期控制、细胞分裂、染色体分割”、“细胞壁/膜/包膜生物发生”、“次生代谢产物的合成转运和分解代谢”和“细胞骨架”等功能表达则相对较低。
3 结论
使用Illumina MiSeq技术对张家界市7个酸鱼样品进行细菌多样性解析发现,优势细菌门为Firmicutes、Actinobacteria和Proteobacteria,三者累计相对含量可达99.74%,优势细菌属主要为Lactobacillus、Tetragenococcus、Staphylococcus、Halanaerobium、Lactococcus、Corynebacterium和Acetobacter,相对含量占细菌属的95.81%。酸鱼样品中含有大量的Lactobacillus、Tetragenococcus及Lactococcus核心类群,含量占总细菌类群的60%左右,且其细菌类群具有较强的氨基酸和碳水化合物转运与代谢功能。
[1]SINGH S S,DE MANDAL S,MATHIPI V,et al.Traditional fermented fish harbors bacteria with potent probiotic and anticancer properties[J].Biocatal Agr Biotechnol,2018,15(7):283-290.
[2]夏秀东,单成俊,刘小莉,等.添加辣椒对酸鱼发酵进程和理化指标的影响[J].江西农业学报,2018,30(8):84-89.
[3]王华娟,刘章武.酸鱼自然发酵工艺研究[J].中国酿造,2015,34(4):165-168.
[4]BAO R,LIU S,JI C,et al.Shortening fermentation period and quality improvement of fermented fish,Chouguiyu,by co-inoculation of Lactococcus lactis M10 and Weissella cibaria M3[J].Front Microbiol,2018,9(12):1-11.
[5]ZANG J H,XU Y S,XIA W S,et al.Dynamics and diversity of microbial community succession during fermentation of Suan yu,a Chinese traditional fermented fish,determined by high throughput sequencing[J].Food Res Int,2018,111(9):565-573.
[6]MELANIE S,IJAZ U Z,ROSALINDA D,et al.Insight into biases and sequencing errors for amplicon sequencing with the Illumina MiSeq platform[J].Nucl Acid Res,2015:43(6):1-16.
[7]WEN C,WU L,QIN Y,et al.Evaluation of the reproducibility of amplicon sequencingwithIlluminaMiSeqplatform[J].PloS One,2017,12(4):e0176716.
[8]智楠楠,宗凯,杨捷琳,等.Illumina Miseq 平台深度测定酸奶中微生物多样性[J].食品工业科技,2016,37(24):78-82.
[9]王玉荣,廖华,赵慧君,等.基于PCR-DGGE 与高通量测序技术的恩施地区腊鱼细菌多样性评价[J].现代食品科技,2018,34(11):208-213.
[10]田建军,张开屏,杨明阳,等.应用Illumina MiSeq 测序技术比较风干肉中细菌多样性和微生物安全性[J].食品科学,2019,40(8):33-40.
[11]YANG H,WU H,GAO L,et al.Effects of Lactobacillus curvatus and Leuconostoc mesenteroides on Suan Cai fermentation in Northeast China[J].J Microbiol Biotechn,2016,26(12):2148-2158.
[12]高航,张欣,赵燕,等.宏基因组测序技术在传统酿造食品微生物群落分析中应用研究进展[J].中国酿造,2020,39(5):1-7.
[13]孙峰,田伟,张菲,等.丹江口库区库滨带植被土壤细菌群落多样性及PICRUSt 功能预测分析[J].环境科学,2019,40(1):421-429.
[14]崔梦君,王玉荣,葛东颖,等.遵义地区莽椒细菌多样性及PICRUSt基因功能预测分析[J].食品与发酵工业,2020,46(4):106-112.
[15]王玉荣,杨成聪,葛东颖,等.扩增区域对鲊广椒细菌MiSeq 测序的影响.食品科学,2019,40(10):134-140.
[16]ZHANG W,LUO Q,ZHU Y,et al.Microbial diversity in two traditional bacterial douchi from Gansu province in northwest China using Illumina sequencing[J].PloS One,2018,13(3):1-16.
[17]王玉荣,折米娜,刘康玲,等.基于MiSeq 高通量测序技术内蒙古地区酸粥细菌多样性研究[J].食品工业科技,2018,39(19):124-129.
[18]FOXMAN B,LUO T,SRINIVASAN U,et al.The effects of family,dentition and dental caries on the salivary microbiome[J].Ann Epidemiol,2016,26(5):348-354.
[19]郭壮,葛东颖,尚雪娇,等.退化和正常窖泥微生物多样性的比较分析[J].食品工业科技,2018,39(22):93-98.
[20]LI G X,MA K M.PICRUSt-based predicted metagenomic analysis of treeline soil bacteria on Mount Dongling,Beijing[J].Acta Ecologica Sinica,2018,38(6):2180-2186.
[21]WANG Y,ZENG X,LIU W.De novo transcriptomic analysis during Lentinula edodes fruiting body growth[J].Gene,2018,641(10):326-334.
[22]单玉鑫,徐文欢,李采婵,等.酸鱼产品中微生物群落结构与品质之间的关系研究[J].食品工业科技,2020,41(6):277-283.
[23]李春生,王悦齐,李来好,等.传统鱼露发酵过程中细菌群落演替及对其挥发性风味形成的影响分析[J].食品科学,2018,39(24):85-92.
[24]游刚,牛改改.复合乳酸菌发酵六齿金线鱼肉的挥发性风味成分探究[J].食品与发酵工业,2016,42(1):167-173.
[25]郑心茹,苏国成,张弦,等.复合乳酸菌发酵鱼肉香肠的工艺及品质研究[J].食品工程,2016,12(4):45-48.