生姜猕猴桃酒是以生姜、猕猴桃为原料,添加一定量白砂糖,以自身附着的微生物在常温条件下自然静置发酵获得的产品。利用微生物对果蔬混合物进行发酵,不仅不会破坏原有的营养物质,而且会使原有的营养物质成倍增加[1]。在发酵过程中,生姜猕猴桃自然酒精发酵醪含有多样性的微生物,包括酵母、细菌等。酵母是发酵过程中重要的功能微生物,影响各种发酵产品的产量和质量[2-4]。
酵母菌是一类真核生物,普遍存在于自然界中。在自然界中目前已发现了1 500多种酵母菌,在比较权威的荷兰微生物菌种保藏中心保藏的酵母菌种类有900种[5]。酵母用于食品和饮料发酵已经上千年,每种特定的发酵过程都有许多不同的酵母菌株,因此许多研究者分析了不同发酵产品中酵母菌的多样性,结果表明,不同的发酵食品酵母菌的分布种属各不相同。蔡燕丽等[6]采用传统分离方法和现代非培养技术聚合酶链反应-变性梯度凝胶电泳(polymerase chain reaction-denatured gradient gel electrophoresis,PCR-DGGE)对不同发酵剂中酵母进行了研究。传统培养方法分离得到的酵母菌5种,分别是酿酒酵母、异常威克汉姆酵母、扣囊复膜孢酵母、德尔布有孢圆酵母和布拉迪酵母。采用PCR-DGGE技术检测到酵母有3种,包括酿酒酵母、异常威克汉姆酵母和扣囊复膜孢酵母。武俊瑞[7]通过选择性纯培养技术方法,从43份传统发酵豆酱样品中,分离筛选出93株酵母菌疑似菌株,进一步采用26S rDNA D1/D2区序列分析对各菌株属种进行鉴定。朱雯娟[8]通过对样品16S rDNA V4可变区的PCR产物的测序结果,进行发酵梅香鱼微生物多样性分析,结果表明,8个门被检测到,分别是酸杆菌门、放线菌门、脱铁杆菌门、厚壁菌门、浮霉菌门、变形菌门、柔膜菌门、及疣微菌门,厚壁菌门是优势菌门。其中乳杆菌属、葡萄球菌属和四联球菌属在梅香鱼发酵中属于优势菌属。
目前关于果蔬发酵以传统的发酵工艺生产为主,受到环境中微生物、发酵体系和季节等条件影响大,容易受到杂菌的污染,发酵产品的质量难以保证[9-11]。传统发酵方式生产的生姜猕猴桃酒是以生姜、猕猴桃为水果原料(氮源),添加大量白砂糖(碳源),仅利用原料自身附着的微生物在常温环境下发酵而制成。因此,为了研究生姜猕猴桃自然酒精发酵醪中的酵母菌系多样性,本实验采用常规培养基、寡营养培养基以及模拟环境培养基从生姜猕猴桃自然酒精发酵醪中分离酵母菌株,并对生姜猕猴桃自然酒精发酵醪酵母菌区系进行多样性研究和生姜猕猴桃自然酒精发酵醪不同时期酵母菌多样性进行分析。研究结果有助于提高生姜猕猴桃酒的质量和风味,为其产业化生产提供理论基础。
1 材料与方法
1.1 材料与试剂
1.1.1 样品
生姜、猕猴桃:市售;生姜猕猴桃自然酒精发酵醪:实验室自制。
1.1.2 化学试剂
无水乙醇(分析纯):国药集团化学试剂有限公司;引物(ITS 1/ITS 4):上海生工生物工程技术服务有限公司合成;酵母脱氧核糖核酸(deoxyribonucleic acid,DNA)提取试剂盒、(2×)Taq PCR Master Mix、4S green核酸染色剂、DNA Marker:上海生工生物工程技术有限公司。
1.1.3 培养基[12]
麦芽膏酵母膏蛋白胨葡萄糖(malt yeast extract peptone glucose,MYPG)琼脂培养基:麦芽浸膏3 g,酵母浸膏3 g,蛋白胨5 g,葡萄糖10 g,琼脂20 g,蒸馏水1 000 mL,pH自然。121 ℃灭菌20 min。
酵母浸出粉胨葡萄糖(yeast extract peptone dextrose,YEPD)培养基:葡萄糖20.0 g,蛋白胨20.0 g,酵母浸膏10.0 g,琼脂20.0 g,蒸馏水1 000 mL,pH 6。121 ℃灭菌20 min。
WL培养基:酵母浸膏4.0 g,胰蛋白胨5.0 g,葡萄糖50.0 g,磷酸二氢钾0.55 g,氯化钾0.425 g,氯化钙0.125 g,硫酸镁0.125 g,氯化铁0.002 5 g,硫酸锰0.002 5 g,琼脂20.0 g,调节pH至6.5。加入溴甲酚绿22.0 mg,蒸馏水1 000 mL。121 ℃灭菌20 min。
麦芽汁琼脂培养基:麦芽浸膏20.0 g,葡萄糖20.0 g,琼脂15.0 g,加热溶于适量水中,蒸馏水1 000 mL,pH自然。115 ℃灭菌30 min。
马铃薯葡萄糖琼脂(potato dextrose agar,PDA)培养基:200 g去皮马铃薯,切成薄片,加入1 L水,煮沸20 min后用纱布过滤。加入20.0 g葡萄糖、15.0 g琼脂,加热溶于适量水中,定容至1 000 mL。pH自然。121 ℃灭菌20 min。
孟加拉红培养基:蛋白胨5.0 g,葡萄糖10.0 g,磷酸二氢钾1.0 g,硫酸镁(MgSO4·7H2O)0.5 g,琼脂20.0 g,加热溶于适量水中,加入1/3 000孟加拉红溶液100 mL。另用少量乙醇溶解氯霉素,加入培养基中,定容至1 000 mL。pH自然。分装后121 ℃灭菌20 min。
酵母粉麦芽糖(yeast maltose,YM)培养基:酪蛋白胨5.0 g,麦芽浸膏3.0 g,葡萄糖10.0 g,琼脂20.0 g,加热溶于适量水中,调节pH至6.0。定容至1 000 mL。分装后115 ℃灭菌30 min。
水琼脂培养基:琼脂粉20.0 g,加热溶于适量蒸馏水中,调节pH至7,定容至1 L。分装后121 ℃灭菌20 min。
1.2 仪器与设备
S1000 PCR扩增仪、Gel Doc XR凝胶成像系统:美国BIO-RAD公司;ABI 3730XL全自动DNA测序仪:美国ABI公司;AllegraX-30RCentrifuge离心机:美国BECKMAN COULTER公司;HWS-250B恒温恒湿培养箱:天津市泰斯特仪器有限公司;GM-K30打浆机:佛山市顺德区格明电器实业有限公司。
1.3 方法
1.3.1 生姜猕猴桃自然酒精发酵醪的制备
生姜、猕猴桃去杂、去坏果,将猕猴桃和生姜清洗后盐水浸泡30 min之后去皮,混合打浆,加入30%的白砂糖后装瓶,在(25±1)℃的温度下自然发酵,生姜猕猴桃自然酒精发酵醪分为发酵初期(plot1)(10 d),发酵中期(plot2)(45 d),发酵后期(plot3)(90 d)3个时期。
1.3.2 酵母菌的分离
取生姜猕猴桃自然酒精发酵醪自然发酵初、中、后3个时期(即发酵第10天、45天、90天)的发酵醪样品10 g,放入盛有90 mL无菌水的三角瓶中,振荡5 min,充分摇匀,稀释制成10-1菌悬液。将制得的菌悬液梯度稀释[12]。分别吸取浓度梯度为10-3、10-4、10-5的菌液100 μL至倒好的平板上。(28±1)℃静置培养2 d,根据菌落大小、形态、颜色、表面的粗细、透明圈、粘稠度、高低、边缘的形状、菌块的质地等表型特征挑取单菌落,转接到相应培养基斜面,(28±1)℃静置培养2 d,用于菌体的制备。
1.3.3 酵母菌5.8S rDNA ITS区序列分析
(1)菌体制备
将于(28±1)℃静置培养24 h左右的酵母斜面菌种,用无菌水洗下菌苔,加入到灭菌离心管中,以10 000 r/min的速度离心15 s,将酵母菌体沉淀物再用无菌水洗涤2次后用于模板制备。
(2)DNA模版制备
使用细菌基因组DNA提取试剂盒,按照说明书方法进行抽提。
(3)5.8S rDNA ITS PCR扩增
以ITS 1(5'-TCC GTA GGT GAA CCT GCG G-3')、ITS 4(5'-TCC TCC GCT TAT TGA TAT GC-3')为引物,以酵母菌总DNA为模板进行PCR扩增。在PCR扩增管中加入DNA模版1 μL,2×Mix13 μL,10 μmol/LITS1 1 μL,10 μmol/L ITS2 1 μL,蒸馏水9 μL,混匀后进行PCR扩增。PCR扩增条件:94 ℃预变性4 min后,94 ℃变性30 s,54 ℃退火30 s,72 ℃延伸1 min,循环30次,再72 ℃延长10 min。
(4)测序方法
DNA测序采用双脱氧终止法。
(5)序列分析
利用DNAman软件拼接序列,启动Seqman,导入目标序列(峰图文件),进行数据修正,采用美国国家生物技术信息中心(nationalcenter for biotechnologyinformation,NCBI)GenBank数据库,下载得分高、序列相似性达到95%以上相关酵母菌株5.8S rDNA ITS序列,选择的菌株尽可能来源于保藏机构。采用MEGA10.0.5软件进行序列比对及聚类分析。
系统发育树构建方法:利用MEGA10.0.5软件的邻接(neighbor-joining,NJ)法重构系统发育树,采用bootstrap检验进行评估,bootstrap检验1 000次。在不同种类的细菌菌株中选取代表菌株,采用NJ法将每一类代表菌株序列与GenBank数据库中的模式菌株序列进行分析,构建系统发育树,获得分离菌株的分类地位和系统发育地位[13-15]。
1.3.4 不同时期生姜猕猴桃自然酒精发酵醪多样性分析
利用R语言(R version 3.5.3),使用Vegan包(Vegan 2.5~4.0)进行生姜猕猴桃自然酒精发酵醪初期、中期、后期的酵母菌群的α多样性分析。分析三个不同发酵阶段的Shannon-Wiener指数、Inverse Simpson指数、Simpson指数、物种累计数、均匀度指数。使用Rattle包(Rattle XXXX 5.2.0)进行生姜猕猴桃自然酒精发酵醪初期、中期、后期的酵母菌群的菌群之间的相关性分析。在https://cloud.tencent.com/developer/article/1474392网页中进行生姜猕猴桃自然酒精发酵醪初期、中期、后期的酵母菌群的β多样性分析。
2 结果与分析
2.1 生姜猕猴桃自然酒精发酵醪期间的酵母菌
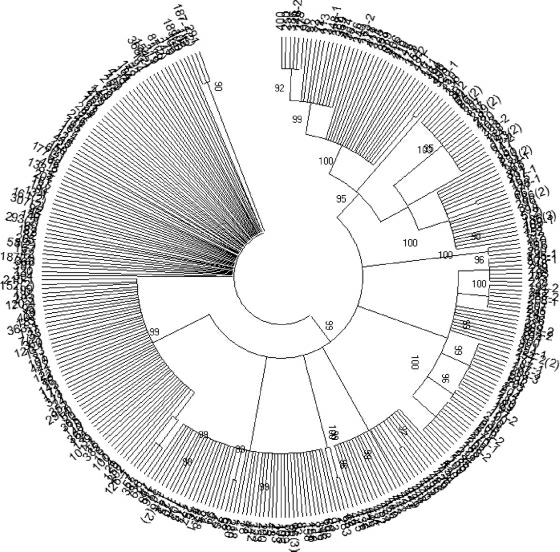
图1 318株酵母菌株基于5.8S rDNA ITS区基因序列的系统发育树
Fig.1 Phylogenetic tree of the 318 yeast strains based on 5.8S rDNA ITS region gene sequence
从生姜猕猴桃自然酒精发酵前期、中期和后期中共分离得到318株酵母菌株,通过对酵母菌株5.8S rDNA序列进行测序,并利用MEGA软件对5.8S rDNA序列进行聚类分析,结果见图1。由图1可知,318株酵母菌株分别聚成了12个不同的独立类群,每个类群进行基本局部比对搜索工具(basic local alignment search tool,BLAST)分析,每个类群的菌株分别和数据库里已知的菌株以95%的相似性聚在一起,12个独立的类群可能归属于12个种。12个独立的类群每一类群选择1个或2个菌株作为代表菌株进行序列比对分析,构建系统发育树,确定其分类地位和系统发育地位,其中选取的酵母菌的代表菌株号分别为290、186(2)、186(3)、136、240、356、452、394、112、1、283、36、208-2、378。
2.2 12个酵母菌类群的鉴定
采用NJ法重建系统发育树将酵母菌的代表菌株进行系统发育分析,结果分别见图2和图3。

图2 酵母菌株基于5.8S rDNA ITS基因序列的系统发育树
Fig.2 Phylogenetic tree of yeast strains based on 5.8S rDNA ITS gene sequence
由图2可知,菌株3-1被鉴定为膜醭毕赤酵母(Pichia membranifaciens),菌株290、186(2)、186(3)被鉴定为克鲁维毕赤酵母(Pichia kluyveri),菌株136被鉴定为梅奇酵母(Metschnikowia ziziphicola),菌株240被鉴定为奥莫柯达酵母(Kodamaea ohmeri),菌株356被鉴定为葡萄汁有孢汉逊酵母(Hanseniaspora uvarum),菌株452被鉴定为酿酒酵母(Saccharomyces cerevisiae),菌株394和菌株112被鉴定为异常毕赤酵母(Wickerhamomyces anomalus),菌株1被鉴定为卡利比克迈耶氏酵母(Meyerozyma caribbica),菌株283被鉴定为德巴利酵母(Debaryomyces nepalensis),菌株36被鉴定为桔假丝酵母(Candida quercitrusa)。其中Myerozyma是由KURTZMAN C P等[16]在对能够产生辅酶Q-9的Debaryomyces、Lodderomyces、Spathaspora、Yamadazyma属酵母以及Pichia、Candida酵母属部分酵母进行了26S rDNA D1/D2区序列分析和系统发育研究后提出的新属,并将Pichia carribica归于此属,称为Myerozyma caribbica[17-18]。
由图3可知,菌株208-2被鉴定为假丝酵母(Candida carpophila),菌株378被鉴定为奥莫柯达酵母(Kodamaea ohmeri)。

图3 菌株208-2和378基于5.8S rDNA ITS基因序列的系统发育树
Fig.3 Phylogenetic tree of strain 208-2 and 378 based on 5.8S rDNA ITS gene sequences
2.3 不同时期生姜猕猴桃自然酒精发酵醪中酵母菌群α多样性分析
2.3.1 α多样性分析
统计不同时期酵母菌株,结果见表1,不同时期酵母菌株通过R语言可视化结果见图4。
表1 不同发酵时期酵母菌株的分布
Table 1 Distribution of yeast strains in different fermentation periods

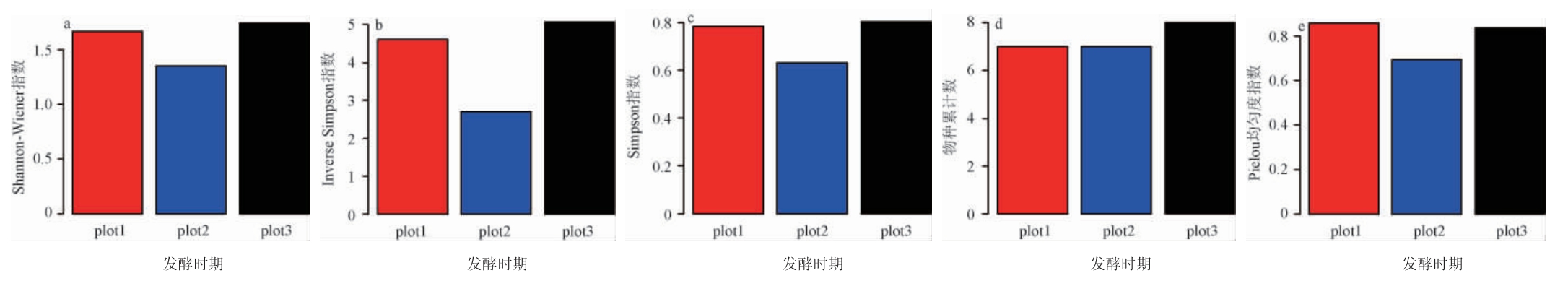
图4 不同组间酵母菌群α多样性指数差异
Fig.4 Differences in α diversity indices of yeast strains between different groups
由图4可知,在生姜猕猴桃酒发酵的不同时期,Shannon-Wiener指数表明生姜猕猴淘酒发酵后期物种多样性较高,中期较低,综合考虑Inverse Simpson 指数和Simpson指数,物种多样性发酵中期>发酵前期>发酵后期。物种累计数(S)表明,生姜猕猴桃酒发酵后期的物种累计数较高。Pielou均匀度指数显示,发酵前期的物种均一度最高,发酵后期的物种均一度较低,发酵中期的物种均一度最低。
2.3.2 β多样性分析
Venn图可用于统计多个样品中所共有的和独有的酵母菌数目。3个不同发酵阶段之间的酵母菌的差异情况见图5。
由图5可知,发酵后期(plot3)酵母菌数目与独有酵母菌数目最多,物种丰富度最高。发酵前期(plot1)和发酵中期(plot2)酵母菌数目与独有酵母菌数目相似。发酵前期(plot1)、发酵中期(plot2)、发酵后期(plot3)共有的酵母菌数为2个,发酵前期(plot1)和发酵中期(plot2)共有的酵母菌株为1个,发酵前期(plot1)和发酵后期(plot3)共有的酵母菌数为1个。
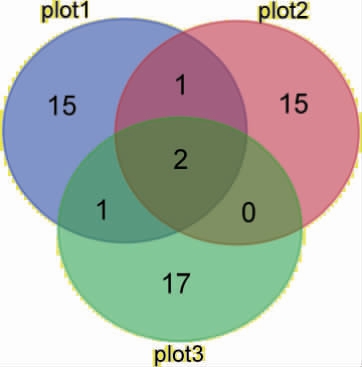
图5 不同发酵时期物种多样性的韦恩图分析
Fig.5 Analysis of Venn diagram of diversity of species in different fermentation periods
3 讨论
酵母在发酵的过程中能产生不同的代谢物质,研究表明Debaryomyces nepalensis产生乙醇、甘油、阿拉伯糖醇和木糖醇代谢物[19]。毕赤酵母(Pichia stipitis)、假丝酵母(Candida shehatae)和嗜鞣管囊酵母(Pachysolen tannophilus)的酵母菌株能够将葡萄糖和木糖发酵成乙醇和木糖醇等代谢物质[20]。经全球代谢物谱分析表明酿酒酵母(Saccharomyces cerevisiae)可以产生丙酮酸、鸟氨酸-1,5-内酰胺、缬氨酸、天冬氨酸、正亮氨酸、甘氨酸、赖氨酸、呋喃-2-羧酸、天冬酰胺和甘氨酰-脯氨酸等代谢物质[21]。在发酵中产生的代谢物质将影响发酵食品的风味,例如代谢物质有机酸、二乙酰,来自支链氨基酸的高级醇和由其衍生的酯等。
通过微生物可培养分析生姜猕猴桃自然酒精发酵醪中的酵母菌群多样性,了解生姜猕猴桃发酵过程中酵母菌系多样性有助于分析之后发酵过程中产生的各种代谢产物来源。为生姜猕猴桃自然酒精发酵醪安全评估提供理论基础,为之后果蔬发酵机制研究提供一定的理论基础和意义。
4 结论
通过5.8S rDNA ITS序列进行多序列比对分析方法对生姜猕猴桃自然酒精发酵醪进行酵母菌多样性分析得出,在分离的318株酵母菌株中,12个细菌类群分属于8属11种,包括膜醭毕赤酵母(Pichia membranifaciens),克鲁维毕赤酵母(Pichia kluyveri),梅奇酵母(Metschnikowia ziziphicola),奥莫柯达酵母(Kodamaea ohmeri),葡萄汁有孢汉逊酵母(Hanseniaspora uvarum),酿酒酵母(Saccharomyces cerevisiae),异常毕赤酵母(Wickerhamomyces anomalus),Meyerozyma caribbica,德巴利酵母(Debaryomyces nepalensis),桔假丝酵母(Candida quercitrusa),假丝酵母(Candida carpophila),说明果蔬自然发酵过程中存在丰富的酵母菌群。
通过对生姜猕猴桃自然酒精发酵醪的不同时期进行多样性分析,α多样性分析结果显示,生姜猕猴桃自然酒精发酵醪发酵中期的酵母多样性最高,发酵后期的物种累计数较高,发酵前期的物种均一度最高。β多样性分析结果显示生姜猕猴桃自然酒精发酵醪中发酵后期的物种丰富度最高,三个发酵阶段共有的酵母菌菌数为2个。
[1]蔡爽.人参术苓酵素的制备及改善肠胃功能研究[D].吉林:吉林大学,2013.
[2]VERBELEN P J,DEKONINCK T M L,SAERENS S M G,et al.Impact of pitching rate on yeast fermentation performance and beer flavour[J]. Appl Microbiol Biotechnol,2009,82(1):155-167.
[3]VERGINER M,LEITNER E,BERG G.Production of volatile metabolites by grape-associated microorganisms[J].J Agr Food Chem,2010,58(14):8344-8350.
[4]CHEN S,XU Y.The influence of yeast strains on the volatile flavour compounds of Chinese rice wine[J].J I Brewing,2010,116(2):190-196.
[5]王庆国,刘天明.酵母菌分类学方法研究进展[J].微生物学杂志,2007,27(3):96-101.
[6]蔡燕丽.新疆桃园酵母菌多样性研究及其资源评估[D].石河子:石河子大学,2018.
[7]武俊瑞.东北传统发酵特色食品中主要微生物多样性研究[D].沈阳:沈阳农业大学,2013.
[8]朱雯娟.发酵梅香鱼微生物种群结构分析及发酵菌株筛选研究[D].广州:广东海洋大学,2015.
[9]蒋增良.天然微生物酵素发酵机理、代谢过程及生物活性研究[D].杭州:浙江理工大学,2012.
[10]CHOI M A.Effects of saccharides and incubation temperature on pH and total acidityof fermented black tea with tea fungus[J].Kor J Food SciTechnol,1996,28(3):405-410.
[11]LIU C H,HSU W H,LEE F L,et al.The isolation and identification of microbes from a fermented tea beverage,Haipao,and their interactions during Haipao fermentation[J].Food Microbiol,1996,13(6):407-415.
[12]薛军侠,徐艳文,杨莹,等.WL 培养基在酿酒酵母筛选中的应用[J].中国酿造,2007,26(9):36-39.
[13]LARKIN M A,BLACKSHIELDS G,BROWN N P,et al.Clustal W and Clustal X version 2.0[J].Bioinformatics,2007,23(21):2947-2948.
[14]TAMURA K,STECHER G,PETERSON D,et al.MEGA6:molecular evolutionary genetics analysis Version 6.0[J]. Mol Biol Evolut,2013,30(12):2725-2729.
[15]MORGULIS A,COULOURIS G,RAYTSELIS Y,et al.Database indexing for production Mega BLAST searches[J].Bioinformatics,2008,24:1757-1764.
[16]KURTZMAN C P,ROBNETT C J.Relationships among genera of the Saccharomycotina (Ascomycota) from multigene phylogenetic analysis of type species[J].Fems Yeast Res,2013,1:23-33.
[17]VAUGHAN-MARTINI A,KURTZMAN C P,MEYER S A,et al.Two new species in the Pichia guilliermondii clade: Pichia caribbica sp.nov.,the ascosporic state of Candida fermentati,and Candida carpophila comb.Nov[J].FEMS Yeast Res,2005,5(4-5):463-469.
[18]KURTZMAN C P,SUZUKI M.Phylogenetic analysis of ascomycete yeasts that form coenzyme Q-9 and the proposal of the new genera Babjeviella,Meyerozyma, Millerozyma, Priceomyces,and Scheffersomyces[J]. Mycoscience,2010,51(1):2-14.
[19]KUMAR S,GUMMADI S N.Metabolism of glucose and xylose as single and mixed feed in Debaryomyces nepalensis NCYC 3413:production of industrially important metabolites[J].Appl Microbiol Biotechn,2011,89(5):1405-1415.
[20]SÁNCHEZ,S,BRAVO V,CASTRO E,et al.The fermentation of mixtures of D-glucose and D-xylose by Candida shehatae,Pichia stipitis or Pachysolen tannophilus to produce ethanol[J]. J Chem Technol Biotechn,2002,77(6):641-648.
[21]SHIN M,KIM J W,YE S,et al.Comparative global metabolite profiling of xylose-fermenting Saccharomyces cerevisiae SR8 and Scheffersomyces stipitis[J].Appl Microbiol Biotechn,2019,103(13):5435-5446.