2,3-丁二醇(2,3-butanediol,2,3-BD)及其衍生物作为一种应用极其广泛的化工原料,社会需求量大[1-3]。一直以来,传统诱变技术存在着正向突变频率低,难以控制其变异的方向和性质,以及回复突变等问题[4-5]。与传统的育种方法相比,使用代谢工程手段改造菌株引起了众多2,3-丁二醇研究者的关注。典型的2,3-丁二醇代谢途径是一个产物以2,3-丁二醇为主,乳酸、乙酸、和琥珀酸等混合酸为副产物的生成过程,因此,混合酸发酵过程中副产物的关键酶基因敲除与目标产物合成支路的关键酶基因强化表达就顺理成章的成为定向改造菌种的常规手段[6-15]。
乳酸是克雷伯氏菌(Klebsiella)以葡萄糖为底物发酵代谢生产2,3-丁二醇的主要副产物之一,其代谢支路是由乳酸脱氢酶(lactate dehydrogenase,LDH)催化丙酮酸而产生,乳酸的大量合成会改变发酵液环境中pH,并对菌体细胞产生毒害作用而影响菌体生长,进而影响2,3-丁二醇的产量[16-17]。因此,减少乃至消除乳酸的产生将是一种通过微生物代谢调控提高2,3-丁二醇产量的理想策略[18-22]。JI X J等[23]采用Red同源重组技术敲除产酸克雷伯氏菌(Klebsiella oxytoca)ME-UD-3中的乙醇脱氢酶基因,获得了乙醇脱氢酶缺失重组菌株。结果表明,乙醇脱氢酶基因敲除后,在以葡萄糖为底物发酵生产2,3-丁二醇的产量为90.75 g/L,同比原始菌株的87.67 g/L提高了3.5%。同时副产物乙偶姻的产量由2.73 g/L下降到0.98 g/L,降低了64%。JUNG M Y等[24]采用Red同源重组技术成功敲除产气肠杆菌(Enterobacter aerogenes)KCTC2190的乳酸脱氢酶基因,通过蔗糖蜜为底物发酵结果显示,在发酵40 h后发酵液中未检出乳酸含量,而2,3-丁二醇的产量为13.25 g/L,对比原始菌株2,3-丁二醇产量10.02 g/L提高了32.3%。
由此可见,利用基因工程手段靶向性地实现2,3-丁二醇高产是一种切实可行的方法。为研究乳酸含量对2,3-丁二醇产量的影响,本研究利用Red同源重组技术敲除乳酸脱氢酶(ldh)基因,企图摸索一套完善的产酸克雷伯氏菌(K.oxytoca)HD79乳酸脱氢酶基因缺失菌株的构建方法,从而为提高2,3-丁二醇的产量及增加工业菌种选择提供依据。
1 材料与方法
1.1 材料与试剂
1.1.1 菌种与质粒
菌种产酸克雷伯氏菌(K.oxytoca)HD79、E.coli DH5α、质粒pGP704-Cm和pKD46:黑龙江大学微生物重点实验室提供;pMDTM18-T:大连宝生物有限公司。
1.1.2 试剂
XhoI、BamHI、BglII、Pfu脱氧核糖核酸(deoxyribonucleic acid,DNA)聚合酶、T4 DNA连接酶、氨苄青霉素、氯霉素和L-阿拉伯糖:大连宝生物有限公司;细菌基因组DNA抽提试剂盒、质粒抽提试剂盒、胶回收试剂盒、核糖核酸(ribonucleic acid,RNA)prep Pure细菌总RNA提取试剂盒、反转录-聚合酶链式反应(reverse transcription-polymerase chain reaction,RT-PCR)试剂盒、RealMasterMix(SYBR Green):北京天根生化科技有限公司。
1.1.3 引物
相关的扩增引物由大连宝生物有限公司合成,如表1所示。
表1 本研究所用引物序列
Table 1 Sequences of primer used in the study

1.1.4 培养基
Luria-Bertani(LB)培养基:蛋白胨10 g/L,酵母提取物5 g/L,NaCl 10 g/L,pH调至7.0,121 ℃灭菌15 min。
发酵培养基:(NH4)2SO4 6.6 g/L,K2HPO4·3H2O 8.7 g/L,KH2PO4 6.8 g/L,MgSO4·7H2O 0.25 g/L,酵母提取物5 g/L,FeSO4·7H2O 0.05 g/L,ZnSO4·7H2O 0.001 g/L,MnSO4·H2O 0.001 g/L,CaCl2·2H2O 0.001 g/L。pH值至7.0,121 ℃灭菌15 min并与108 ℃灭菌20 min的150 g/L葡萄糖粉末在无菌条件下混合。
1.2 仪器与设备
JT-150双人超净工作台:沈阳医用净化设备厂;MLS-3780高压蒸汽灭菌锅:日本SANYO公司;ZHWY-211C全温度恒温摇床:上海智城分析仪器制造有限公司;Beckman Coulter Allegar® X-15R离心机:德国Beckman公司;HH-II-420-S恒温培养箱:上海跃进医疗器械厂;HPX-87H色谱柱(300mm×7.8mm)、GenePulserXcell电穿孔仪:美国BIO-RAD公司;SCL-10A高效液相色谱(high performance liquid chromatography,HPLC)仪:日本岛津公司;ABI7500荧光定量PCR仪:美国Life Technologies公司。
1.3 方法
1.3.1 基因元件的克隆
以K.oxytoca HD79基因组和质粒pGP704-Cm为模板,ldh-L1/ldh-L2、ldh-R1/ldh-R2和Cm-F/Cm-R为引物分别对乳酸脱氢酶和氯霉素抗性基因进行PCR扩增。PCR扩增体系:模板DNA 2 μL,上下游引物(1 pmol/μL)各1 μL,PrimeSTAR Max Premix(2X)12.5 μL,ddH2O 8.5 μL。PCR扩增程序:94 ℃预变性3 min;98 ℃变性60 s,57 ℃退火60 s,72 ℃延伸60 s,共30个循环;72 ℃终延伸5 min。
1.3.2 线性片段ldhL-Cmr-ldhR的构建
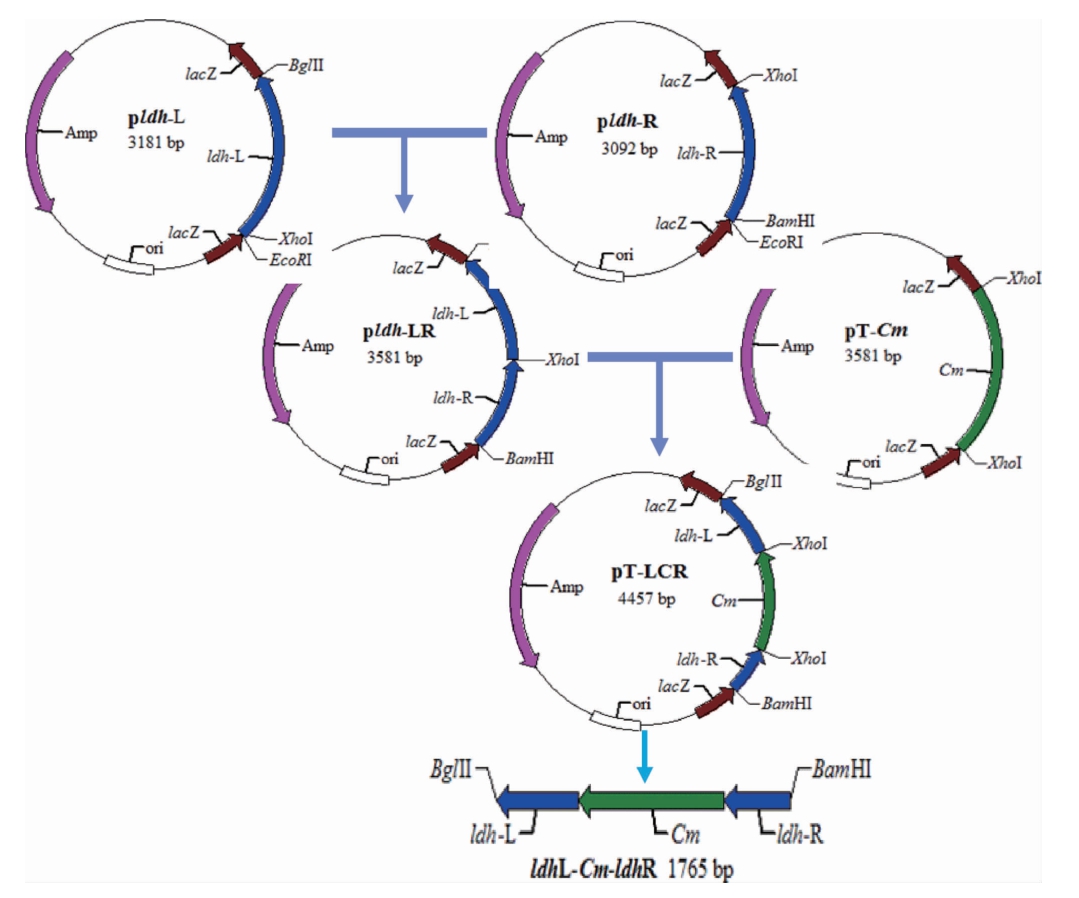
图1 同源重组片段ldhL-Cmr-ldhR完整构建策略
Fig.1 Whole construction strategy of homologous recombination fragment ldhL-Cmr-ldhR
线性片段ldhL-Cmr-ldhR的构建思路如图1所示,先行构建质粒pldh-LR和pT-LCR,其中pldh-LR是将ldh基因的左右同源臂拼接的重组质粒;而pT-LCR是以pldh-LR为骨架,在ldh基因的同源臂中间插入替换基因Cmr;然后通过酶切获得线性片段ldhL-Cmr-ldhR,用于后续的电转化试验。
1.3.3 电转化
(1)同源辅助质粒pKD46转化K.oxytoca HD79
pKD46转化K.oxytoca HD79的具体实验方法见参考文献[25]。
(2)线性片段ldhL-Cmr-ldhR转化K.oxytocaHD79/pKD46
将ldhL-Cmr-ldhR转化到经5 mmol/L L-阿拉伯糖诱导的含有质粒pKD46的K.oxytoca HD79宿主菌中,使其与宿主菌中的ldh基因发生同源重组,以达到敲除ldh基因的目的。ldhL-Cmr-ldhR转化K.oxytoca HD79/pKD46方法同上。
1.3.4 同源重组阳性转化子的检测
在终质量浓度为150 μg/mL的氯霉素平板上挑取4个白色菌落,接种到20 mL LB液体培养基,30 ℃、150 r/min培养过夜。以阳性转化子菌液为模板进行ldhL-Cmr-ldhR的两端引物ldh-L1和ldh-R2验证,并将正确的PCR产物送交哈尔滨博仕生物科技公司进行测序分析。
1.3.5 荧光定量PCR测定重组菌株ldh基因表达
设置供试菌株对照组和ldh敲除实验组,各组分别取2μL互补脱氧核糖核酸(complementary deoxyribonucleic acid,cDNA)作模板,以16S rRNA和ldh-R特异性引物进行荧光定量-聚合酶链式反应(fluorogenic quantitative-polymerase chain reaction,FQ-PCR),获得各样品的临界循环数(cycle threshold,CT)值,采用2-ΔΔCT相对定量法分析,以内参基因16S rRNA校正[26]。ΔCT=待测样品的目的基因CT均值—对应内参基因的CT均值;ΔΔCT的计算公式如下:
ΔΔCT=各组ΔCT—对照组ΔCT
经2-ΔΔCT计算后,将对照组中目的基因表达量设为1,其余各组相对于对照组的基因表达量的2-ΔΔCT。得出待测样品的CT值,以CT值以“平均数±标准差”表示。
1.3.6 重组菌株发酵产醇能力测定
重组菌株和供试菌株分别接种到种子液培养基中,30 ℃、150 r/min培养至对数生长期,以5%接种量分别接种到装液量为150 mL/500 mL发酵培养基中,底物葡萄糖150 g/L,30 ℃、150 r/min发酵156 h后结束。间隔12 h取样,样品经处理后,经高效液相色谱检测乳酸、2,3-丁二醇及其他副产物的产量。
HPLC检测条件:HPX-87H色谱柱(300 mm×7.8 mm),RID-10A示差检测器,进样量20 μL,以0.005 mol/L稀硫酸为流动相,流速0.8 mL/min,示差检测器温度40 ℃,色谱柱柱温65 ℃,分析时间18 min。
1.3.7 数据处理
试验处理均设置3个重复,数据以“平均值±标准差”形式表示,统计检验的显著水平设定为0.05,极显著水平设定为0.01。利用Duncan's新负极差进行分析及多重比较,不同字母表示处理间差异显著。
2 结果与分析
2.1 基因元件的克隆
1%琼脂糖凝胶电泳检测结果如图2a-c所示:PCR产物ldh-L、ldh-R和Cmr与目的片段设计大小相符,分别为489 bp、400 bp和876 bp。通过测序发现扩增所得的核苷酸序列与美国国家生物技术信息中心(national center of biotechnology information,NCBI)数据平台上公布的基因序列比对一致,确保扩增序列无突变。

图2 ldh-L片段(a)、ldh-R片段(b)、Cmr片段(c)及ldhL-Cmr-ldhR(d)PCR扩增结果
Fig.2 PCR amplification results of ldh-L (a), ldh-R (b), Cmr (c) and ldhL-Cmr-ldhR (d) fragment
2.2 阳性转化子筛选
辅助质粒pKD46电转化至K.oxytoca HD79筛选结果如图3a如示,质粒pKD46转化效率良好。线性片段ldhL-CmrldhR电转化至K.oxytoca HD79/pKD46筛选结果如3b所示,在终质量浓度150 μg/mL的氯霉素平板上随机挑选4个阳性转化子,再次接种在新的终质量浓度150 μg/mL的氯霉素平板上,结果仍为阳性。

图3 电转化筛选结果
Fig.3 Screening results of electrotransformation
(a)质粒pKD46转化K.oxytoca HD79;(b)ldhL-Cmr-ldhR转化K.oxytoca HD79/pKD46。
2.3 ldh基因缺失突变株鉴定
PCR验证结果如图2d所示,泳道1~4均在约2 000 bp处有一清晰条带,与ldhL-Cmr-ldhR即ldh-LR、Cmr片段大小之和相符,为1 765 bp。结合测序结果比对分析表明重组片段ldhL-Cmr-ldhR整合到宿主菌K.oxytoca HD79的基因组上;初步说明ldh基因缺失突变株构建成功。
2.4 荧光定量PCR测定重组菌株ldh基因表达
ldh基因在重组菌株中相对表达量的结果如图4所示,重组菌株中的ldh基因相对表达量在24 h、48 h和72 h均明显低于供试菌株,同比分别降低了76%、74%和66%,差异显著(P <0.05)。说明了敲除基因ldh能有效地降低基因ldh的表达。

图4 FQ-PCR测定不同时间处理组中ldh基因的相对表达
Fig.4 Relative expression of ldh gene in different time treatment groups by FQ-PCR
2.5 重组菌株摇瓶发酵产醇能力测定
摇瓶发酵产物质量浓度通过高效液相色谱(HPLC)法测定结果如表2所示,重组菌株相对供试菌株来说,其乳酸产量明显降低,而2,3-丁二醇的产量增加,说明试验基本达到预期敲除ldh基因的目的。重组菌株2,3-丁二醇最高产量比供试菌株提高了26.8%,2,3-丁二醇最高产量的积累时间也提前了12 h,而乳酸的最高产量同比则降低了49.3%。有趣的是,1,3-丙二醇的最高产量由5.33 g/L增加到8.04 g/L,提高了50.9%。此外,其他副产物乙酸、琥珀酸及乙醇的产量变化不大,差异不显著。
表2 供试菌株和重组菌株的摇瓶发酵代谢产物峰值
Table 2 Peak value of metabolites of tested strain and recombinant strain by flask fermentation

3 讨论
同源辅助质粒pKD46在线性片段ldhL-Cmr-ldhR转化至宿主菌中起至关重要的作用。pKD46表达λRed系统发生同源重组所需要的Exo、Bet与Gam三种蛋白因子,而Gam蛋白作为一种保护外源DNA的多肽分子,可与宿主菌中自身重组系统的RecBCD核酸外切酶结合,抑制其对外源DNA的降解。此外,pKD46是一种温敏型质粒,当温度超过37 ℃时,容易自动丢失,因此,为了获得阳性转化菌株,实验培养温度控制在30 ℃。
菌体代谢途径关键酶基因敲除往往会在一定程度上对菌体生长产生影响,这在许多关于克雷伯氏菌基因敲除研究报道中得到证实[27]。本研究中对产酸克雷伯氏菌乳酸脱氢酶基因的敲除出现了相类似的情况,即获得的乳酸脱氢酶缺失重组菌株相对供试菌株生长缓慢且2,3-丁二醇产量较低。微生物发酵法生产2,3-丁二醇,其产量受到很多因素的影响和限制,其中影响较大的有pH值、菌种、发酵方式和发酵培养基成分配比等,尤其是培养基成分中的磷酸盐配比对酸性产物缺失的重组菌株生长及2,3-丁二醇产量有重要的影响[28-29]。必须通过对培养基成分进行优化,获得最佳的成分配比,以提高目的产物的底物转化率和生产强度。基于课题组前期的研究工作基础,通过改变磷酸盐的比例,确立了最佳培养基成份配比(详见1.1.4发酵培养基),极大地改善重组菌株的生长及2,3-丁二醇的产量。
乳酸作为产酸克雷伯氏菌生产2,3-丁二醇过程中的主要副产物之一,并与2,3-丁二醇代谢支路竞争还原型烟酰胺腺嘌呤二核苷酸(nicotinamide adenine dinucleotide_reduced,NADH),在一定程度上降低了2,3-丁二醇的产量。本研究利用Red同源重组技术将K.oxytoca HD79的乳酸脱氢酶基因敲除,从根本上减少乳酸的积累,以期能提高2,3-丁二醇的产量。但最终所构建成功的ldh缺失重组菌株在以150 g/L葡萄糖为底物摇瓶发酵试验中发现,乳酸的产量只比供试菌株降低了49.3%,并没有表现出产乳酸功能完全丧失的预期现象,其原因可能是产酸克雷伯氏菌中存在乳酸脱氢酶同工酶。结合国外的研究发现,缺失重组菌株基于维持其正常的生理代谢功能,所以基因敲除的效果往往不一定表现出相关产物合成能力完全丢失的性状,可能是相关产物产量处于一个较低的合成水平[30-31]。
4 结论
本研究通过构建K.oxytoca HD79乳酸脱氢酶基因缺失重组菌株,以考察阻断乳酸代谢途径对2,3-丁二醇产量的影响。对K.oxytoca HD79乳酸脱氢酶基因缺失菌株进行摇瓶发酵实验发现,其比供试菌株的2,3-丁二醇产量提高26.8%,而乳酸产量则降低了49.3%,说明ldh基因的缺失对K.oxytoca HD79的2,3-丁二醇产量有影响,对构建高产2,3-丁二醇基因工程菌株扮演着重要角色。
[1]BIALKOWSKA A M.Strategies for efficient and economical 2,3-butanediol production:New trends in this field[J].World J Microbiol Biotechn,2016,32(12):200-210.
[2]王家旺,邓利廷,孙健.等.产2,3-丁二醇的酿酒酵母菌的筛选及其鉴定[J].中国酿造,2020,39(1):21-25.
[3]叶广彬,郭睿,孙珊珊,等.λRed 技术构建产酸克雷伯氏菌乙酸激酶基因缺失突变株[J].黑龙江大学自然科学学报,2018,35(3):334-341.
[4]CHO S,KIM T,WOO H M,et al.High production of 2,3-butanediol from biodiesel-derived crude glycerol by metabolically engineered Klebsiella oxytoca M1[J].Biotech Biofuels,2015,146(8):2-12.
[5]XIE N Z,CHEN X R,WANG Q Y,et al.Microbial routes to(2R,3R)-2,3-butanediol:Recent advances and future prospects[J].Curr Top Med Chem,2017,48(11):1610-1617.
[6]DAI J Y,ZHAO P,CHENG X L,et al.Enhanced production of 2,3-butanediol from sugarcane molasses[J]. Appl Biochem Biotech,2015,175(6):3014-3024.
[7]JI X J,LIU L G,SHEN M Q,et al.Constructing a synthetic metabolic pathway in Escherichia coli to produce the enantiomerically pure(R,R)-2,3-butanediol[J].Biotechnol Bioeng,2015,112(5):1056-1059.
[8]CHO S,KIM T,WOO H M,et al.Enhanced 2,3-butanediol production by optimizing fermentation conditions and engineering Klebsiella oxytoca M1 through overexpression of acetoin reductase[J]. PLoS One,2015,10(9):1-16.
[9]ZHANG L,CAO C,JIANG R F,et al.Production of R,R-2,3-butanediol of ultra-high optical purity from Paenibacillus polymyxa ZJ-9 using homologous recombination[J]. Bioresource Technol,2018,261:272-278.
[10]郭睿,曹春振,李娜,等.合成2,3-丁二醇的肺炎克雷伯氏菌菌株的筛选及改造[J].黑龙江大学自然科学学报,2019,36(3):313-319.
[11]YAMADA R,WAKITA K,MITSUI R,et al.Efficient production of 2,3-butanediol by recombinant Saccharomyces cerevisiae through modulation of gene expression by cocktail δ-integration[J]. Bioresource Technol,2017,245(Part B):1558-1566.
[12]YANG Z L,ZHANG Z S.Production of (2R,3R)-2,3-butanediol using engineered Pichia pastoris:strain construction,characterization and fermentation[J].Biotech Biofuels,2018,11(35):2-16.
[13]KIM S J,KIM J W,LEE Y G,et al.Metabolic engineering of Saccharomyces cerevisiae for 2,3-butanediol production[J]. Appl Microbiol Biot,2017,101(6):2241-2250.
[14]刘德安,王长丽,丁昊,等.代谢工程改造酿酒酵母生产2,3-丁二醇的研究进展[J].中国酿造,2018,37(9):6-11.
[15]TONG Y J,JI X J,SHEN M Q,et al.Constructing a synthetic constitutive metabolic pathway in Escherichia coli for (R,R)-2,3-butanediol production[J].Appl Microbiol Biot,2016,100(2):637-647.
[16]GAO Y B,HUANG H H,CHEN S W,et al.Production of optically pure 2,3-butanediol from Miscanthus floridulus hydrolysate using engineered Bacillus licheniformis strains[J].World J Microbiol Biotechn,2018,34(5):1-10.
[17]SUN S,ZHANG H,LU S,et al.The metabolic flux regulation of Klebsiella pneumoniae based on quorum sensing system[J].Sci Rep,2016,6(1):1-9.
[18]YANG M,YUN J,ZHANG H,et al.Genetically engineered strains:Application and advances for 1,3-propanediol production from glycerol[J].Food Technol Biotech,2018,56(1):3-15.
[19]NGUYEN A D,HWANG I Y,LEE O K,et al.Systematic metabolic engineering of Methylomicrobium alcaliphilum 20Z for 2,3-butanediol production from methane[J].Metab Eng,2018,47:323-333.
[20]PARK J M,RATHNASINGH C,SONG H.Enhanced production of (R,R)-2,3-butanediol bymetabolicallyengineered Klebsiella oxytoca[J].J Ind Microbiol Biot,2015,42(10):1419-1425.
[21]JEONG D,YANG J,LEE S,et al.Deletion of the budBAC operon in Klebsiella pneumoniae to understand the physiological role of 2,3-butanediol biosynthesis[J].Prep Biochem Biotech,2016,46(4):410-419.
[22]YANG T W,RAO Z M,ZHANG X A,et al.Metabolic engineering strategies for acetoin and 2,3-butanediol production:Advances and prospects[J].Crit Rev Biotechnol,2017,37(8):990-1005.
[23]JI X J,HUANG H,ZHU J G,et al.Engineered Klebsiella oxytoca for efficient 2,3-butanediol production through insertional inactivation of ethanol dehydrogenation gene[J].Appl Microbiol Biotechnol,2010,85(6):1751-1758.
[24]JUNG M Y,PARK B S,LEE J W,et al.Engineered Enterobacter aerogenes for efficient utilization of sugarcane molasses in 2,3-butanediol production[J].Appl Microbiol Biot,2013,139(1):21-27.
[25]SAMBROOK J,RUSSELL D W.分子克隆实验指南(第三版)(上册)[M].北京:科学出版社,2002:99-102.
[26]贺婷婷,宋婷,王超,等.短小芽孢杆菌实时荧光定量PCR 分析中内参基因的筛选[J].生物技术通报,2016,32(11):99-106.
[27]郭欣坤,方慧英,诸葛斌,等.2,3-丁二醇代谢途径关键酶基因敲除对克雷伯氏菌发酵产1,3-丙二醇的影响[J].生物工程学报,2013,29(9):1290-1300.
[28]REBECCHI S,PINELLI D,ZANAROLI G,et al.Effect of oxygen mass transfer rate on the production of 2,3-butanediol from glucose and agroindustrial byproducts by Bacillus licheniformis ATCC9789[J]. Biotech Biofuels,2018,45(4):613-616.
[29]EUN J C,KIM S J,KIM J W,et al.Enhanced production of 2,3-butanediol in pyruvate decarboxylase-deficient Saccharomyces cerevisiae through optimizing ratio of glucose/galactose[J]. Botechnol J,2016,11(11):1424-1432.
[30]GE J P,WANG J W,YE G B,et al.Disruption of the lactate dehydrogenase and acetate kinase genes in Klebsiella pneumoniae HD79 to enhance 2,3-butanediol production,and related transcriptomics analysis[J].Biotechnol lett,2020,42(4):537-549.
[31]KUIT W,MINTON N P,LOPEZ-CONTRELAS A M.Disruption of the acetate kinase(ack)gene of Clostridium acetobutylicum results in delayed acetate production[J].Appl Microbiol Biot,2012,94:729-741.