酪乳和酸奶作为发酵牛奶制品香甜可口,营养丰富,含多种人体必需的蛋白质、脂肪、碳水化合物、氨基酸、维生素、矿物质等。其发酵风味不仅取决于鲜奶的种类和品质,同样也与发酵过程中的微生物代谢息息相关[1-2]。微生物够利用鲜奶中的各种营养物质进行发酵活动,进而产生各种不同的风味[3-5]。西藏林芝地区的传统发酵制品在以肉为主的林芝人日常生活中扮演着不可或缺的角色[6]。但目前为自制酸奶,人尽皆知的是自制酸奶的环境是相对分散和开放的,环境中存在大量微生物,有一些微生物并不能对酸奶的发酵产生有利的影响,环境存在的某些有害微生物反而会导致酸奶的品质下降,影响食用甚至造成危害[7]。酸奶发酵主要用的微生物是乳酸菌,而传统的乳酸菌检测方法主要是通过形态观察、革兰氏染色和生化反应等进行分析,操作简单容易得到结果。但无法用于细菌大批量分析鉴定[8-9]。随着测序技术的不断发展,高通量测序(highthroughput sequencing,HTS)技术被称为下一代测序技术,由于成本低、可读量和质量高,是目前评估微生物多样性的有力工具[10-11]。该技术越来越受到国内外学者的重视,梁明明等[12]通过研究微生物的遗传稳定性结合感官评价机制成功筛选出了优质乳酸菌株,杨行等[13]通过基因比对技术分析出新疆喀什地区的酸奶中的优势菌种为屎肠球菌(Enterococcus faecium),吴均等[14-15]通过对分离出的乳酸菌株进行16S核糖体脱氧核糖核苷酸(16S ribosome deoxyribonucleic acid,16S rDNA)鉴定得出菌株TG1-1为干酪乳杆菌或副干酪乳杆菌,菌株TG1-11为副干酪乳杆菌。
西藏林芝地理位置优越,生物资源丰富,生态地理环境复杂多样,蕴含着丰富的微生物资源,目前未见高通量测序技术对林芝传统发酵型酸奶及酪乳相关的研究。为了更好地利用这些微生物资源,本研究采用Illumina Miseq高通量测序技术,对林芝牧民所食用的传统酸奶和酪乳中微生物进行分析揭示酸奶中的微生物多样性,并对细菌群落类型、结构和优势菌群进行研究和分析,为优化西藏传统酸奶发酵菌种和工艺提供理论基础。
1 材料与方法
1.1 材料与试剂
传统发酵酸奶(编号为sn)、酪乳样品(编号为nl):购买于西藏林芝市巴宜区觉木沟农牧民家庭(E 94.36°,N 29.64°,海拔2 988 m)。
1.2 仪器与设备
YP3002N电子天平:上海菁海仪器有限公司;TH-86-150-WA超低温冰箱:北京天地精仪科技有限公司;ZD-F12冷冻干燥机:郑州比朗仪器有限公司;Eppendorf 5424R高速台式冷冻离心机:德国Eppendorf 公司;ABI GeneAmp® 9700 型PCR仪:美国ABI公司;Illumina Miseq MISEQ测序仪:美国Illumina公司。
1.3 试验方法
1.3.1 样品处理
发酵酸奶及酪乳样品原料牛乳为犏牛牛乳,分别编号为sn和nl,每个样品做3个平行。样品购买后装入无菌采样袋中,放入冰盒内,于6 h内带回西藏农牧学院食品科学与工程实验室置于-80 ℃冰箱内保藏备用。称取100 g酸奶及酪乳样品冷冻干燥后送样至上海美吉生物有限公司进行后续实验。
1.3.2 DNA抽提和PCR扩增
脱氧核糖核苷酸(deoxyribonucleic acid,DNA)抽提和聚合酶链式反应(polymerase chain reaction,PCR)扩增参考文献[16-17],具体如下:根据细菌基因组DNA提取试剂盒(天根DP302)说明书进行总DNA抽提,DNA浓度和纯度利用NanoDrop2000进行检测,利用1%琼脂糖凝胶电泳检测DNA提取质量;使用细菌通用引物:338F(5'-ACTCCTACGGGAGGCAGCAG-3')和806R(5'-GGACTACHVGGGTWTCTAAT-3')对V3-V4可变区进行PCR扩增,PCR扩增体系为20 μL,4μL5×FastPfu 缓冲液,2 μL2.5 mmol/L脱氧核糖核苷三磷酸(deoxy-ribonucleoside triphosphate,dNTPs),0.8 μL引物(5 μmol/L),0.4 μL FastPfu 聚合酶,10 ng DNA模板。PCR扩增程序为:95 ℃预变性3 min,27个循环(95 ℃变性30 s,55 ℃退火30 s,72 ℃延伸30 s),最后72 ℃延伸10 min。
1.3.3 Illumina Miseq高通量测序
利用Illumina公司的Miseq PE300平台进行测序。
1.3.4 数据处理
在上海美吉生物医药科技有限公司云平台上进行在线分析(www.i-sanger.com),采用FastTree软件构建进化树,使用R语言作图绘制进化树。结果可以通过进化树与reads丰度组合图的形式呈现。其他数据处理采用SPSS 20.0软件和Prism 5.0软件。
2 结果与分析
2.1 测序数据统计
对原始测序数据、质控后的优化数据及操作分类单元(operational taxonomic units,OTU)统计见表1。由表1 可知,酪乳(nl)样品和酸奶(sn)样品总计测得有效序列条数分别为(58 455±1 145)条和(57 068±1 263)条,共产生(63±2)个和(25±3)个OTU,分别包含7个门、9纲、22目、34科、46属、61种和7个门、9纲、22目、34科、46属、61种。
表1 酸奶和酪乳样品测序数据统计
Table 1 Sequence data statistics of yoghurt and buttermilk samples

不同样本的OTU韦恩图见图1。由图1可知,2个样本中共同含22个OTU,其中林芝酸奶样品独有3个OTU,酪乳样品独有41个OTU,分别占各自OTU总数的12%和65%,说明酪乳样品细菌多样性较丰富。

图1 酸奶和酪乳样品OTU韦恩图
Fig.1 OTU Venn diagram of yoghurt and buttermilk samples
2.2 稀释曲线
以多样性指数为表征对实际观测到的物种数目做稀释曲线见图2。由图2可知,sn和nl样品的稀释曲线均基本趋于平缓,说明取样数量合理,所得序列可基本反映酸奶和酪乳样品的细菌群落结构。
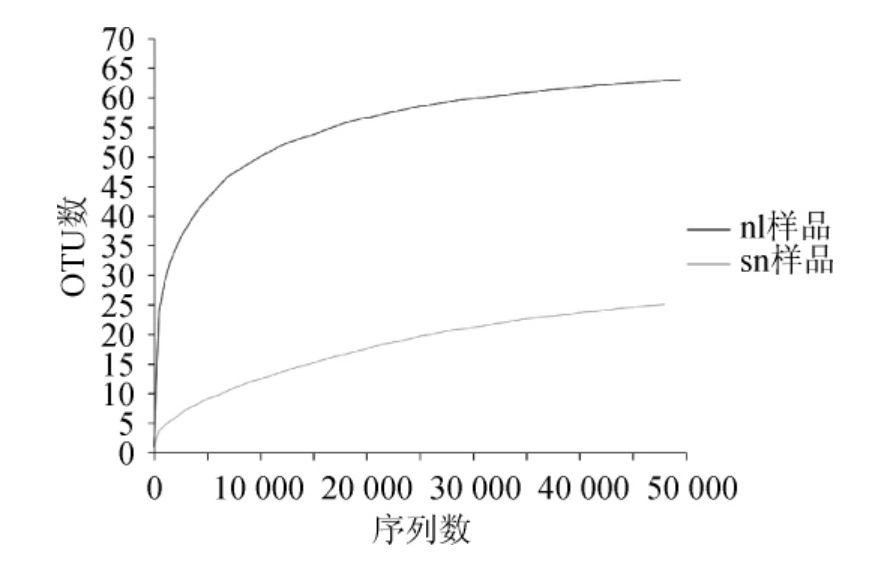
图2 酸奶和酪乳样品稀释曲线
Fig.2 Dilution curve of yoghurt and buttermilk samples
2.3 样品Alpha多样性分析
酸奶和酪乳样品中的Alpha多样性指标见表2。由表2可知,nl样品的Sobs指数、ACE 指数和Chao1指数分别为63.00±4.13、65.80±2.14和64.25±3.13,都比sn样品中相应的指数值大,说明nl样品具有较高的微生物群落丰富度。
表2 酸奶和酪乳样品Alpha多样性指数统计结果
Table 2 Statistics results of alpha diversity indexes of yoghurt and buttermilk samples

2.4 门水平样本细菌群落结构
酸奶和酪乳样品门水平细菌群落结构结果见表3。
表3 酸奶和酪乳样品细菌在门水平群落结构
Table 3 Bacterial community structure of yoghurt and buttermilk samples at phylum level
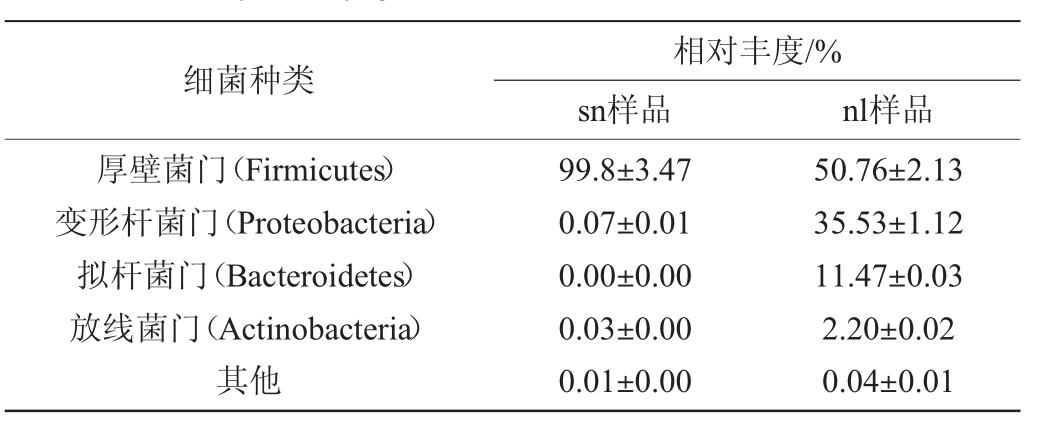
由表3可知,在门水平分布来看,sn和nl样品中厚壁菌门(Firmicutes)都排在第一位,其相对丰度分别为(99.80±3.47)%和(50.76±2.13)%,厚壁菌门(Firmicutes)为优势细菌门,这是因为牦牛酸奶中富含大量的乳酸菌,乳酸菌均为厚壁菌门。sn和nl样品排在第二位的是变形菌门(Proteobacteria),其相对丰度分别为(0.07±0.01)%和(35.53±1.12)%,变形菌门是植物土壤中常有的微生物菌门[18-19],牧民生活环境恶劣,常常以地为桌,做好的酸奶普遍放在地上,这可能是样品中均检测出少量这些微生物的原因[20]。在sn样品中并未检测到拟杆菌门(Bacteroidetes),而在nl样品中其相对丰度为(11.47±0.03)%。
2.5 属水平样本细菌群落结构
酸奶和酪乳样品属水平细菌群落结构见表4。由表4可知,细菌在属水平分布来看,乳杆菌属(Lactobacillus)、链球菌属(Streptococcus)和醋杆菌属(Acetobacter)在sn样品位于前三位,乳杆菌属相对丰度最高,占据总样本数量的(74.32±1.23)%,为优势菌属。其次为链球菌属(Streptococcus),占据总样本数量的(25.55±0.45)%,说明链球菌属在牦牛酸奶发酵过程中可能起着较为重要的作用[20]。在nl样品的属水平细菌群落结构中相对丰度由高到低的排序为:乳球菌属(Lactococcus)[(48.44±2.13)%]>不动杆菌属(Acinetobacter)[(16.18±1.12)%]>假单胞菌属(Pseudomonas)[(15.69±0.89)%]>金黄杆菌属(Chryseobacterium)[(11.22±0.56)%]。
表4 酸奶和酪乳样品细菌在属水平群落结构
Table 4 Bacterial community structure of yoghurt and buttermilk samples at genus level

2.6 属水平聚类分析
将林芝传统发酵酪乳(nl)和酸奶(sn)进行属水平的细菌群落TOP50热图分析,结果见图3。由图3可知,nl和sn样品差别较大,共显示30个细菌属,排名前十的分别是:乳杆菌属(Lactobacillus)、链球菌属(Streptococcus)、醋杆菌属(Acetobacter)、乳球菌属(Lactococcus)、金黄杆菌属(Chryseobacterium)、不动杆菌属(Acinetobacter)、假单胞菌属(Pseudomonas)、丛毛单胞菌属(Comamonas)、考克氏菌属(Kocuria)、明串珠菌属(Leuconostoc)。根据样品的热图可知,林芝传统发酵酪乳(nl)和酸奶(sn)样品在属水平的群落结构具有较大差异性。

图3 酸奶和酪乳样品属水平物种丰富度热图
Fig.3 Heatmap of species richness of yoghurt and buttermilk samples at genus level
横坐标为样本名(或分组名),纵坐标为物种名,通过色块颜色梯度来展示样本中不同物种的丰度变化情况,图中右侧为颜色梯度代表的数值。
3 结论
本研究利用Illumina Miseq测序技术对西藏林芝农牧民家经传统自然发酵的牦牛酸奶及酪乳细菌群落结构进行分析。结果表明,酪乳(nl)样品和酸奶(sn)样品总计测得有效序列条数分别为(58 455±1 145)条和(57 068±1 263)条,共产生(63±2)个和(25±3)个OTU,分别包含7门、9纲、22目、34科、46属、61种和7门、9纲、22目、34科、46属、61种。通过样品的Alpha多样性指数分析表明,虽然与酸奶样品相比,酪乳样品具有更高的群落丰富度,但是两个样品在门水平上的优势门均是厚壁菌门(Firmicutes),属水平上,sn样品的优势属为乳杆菌属(Lactobacillus),其相对含量为(74.32±1.23)%,nl样品中细菌在属水平上乳球菌属(Lactococcus)为优势属,另外nl样本中不动杆菌属(Acinetobacter)、假单胞菌属(Pseudomonas)、金黄杆菌属(Chryseobacterium)相对含量较高,说明这三属在牦牛酪乳发酵过程中可能起着较为重要的作用。结果表明,林芝传统发酵酪乳(nl)和酸奶(sn)样品在属水平的群落结构具有较大差异性。
[1]郭琳,孟梦,郭淼,等.接种乳酸菌改善辣椒酱风味的研究[J].中国调味品,2016,41(5):1-7.
[2]白露露,胡文忠,姜爱丽,等.辣椒加工工艺及其设备的应用现状[J].食品工业科技,2014,35(15):369-372,376.
[3]张琪.饮用性干酪风味酸奶的影响因素研究[D].成都:西华大学,2017.
[4]陈南,徐乾达,高浩祥,等.共发酵工艺对酸乳贮藏品质的影响及作用机理[J].乳业科学与技术,2018,41(5):6-11.
[5]刘凝,岳田利.乳品厂酸奶生产质量的控制方法分析[J].中小企业管理与科技,2016(4):16-17.
[6]周松文,方江平.林芝地区酸奶及灭菌乳市场调查分析[J].农产品加工(学刊),2013(22):57-58,61.
[7]王丽.酸奶生产加工中的微生物污染原因和控制方法[J].食品安全导刊,2016,33(11):68.
[8]刘艳容,舒永红,杨佳玮,等.食品中乳酸菌定量检测方法研究进展[J].食品工业科技,2020,41(12):364-371.
[9]赵月杰,程海芳,范雯婷,等.乳酸菌及其检测方法的研究进展[J].现代食品,2018(17):1-3,17.
[10]包秋华.甘肃和四川省牦牛奶制品中乳酸菌的多样性研究[D].呼和浩特:内蒙古农业大学,2012.
[11]翟云,托娅.益生菌的免疫调节作用及其相关应用研究进展[J].中国微生态学杂志,2018,30(2):235-239.
[12]梁明明,曹菲薇,杨欢东,等.传统藏式牦牛酸奶中优良乳酸菌筛选与产香性能分析[J].食品工业科技,2020,41(16):108-113.
[13]杨行,王莉,郭丽君,等.新疆喀什地区传统酸奶中乳酸菌的分离鉴定及产酸能力评价[J].食品与发酵工业,2020,46(2):102-107.
[14]吴均,赵晓娟,陈佳昕,等.西藏、川西青藏高原牧区自然发酵牦牛酸奶中优良乳酸菌的筛选及鉴定[J].食品科学,2013,34(17):150-155.
[15]董成.益生菌L.casei Zhang 在干酪中的应用研究[D].呼和浩特:内蒙古农业大学,2008.
[16]陈梅春,朱育菁,刘波,等.基于宏基因组的茉莉花内生细菌多样性分析[J].热带亚热带植物学报,2018,26(6):633-643
[17]陈奇,闫峻,王玖,等.食品级二氧化钛对幼鼠肠道菌群及血清炎性因子的影响[J].解放军预防医学杂志,2019,37(9):3-6.
[18]金奇.鄱阳湖典型湿地土壤微生物群落特征及对环境变化的响应研究[D].南昌:江西师范大学,2017.
[19]宋宇,王鹏,韦月平.不同稻田共作模式对土壤细菌群落结构的影响[J].西北农业学报,2020,29(2):216-223.
[20]孙思雨,陈炼红,王琳琳.应用Illumina Miseq 测序分析川西高原传统牦牛发酵酸奶中细菌多样性[J].食品工业科技,2019,439(23):98-103.