藠头(Allium chinense G.Don)又称荞头和薤等,是百合科的多年生宿根性草本植物,因含有大蒜素等活性成分而具有辛辣感,故常加热或腌制后食用。腌制后的藠头具有良好的风味和保健功能[1],其腌制工艺也相对简单,将去尾的藠头、碎辣椒和盐放于坛中密封发酵2个月,随后加入熬制的红糖、酱油、花椒水和白酒继续密封发酵1个月即可。泡菜可分为泡渍泡菜和盐渍泡菜两大类[2],其中四川和东北地区的泡菜多以泡渍泡菜为主[3],而腌制藠头应属于盐渍泡菜的范畴。作为厌氧自然发酵的蔬菜制品,泡菜具有丰富的微生物群系,且以乳酸菌为优势类群[4]。采用纯培养技术,汤水平等[5-6]研究发现,藠头发酵液中含有短乳杆菌(Lactobacillus brevis)、发酵乳杆菌(Lactobacillus fermenti)和植物乳球菌(Lactobacillus plantarum)等7种乳酸菌,其中L.brevis的产酸能力与耐盐性均较强,可作为腌制藠头制作的潜在使用菌株。
较之纯培养技术,近年兴起的高通量测序技术日趋完善、测序成本逐渐降低、使用范围也拓展至病原微生物[7]、肠道微生物[8]和发酵食品微生物[9]多样性的解析中,实现了不同样本间微生物类群的平行比较分析[10]。泡菜中的微生物主要来源于环境和原料表面,对泡菜风味品质的形成具有重要的作用[11],其群系结构与原料[12]、制作方式[13]和地理环境[14]等因素均息息相关。恩施土家族苗族自治州来凤县,地处鄂、湘和渝三省市交界处,为湖北省的西大门,是一个以土家族为主的多民族聚居县,境内盛产藠头、藤茶和凤头姜等多种特色农产品,且具有加工和食用盐渍泡菜的习俗。目前,关于MiSeq高通量测序技术在盐渍藠头微生物多样性解析中应用的报道尚少,有关土家族等少数民族聚居地区盐渍藠头细菌类群的研究更是尚未开展。
本研究采用MiSeq高通量检测技术对来凤地区盐渍藠头盐水中细菌菌群进行解析,同时使用PICRUSt技术对其基因功能进行预测,以期为后续盐渍藠头等特色泡菜制品的微生物多样性研究和产业化提供一定的数据参考。
1 材料与方法
1.1 材料与试剂
1.1.1 材料
盐渍藠头盐水:采自湖北省恩施土家族苗族自治州来凤县(E109°00′~109°27′,N29°06′~29°40′)的城南农贸大市场、大河镇菜市场和鄂湘渝边贸大市场,共采集6个样品,记为JT1~JT6,腌制时间均在3.5个月左右。
1.1.2 试剂
DNeasy mericon Food Kit提取试剂盒:德国QIAGEN公司;338F/806R双向引物:武汉天一辉远生物科技有限公司;5×Fast Pfu Buffer、2.5 mmol/L脱氧核糖核苷三磷酸(deoxyribonucleoside triphosphate,dNTP)mix、rTaq酶(5 U/μL)、DL 2000脱氧核糖核酸(deoxyribonucleic acid,DNA)Marker:宝生物工程大连有限公司;MiSeq测序配套试剂:美国Illumina公司。
1.2 仪器与设备
Veriti FAST梯度聚合酶链式反应(polymerase chain reaction,PCR)仪:美国ABI公司;164-5050基础电泳仪:美国Bio-Rad公司;UVPCDS8000凝胶成像分析系统:美国Protein Simple公司;ND-2000C微量紫外分光光度计:美国Thermo公司;5810R台式高速冷冻离心机:德国Eppendorf公司;MiSeq PE250高通量测序平台:美国Illumina公司;R930机架式服务器:美国DELL公司。
1.3 实验方法
1.3.1 样品采集
样品采集时间为2019年12月下旬,选择藠头种植和制作地均为来凤县的样品进行采集。样品采集时从腌制藠头的坛中取100 mL盐水,装入样品瓶中低温运回实验室。
1.3.2 MiSeq高通量测序
采集的盐水300 r/min离心10 min,去除藠头残渣,上清液8 000 r/min离心10 min,去上清,使用DNeasy mericon Food Kit试剂盒对沉淀菌泥的宏基因组DNA进行提取,并以其为模板,参照尚雪娇等[15]的方法对16S rRNA V3-V4区序列进行PCR扩增,扩增产物委托上海美吉生物医药科技有限公司采用MiSeq PE250平台进行高通量测序。
1.3.3 细菌类群解析
使用实验室自有脚本去除序列核苷酸标签(barcode)和引物后[16],将6个样品序列合并为1个fna文件,提交至QIIME(v1.7.0)平台[17]进行序列对齐、两步UCULST分类操作单元(operational taxonomic units,OTU)划分[18]、Chimera Slayer嵌合体检查[19]和序列比对[20],其中OTU划分所采用的序列相似度参数为100%和97%[21]。将鉴定出的细菌门或属按照平均相对含量从大到小进行排序,排在前3位的则定义为优势细菌门或属[16]。
1.3.4 基因功能预测
使用PICRUSt软件对1.3.3中fna文件中基因的功能进行预测[22],并参照蛋白质直系同源簇(clusters of orthologous groups of proteins,COGs)数据库进行注释[23]。
1.3.5 图形绘制及数据分析
使用Origin8.5软件绘制柱形图;使用Excel2017软件绘制瀑布图;使用R软件的waterfall、UpSetR、corrplot和pheatmap软件包绘制其他图,其中相关性热图中的相关系数采用Pearson 相关性分析计算。
2 结果与分析
2.1 测序深度分析
本研究使用MiSeq PE250平台对6个盐渍藠头盐水中细菌类群进行高通量测序,经质控后共产生288 495条高质量16S rRNA V3-V4区序列,平均每个样品测序深度为48 083条。切除barcode和引物后序列的碱基长度分布情况见图1。
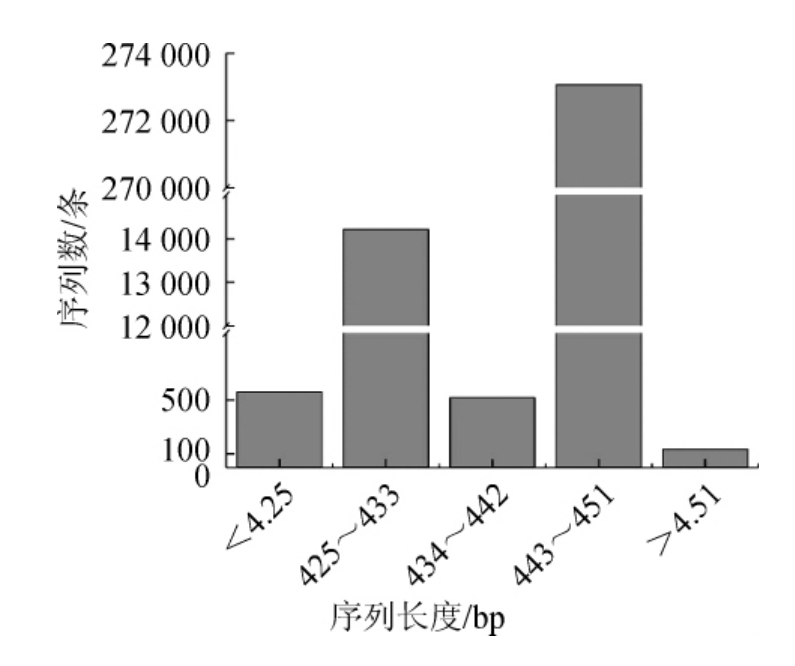
图1 序列长度的分布
Fig.1 Distribution of sequences length
由图1可知,有273 067条序列长度在443~451 bp之间,占总序列数的94.65%;有14 211条序列长度在425~433 bp,占总序列数的4.93%;亦有部分序列长度<433bp或>451bp,但其数量较少,仅占总序列数的0.42%。由此可知,切除barcode和引物后,本研究99.58%的序列长度在425~451 bp,可基本覆盖16S rRNA V3-V4区序列,因而后续可在分类学地位“门”和“属”水平上对样品细菌多样性进行分析。
2.2 基于门和属水平的细菌群落结构分析
根据97%相似性将273 067条序列划分为13 417个OTU,经ChimeraSlayer检查并无嵌合体存在,经比对后将所有序列鉴定为28个门和561个属。为保证分析结果的准确性,仅对平均相对含量>1.0%的门和属进行展示[24]。盐渍藠头盐水中平均相对含量>1.0%的细菌门见图2。

图2 盐渍藠头盐水中平均相对含量>1.0%的细菌门
Fig.2 Bacterial phyla with average relative content more than 1.0%in saline Allium chinese G.Don brine samples
由图2可知,盐渍藠头盐水中细菌主要隶属于硬壁菌门(Firmicutes)、变形菌门(Proteobacteria)、放线菌门(Actinobacteria)和拟杆菌门(Bacteroidetes),平均相对含量分别为43.94%、33.12%、18.21%和2.85%,其中Firmicutes、Proteobacteria和Actinobacteria为优势细菌门。盐渍藠头盐水中平均相对含量>1.0%的细菌属见图3。
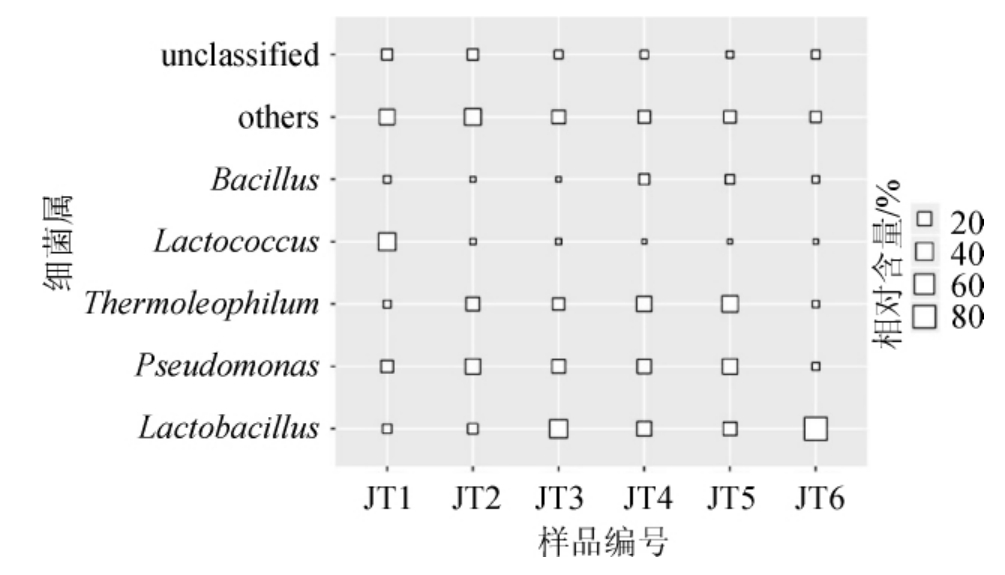
图3 盐渍藠头盐水中平均相对含量>1.0%的细菌属
Fig.3 Bacterial genera with average relative content more than 1.0%in saline Allium chinese G.Don brine samples
由图3可知,盐渍藠头盐水中存在5个平均相对含量>1.0%的细菌属,分别为隶属于Firmicutes的乳杆菌属(Lactobacillus)、芽孢杆菌属(Bacillus)和乳球菌属(Lactococcus),以及隶属于Proteobacteria的假单胞菌属(Pseudomonas)和隶属于Actinobacteria的嗜热油菌属(Thermoleophilum),平均相对含量依次为30.28%、6.94%、2.94%、18.21%和16.13%。由此可见,Lactobacillus、Pseudomonas和Thermoleophilum为盐渍藠头盐水中的优势细菌属。本研究发现,盐渍藠头盐水中的乳酸菌主要为Lactobacillus和Lactococcus,有研究报道指出韩国泡菜Kimchi中的乳酸菌主要为Lactobacillus、魏斯氏菌属(Weissella)和明串珠菌属(Leuconostoc)[25],而四川地区泡菜中的乳酸菌主要为Lactobacillus、Pediococcus、Lactococcus和Leuconotoc[26-27],造成不同泡菜中乳酸菌类群存在差异的原因除与原料种类有关外,还与地域、制作工艺和添加食盐量等诸多因素有关[28]。作为条件治病菌,Pseudomonas中的部分种群可导致炎症的发生[29],但本研究发现其为盐渍藠头盐水中的优势细菌,李欣蔚等[30]采用高通量测序技术对东北地区泡菜细菌类群进行解析亦发现了与本研究相同的现象,Pseudomonas在样品中的平均相对含量为39.6%~77.7%。由此可见,开展对盐渍藠头盐水中细菌类群的研究,对后续产品食用安全品质的提升具有积极的意义。
2.3 基于OTU水平的细菌群落结构分析
若某一OTU在6个盐渍藠头盐水样品中均存在,则将其定义为核心OTU[31],OTU在每个样品中的分布情况见图4。

图4 基于OTU的UpSet分布图
Fig.4 Distribution map of UpSet based on OTU
由图4可知,样品JT1~JT6分别含有3 256、2 843、3 272、4 296、2 636和2 980个OTU,其中219个为核心OTU,占OTU总数的1.63%,但包含了217998条序列,占总序列数的75.56%,14个核心OTU的平均相对含量>1.0%,累计平均相对含量为43.14%,其平均相对含量的分布见图5。

图5 平均相对含量>1.0%核心OTU的瀑布图
Fig.5 Waterfall plot of core OTUs with average relative content more than 1.0%
由图5可知,分别有5、3、3、2和1个OTU被鉴定隶属于Thermoleophilum、Lactobacillus、Pseudomonas、Bacillus 和Lactococcus,累计平均相对含量分别为7.98%、15.58%、11.96%、2.27%和5.35%。平均相对含量>1.0%核心OTU相关性的热图见图6。
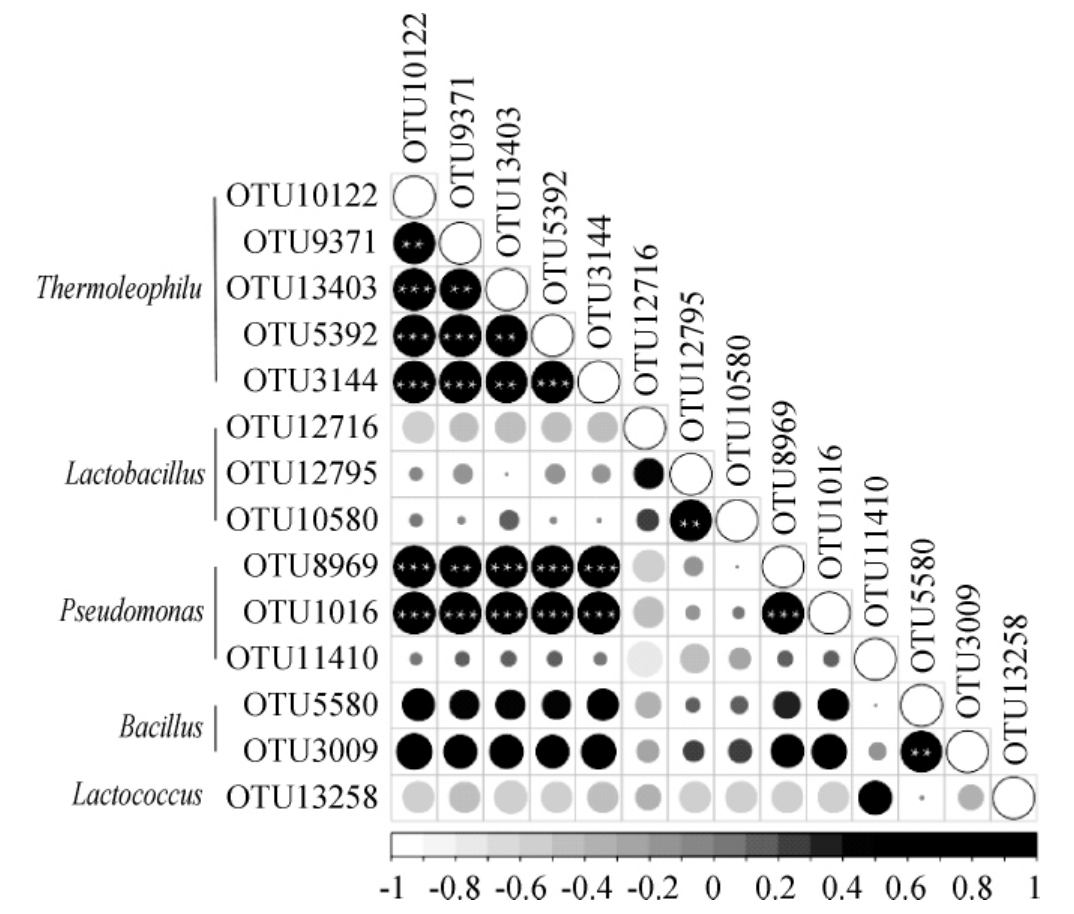
图6 平均相对含量>1.0%核心OTU相关性的热图
Fig.6 Heatmap of correlation of core OTUs with average relative content more than 1.0%
“**”表示相关性非常显著(P<0.01);“***”表示相关性极显著(P<0.001);圆圈的半径越大表示相关系数的绝对值越大;圆圈的颜色越深表示相关系数越接近于1.0,颜色越浅表示相关系数越接近于-1.0。
由图6可知,隶属于Pseudomonas的OTU8969和OTU1016与5个隶属于Thermoleophilum的核心OTU均呈现非常显著(P<0.01)或极显著(P<0.001)正相关,而隶属于Lactobacillus和Lactococcus的核心OTU与其他属的核心OTU相关性均不显著(P>0.05)。此外,Thermoleophilum、Lactobacillus、Pseudomonas或Bacillus等同属内的部分核心OTU之间亦存在一定的正相关,且相关性非常显著(P<0.01)或极显著(P<0.001)。由此可见,6个盐渍藠头盐水共有大量的细菌类群,且不同类群间可能有一定的共生关系。
2.4 基因功能预测
在对盐渍藠头盐水细菌类群解析的基础上,进一步对其基因功能进行了预测,结果见图7。
由图7可知,所有序列共注释到隶属于23个功能大类的4 305个蛋白质直系同源簇(clusters of orthologous groups of proteins,COGs)。经聚类分析发现,功能E、K、R和S形成1个聚类,即盐渍藠头盐水中细菌具有较强的转录及氨基酸转运和代谢功能;其他19个功能形成1个聚类,相关基因表达功能均较弱。采用超高效液相色谱-四极杆飞行时间质谱技术,HE Q等[32]研究发现,藠头富含8种人体必需氨基酸和5种半必需氨基酸,两者累计含量占总氨基酸的64.9%,这可能是造成盐渍藠头盐水中细菌氨基酸转运和代谢功能较强的原因。

图7 基因功能预测热图
Fig.7 Heatmap of gene functional prediction
A,RNA处理和修饰;B,染色质结构和动力学;C,能量生产和转换;D,细胞周期控制,细胞分裂,染色体分割;E,氨基酸运输和代谢;F,核苷酸运输和代谢;G,碳水化合物运输和代谢;H,辅酶运输和代谢;I,脂质转运与代谢;J,翻译、核糖体结构和生物发生;K,转录;L,复制、重组和修复;M,细胞壁/膜/包膜生物发生;N,细胞运动;O,翻译后修饰、蛋白质转换、伴随蛋白;P,去氧离子运输和代谢;Q,次生代谢物的生物合成、运输和分解代谢;R,一般功能预测;S,未知功能;T,信号转导机制;U,细胞内运输、分泌和囊泡运输;V,防御机制;Z,细胞支架。
3 结论
通过MiSeq高通量测序分析发现,来凤地区盐渍藠头盐水中优势细菌门为硬壁菌门(Firmicutes)、变形菌门(Proteobacteria)和放线菌门(Actinobacteria),平均相对含量分别为43.94%、33.12%和18.21%;优势细菌属为乳杆菌属(Lactobacillus)、假单胞菌属(Pseudomonas)和嗜热油菌属(Thermoleophilum),平均相对含量分别为30.28%、18.21%和16.13%;共有219个核心操作分类单元(OTU),包含的序列数占总序列数的75.56%;盐渍藠头盐水中可能共有大量具有较强氨基酸转运和代谢功能的细菌类群。
[1]周向荣,夏延斌,周跃斌,等.藠头的主要功能成分及其作用的研究进展[J].食品与机械,2006,22(3):73-75.
[2]赵楠.四川泡菜的主要特性及其成因分析[D].无锡:江南大学,2017.
[3]曹佳璐.传统四川泡菜盐水乳酸菌多样性的研究[D].北京:中国农业大学,2017.
[4]XIAO Y,XIONG T,PENG Z,et al.Correlation between microbiota and flavours in fermentation of Chinese Sichuan Paocai[J].Food Res Int,2018,114(12):123-132.
[5]汤水平,张青燕,谢伟岸,等.盐渍藠头发酵过程中乳酸菌的分离及特性研究[J].中国酿造,2007,26(8):61-64.
[6]汤水平,朱泽瑞,谢伟岸,等.腌制藠头发酵过程中乳酸菌的鉴定及生物学特性比较研究[J].食品工业科技,2008,30(5):128-130.
[7]YIN X,GU X,YIN T,et al.Study of enteropathogenic bacteria in children with acute diarrhoea aged from 7 to 10 years in Xuzhou,China[J].Micro-bial Pathogen,2016,91(2):41-45.
[8]MOROWITZ M J,POROYKO V,CAPLAN M,et al.Redefining the role of intestinal microbes in the pathogenesis of necrotizing enterocolitis[J].Pediatrics,2010,125(4):777-785.
[9]LIANG H,YIN L,ZHANG Y,et al.Dynamics and diversity of a microbial community during the fermentation of industrialized Qingcai paocai,a traditional Chinese fermented vegetable food,as assessed by Illumina MiSeq sequencing,DGGE and qPCR assay[J].Ann Microbiol,2018,68(2):111-122.
[10]TANG Y,ZHOU X,HUANG S,et al.Microbial community analysis of different qualities of pickled radishes by Illumina MiSeq sequencing[J]. J Food Safe,2019,39(2):e12596.
[11]代富英,戴雨珂,王海娟,等.中国泡菜乳杆菌种质资源调查[J].食品研究与开发,2016,37(18):162-165.
[12]LIANG H,ZHANG A,WU Z,et al.Characterization of microbial community during the fermentation of Chinese homemade paocai,a traditional fermented vegetable food[J]. Food Sci Technol Res,2016,22(4):467-475.
[13]佟婷婷,田丰伟,王刚,等.基于宏基因组分析四川泡菜母水作引子的泡菜发酵过程中细菌多样性变化[J].食品工业科技,2015,36(21):173-177.
[14]申文熹,陈功,唐垚,等.不同盐质量分数泡白菜发酵过程中乳酸菌群落结构的变化[J].食品科学,2017,38(10):111-117.
[15]尚雪娇,王玉荣,杨江,等.应用Illumina MiSeq 高通量测序技术解析梅干菜中细菌多样性[J].中国酿造,2019,38(1):140-144.
[16]郭壮,葛东颖,尚雪娇,等.退化和正常窖泥微生物多样性的比较分析[J].食品工业科技,2018,39(22):93-98.
[17]CAPORASO J G,KUCZYNSKI J,STOMBAUGH J,et al.QIIME allows analysis of high-throughput community sequencing data[J]. Nat Meth,2010,7(5):335-336.
[18]EDGAR R C.Search and clustering orders of magnitude faster than BLAST[J].Bioinformatics,2010,26(19):2460-2461.
[19]EDGAR R C,HAAS B J,CLEMENTE J C,et al.UCHIME improves sensitivity and speed of chimera detection[J]. Bioinformatics,2011,27(16):2194-2200.
[20]MAIDAK B L,COLE J R,LILBURN T G,et al.The RDP (ribosomal database project)continues[J].Nucl Acid Res,2000,28(1):173-174.
[21]尚雪娇,折米娜,邓长阳,等.恩施市3 种泡辣椒样品中细菌多样性研究[J].中国酿造,2019,38(2):53-57.
[22]WILKINSON T J,HUWS S A,EDWARDS J E,et al.CowPI:a rumen microbiome focussed version of the PICRUSt functional inference software[J].Front Microbiol,2018,9(5):1095.
[23]GALPERIN M Y,KRISTENSEN D M,MAKAROVA K S,et al.Microbial genome analysis:the COG approach[J].Brief Bioinformat,2019,20(4):1063-1070.
[24]沈馨,王艳,代凯文,等.基于Miseq 高通量测序技术的辣椒酱核心细菌类群研究[J].食品研究与开发,2018,39(10):151-157.
[25]JEONG S H,LEE S H,JUNG J Y,et al.Microbial succession and metabolite changes during long-term storage of kimchi[J].J Food Sci,2013,78(5):763-769.
[26]CAO J,YANG J,HOU Q,et al.Assessment of bacterial profiles in aged,home-made Sichuan paocai brine with varying titratable acidity by PacBio SMRT sequencing technology[J].Food Control,2017,78(8):14-23.
[27]ZHAO N,ZHANG C,YANG Q,et al.Multiple roles of lactic acid bacteria microflora in the formation of marker flavour compounds in traditional Chinese paocai[J].RSC Adv,2016,6(92):89671-89678.
[28]FANG R S,DONG Y C,CHEN F,et al.Bacterial diversity analysis during the fermentation processing of traditional Chinese yellow rice wine revealed by 16S rDNA 454 pyrosequencing[J].J Food Sci,2015,80(10):M2265-M2271.
[29]LI X,CHEN Y,GAO W,et al.A 6-year study of complicated urinary tract infections in southern China:prevalence,antibiotic resistance,clinical and economic outcomes[J].Therapeut ClinRisk Manag,2017,13(11):1479-1487.
[30]李欣蔚,丛敏,武俊瑞,等.基于16S rRNA 基因V3-V4 区高通量测序分析东北自然发酵酸菜中细菌群落结构[J].现代食品科技,2017,33(2):69-75.
[31]ZHANG J,GUO Z,XUE Z,et al.A phylo-functional core of gut microbiota in healthy young Chinese cohorts across lifestyles,geography and ethnicities[J]. ISME J,2015,9(9):1979-1990.
[32]HE Q,HUANG S,WU Y,et al.Comparative study on the composition of free amino acids and derivatives in the two botanical origins of an edible Chinese herb"Xiebai",i.e.,Allium chinese G.Don and Allium macrostemon Bunge species[J].Food Res Int,2018,106(4):446-457.