泡菜作为我国最具代表性的发酵蔬菜制品,常以白菜、萝卜、豇豆和辣椒等新鲜蔬菜为主料,并辅以姜、蒜和花椒等佐料浸于低浓度盐水中自然发酵而成[1]。因泡制后口感清脆酸爽,豇豆(Vigna unguiculata(Linn.)Walp)成为了制作泡菜的主要原料。湖北省恩施土家族苗族自治州被誉为“世界硒都”,蕴藏着较为丰富的硒元素,建始县作为其州辖县,境内少数民族众多,饮食文化也独具地方特色,发酵食品大体可分为“酸、辣、干、咸”四大类,其中酸豇豆作为酸味的代表在当地居民饮食中是不可或缺的。
虽然泡菜坛的坛沿常用水封以达到隔绝空气进入的目的,但在取食泡菜的过程中会反复开盖,从而导致其发酵过程并不是完全的厌氧过程,加之泡菜原料和制作器皿自身携带微生物,因而泡菜盐水中的微生物类群是较为复杂的[2]。目前,已有众多关于泡菜中微生物类群研究的报道,在不同食盐浓度下乳酸菌均为泡菜发酵过程中的主导菌系[3];蔡炯等[4]从四川腐败泡菜中发现甲基营养型芽孢杆菌、枯草芽孢杆菌和地衣芽孢杆菌为主要的致腐败细菌。高通量测序技术的出现为泡菜微生物多样性的解析提供了新的契机,LIANG H等[5]采用Illumina MiSeq高通量测序技术发现,乳酸杆菌、弧菌属、假单胞菌属和盐单胞菌属为青菜泡菜中的优势细菌;CAO J等[6]采用单分子实时测序技术发现,耐酸乳杆菌和短乳杆菌为四川地区泡菜盐水中的优势细菌。使用高通量技术对微生物多样性进行解析时,常仅对平均相对含量>1.0%的细菌类群进行重点讨论以保证研究结果的可信性,同时亦较关注在不同样品中均存在的核心微生物类群,并认为其对发酵食品品质的形成具有直接的作用[7]。
本研究在采用高通量测序技术对建始地区酸豇豆盐水中细菌多样性进行解析的基础上,甄别了其核心细菌类群,同时对盐水中蕴含的乳酸菌菌种分离鉴定,以期为华中地区泡菜制品微生物群落结构研究提供数据支持,同时为泡菜发酵剂的制备提供菌株支持。
1 材料与方法
1.1 材料与试剂
酸豇豆盐水:5份酸豇豆盐水样品均于湖北省恩施土家族苗族自治州建始县(E109°32'~110°12',N30°32'~30°54')龙坪乡和高坪镇采集,编号分别为JD1、JD2、JD3、JD4和JD5。采样时使用不锈钢酒提从泡菜坛中舀取约200 mL盐水装入洁净样品瓶中,带回实验室置于4 ℃备用。
MRS培养基、LB培养基:青岛海博生物技术有限公司;Biowest琼脂糖:上海贝晶生物技术有限公司;Axygen清洁试剂盒:康宁生命科学吴江有限公司;DNeasy mericon Food Kit脱氧核糖核酸(deoxyribonucleic acid,DNA)基因组提取试剂盒:德国QIAGEN公司;10×Buffer、脱氧核糖核苷三磷酸(deoxy-ribonucleoside triphosphates,dNTPs)Mix 和DNA聚合酶:北京全式金生物技术有限公司;引物338F/806R和M13F/M13R:天一辉远(武汉)生物科技有限公司;大肠杆菌(Escherichia coli)top10:鄂西北传统发酵食品研究所自制。
1.2 仪器与设备
DG250型厌氧工作站:英国DonWhitley公司;VeritiFAST梯度聚合酶链式反应(polymerase chain reaction,PCR)仪:美国ABI公司;UVPCDS8000凝胶成像分析系统:美国Protein Simple公司;Illumina MiSeq高通量测序平台:美国Illumina公司;R920机架式服务器:美国DELL公司。
1.3 试验方法
1.3.1 宏基因组提取、PCR扩增和高通量测序
参照基因组提取试剂盒中的方法提取酸豇豆盐水样品的总脱氧核糖核酸(DNA)。参照文献[8]中的方法,对细菌16S rRNA V3-V4区进行PCR扩增。PCR扩增体系:10×Buffer 5 μL,dNTPs Mix 4 μL,正反向引物各0.8 μL,DNA聚合酶0.4 μL,DNA模板10 ng;PCR扩增程序:94 ℃、3 min预变性;95 ℃变性45 s,55 ℃退火30 s,75 ℃延伸45 s,循环30 次后;72 ℃延伸10 min。将扩增合格的PCR产物寄至上海美吉生物医药科技公司进行测序。
1.3.2 生物信息学分析
采用QIIME(V1.7.0)平台对细菌多样性进行分析评价。参照文献[8]的方法将测序得到的序列拼接对齐后,按照97%相似度对序列进行划分构建操作分类单元(operational taxonomic units,OTU)[9]并进行嵌合体检查[10],将含有嵌合体的OTU删除,选择余下OTU代表性序列进行同源性比对并注释其分类学地位[11],同时对各样品细菌的α多样性进行计算分析[12]。
1.3.3 乳酸菌的分离和鉴定
采用倍比稀释涂布法对酸豇豆盐水中乳酸菌进行分离。量取10 g酸豇豆盐水装入90 mL生理盐水中稀释到10-1梯度,置于37 ℃摇床摇30 min,吸取0.5 mL 10-1梯度稀释液于4.5 mL生理盐水中,稀释到10-2梯度,按照此法依次稀释到10-6梯度,分别吸取10-4、10-5和10-6梯度稀释液100 μL涂布于含有1.0%CaCO3的MRS固体培养基中,37 ℃厌氧培养48 h,挑取含有透明圈的单菌落进行2~3 次划线纯化,选择过氧化氢酶阴性且革兰氏阳性的菌株使用30%的甘油保藏于-80 ℃备用[13]。参照参考文献[13]中的方法进行乳酸菌菌株基因组DNA提取和16S rRNA基因PCR扩增后,使用清洁试剂盒对扩增成功的PCR产物进行清洁纯化,最后进行连接和转化,挑取阳性克隆子送至天一辉远(武汉)生物科技有限公司测序。
1.3.4 统计分析
利用Origin2017软件和R软件(V3.6.2)进行图的绘制。
2 结果与分析
2.1 测序结果与α多样性分析
基于MiSeq高通量测序技术,5 个样品测序共得到了168 808 条有效序列,将序列按97%相似度划分得到9 036 个OTU,各样品的测序结果及各分类学地位数量如表1所示。
表1 样品的测序结果与分类学地位
Table 1 Sequencing results and taxonomic status of samples
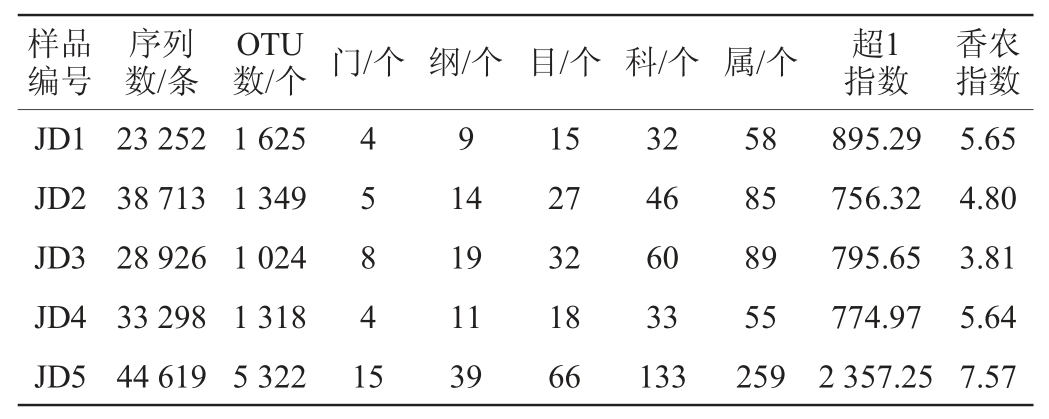
注:所有样品的超1指数和香农指数均在测序深度为28 010 条序列时计算得到。
由表1可知,样品JD1、JD2、JD3、JD4和JD5的序列数分别为23 252、38 713、28 926、33 298和44 619 条,OTU数量分别为1 625、1 349、1 024、1 318和5 322 个。对每个OTU的代表性序列进行同源性比对后,所有序列被鉴定到15个门,39个纲,67个目,137个科和283个属,不能被鉴定到门和属水平的序列数分别占总序列数的0.034%和3.96%。由表1亦可知,经α多样性计算,样品JD5的超1和香农指数均为所有样品中最高,分别为2 357.25和7.57,而其他样品的超1指数均低于1 000,香农指数均低于6。由此可见,不同酸豇豆盐水中细菌丰度和多样性存在一定的差异。
2.2 基于门和属水平的细菌多样性分析
本研究从门和属水平上进一步对酸豇豆盐水中细菌多样性进行了解析,其中平均相对含量大于1.0%的细菌门如图1所示。
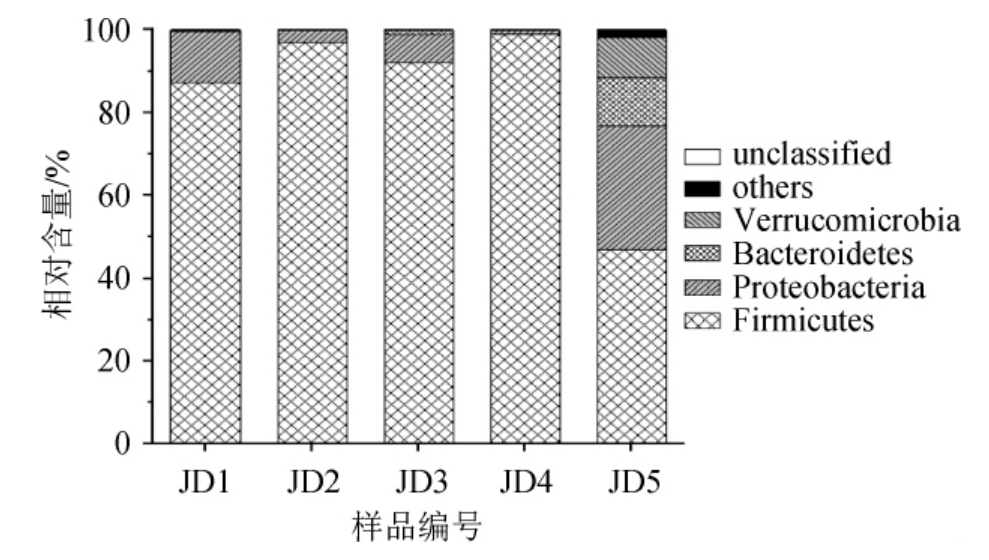
图1 平均相对含量>1.0%的细菌门
Fig.1 Bacterial phylum with an average relative content more than 1.0%
由图1可知,硬壁菌门(Firmicutes)、变形菌门(Proteobacteria)、拟杆菌门(Bacteroidetes)和疣微菌门(Verrucomicrobia)为酸豇豆中平均相对含量>1.0%的细菌门,分别为84.41%、10.52%、2.63%和1.94%,其中Firmicutes和Pro teobacteria在所有样品中均存在,而Bacteroidetes和Verrucomicrobia只在样品JD5中占有较高的比例,分别为11.72%和9.68%。酸豇豆中平均相对含量大于1.0%的细菌属如图2所示。

图2 平均相对含量>1.0%的细菌属
Fig.2 Bacterial genus with an average relative content more than 1.0%
由图2可知,酸豇豆盐水中共有8个平均相对含量大于1.0%的细菌属,分别为隶属于Firmicutes的乳酸杆菌属(Lactobacillus,57.02%)、魏斯氏菌属(Weissella,9.33%)、葡萄球菌属(Staphylococcus,7.07%)、片球菌属(Pediococcus,3.40%)、明串珠菌属(Leuconostoc,1.94%)和索丝菌属(Brochothrix,1.07%),隶属于Proteobacteria的假单胞菌属(Pseudomonas,4.73%),隶属于Verrucomicrobia的艾克曼菌属(Akkermansia,2.54%)。乳酸菌是泡菜发酵过程中主要的优势菌群[2],其中短乳杆菌(L.brevis)被认为是影响泡菜感官品质的重要菌属之一[14],而本研究的酸豇豆盐水中属于乳酸菌菌群的Lactobacillus、Weissella、Pediococcus和Leuconostoc的累计平均相对含量达71.69%。然而,在样品JD1和JD5中分别存在着少量的Staphylococcus和Pseudomonas,有报道指出,隶属于金黄色葡萄球菌(Staphylococcus aureus)[15]和铜绿假单胞菌(Pseudomonas aeruginos)[16]的部分“种”对人体均具有条件致病性。由此可见,在对建始地区酸豇豆盐水中乳酸菌资源进行收集的同时,通过改善生产环境从而降低部分样品中条件致病菌的含量亦具有积极意义。
2.3 基于OTU水平的细菌多样性分析
在解析门和属含量的基础上,本研究进一步从OTU水平上对细菌多样性进行了分析,各样品中OTU的分布如图3所示。

图3 各样品中OTU分布情况
Fig.3 Distribution of OTUs in each sample
由图3可知,样品JD1~JD5中特有的OTU 个数分别为1 246、892、485、888和4 479个,各包含了5 463、5 539、847、3 186和19 124条序列,分别占各样品总序列数的23.50%、14.31%、2.93%、9.57%和42.86%。由此可见,样品JD5的细菌类群与其他样品相比可能较为特殊,但每个OTU包含的序列量仅占总测序量的0.009 6%,因而其差异主要体现在若干相对含量极低的种系型上。
若某个OTU在5个酸豇豆盐水样品中均存在,则将其定义为核心OTU[17]。本研究共发现了44个核心OTU,包含了42 307 条序列,累计包含序列数占总序列数的25.06%。核心OTU中分别有6个、11个和13个被鉴定为Pseudomonas、Weissella和Lactobacillus,累计平均含量分别为4.61%、4.97%和14.46%;有2个OTU被鉴定为不动菌属(Acinetobacter),累计平均相对含量为0.08%;各有1个OTU被鉴定为鞘氨醇杆菌属(Sphingobacterium)、沙雷氏菌属(Serratia)、根瘤菌属(Rhizobium)、果胶杆菌属(Pectobacterium)、紫色杆菌属(Janthinobacterium)、肠球菌属(Enterococcus)、布丘氏菌属(Buttiauxella)、杆菌属(Bacillus)和明串珠菌属(Leuconos toc),但其累计平均相对含量均<0.1%;有3个OTU不能鉴定到属水平,累计平均相对含量为0.49%。相对含量>1.0%的核心OTU如图4所示。
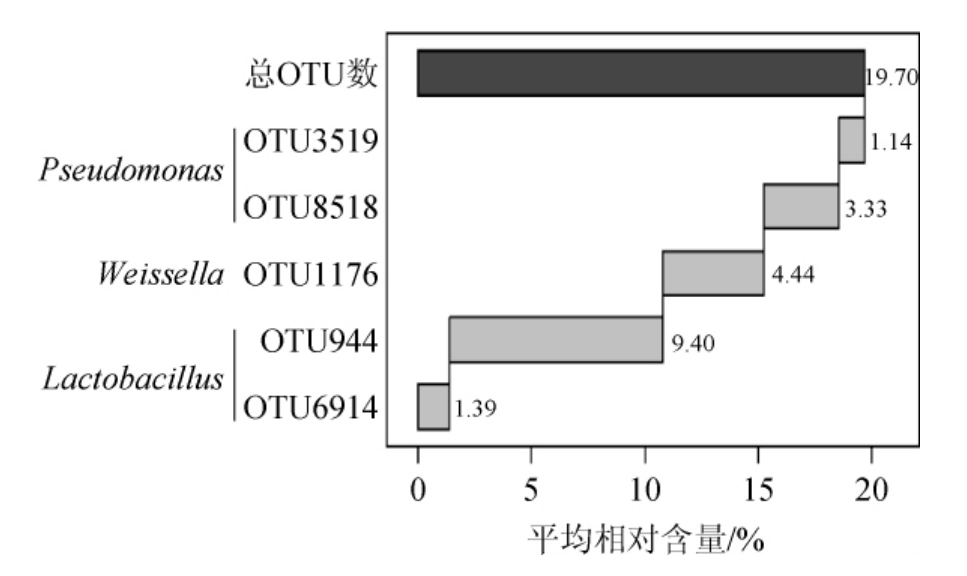
图4 平均相对含量>1.0%的核心OTU
Fig.4 Core OTUs with average relative content more than 1.0%
由图4可知,OTU944和OTU6914被鉴定为Lactobacillus,平均相对含量分别为9.40%和1.39%;OTU8518和OTU3519被鉴定为Pseudomonas,平均相对含量分别为3.33%和1.14%;OTU1176被鉴定为Weissella,平均相对含量为4.44%;该结果进一步证实了,建始地区酸豇豆盐水中乳酸菌以Lactobacillus和Weissella为主。
2.4 乳酸菌的分离鉴定
由测序结果可知,建始地区酸豇豆盐水中蕴含了极为丰富的乳酸菌资源,为进一步明确其中的乳酸菌类群,本研究采用纯培养的方式对其中乳酸菌进行了分离鉴定,鉴定结果如表2所示。
表2 乳酸菌鉴定结果
Table 2 Identification results of lactic acid bacteria

由表2可知,从5 个样品中共分离出了13 株乳酸菌,除JD2-1被鉴定为发酵乳杆菌(L.fermentum)以外,其他12 株乳酸菌均被鉴定为植物乳杆菌(L.plantarum),占分离株的92.3%。由此可见,L.plantarum为建始地区酸豇豆盐水中的优势乳酸菌。本研究团队曾从采集自恩施土家族苗族自治州恩施市的6份酸豇豆盐水样品中分离到鼠李糖乳杆菌(L.rhamnosus)、L.fermentum和L.plantarum 3个种的23株乳酸菌,其中21 株为L.plantarum[18],相同的原料、相似的地理生态环境和制作工艺,可能是导致两个研究结果相同的根本原因之一。此外,样品JD5中共分离了4 株L.plantarum,明显高于其他4 个样品,这可能与该样品具有较高的细菌多样性和丰度有关。虽然MiSeq高通量测序结果表明,Weissella亦为酸豇豆盐水中的优势乳酸菌,但是采用纯培养技术并未获得其分离株。有研究表明泡菜盐水群落结构会随着泡菜的发酵进程而产生明显变化,发酵后期酸耐受性较强的Lactobacillus成为优势菌,而不耐酸的Weissella和Leuconostoc则逐渐消失[19]。MiSeq高通量测序技术是基于细菌16S rRNA的某一个或几个可变区间进行序列测定,因而即使微生物在发酵基质中已经死亡或失去活性,但只要其基因组DNA没有降解,则可实现成功扩增[20],从而导致了本研究现象的发生。此外,无法分离出Weissella菌株的原因还可能与所使用的培养基和培养条件有关,虽然Weissella的分离培养基亦为MRS培养基[21],但适当调整其成分构成有可能实现菌株的纯培养。
3 结论
建始地区酸豇豆盐水中乳酸菌的多样性较高,乳酸菌群系主要由乳酸杆菌属(Lactobacillus)、魏斯氏菌属(Weis sella)、片球菌属(Pediococcus)和明串珠菌属(Leuconostoc)。Lactobacillus亦是酸豇豆盐水中的优势菌群,平均相对含量达到57.02%,且主要以植物乳杆菌(L.plantarum)为主。
[1]陈功,余文华,张其圣,等.四川泡菜与乳酸菌的研究[J].中国酿造,2012,31(9):151-154.
[2]ZHANG F,TANG Y,REN Y,et al.Microbial composition of spoiled industrial-scale Sichuan paocai and characteristics of the microorganisms responsible for paocai spoilage[J].Int J Food Microbiol,2018,275(6):32-38.
[3]熊涛,李军波,彭飞,等.食盐质量浓度对传统自然发酵圆白菜的菌系结构和代谢的影响[J].食品科学,2015,36(11):172-176.
[4]蔡炯,迟原龙,汪冬冬,等.腐败四川泡菜菌相构成及主要腐败菌特性分析[J].食品科技,2017,42(1):279-283.
[5]LIANG H,YIN L,ZHANG Y,et al.Dynamics and diversity of a microbial community during the fermentation of industrialized Qingcai paocai,a traditional Chinese fermented vegetable food,as assessed by Illumina MiSeq sequencing,DGGE and qPCR assay[J].Ann Microbiol,2018,68(2):111-122.
[6]CAO J,YANG J,HOU Q,et al.Assessment of bacterial profiles in aged,home-made Sichuan paocai brine with varying titratable acidity by PacBio SMRT sequencing technology[J].Food Control,2017,78(8):14-23.
[7]HUANG Z R,HONG J L,XU J X,et al.Exploring core functional microbiota responsible for the production of volatile flavour during the traditional brewing of Wuyi Hong Qu glutinous rice wine[J].Food Microbiol,2018,76(12):487-496.
[8]折米娜,望诗琪,赵慧君,等.酸菜水中细菌多样性研究[J].中国调味品,2019,44(1):26-31.
[9]CALLAJAN B J,MCMURDIE P J,HOLMES S P.Exact sequence variants should replace operational taxonomic units in Marker-gene data analysis[J].ISME J,2017,11(12):2639-2643.
[10]HAAS B J,GEVERS D,EARL A M,et al.Chimeric 16S rRNA sequence formation and detection in Sanger and 454-pyrosequenced PCR amplicons[J].Genome Res,2011,21(3):494-504.
[11]COLE J R,WANG Q,FISH J A,et al.Ribosomal Database Project:data and tools for high throughput rRNA analysis[J]. Nucl Acid Res,2014,42(D1):D633-D642.
[12]ZHANG J,WANG X,HUO D,et al.Metagenomic approach reveals microbial diversity and predictive microbial metabolic pathways in Yucha,a traditional Li fermented food[J].Sci Rep,2016,6(1):1-9.
[13]代程洋,陈江红,郭壮,等.枣阳酸浆水来源乳酸菌对泡菜及泡菜水发酵品质的影响[J].中国酿造,2019,38(5):54-58.
[14]XIA Y,LIU X,WANG G,et al.Characterization and selection of Lacto-bacillus brevis starter for nitrite degradation of Chinese pickle[J]. Food Control,2017,78(8):126-131.
[15]THOMER L,SCHNEEWIND O,MISSIAKAS D.Pathogenesis of Staphylococcus aureus bloodstream infections[J].Ann Rev Pathol,2016,11(3):343-364.
[16]MAYER-HAMBLETT N,RETSCH-BOGART G,KLOSTER M,et al.Azithromycin for early Pseudomonas infection in cystic fibrosis.The OPTIMIZE randomized trial[J].Am J Respir Crit Care Med,2018,198(9):1177-1187.
[17]ZHANG J,GUO Z,XUE Z,et al.A Phylo-functional core of gut microbiota in healthy Young Chinese cohorts across lifestyles,geography and ethnicities[J].ISME J,2015,9(9):1979-1990.
[18]舒娜,张芮,宋玥晗,等.恩施市酸豇豆中乳酸菌多样性解析及其分离株发酵特性评价[J].中国食品添加剂,2019,30(2):64-70.
[19]XIONG T,GUAN Q,SONG S,et al.Dynamic changes of lactic acid bacteria flora during Chinese sauerkraut fermentation[J].Food Control,2012,26(1):178-181.
[20]TIAN S,TIAN Z,YANG H,et al.Detection of viable bacteria during sludge ozonation by the combination of ATP assay with PMA-Miseq sequencing[J].Water,2017,9(3):166-170.
[21]COLLINS M D,SAMELIS J,METAXOPOULOS J,et al.Taxonomic studies on some Leuconostoc-like organisms from fermented sausages:description of a new genus Weissella for the Leuconostoc paramesenteroides group of species[J].J Appl Bacteriol,1993,75(6):595-603.