鲊广椒是我国云南、贵州、四川等地区的特色传统发酵食品,湖北省咸丰市、襄阳市、当阳市和荆门市亦有制作和食用鲊广椒的习俗[1]。鲊广椒通常以粉碎的大米面为主要原料,同时以鲜红辣椒、玉米面和食盐作为辅料,上述物料混合均匀后装入坛中,同时塞入稻草并将坛子倒扣于水盆中发酵而成[2]。由于鲊广椒的发酵原料为蔬菜和淀粉质食材,发酵方式为厌氧固态发酵,制作方法与酸奶、泡菜及腊肉等发酵食品均存在较大的差异,形成了其有别于其他发酵食品的独特菌群结构[3]。
目前已有关于鲊广椒菌群多样性的研究,王玉荣等[4]采用MiSeq高通量测序技术对当阳地区鲊广椒微生物多样性进行解析,结果发现当阳地区鲊广椒优势细菌属包括乳酸杆菌属(Lactobacillus)、魏斯氏菌属(Weissella)、片球菌属(Pediococcus)、葡萄球菌属(Staphylococcus)、肉胞菌属(Carnimonas)等,其中乳酸菌的相对含量高达81.75%;雷炎等[5]通过对鲊广椒中的乳酸菌进行分离鉴定,从中共分离鉴定出植物乳杆菌(Lactobacillus plantarum)10株、发酵乳杆菌(Lactobacillus fermentum)3株、副干酪乳杆菌(Lactobacillus paracasei)和食品乳杆菌(Lactobacillus alimentarius)各1株,表明植物乳杆菌是鲊广椒中的优势乳酸菌。然而,不同地区生产鲊广椒所使用的各物料比例及制作工艺不尽相同,导致产品风味和菌群结构可能存在较大区别[6],鲊广椒产品细菌群落结构在不同地区间是否存在差异仍未得到有效回答。
为了揭示不同地区鲊广椒产品细菌群落结构特点及彼此间的差异,本研究采集了11份湖北省咸丰市的鲊广椒样品,并采用Illumina MiSeq高通量测序技术全面揭示了咸丰地区鲊广椒的菌群结构。同时通过结合王玉荣等[4]采集自当阳地区的鲊广椒的测序数据,探究两地区鲊广椒菌群结构的异同。本研究将进一步丰富人们对不同地区鲊广椒细菌群落结构的认识,同时为后续鲊广椒的工业化生产奠定基础。
1 材料与方法
1.1 材料与试剂
1.1.1 材料
鲊广椒样品:本研究11份样品(XF1~XF11)于2018年10月采集自湖北省咸丰市不同农户家。所有样品均由红辣椒和大米发酵制作,发酵时间在一个月左右,无酸臭味且无霉斑,色泽微红,外观无明显差异。样品采集后装入采样袋并迅速置于含有冰袋的采样箱,冷链运送回实验室于24 h内完成脱氧核糖核酸(deoxyribonucleic acid,DNA)提取。
1.1.2 试剂
QIAGENDNeasymericon Food试剂盒:德国QIAGEN公司;脱氧核糖核苷三磷酸(deoxy-ribonucleoside triphosphates,dNTPs)、聚合酶链式反应(polymerase chain reaction,PCR)缓冲液、脱氧核糖核酸(deoxyribonucleic acid,DNA)聚合酶(5 U/μL):北京全式金生物技术有限公司;16S rRNA V3-V4区引物:武汉天一辉远生物科技有限公司。
1.2 仪器与设备
CT15RE台式冷冻离心机:日本HITACHI公司;2100芯片生物分析仪:美国Agilent公司;ND-2000C微量紫外分光光度计:美国Thermo公司;vetiri梯度基因扩增仪:美国AB公司;UVPCDS8000凝胶成像分析系统:美国ProteinSimple公司;MiSeqPE300高通量测序平台:美国Illumina公司;R930机架式服务器:美国DELL公司。
1.3 实验方法
1.3.1 DNA提取、PCR扩增及高通量测序
使用QIAGEN DNeasy maricon Food试剂盒分别提取11份鲊广椒样品的宏基因组DNA,以其为模板,使用引物338F(5'-ACTCCTACGGGAGGCAGCAG-3')和806R(5'-GGACTACHVGGGTWTCTAAT-3')对样品16S rRNA V3-V4区域进行PCR扩增。PCR扩增体系(20 μL):10×PCR缓冲液(含Mg2+)4 μL,dNTPs(2.5 mmol/L)2 μL,正反向引物(5 μmol/L)各0.8μL,DNA聚合酶0.4μL,DNA模板10ng,双蒸水(ddH2O)补充至20 μL。PCR扩增条件:95 ℃预变性3 min;95 ℃变性30 s,55 ℃退火30 s,72 ℃延伸45 s,共35个循环;72 ℃末端延伸10 min。PCR扩增产物由上海美吉Illumina MiSeq高通量测序。
1.3.2 细菌多样性分析
对测序后的序列进行质控(目的片段碱基长度>300bp,引物和标签序列完全匹配,整条序列质量>20的碱基所占比例>93%)。测序数据经过质控后,使用QIIME(V1.9.1)平台进行后续生物信息学分析[7],主要分析步骤如下:(1)使用PyNAST校正对齐序列[8];(2)使用Uclust工具在100%相似度条件下对操作分类单元(operational taxonomic units,OTU)进行划分得到非重复的测序序列集;(3)进一步在97%相似度条件下对步骤(2)得到的序列集进行OTU划分[9];(4)使用Vsearch去除属于嵌合体序列的OTU[10];(5)使用RDP classifier结合目前最常用的RDP(Relace 11.5)[11]、Greengene(Version 13.8)[12]和Sliva(Version 132)[13]数据库进行同源性比对确定各OTU的分类学地位[14],使用内部脚本整合3个数据库的注释结果,确定各OTU的最终分类学地位;(6)基于各OTU的代表性序列,使用FastTree软件构建进化树;(7)标准化各样品测序序列数后,在步骤(6)的基础上进行α多样性和β多样性分析。通过绘制稀疏曲线和香农(Shannon)多样性曲线确定本研究各样品测序深度是否足够代表样品中的菌群组成。通过计算相同测序深度下的OTU数量指数和Shannon多样性指数评估样品中相应菌群的丰度和多样性,具体操作为以测序量最低的样品为基准,各样品随机抽取相同的序列数,并计算上述两个α多样性指标,重复该过程1 000次,取各指标1 000次计算得到的平均值作为该样品最终的α多样性指数值。
1.3.3 数据处理
使用非参数的Mann-Whitney秩和检验对不同地区鲊广椒样品菌群组成进行差异显著性检验,使用Benjaminiand Hochberg的方法校正多次检验的假阳性率[15]。使用基于非加权(Unweighted)UniFrac距离和加权(Weighted)UniFrac距离的主坐标分析(principal coordinates analysis,PCoA)直观展示不同地区鲊广椒样品菌群结构的差异。使用置换多元方差分析(permutational multivariate analysis of variance,PERMANOVA)检验地域对鲊广椒样品影响的显著性。使用Spearman相关性分析确定鲊广椒菌群彼此间的相关性。使用Origin(V8.6)软件和R语言软件(V3.5.0)对数据进行可视化。
2 结果与分析
2.1 咸丰地区鲊广椒样品测序结果及α多样性分析
对11份采集自湖北省咸丰市的鲊广椒样品细菌16S rRNA V3-V4区基因序列进行Illumina MiSeq高通量测序,共得到383 028条高质量序列,平均每份样品测序量为34 821条序列(表1)。为了反映当前测序量是否足够代表样品中的菌群组成,本研究使用稀疏曲线和Shannon多样性曲线对各样品测序量进行了评估,结果见图1。由图1a可知,随着测序量增加,稀疏曲线斜率下降,但始终未达到水平状态,但由图1b可知,随着测序量增加,所有样品的Shannon多样性曲线均已趋向水平,表明随着测序量增加虽然有可能发现新的种,但当前测序量足以揭示出样品中主要菌群的组成[16]。

图1 各鲊广椒样品细菌菌群的稀疏曲线(a)和Shannon曲线(b)
Fig.1 Rarefaction (a) and Shannon (b) curves of bacterial community of Zhaguangjiao samples
α多样性指标可以反映不同样品中菌群的丰度和多样性差异。本研究使用获得的OTU数量指数反映菌群的丰度,选取Shannon指数反映样品中菌群的多样性,结果见表1。
表1 鲊广椒样品的测序序列信息和α多样性指数
Table 1 Sequence information and α-diversity index of Zhaguangjiao samples
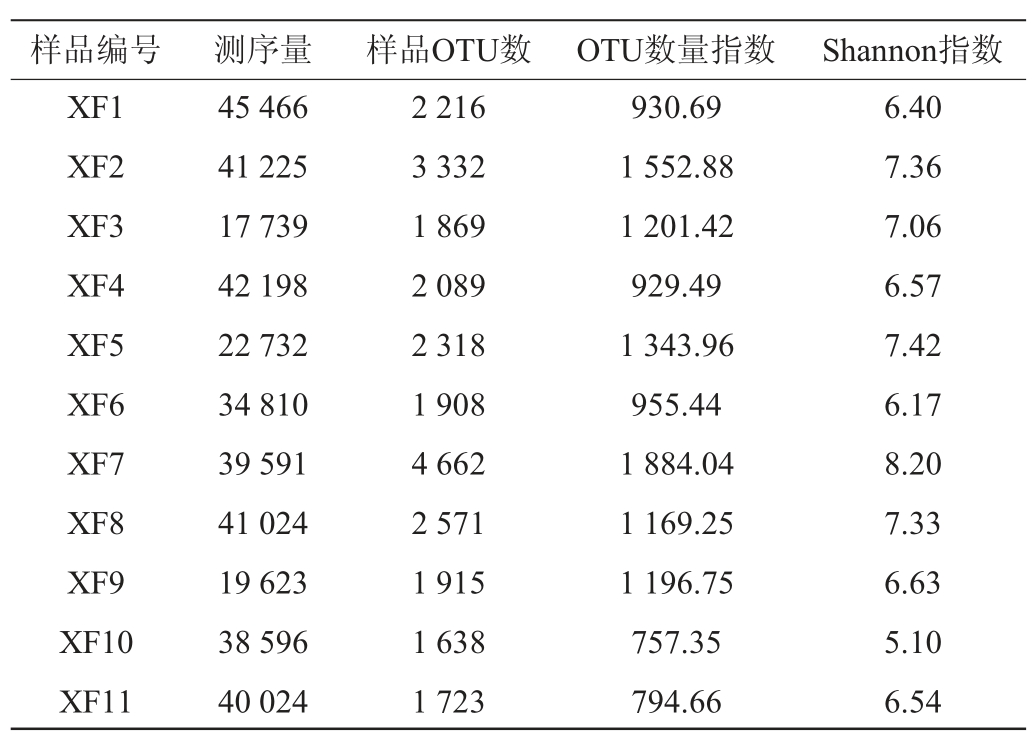
由表1统计发现,湖北省咸丰市的鲊广椒样品OTU数量指数和Shannon多样性指数分别为1 156.0±102.6和6.8±2.0,整体拥有较高的细菌丰度和多样性,但不同样本间存在一定的差异,这种差异性可能与不同农户生产过程的卫生条件不同、制作材料的差异等有很大关系。
2.2 咸丰地区鲊广椒细菌群落结构组成分析
咸丰地区鲊广椒样品中共注释得到25个细菌门和451个细菌属,本研究将平均相对含量>1%的细菌门和细菌属分别定义为优势细菌门和优势细菌属。其中优势细菌门包括硬壁菌门(Firmicutes)、变形菌门(Proteobacteria)和放线菌门(Actinobacteria),平均相对含量分别为85.33%、10.91%和2.49%。优势细菌属共有6个,结果见图2。
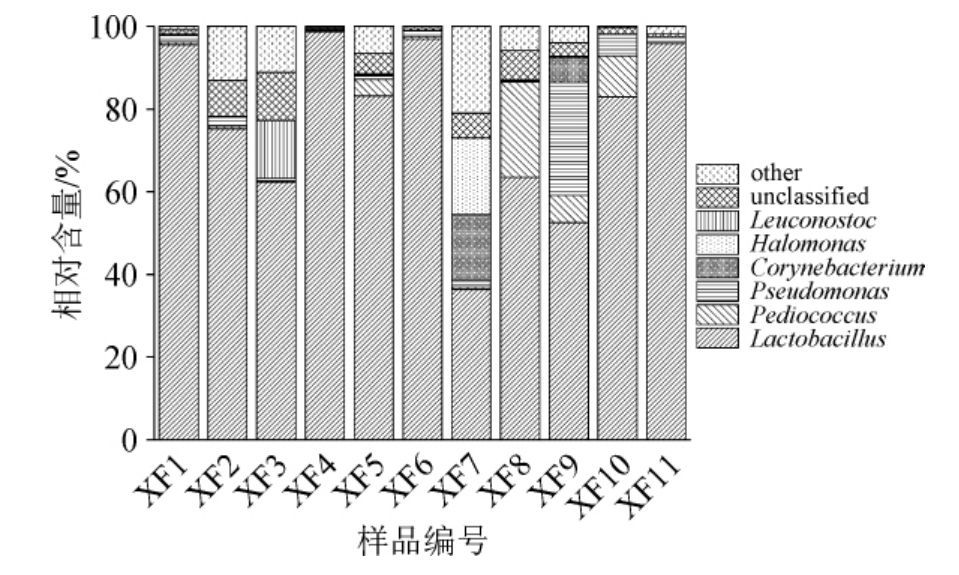
图2 咸丰地区鲊广椒属水平主要细菌的相对丰度
Fig.2 Relative abundances of major bacteria in Zhaguangjiao from Xianfeng region at the genus level
由图2可知,咸丰地区鲊广椒样品中优势细菌属分别为乳酸杆菌(Lactobacillus)、片球菌属(Pediococcus)、假单胞菌属(Pseudomonas)、棒状杆菌属(Corynebacterium)、盐单胞菌属(Halomonas)和明串珠菌属(Leuconostoc),平均相对含量分别为76.63%、4.25%、3.87%、2.02%、1.74%、1.36%。由图2亦可知,乳酸杆菌是所有鲊广椒样品中的主要菌属,而不同的样品间仍表现出了一定的菌群差异,如棒状杆菌属和盐单胞菌属主要在样品XF7中有分布,而假单胞菌属仅在样品XF9中相对含量较高。值得注意的是,本研究分别在91%、81%和73%的样本中检测到了肠杆菌属(Enterobacter)、克雷伯氏菌属(Klebsiella)和芽孢杆菌属(Bacillus),并且在64%的样本中同时检测到了上述三种菌属。
乳酸杆菌属和片球菌属是咸丰地区鲊广椒中的主要优势菌属,这与王玉荣等[4]基于当阳地区采集的鲊广椒样品得到的结论一致。乳酸杆菌属和片球菌属均属于乳酸菌的范畴,乳酸菌具有高效的乳酸产生能力和细菌素(如乳酸链球菌肽)分泌能力[17],其本身也具有较高的酸耐受能力,同时可以抑制多种类型有害微生物的生长,乳酸菌的上述特性对于保证发酵食品的安全性亦具有十分重要的作用[18],同时这可能也是两者在鲊广椒中相对含量最高的原因。此外,乳酸杆菌属的细菌长期以来被授予普遍认为是安全的(generally recognized as safe,GRAS)地位[19],该菌属的众多菌株在目前的益生菌市场广泛应用。然而,目前产业化生产中使用的乳酸杆菌属的菌株多分离筛选自传统发酵乳制品和发酵蔬菜制品等[20],而鲊广椒作为我国华中及西南地区流行的传统发酵食品,一直未得到充分的关注和研究,加之其发酵基质和制作工艺与乳制品和发酵蔬菜等均存在较大差异,导致其所蕴含的潜在益生菌资源未得到很好的挖掘。因此,在后续研究中进一步加强对鲊广椒中乳酸菌的分离保藏和筛选将会对发酵食品的产业化推动具有重要意义。本研究结果还发现在部分样品中假单胞菌属占有较高的比例,该菌属是引起皮肤、肠道、上下呼吸道和直肠周围区域的感染的常见菌属[21]。同时在大量样本中检测到了肠杆菌属、克雷伯氏菌属和芽孢杆菌属的存在,这三类菌属均属于致病共栖菌的范畴[22-23],对人体健康构成潜在的威胁。表明鲊广椒家庭作坊相对开放的制作环境引入了一些环境中的潜在有害菌,进一步优化鲊广椒的制作工艺尤为重要。
2.3 鲊广椒中细菌核心菌群组成分析
为了揭示对鲊广椒发酵可能起到关键作用的菌群,本研究将在咸丰地区和当阳地区[4]90%以上样品中均出现的OTU定义为鲊广椒的核心OTU,鲊广椒中核心OTU的分布结果见表2。
表2 鲊广椒核心OTU的分布
Table 2 Distribution of core OTU in Zhaguangjiao
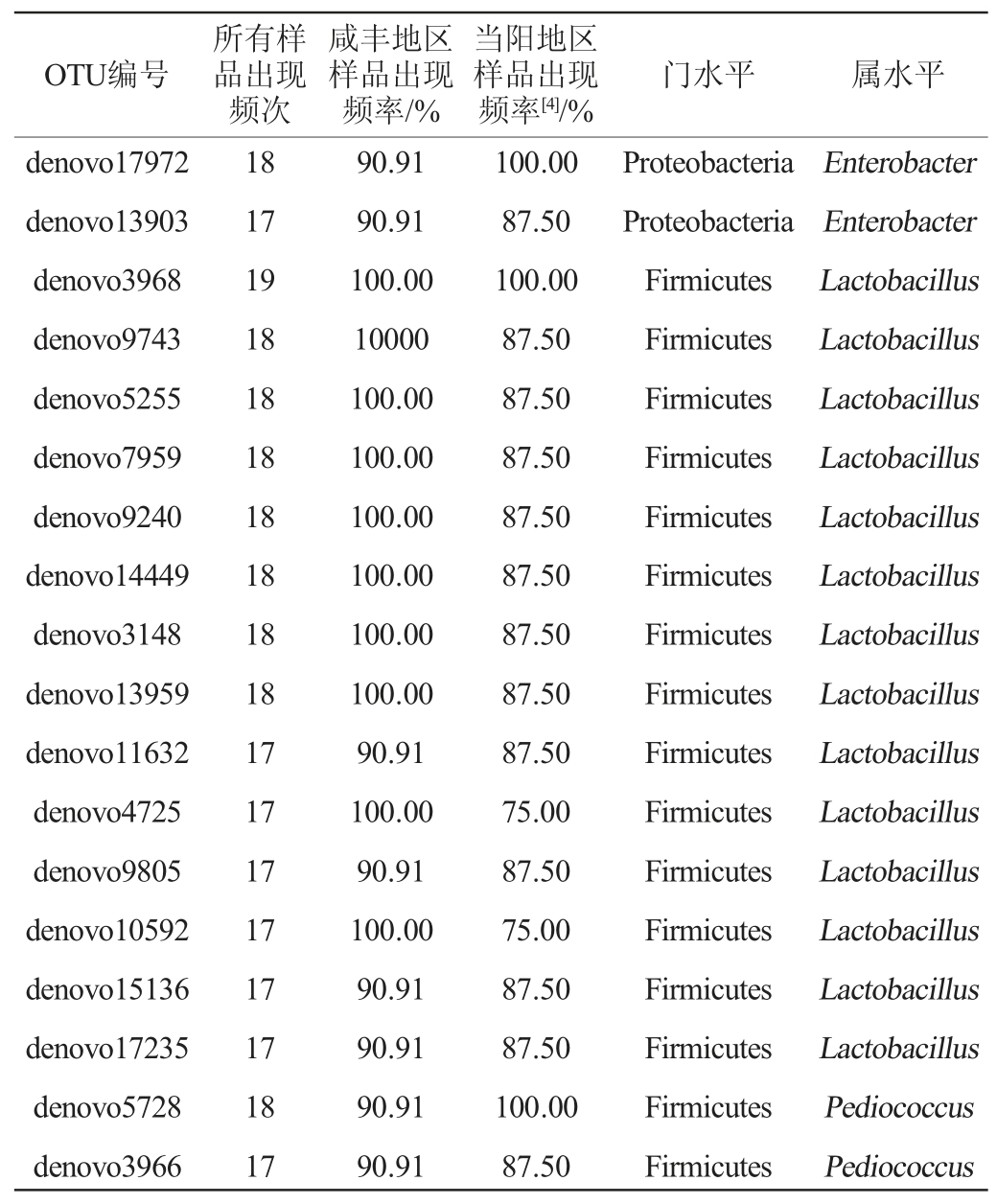
由表2可知,共有18个OTU满足该阈值条件,其中有2个OTU注释结果分别为片球菌属和肠杆菌属(Enterobacter),其余核心OTU注释结果均为乳酸杆菌属,再次表明乳酸杆菌属和片球菌属的细菌在鲊广椒发酵中可能起到了主导作用。而肠杆菌属是一种机会性的产生内毒素的病原体[24],有研究报道该菌属占患有糖尿病、高血压和其他严重代谢恶化的病态肥胖者肠道细菌的35%[25]。该菌属在鲊广椒产品中广泛存在,提示人们需要进一步加强对鲊广椒生产过程的质量控制。
2.4 咸丰与当阳地区鲊广椒样品细菌菌群结构比较分析
本研究基于加权和非加权UniFrac距离对咸丰和当阳地区收集的鲊广椒样品进行了主坐标分析,结果见图3。
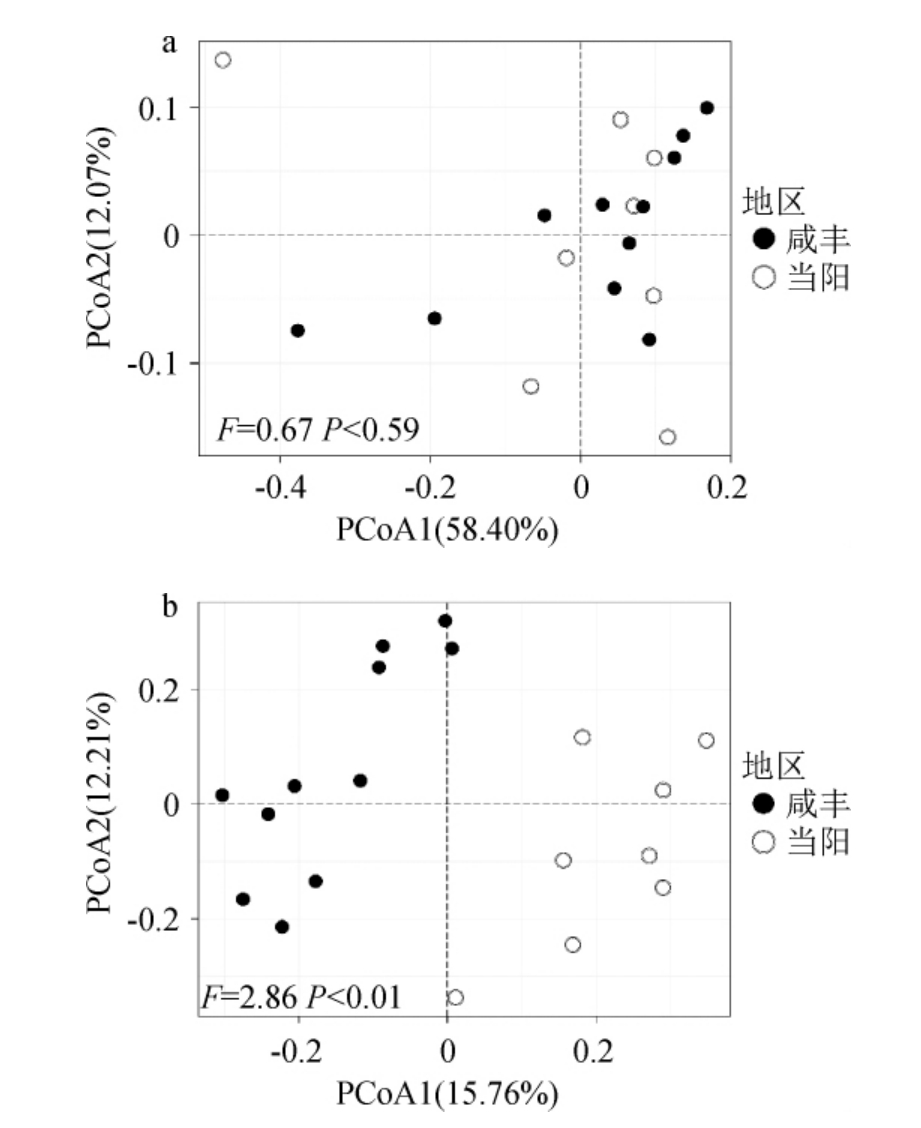
图3 基于加权UniFrac距离(a)和非加权UniFrac距离(b)咸丰与当阳地区鲊广椒样品细菌菌群结构主坐标分析结果
Fig.3 Results of principal coordinate analysis of bacterial community structure in Xianfeng and Dangyang based on weighted UniFrac distance (a) and unweighted UniFrac distance (b)
由图3可知,仅在基于非加权UniFrac距离进行主坐标分析时两个地区的样品存在明显的聚类趋势。此外,基于非加权UniFrac距离的置换多元方差分析的结果亦显示两个地区的鲊广椒样品菌群结构存在极显著差异(P<0.01)。结果表明,咸丰和当阳地区生产的鲊广椒样品中高丰度的菌群组成较为相似,而一些低丰度的菌群存在显著差异。
通过进一步使用OTU数量指数和Shannon指数对咸丰和当阳地区采集的鲊广椒样品进行菌群丰度和多样性评估,结果见图4。

图4 咸丰和当阳地区鲊广椒样品细菌α多样性比较
Fig.4 Comparison of α-diversity of bacteria in Zhaguangjiao samples from Xianfeng and Dangyang
“**”表示差异极显著(P<0.01)。
由图4可知,咸丰地区鲊广椒样品中菌群的OTU数量指数和Shannon指数均极显著高于当阳地区样品(P<0.01),表明咸丰地区鲊广椒样品中细菌的丰度和多样性极显著高于当阳地区(P<0.01)。为了揭示咸丰和当阳地区鲊广椒样品菌群的具体差异,本研究进一步使用Mann-Whitney秩和检验在属水平对两个地区鲊广椒样品的细菌菌群组成进行差异显著性检验,发现在两个地区共有35个细菌属的相对含量存在显著差异(P<0.05)。其中铜绿假单胞菌属(Pseudomonas)在咸丰地区样品中的相对含量(3.87%)显著高于当阳地区样品(0.38%)(P<0.01),而红球菌属(Rhodococcus)和芽孢杆菌属(Bacillus)则在当阳地区相对含量(0.30%、0.16%)显著高于咸丰地区(0.01%、0.06%)(P<0.01),其他存在显著差异的菌属在样品中的相对含量均比较低(平均相对含量<0.1%),这与主坐标分析结果相一致。然而,导致众多低丰度菌属在两地区鲊广椒样品中存在显著差异的原因尚不可知,本研究推测可能与两个地区居民在鲊广椒制作过程中使用的物料比例、制作工艺和加工环境存在一定差异有关[26]。
2.5 鲊广椒样品细菌菌群关联性分析
为了进一步揭示鲊广椒菌群彼此间共生或拮抗关系,本研究使用Spearman相关性分析在属水平检验了鲊广椒样品细菌之间的相关关系,其中具有显著相关性的菌群见图5。
由图5可知,乳杆菌属与片球菌属、葡萄球菌属(Staphylococcus)、棒状杆菌属和盐单胞菌属具有极显著负相关关系(P<0.01);葡萄球菌属与盐单胞菌属、肠杆菌属和棒状杆菌属之间具有显著正相关关系(P<0.05)。此外,魏斯氏菌属(Weissella)与明串珠菌属和肠杆菌属之间亦存在显著正相关关系(P<0.05)。鲊广椒的生产需多种微生物菌群协调作用,对这些微生物菌群相互作用关系的解析将有利于定向控制发酵过程中微生物种类,提高生产效率,改善鲊广椒产品风味和保障产品质量安全。
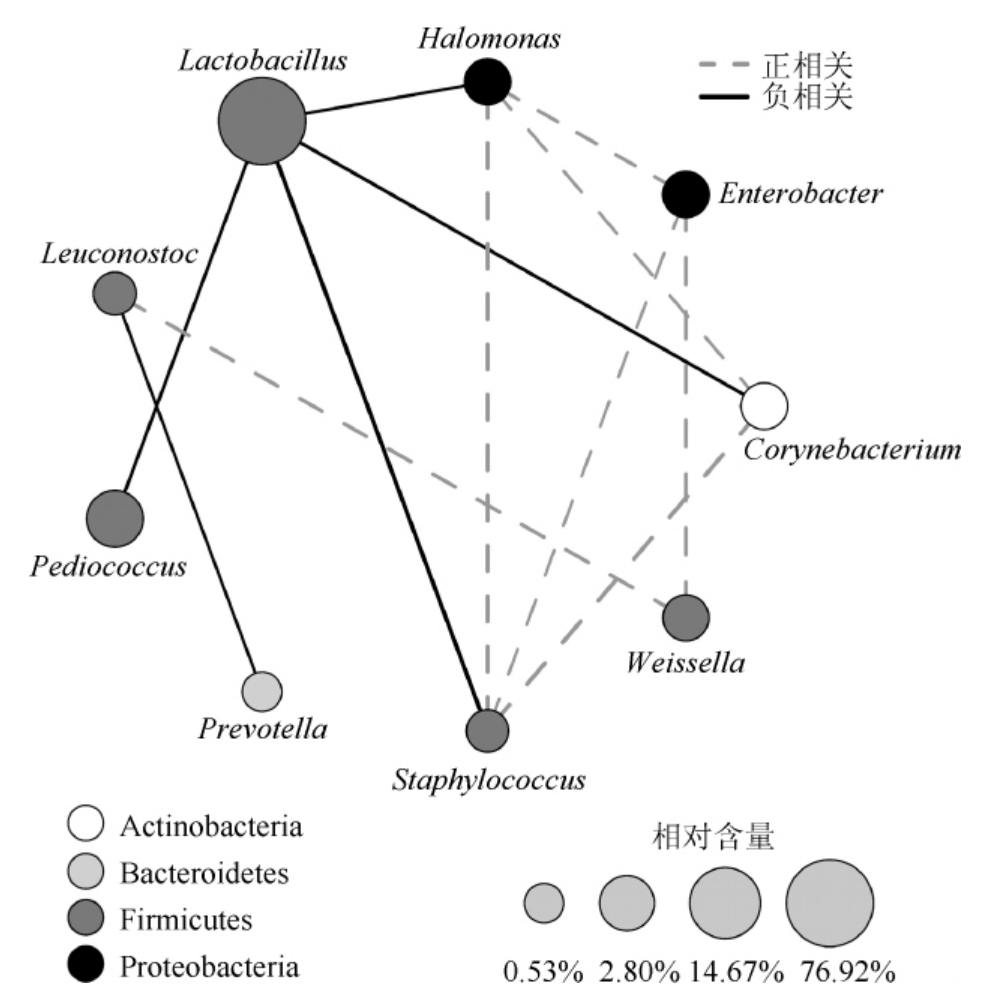
图5 鲊广椒样品细菌属之间的相关性
Fig.5 Correlation of bacteria at genus level in Zhaguangjiao
3 结论
本研究采集了11份湖北省咸丰市的鲊广椒样品,在对咸丰地区鲊广椒细菌群落结构分析的基础之上,通过结合之前研究报道的当阳地区鲊广椒样品分析数据,探究上述两地区鲊广椒菌群结构的异同。结果表明,咸丰地区鲊广椒样品优势细菌门为硬壁菌门、变形菌门和放线菌门,平均相对含量分别为85.33%、10.91%和2.49%;优势细菌属分别为乳酸杆菌、片球菌属、假单胞菌属、棒状杆菌属、盐单胞菌属和明串珠菌属,平均相对含量分别为76.63%、4.25%、3.87%、2.02%、1.74%、1.36%。咸丰地区鲊广椒中细菌的丰度和多样性均极显著高于当阳地区(P<0.01),一些低丰度的菌群在两个地区的鲊广椒样品中存在显著差异(P<0.05)。乳杆菌属和片球菌属是两个地区鲊广椒中的主要优势菌属和核心菌属,同时鲊广椒中广泛分布有肠杆菌属、克雷伯氏菌属和芽孢杆菌属等致病共栖菌,进一步加强鲊广椒生产过程的质量控制是提升鲊广椒品质和质量安全亟待解决的问题。相关性分析发现,鲊广椒发酵菌群彼此间存在密切的相关关系,表明鲊广椒的生产需要多种微生物的协调作用。
[1]王巧碧,王丹,赵欠,等.SDE 和SPME 法对鲊海椒发酵中香气组成的比较分析[J].食品科学,2016,37(4):108-114.
[2]王微,阚建全.响应面法优化鲊辣椒的纯种发酵工艺[J].食品科学,2014,35(7):143-148.
[3]张逸舒,王玉荣,陈芸曼,等.保康鲊广椒中乳酸菌的分离鉴定及其发酵特性的评价[J].食品研究与开发,2019,40(16):159-165.
[4]王玉荣,沈馨,董蕴,等.鲊广椒细菌多样性评价及其对风味的影响[J].食品与机械,2018,34(4):25-30.
[5]雷炎,马佳佳,雷敏,等.恩施鲊广椒乳酸菌的分离鉴定及其对挥发性风味物质的影响[J].中国酿造,2019,38(7):126-130.
[6]葛平珍,王丹,周才琼.不同淀粉源对鲊海椒发酵过程中功能成分的影响[J].食品科学,2015,36(21):191-195.
[7]CAPORASO J G,KUCZYNSKI J,STOMBAUGH J,et al.QIIME allows analysis of high-throughput community sequencing data[J]. Nat Method,2010,7(5):335-336.
[8]CAPORASO J G,BITTINGER K,BUSHMAN F D,et al.PyNAST:a flexible tool for aligning sequences to a template alignment[J].Bioinformatics,2010,26(2):266-267.
[9]EDGAR R C.Search and clustering orders of magnitude faster than BLAST[J].Bioinformatics,2010,26(19):2460-2461.
[10]ROGNES T,FLOURI T,NICHOLS B,et al.VSEARCH:a versatile open source tool for metagenomics[J].Peer J,2016,4:e2584.
[11]COLE J R,CHAI B,FARRIS R J,et al.The ribosomal database project(RDP-II):introducing myRDP space and quality controlled public data[J].Nucl Acid Res,2007,35:169-172.
[12]DESANTIS T Z,HUGENHOLTZ P,LARSEN N,et al.Greengenes,a chimera-checked 16S rRNA gene database and workbench compatible with ARB[J].Appl Environ Microbiol,2006,72(7):5069-5072.
[13]QUAST C,PRUESSE E,YILMAZ P,et al.The SILVA ribosomal RNA gene database project:improved data processing and web-based tools[J].Nucl Acid Res,2013,41:590-596.
[14]COLE J R,WANG Q,FISH J A,et al.Ribosomal Database Project:data and tools for high throughput rRNA analysis[J].Nucl Acid Res,2014,42:633-642.
[15]BENJAMINI Y,HOCHBERG Y.Controlling the false discovery rate:a practical and powerful approach to multiple testing[J].J Royal Stat Soc Series B,1995,57(1):289-300.
[16]侯强川,郭壮,张家超,等.俄罗斯卡尔梅克共和国发酵蔬菜中细菌多样性研究[J].食品与发酵工业,2014,40(7):16-22.
[17]朱文淼,刘稳.乳酸菌细菌素的分子生物学研究进展[J].中国微生态学杂志,2000(2):55-57,65.
[18]CLEVELAND J,MONTVILLE T J,NES I F,et al.Bacteriocins:safe,natural antimicrobials for food preservation[J]. Int J Food Microbiol,2001,71(1):1-20.
[19]SALMINEN S,DEIGHTON MA,BENNO Y,et al.Lactic acid bacteria in health and disease[M].Lactic acid bacteria:microbiology and functional aspects,2nd ed.New York:Marcel Dekker Inc,1998:211-253.
[20]WU R,WANG L,WANG J,et al.Isolation and preliminary probiotic selection of lactobacilli from koumiss in Inner Mongolia[J].J Basic Microbiol,2009,49(3):318-326.
[21]ROSENZWEIG S D,HOLLAND S M.Phagocyte immunodeficiencies and their infections[J].J Allergy Clin Immunol,2004,113(4):620-626.
[22]ROUND J L,MAZMANIAN S K.The gut microbiota shapes intestinal immune responses during health and disease[J].Nat Rev Immunol,2009,9(5):313-323.
[23]NAKAMOTO N,SASAKI N,AOKI R,et al.Gut pathobionts underlie intestinal barrier dysfunction and liver T helper 17 cell immune response in primary sclerosing cholangitis[J].Nat Microbiol,2019,4(3):492-503.
[24]SANDERS W,SANDERS C C. Enterobacter spp.:pathogens poised to flourish at the turn of the century[J].Clin Microbiol Rev,1997,10(2):220-241.
[25]KANG Y,ZHANG X,CAI Y,et al.Gut microbiota and metabolic disease:from pathogenesis to new therapeutic strategies[J].Rev Med Microbiol,2016,27(4):141-152.
[26]王慧琳,周炜城,任聪,等.传统发酵食品微生物学研究进展[J].生物学杂志,2018,35(6):1-5.