我国大豆发酵制品种系繁多,常见的酱油、豆豉、甜面酱和腐乳等作为调味品已成为我国居民日常饮食的重要组成部分[1]。较之泡菜和酸奶等发酵植物及发酵乳制品,由于制作环境更为开放且pH值偏于中性,因而发酵豆制品具有更高的微生物丰度及多样性[2]。李晓然等[3]研究发现,肠膜明串珠菌和金黄色葡萄球菌为云南地区豆豉中的优势细菌,而耐盐假丝酵母菌为优势真菌;续丹丹等[4]研究表明,腐乳在发酵过程中真菌微生物群系存在动态变化,前期以丝孢菌属、放线菌属和隐球菌属为主,而后期以曲霉属为主。发酵豆制品的微生物群系受原料、制作工艺、制作环境和发酵时间等诸多因素的影响。与常见发酵豆制品不同,渣豆酱是以豆渣为原料制作而成,用食用油煎炸后常作为火锅配菜食用,在湖北省恩施土家族苗族自治州部分县市颇为流行。渣豆酱的制作工艺颇具特色,首先使用铁锅将豆渣炒至颗粒松散,置于“簝箕”摊开晾凉后,于柜中发酵7~10 d左右,发酵完成后添加适量食盐并将其捏成饼状,最后烟熏炕干以便长期保存食用。特殊的原料和制作工艺,可能使渣豆酱具有较为独特的微生物类群,然而目前相关报道尚少。
研究人员常采用纯培养、变性梯度凝胶电泳和高通量测序等技术对发酵食品的微生物类群进行解析,其中MiSeq作为第二代高通量测序技术的代表,具有通量高、成本低和结果准确的优点[5],在发酵乳制品[6]、发酵豆制品[7]及发酵蔬菜制品[8]等相关研究中被广泛应用。恩施土家族苗族自治州鹤峰县作为巴文化的重要发祥地,毗邻湖南省,是湖北省的高山县之一,境内聚居着土家族和苗族等诸多少数民族,饮食文化和生态环境均较为独特。本研究采用MiSeq高通量测序技术对鹤峰地区渣豆酱中优势细菌和真菌类群进行解析,以期为我国特色发酵豆制品微生物多样性研究及产业化推动提供数据支撑。
1 材料与方法
1.1 材料与试剂
1.1.1 材料
渣豆酱:市售。
1.1.2 试剂
食品微生物脱氧核糖核酸(deoxyribonucleic acid,DNA)基因组提取试剂盒:德国QIAGEN公司;DNA聚合酶(5万U/μL)和脱氧核糖核苷三磷酸(deoxy-ribonucleoside triphosphate,dNTP)Mix:北京全式金生物技术有限公司;pMD18-T载体:大连宝生物技术有限公司;引物对338F/806R和SSU0817F/SSU1196R:天一辉远(武汉)生物科技有限公司;Axygen清洁试剂盒:康宁生命科学吴江有限公司。
1.2 仪器与设备
HBM-400B拍击式无菌均质器:天津市恒奥科技发展有限公司;PTC-100型聚合酶链式反应(polymerase chain reaction,PCR)仪、164-5050基础电泳仪:美国BIO-RAD公司;ND-2000C微量紫外分光光度计:美国Nano Drop公司;Fluor Chem FC3型化学发光凝胶成像系统:美国Protein Simple公司;Illumina MiSeq PE250高通量测序平台:美国Illumina公司。
1.3 试验方法
1.3.1 样品采集
渣豆酱样品均于2018年12月下旬采自湖北省恩施土家族苗族自治州鹤峰县(E109°45′~110°38′,N29°38′~30°14′)容美镇海通农贸市场,所有样品均在鹤峰县本地制作销售、无异味且无明显霉变。样品采集后,编号分别为DJ1、DJ2和DJ3,单独置于采样袋中常温带回实验室。为便于区分,细菌测序时编号记为BDJ1、BDJ2和BDJ3,真菌测序时编号记为FDJ1、FDJ2和FDJ3,其中BDJ1和FDJ1为同一样品,以此类推。
1.3.2 渣豆酱宏基因组DNA提取、PCR扩增和MiSeq高通量测序
DNA提取:称取渣豆酱样品10 g,加入40 mL无菌超纯水,使用拍击器拍击5 min后,300 r/min离心10 min,吸取上清液,参照DNA基因组提取试剂盒提取样品的总DNA。
PCR扩增:将检验合格的DNA使用加入了7 个核苷酸标签(barcode)的引物进行PCR扩增,其中细菌16S rRNA V3-V4区PCR扩增参照文献9中的方法,真菌18S rRNA V4-V5区PCR扩增参照文献[9]中的方法。
MiSeq高通量测序:由上海美吉生物医药科技有限公司采用Illumina MiSeq PE250高通量测序平台完成测序,每个样品测序深度不低于30 000 条reads(条带)。
1.3.3 MiSeq高通量测序数据的质控及生物信息学分析
参照文献[10]中的方法质控后,将序列文件使用QIIME(v1.70)平台进行微生物类群分析。序列排齐(align)后按100%和97%相似性构建分类操作单元(operationaltaxonomic units,OTU)矩阵[11],同时去除含有嵌合体序列的OTU,余下细菌OTU各选择1条代表性序列在核糖体数据库项目(ribosomal database project,RDP)[12]、Greengenes[13]和Silva[14]数据库中进行同源性比对确定其分类学地位,若存在3个数据库比对结果不一致的情况则将其视为未鉴定的细菌属;真菌序列在Silva数据库中比对。序列比对后,建立系统发育树[15],并在此基础上进行超1和香农等α多样性指数计算[16]。将平均相对含量>1.0%的门、属和OTU分别定义为优势门、属和OTU,将在3 个样品中均存在的门、属和OTU分别定义为核心门、属和OTU[17]。
1.3.4 多元统计分析
利用SAS软件中Pearson相关系数法计算优势细菌属与真菌属间的相关系数,采用R语言(v3.6.2)对测序数据进行作图分析。
2 结果与分析
2.1 基于门和属水平的细菌多样性分析
本研究采用MiSeq高通量测序技术对3 个渣豆酱样品进行了测序,共得到129 187 条序列,经100%和97%相似度划分后,产生5 613 个OTU,未检测到嵌合体的存在,每个OTU选择1 条代表性序列进行同源性比对并确定了其分类学地位,优势细菌门如图1所示。
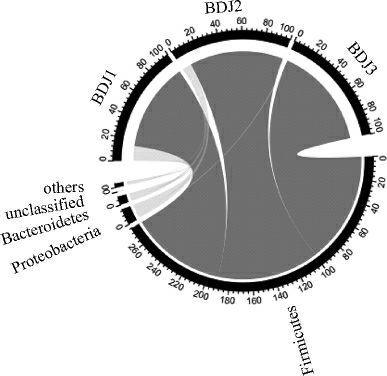
图1 优势细菌门的相对含量
Fig.1 Relative contents of dominate bacterial phyla
由图1可知,优势细菌门为硬壁菌门(Firmicutes)、变形菌门(Proteobacteria)和拟杆菌门(Bacteroidetes),其平均相对含量分别为92.0%、4.46%和2.35%,其中Firmicutes在所有样品中均存在,为唯一的核心细菌门,在样品DJ3中的相对含量甚至高达99.19%。本研究进一步从属水平上对细菌类群进行分析,共鉴定出5个优势属,如图2所示。
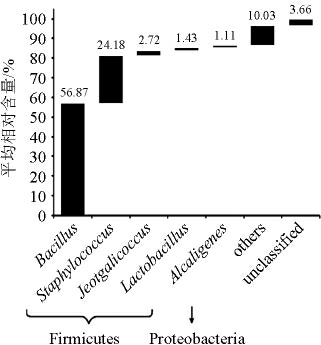
图2 优势细菌属的相对含量
Fig.2 Relative contents of dominate bacterial genera
由图2可知,芽孢杆菌属(Bacillus)(56.87%)、葡萄球菌属(Staphylococcus)(24.28%)、咸海鲜球菌属(Jeotgalicoccus)(2.72%)、乳杆菌属(Lactobacillus)(1.43%)和产碱菌属(Alcaligenes)(1.11%)为优势细菌属。由此可见,渣豆酱中的细菌主要由隶属于硬壁菌门(Firmicutes)的Bacillus和Staphylococcus构成,两者累计平均相对含量高达81.1%。张燕鹏等[18]采用纯培养的方法从江西地区霉豆渣中分离出了3株枯草芽孢杆菌(B.subtilis),并证明了其为霉豆渣中产蛋白酶的主要菌属之一。值得一提的是,渣豆酱中还含有少量的Lactobacillus和Alcaligenes,李伟伟等[19]在对联合发酵豆渣的研究中发现Lactobacillus有降低豆渣中脂肪含量的作用,ZHANG H H等[20]的研究表明粪便产碱菌(A.faecalis)具有降解赭曲霉毒素A的特性。除此之外,渣豆酱中还含有大量Staphylococcus,有报道指出,金黄色葡萄球菌(S.aureus)是感染性心内膜炎的主要病因之一[21],这种现象的产生可能是与其制作环境较为开放有关,因而积极开展渣豆酱中细菌类群的研究,对后续其食用安全品质的提升具有积极的意义。
2.2 基于OTU水平细菌多样性分析
渣豆酱样品细菌OTU的分布结果如图3所示。
由图3可知,样品BDJ1、BDJ2和BDJ3分别含有2 638、2 262和1 211个OTU,各包含了51 831、41 432和35 924条序列,其中有95个OTU在所有样品中均存在,共包含了89 275条序列,占序列总数的69.13%。值得一提的是,在7个平均相对含量>1.0%的核心OTU中有6个隶属于Bacillus,其累计相对含量达44.59%;1个隶属于Staphylococcus,其相对含量为15.15%。由此可见,渣豆酱样品共有大量的核心细菌类群,而这些类群主要隶属于Bacillus和Staphylococcus。
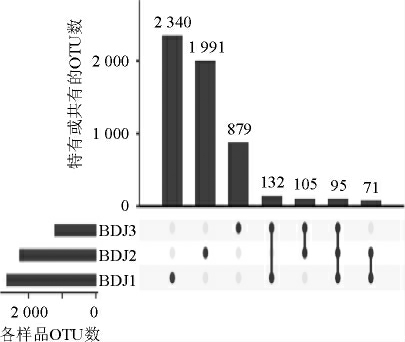
图3 细菌OTU在样品中的分布
Fig.3 Distribution of bacterial OTU in samples
2.3 基于门和属水平的真菌多样性分析
朱蕴兰等[22]使用蛹拟青霉(Paecilomyces militaris)发酵豆渣的方法制备出了豆渣小肽,董伟亮[23]则利用米曲霉(Aspergillus oryzae)发酵豆渣制备出了大豆多肽和多糖,而谢欢等[24]的研究表明,黑曲霉(A.niger)发酵豆渣后能使其中含有高可溶性的膳食纤维。由此可见,真菌在豆渣发酵过程中具有积极的作用。本研究进一步对鹤峰地区渣豆酱中的真菌多样性进行了解析,MiSeq高通量测序共产生了130 377 条序列,按100%和97%相似性划分得到1 817个OTU,未检测到嵌合体的存在,经同源性比对后,其真菌门的含量如图4所示。
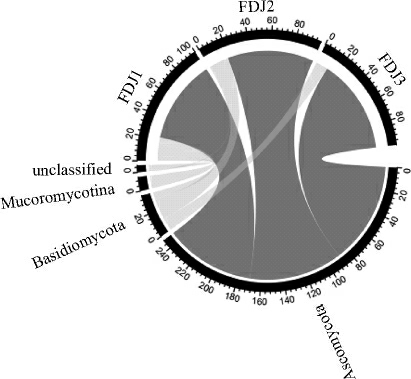
图4 优势真菌门的相对含量
Fig.4 Relative contents of dominate fungal phyla
由图4可知,子囊菌门(Ascomycota)、担子菌门(Basidiomycota)和毛霉亚门(Mucoromycotina)为渣豆酱中的优势真菌门,平均相对含量分别为82.57%、11.86%和3.71%,其中Ascomycota和Basidiomycota存在于所有样品中,而Mucoromycotina仅存在于样品FDJ1中,其相对含量为10.98%。优势真菌属含量分布如图5所示。
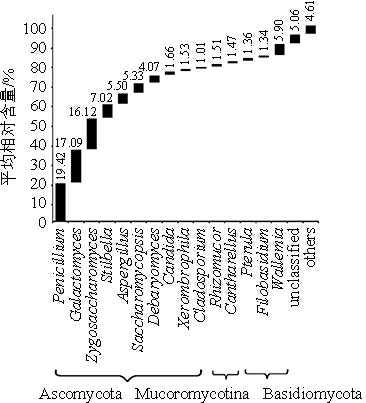
图5 优势真菌属的相对含量
Fig.5 Relative contents of dominate fungal genera
由图5可知,渣豆酱中优势真菌属分别为隶属于子囊菌门(Ascomycota)的青霉属(Penicillium)(19.42%)、耐碱酵母属(Galactomyces)(17.09%)、接合酵母属(Zygosaccharomyces)(16.12%)、束梗孢属(Stilbella)(7.02%)、曲霉属(Aspergillus)(5.50%)、囊复膜孢酵母属(Saccharomycopsis)(5.33%)、德巴利酵母属(Debaryomyces)(4.07%)、念珠菌属(Candida)(1.66%)、Xerombrophila(1.53%)和枝孢属(Cladosporium)(1.01%);隶属于Mucoromycotina的根毛霉属(Rhizomucor)(1.51%)、鸡油菌属(Cantharellus)(1.47%);隶属于Basidiomycota的龙须菌属(Pterula)(1.36%)、丝状黑粉菌属(Filobasidium)(1.34)%和节担菌属(Wallemia)(5.90%)。渣豆酱中共发现了15个优势真菌属,而优势细菌属仅发现了5个,故而细菌和真菌的丰度和多样性可能存在一定的差异。本研究采用超1指数和香农指数对该推论进行了验证,结果发现在测序量均为34 010 条序列时,细菌和真菌的超1指数分别为904±291和2 218±125,经Mood-median检验发现其差异显著(P<0.05),而香农指数分别为4.55±1.32和4.41±0.19,经秩和检验发现其差异不显著(P>0.05)。由此可见,渣豆酱中的真菌主要由隶属于Ascomycota 的Penicillium、Galactomyces和Zygosaccharomyces构成,三者平均累计相对含量为52.63%,且真菌丰度要显著高于细菌。
2.4 基于OTU水平的真菌多样性分析
渣豆酱样品真菌OTU的分布结果如图6所示。
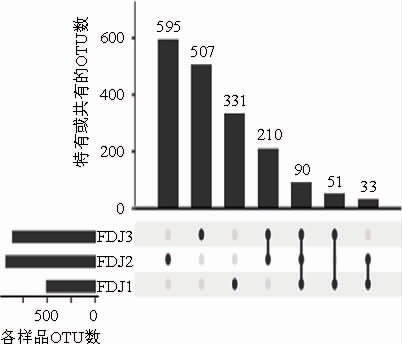
图6 真菌OTU在样品中的分布
Fig.6 Distribution of fungal OTU in samples
由图6可知,样品DJ1中共产生了505个OTU,其中包含39 415 条序列;DJ2共产生了928 个OTU,包含41 101条序列;DJ3共产生了858个OTU,包含49 861条序列。所有样品共有的OTU有90个,其中11个相对含量>1.0%,累计平均相对含量为63.03%。11个OTU中分别有3个隶属于Zygosaccharomyces,2 个隶属于Galactomyces,各有1 个隶属于Xerombrophila、Stilbella、Saccharomycopsis、Penicillium、Debaryomyces和Aspergillus,其中隶属于Penicillium、Zygosaccharomyces和Galactomyces的OTU相对含量分别为18.90%、14.38%和13.70%,3类OTU的累计平均相对含量达到46.98%。由此可见,渣豆酱样品亦共有大量的核心真菌类群,而这些类群主要隶属于Penicillium、Zygosaccharomyces和Galactomyces。
2.5 基于优势细菌和真菌相关性分析
本研究进一步对优势细菌属和真菌属之间的相关性进行了解析,如图7所示。
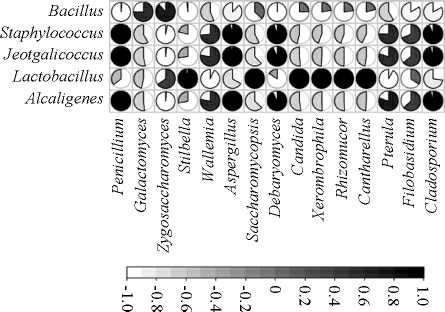
图7 相对含量>1.0%细菌属和真菌属之间的相关性
Fig.7 Correlations among bacterial and fungal genera with relative abundance more than 1.0%
圆圈颜色越深表明越趋向正相关,圆圈颜色越浅表明越趋向负相关,圆圈越完整表示相关性越大。
由图7可知,芽孢杆菌数(Bacillus)与德巴利氏酵母属(Debaryomyces)呈显著负相关(R=-0.997,P<0.05);乳酸菌(Lactobacillus)与假丝酵母(Candida)和喇叭菌属(Cantharellus)均呈显著正相关(P<0.05),相关系数均大于0.999;而乳酸菌(Lactobacillus)与Xerombrophila和毛霉属(Rhizomucor)均呈极显著正相关(P<0.001),相关系数亦均大于0.999。由此可见,渣豆酱中的乳酸菌与部分真菌之间存在着共生关系,这一现象可能与乳酸菌和真菌联合发酵豆渣能降解其中不溶性膳食纤维、蛋白质和脂肪等大分子的特性有关[19],而Bacillus与Debaryomyces之间则可能存在着一定的竞争关系。
3 结论
采用MiSeq高通量测序技术对鹤峰地区渣豆酱中细菌和真菌多样性进行解析,结果发现细菌主要为隶属于Firmicutes门的Bacillus属和Staphylococcus属,而真菌主要为隶属于Ascomycota门的Penicillium属、Galactomyces属和Zygosaccharomyces属。
[1]喻世哲,范熠,韩北忠,等.低盐腐乳生产过程及加工环境中的微生物分析[J].中国酿造,2017,36(12):18-22.
[2]张鹏飞,乌日娜,武俊瑞.传统发酵大豆制品挥发性成分和微生物多样性的研究进展[J].中国酿造,2018,37(12):1-6.
[3]李晓然,刘晓峰,张海燕,等.利用高通量测序分析云南两个地区豆豉微生物群落的多样性[J].现代食品科技,2014,30(12):61-67.
[4]XU D,WANG P,ZHANG X,et al.High-throughput sequencing approach to characterize dynamic changes of the fungal and bacterial communities during the production of sufu,a traditional Chinese fermented soybean food[J].Food Microbiol,2020,86(4):103340.
[5] CAPORASO J G,LAUBER C L,WALTERS W A,et al.Ultra-highthroughput microbial community analysis on the Illumina HiSeq and MiSeq platforms[J].ISME J,2012,6(8):1621-1624.
[6] BASSI D,PUGLISI E,COCCONCELLI P S.Understanding the bacterial communities of hard cheese with blowing defect[J].Food Microbiol,2015,52(12):106-118.
[7]SUN X,LYU G,LUAN Y,et al.Analyses of microbial community of naturally homemade soybean pastes in Liaoning province of China by Illumina Miseq sequencing[J].Food Res Int,2018,111(9):50-57.
[8]LIANG H,YIN L,ZHANG Y,et al.Dynamics and diversity of a microbial community during the fermentation of industrialized Qingcaipaocai,a traditional Chinese fermented vegetable food,as assessed by Illumina MiSeqsequencing,DGGE and qPCR assay[J].Ann Microbiol,2018,68(2):111-122.
[9]王玉荣,折米娜,刘康玲,等.内蒙古鄂尔多斯地区酸粥真菌多样性研究[J].中国酿造,2018,37(8):50-55.
[10]郭壮,葛东颖,尚雪娇,等.退化和正常窖泥微生物多样性的比较分析[J].食品工业科技,2018,39(22):93-98.
[11]CALLAHAN B J,MCMURDIE P J,HOLMES S P.Exact sequence variants should replace operational taxonomic units in marker-gene data analysis[J].ISME J,2017,11(12):2639.
[12]COLE J R,WANG Q,FISH J A,et al.Ribosomal Database Project:data and tools for high throughput rRNA analysis[J].Nucl Acid Res,2014,42(D1):D633-D642.
[13] ANUKAM K.Effects of ampicillin on the gut microbiome of an adult male as determined by 16S rRNA V4 metagenomics sequencing and Greengenes bioinformatics suite[J].J Adv Microbiol,2017,7(4):1-18.
[14]QUAST C,PRUESSE E,YILMAZ P,et al.The SILVA ribosomal RNA gene database project:improved data processing and web-based tools[J].Nucl Acid Res,2012,41(D1):D590-D596.
[15]PRICE M N,DEHAL P S,ARKIN A P.FastTree:computing large minimum evolution trees with profiles instead of a distance matrix[J].Mol Biol Evolut,2009,26(7):1641-1650.
[16] COMINACINI L,GARBIN U,DAVOLI A,et al.A simple test for predisposition to LDL oxidation based on the fluorescence development during copper-catalyzed oxidative modification[J].J Lipid Res,1991,32(2):349-358.
[17]ZHANG J,GUO Z,XUE Z,et al.A phylo-functional core of gut microbiota in healthy young Chinese cohorts across lifestyles,geography and ethnicities[J].ISME J,2015,9(9):1979-1990.
[18]张燕鹏,杨瑞金,王贺,等.传统豆渣菌的菌相分析及蛋白酶和纤维素酶主要产生菌株的鉴定[J].食品工业科技,2012,33(1):171-174.
[19]李伟伟,曲俊雅,周才琼.真菌及乳酸菌联合发酵对豆渣膳食纤维及理化特性的影响[J].食品与发酵工业,2018,44(11):159-166.
[20]ZHANG H H,WANG Y,ZHAO C,et al.Biodegradation of ochratoxin a by Alcaligenes faecalis isolated from soil[J].J Appl Microbiol,2017,123(3):661-668.
[21] HAN S M,SORABELLA R A,VASAN S,et al.Influence of Staphylococcus aureus on outcomes after valvular surgery for infective endocarditis[J].J Cardiothor Surg,2017,12(1):57.
[22]朱蕴兰,陈宏伟,陈安徽,等.蛹拟青霉固态发酵法制备大豆渣小肽及其理化特性[J].中国油脂,2019,44(12):86-91.
[23]董伟亮.米曲霉发酵豆渣制备大豆多肽和多糖[D].哈尔滨:哈尔滨工业大学,2018.
[24]谢欢,涂宗财,张露,等.黑曲霉发酵制备高可溶性膳食纤维豆渣工艺优化及其水合性质研究[J].中国粮油学报,2017,32(4):116-121.