醋酸菌属于醋杆菌科(Acetobacteriaceae),目前有11个属中的46种被认为是醋酸菌[1],其中绝大部分的醋酸菌存在于发酵食品中的醋、酸奶、泡菜和葡萄酒中[2-5]。醋酸菌是革兰氏阴性专性好氧菌,是葡萄酒生产过程中的有害微生物之一,它能够将乙醇氧化成乙醛和醋酸,使酒中的挥发酸含量增高,受醋酸菌污染的葡萄酒明显会有醋酸味产生,因此控制葡萄酒中的醋酸菌显得尤为重要[6-9]。目前国内醋酸菌的检测方法通常是在固体选择培养基上分离,然后鉴定其生理生化特性[10],还有一些方法是针对醋酸菌中的个别物种设计了实时荧光聚合酶链式反应(real-timepolymerase chain reaction,RT-PCR)的方法,GAMMON K S等[11]成功运用RT-PCR技术检测了电解质饮料中微量的葡萄糖杆菌,VALERA M J等[12]设计16S~23S r RNA间区序列的2个特异性探针,成功定量检测了醋酸菌Acetobacter malorum和Acetobacter cerevisiae的数量。但大多数的醋酸菌却不能在食品和环境样品中被检测到。
实时荧光PCR技术特异性强,灵敏度高,重复性好,已被广泛应用到检测过程中[13]。而在实验过程中发现,在葡萄酒中存在大量的单宁、多糖和色素,这些成分作PCR抑制因子在进行RT-PCR时可能会成为干扰实验准确性的存在[14]。PCR抑制剂通常通过与脱氧核糖核酸(deoxyribonucleic acid,DNA)直接相互作用或对热稳定DNA聚合酶的干扰而发挥作用,会影响PCR的扩增效率,所以在实际样品检测过程中,应使用生理盐水对葡萄酒进行洗脱,以排除干扰因素对基因组的提取[15]。
在本研究中,参考了KIM D H等[16]设计的引物序列,利用SYBR Green I染料法[17],建立了一种应用到葡萄酒中醋酸菌检验的实时荧光定量聚合酶链式反应(real-time fluorescent quantative polymerase chain reaction,RT-FQPCR)检测方法来快速检测葡萄酒中是否含有醋酸菌群,以此来对葡萄酒酿造过程中的醋酸菌群进行快速检测,并做出预警分析。
1 材料与方法
1.1 材料与试剂
1.1.1 菌株和检测样品
醋酸菌标准菌株7株,分别为巴氏醋杆菌(Acetobacter pasteurianus)、食蔗糖驹形氏杆菌(Acetobacter pasteurianus)、氧化葡萄糖杆菌(Gluconobacter oxydans)、日本葡糖杆菌(Gluconobacter japonicas)、蜡状葡糖杆菌(Gluconobacter cerinus)、醋化醋杆菌(Acetobacter aceti)及热带醋杆菌(Acetobacter tropicalis);其他非醋酸菌株7株,分别为金葡萄球菌(Staphylococcus aureus)、单核细胞增生李斯特氏菌(Listeria monocytogenes)、大肠杆菌(Escherichia coli)、志贺菌(Shigella sonnei)、鼠伤寒沙门菌(Salmonella typhimurium):中国工业微生物菌种保藏管理中心(China center of indus trial culture collection,CICC)和中国普通微生物菌种保藏管理中心(China general microbiological culture collection center,CGMCC);肠膜明串珠菌(Leuconostoc mesenteroides)和乳酸乳球菌(Lactococcus lactis)来自实验室分离菌种。
红葡萄酒(赛曦吉普斯兰黑比诺、麦格根·黑牌、2015拜伦湾西拉子),白葡萄酒(爱在深秋、骏马酒庄霞多丽、骏马酒庄霞多丽、卡聂高金伯爵、康斯汀特),桃红葡萄酒(拉亚莫斯卡托),冰酒(VQA维达尔):市场抽样。
1.1.2 试剂
细菌基因组脱氧核糖核酸(deoxyribonucleic acid,DNA)提取试剂盒:天根生化科技(北京)有限公司。
1.1.3 培养基
醋酸菌培养基:北京陆桥技术股份有限公司。
1.2 仪器与设备
X3R高速冷冻离心机:美国Thermo公司;PCR System 7900基因扩增仪、StepOnePlus实时定量荧光PCR仪、SYBR Green Master Mix:美国ABI公司。
1.3 方法
1.3.1 醋酸菌培养条件
7株醋酸菌在无菌环境下各挑取一菌环于醋酸菌培养基上划线,在需氧条件下30 ℃培养4~5 d。
1.3.2 基因组DNA的提取
7株醋酸菌经过恢复培养后,将长势处于指数生长期的醋酸菌,参考细菌基因组DNA提取试剂盒提取核酸[18]。其他非醋酸菌也参考细菌基因组DNA提取试剂盒提取核酸。
1.3.3 引物组
引物组:K-AAB_F:5'-AAGGGGGCTAGCGTTGCTCG-3';K-AAB_R:5'-ACCGCCTACACGCCCTTTACG-3'。
1.3.4 RT-FQPCR反应体系
RT-FQPCR反应体系(25 μL):SYBR Green Master Mix 12.5 μL,上游引物K-AAB_F 0.5 μL,下游引物K-AAB_R 0.5 μL,DNA模板2 μL,超纯水9.5 μL。
RT-FQPCR反应程序:95 ℃预变性30 s;95 ℃、5 s;62 ℃、34 s;循环45次,熔解曲线测定程序温度(0.5 ℃/s)速率从65 ℃升至95 ℃,同时收集荧光数据。
1.3.5 葡萄酒中DNA的提取
吸取1 mL葡萄酒样,8 000 r/min离心2 min,弃去上清液,加入200 μL生理盐水(0.85%)漩涡混匀,12 000 r/min离心2 min,所得沉淀按上述方法洗涤2次。
2 结果与分析
2.1 引物特异性实验结果
利用醋酸菌通用引物,以7种醋酸菌的DNA模板为阳性对照,剩余5种常见病原菌(金葡萄球菌(Staphylococcus aureus)、单核细胞增生李斯特氏菌(Listeria monocytogenes)、大肠杆菌(Escherichia coli)、志贺菌(Shigella sonnei)、鼠伤寒沙门氏菌(Salmonella typhimurium))和2种益生乳酸菌(肠膜明串珠菌(leuconostoc mesenteroides)和乳酸乳球菌(lactococcus lactis))的DNA为模板为阴性对照,以无菌去离子水为空白对照,按照1.3.4中的反应体系进行特异性检测,结果如图1所示。
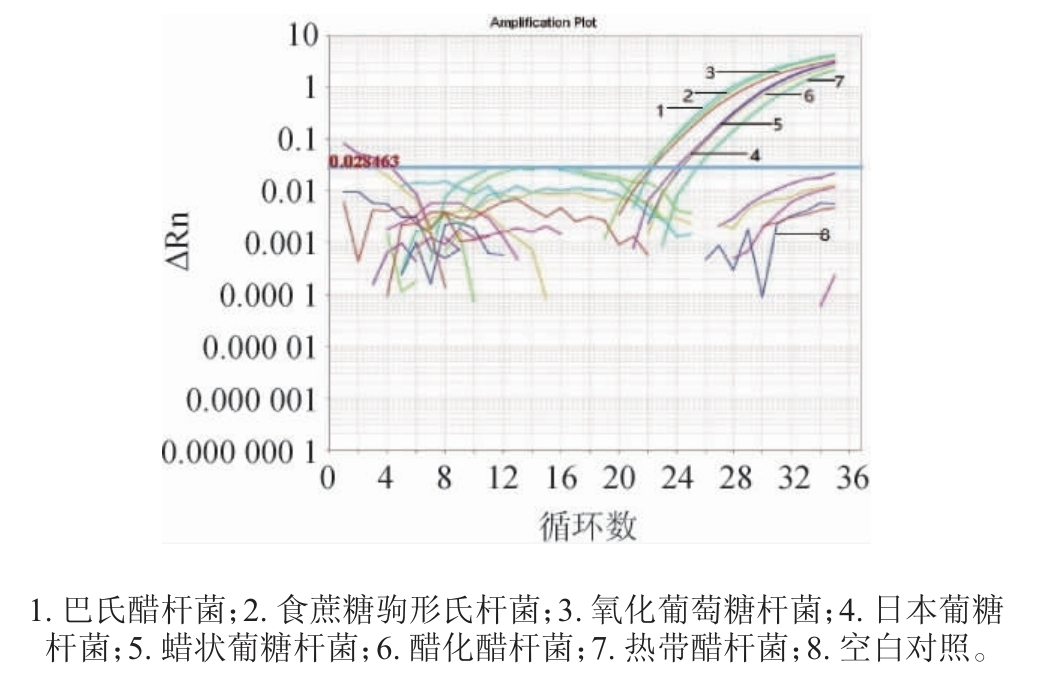
图1 实时荧光定量聚合酶链式反应特异性扩增曲线
Fig. 1 Amplification curve of real-time fluorescence quantitative polymerase chain reaction specific
由图1可知,7株醋酸菌出现阳性扩增曲线,其他非醋酸菌荧光信号均在阈值以内,空白对照也无扩增曲线,说明引物特异性良好。葡萄酒添加实验过程中的阴性对照和空白对照在扩增曲线中出现少量翘尾现象,分析得DNA模板提取过程中存在污染现象或成品酒中存在少量醋酸菌被检测出。由于出现的量很少,不干扰实验的进行。
2.2 不同菌株灵敏度实验结果
为了验证不同菌株的灵敏度,对7种醋酸菌用生理盐水进行梯度稀释,菌液浓度稀释至10-7,按照1.3.4的反应体系进行RT-FQPCR扩增,同时进行平板计数。活菌计数(CFU/mL)=同一稀释度三次重复的平均菌落数×稀释倍数×5,计算得到最低检出限,结果见表1。
表1 菌落计数结果
Table 1 Results of colony count
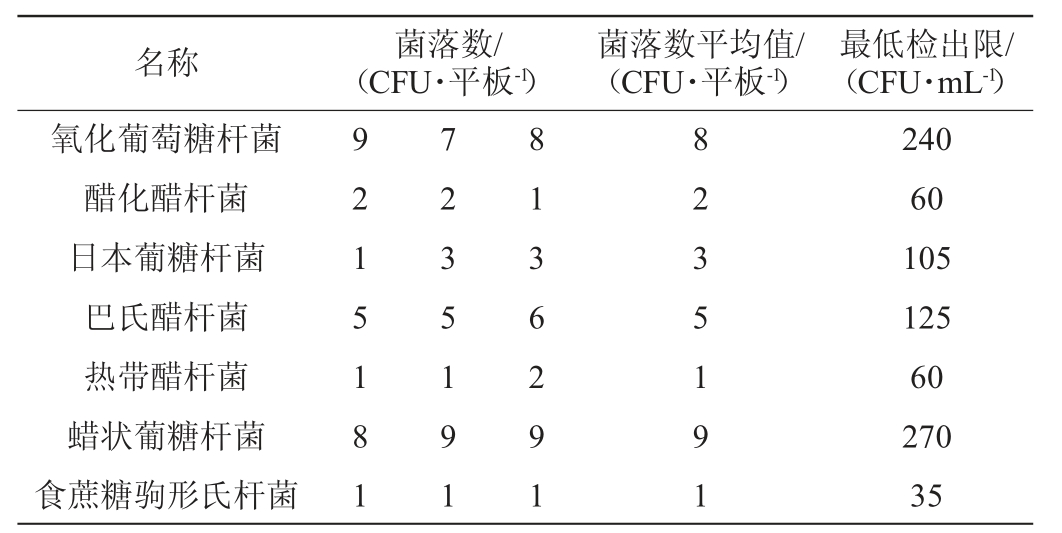
名称 菌落数/(CFU·平板-1)菌落数平均值/(CFU·平板-1)最低检出限/(CFU·mL-1)氧化葡萄糖杆菌醋化醋杆菌日本葡糖杆菌巴氏醋杆菌热带醋杆菌蜡状葡糖杆菌食蔗糖驹形氏杆菌9 2 1 5 1 8 1 7 2 3 5 1 9 1 8 1 3 6 2 9 1 8 2 3 5 1 9 1 240 60 105 125 60 270 35
由表1可知,7株醋酸菌在不同菌液稀释倍数下的菌落计数结果值各不相同,选取每种醋酸菌能被活菌计数的最低稀释度得到结果:氧化葡萄糖杆菌最低检出限240 CFU/mL、醋化醋杆菌最低检出限60 CFU/mL、日本葡糖杆菌最低检出限105 CFU/mL、巴氏醋杆菌最低检出限125 CFU/mL热带醋杆菌最低检出限60 CFU/mL、蜡状葡糖杆菌最低检出限270 CFU/mL和食蔗糖驹形氏杆菌最低检出限35 CFU/mL,综合检测限为124 CFU/mL,符合活菌计数标准范围内。
2.3 标准曲线的建立[19-20]
根据扩增产物的荧光信号达到设定的荧光阈值时所
对应的扩增循环数(cycle threshold,Ct)值(y)与菌液稀释
倍数的对数值(x)制作醋酸菌标准曲线,结果如图2所示。标准曲线回归方程、相关系数见表2。
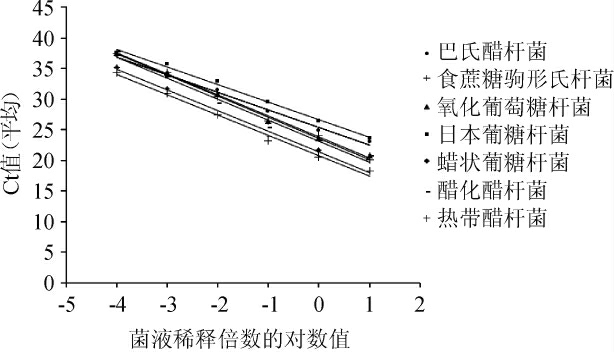
图2 7株醋酸菌标准曲线
Fig. 2 Standard curves of 7 acetic acid bacteria strains
表2 7株醋酸菌的标准曲线回归方程及相关系数
Table 2 Regression equation and correlation coefficient of standard curves of 7 acetic acid bacteria strains
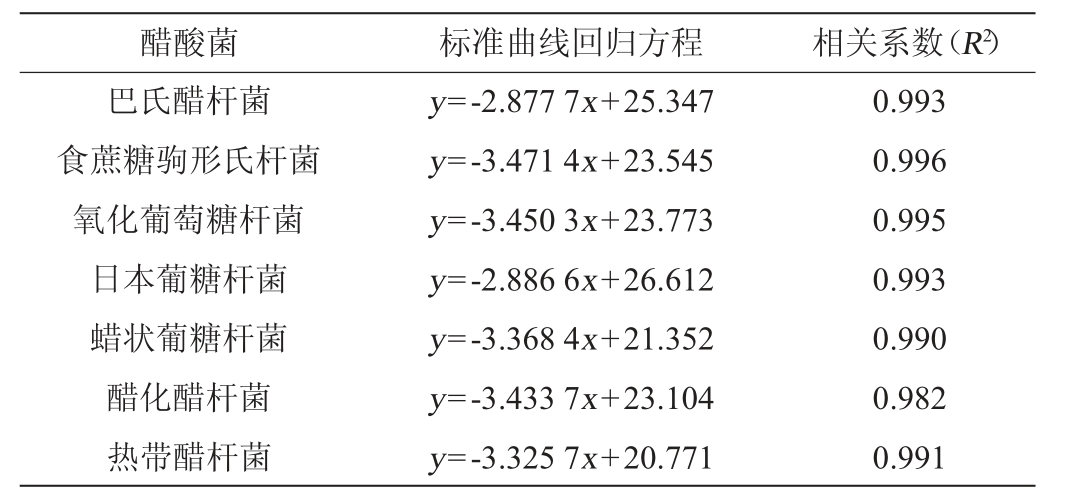
醋酸菌 标准曲线回归方程 相关系数(R2)巴氏醋杆菌食蔗糖驹形氏杆菌氧化葡萄糖杆菌日本葡糖杆菌蜡状葡糖杆菌醋化醋杆菌热带醋杆菌y=-2.877 7x+25.347 y=-3.471 4x+23.545 y=-3.450 3x+23.773 y=-2.886 6x+26.612 y=-3.368 4x+21.352 y=-3.433 7x+23.104 y=-3.325 7x+20.771 0.993 0.996 0.995 0.993 0.990 0.982 0.991
由图2及表2可知,检测到7株醋酸菌标准曲线回归方程线性关系良好,相关系数R2均可达到0.9以上,检测结果可靠,可以用作定量分析。
2.4 醋酸菌株添加葡萄酒中检测结果
以不加醋酸菌的葡萄酒为阴性对照,无菌去离子水为空白对照,参考表1中每株醋酸菌在不同菌浓下的检测限,混匀吸取5 mL醋酸菌菌液加入45 mL葡萄酒中,按照1.3.4的反应体系进行RT-FQPCR扩增,结果见表3。
表3 醋酸菌株添加葡萄酒中检测结果
Table 3 Determination results of acetic acid bacteria strains addition in wines
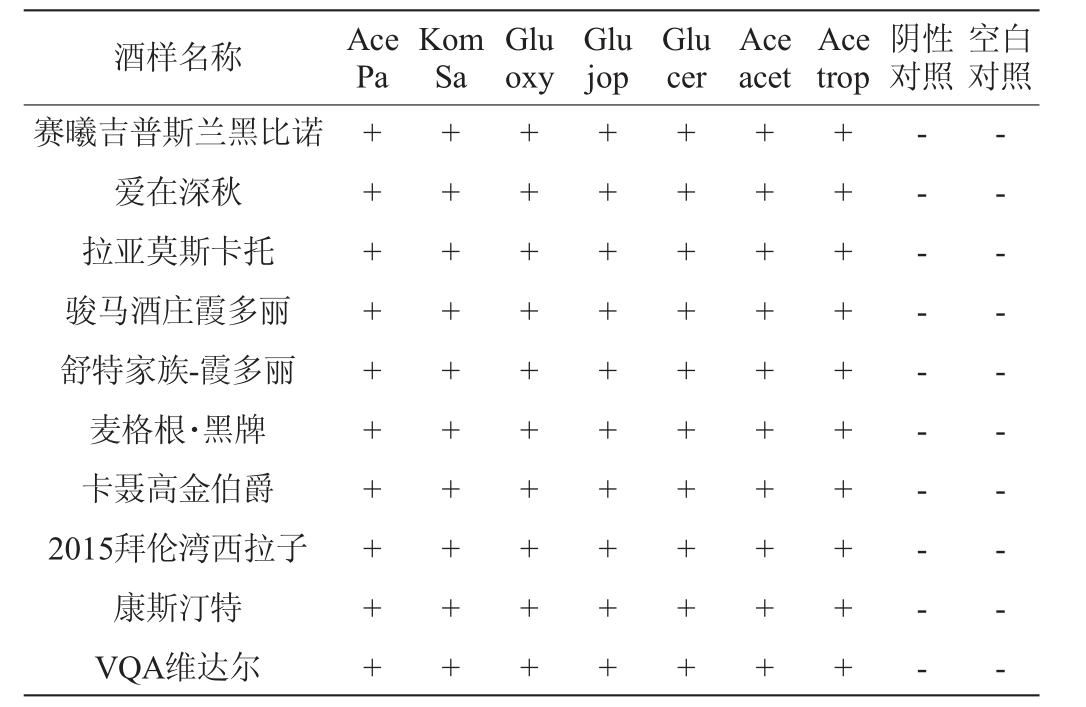
注:Ace Pa为巴氏醋杆菌;Kom Sa为食蔗糖驹形氏杆菌;Glu oxy为氧化葡萄糖杆菌;Glu jop为日本葡糖杆菌;Glu cer为蜡状葡糖杆菌;Ace acet为醋化醋杆菌;Ace trop为热带醋杆菌;“+”为检出;“-”为未检出。
酒样名称 Ace Pa Kom Sa Glu oxy Glu jop Glu cer Ace acet Ace trop阴性对照空白对照赛曦吉普斯兰黑比诺爱在深秋拉亚莫斯卡托骏马酒庄霞多丽舒特家族-霞多丽麦格根·黑牌卡聂高金伯爵2015拜伦湾西拉子康斯汀特VQA维达尔++++++++++++++++++++++++++++++++++++++++++++++++++++++++++++++++++++++-- - - - - - - - --- - - - - - - - -
由表3可知,7株醋酸菌添加到成品葡萄酒中均可被检测出,而在不添加醋酸菌的葡萄酒和空白对照中均未检出,表明此方法可用于实际生产过程中。
3 结论
本研究依据醋酸菌通用引物进行反应条件的优化并通过7株醋酸菌,进行实时荧光定量PCR扩增。结果表明此方法综合灵敏度可达124 CFU/mL,该方法对醋酸菌具有良好的特异性,耗时短,通用性强在实际样品中的实验结果也真实可靠,能够快速检测葡萄酒酿造过程中的醋酸菌,可应用于实验室检测或者实际生产过程,具有实际意义。
[1]CLEENWERCK I,DE VOS P.Polyphasic taxonomy of acetic acid bacteria:an overview of the currently applied methodology[J].Int J Food Microbiol,2008,125(1):2-14.
[2]YAMADA Y,YUKPHAN P.Genera and species in acetic acid bacteria[J].Int J Food Microbiol,2008,125(1):15-24.
[3]马雷,陈晓艺,孙玉梅.应用于葡萄酒酿造中的抑菌剂[J].中国酿造,2017,36(1):29-34.
[4]李啸.我国传统泡菜自然发酵与单菌发酵微生物及代谢特性研究[D].南昌:南昌大学,2014.
[5]李月华.LAMP 与SYBR GREEN I 实时荧光定量PCR 方法检测酸奶中葡糖杆菌的研究[D].保定:河北农业大学,2015.
[6]王树庆,姜薇薇,李保国,等.葡萄酒酿造过程中的有害微生物[J].酿酒,2019(3):19-22.
[7] BARTOWSKY E J, HENSCHKE P A. Acetic acid bacteria spoilage of bottled red wine-a review[J].Int J Food Microbiol,2008,125(1):60-70.
[8]OUBA L I I,KANDO C,PARKOUDA C,et al.The microbiology of Bandji,palm wine of Borassus akeassii from Burkina Faso:identification and genotypic diversity of yeasts,lactic acid and acetic acid bacteria[J].J Appl Microbiol,2012,113(6):1428-1441.
[9]陈苏娟.啤酒酿造过程中醋酸菌的检测与鉴定[J].啤酒科技,2006(4):59-61.
[10]ZHOU W,ZHANG Y,WANG S,et al.LAMP,PCR,and real-time PCR detection of Acetobacter aceti in yogurt[J]. Food Sci Biotechnol, 2017,26(1):153-158.
[11] GAMMON K S, LIVENS S, PAWLOWSKY K, et al. Development of real-time PCR methods for the rapid detection of low concentrations of Gluconobacter and Gluconacetobacter species in an electrolyte replacement drink[J].Lett Appl Microbiol,2007,44(3):262-267.
[12] VALERA M J, TORIJA M J, MAS A, et al. Acetobacter malorum and Acetobacter cerevisiae identification and quantification by real-time PCR with TaqMan-MGB probes[J].Food Microbiol,2013,36(1):30-39.
[13]魏秀萍,李金山,吕国平.菌液SYBR 实时荧光PCR 快速检测金黄色葡萄球菌[J].医学动物防制,2014(5):579-582.
[14]TESSONNIÈRE H,VIDAL S,BARNAVON L,et al.Design and performance testing of a real-time PCR assay for sensitive and reliable direct quantification of Brettanomyces in wine[J].Int J Food Microbiol,2009,129(3):237-243.
[15]BESSETTI J.An introduction to PCR inhibitors[J].J Microbiol Methods,2007,28:159-67.
[16] KIM D H, LIM H W, KIM S H, et al. Development of a real-time PCR assay for rapid screening of acetic acid bacteria as a group in food products[J].Food Control,2019,100:78-82.
[17]钱云开,王海洋,崔宗岩,等.实时荧光PCR 法快速检测肉类中貉源性成分[J].中国食品卫生杂志,2016,28(5):602-605.
[18]侯晓阳.多粘类芽孢杆菌SC2 形态分化的分子机制研究[D].泰安:山东农业大学,2015.
[19]刘夏,王传现,吴美琪,等.葡萄酒中酒香酵母实时荧光PCR 检测方法的研究[J].中国卫生检验杂志,2016,26(11):1591-1593.
[20]王青龙,蔡雪凤,周艳霞,等.实时荧光PCR 法检测食品中亚利桑那沙门氏菌[J].中国酿造,2018,37(6):40.