石花酒是用高粱、糯米为原料,以豌豆、大麦制曲,进行地缸发酵、分段取酒,最后勾兑而成的清香型白酒,是湖北省名酒之一。大曲中丰富的微生物菌落是酒体风味品质形成的关键因素。近年来,研究人员对全国各类大曲中微生物多样性展开了广泛的研究。刘桂君等[1]从牛栏山酒厂清香型大曲和酒醅中分离出23株芽孢杆菌,通过生理生化试验鉴定12株为地衣芽孢杆菌(Bacillus licheniformis);李增胜[2]研究发现,汾酒酒醅中的酵母菌以酵母菌属(Saccharomyces)为主,其产酒精能力最强,其次是具有一定产香和产酒精能力的汉逊酵母(Hansenula)和假丝酵母(Candida)及产酒精不强的拟内孢霉(Endomycopsis),还有极少量的毕赤酵母(Pichia)等产膜酵母;兰玉倩等[3]利用聚合酶链式反应-变性梯度凝胶电泳(polymerase chain reaction-denaturing gradient gel electrophoresis,PCR-DGGE)指纹技术分析了清香型大曲在生产过程中酵母类微生物的演替规律。共检测到伊萨酵母属(Issatchenkia)、毕氏酵母(Pichia)、酵母菌属(Saccharomyces)、复膜孢酵母属(Saccharomycopsis)、地霉属(Geotrichum)和根霉属(Rhizopus)6个真菌微生物属,其中东方伊萨酵母(Issatchenkia orientalis)在大曲整个生产过程中占绝对优势;王彩虹[4]采用克隆文库技术与PCR-限制性片段长度多态性(restricted fragment length polymorphisms,RFLP)技术相结合的手段研究发现,3种汾酒清香型大曲中细菌均分布于芽孢杆菌纲(Bacillibacteria)、放线菌纲(Actinobacteria)及变形菌纲(Alphaproteobacteria),且3种大曲的优势细菌群均含有乳杆菌属。以上研究成果为酒曲中优势菌种的开发及进一步深入研究的开展奠定了良好的基础。石花酒大曲(以下简称石花大曲)中微生物丰富,群落结构复杂,种类繁多,然而目前未有关于石花大曲微生物多样性研究的报道。
下一代测序(next generation sequencing,NGS)一方面可快速准确地提供高通量测序信息,另一方面可为分析提供定位结果[5],其中Illumina MiSeq高通量测序技术已被广泛应用于各项研究中。HU X L等[6]使用Illumina MiSeq对浓香型白酒窖泥微生物信息进行测序分析,结果表明,窖泥微生物群落与窖泥品质和环境因素有关。YANG H Y等[7]根据Illumina MiSeq测序分析发现,东北地区酸菜发酵的过程中出现了硬杆菌、变形杆菌、拟杆菌、放线菌、蓝藻、梭杆菌和疣菌,以及其他未分类的细菌;沈馨等[8]利用Illumina MiSeq高通量测序技术发现,孝感地区3个凤窝酒曲共有大量的核心细菌菌群;POLKA J等[9]利用Illumina MiSeq测序在意大利蒜味咸肠中鉴定出32种不同的葡萄球菌和33种乳酸杆菌,其中23个存在于至少10%的调查样品中。综上,Illumina MiSeq高通量测序技术能够更准确全面地获取样品中微生物群落结构信息。
本研究以采集自湖北襄阳市石花酒厂的石花大曲为研究对象,提取样品宏基因组,采用Illumina MiSeq高通量测序技术对其微生物多样性进行解析,以期为石花大曲中微生物资源的开发奠定一定的理论基础。
1 材料与方法
1.1 材料与试剂
1.1.1 样品
石花大曲:襄阳石花酒厂;采集自湖北襄阳市石花酒厂,4个样品分别命名为SHQ01、SHQ02、SHQ03、SHQ04。
1.1.2 试剂
脱氧核糖核苷三磷酸(deoxy-ribonucleosidetriphosphate,dNTPs)Mix、5×TransStartTM、FastPfu Buffer和FastPfu Fly脱氧核糖核酸(deoxyribonucleicacid,DNA)Polymerase(5 U/μL):北京全式金生物技术有限公司;10×PCR Buffer、Solution I、pMD18-T vector、蛋白酶K(20 U/μg)、溶菌酶(400 U/μg):宝生物工程(大连)有限公司;QIAGEN DNeasy mericon Food Kit试剂盒:德国QIAGEN公司;乙二胺四乙酸二钠、十二烷基硫酸钠、氯仿、十六烷基三甲基溴化铵、异丙醇、乙醇、三轻甲基氨基甲烷、酚、异戊醇、氯化钠和乙酸钠等(均为分析纯):国药集团化学试剂有限公司。
1.2 仪器与设备
DYY-12电泳仪:北京六一仪器厂;ND-2000C微量紫外分光光度计:美国NanoDrop公司;UVPCDS8000凝胶成像分析系统:美国BIO-RAD公司;Vetiri梯度基因扩增仪:美国AB公司;R920机架式服务器:美国DELL公司;Miseq高通量测序平台:美国Illumina公司;2100芯片生物分析仪:美国Agilent公司;5810R台式高速冷冻离心机:德国Eppendorf公司。
1.3 实验方法
1.3.1 样品处理
将采集到的石花大曲样品置于有干冰的采样箱中于24 h内运回实验室,使用洁净无菌的研钵研成粉末后使用。
1.3.2 石花大曲中微生物宏基因组DNA提取
使用QIAGEN DNeasy mericon Food Kit试剂盒提取石花大曲样品中的微生物总DNA,并用微量紫外分光光度计检测提取的DNA纯度及浓度,以此获得高品质和高浓度的微生物宏基因组DNA。
1.3.3 细菌和真菌PCR扩增
按文献[10]所述方法对样品中细菌16S rRNA的V3-V4区进行PCR扩增。按文献[11]所述方法对样品中真菌18SrRNA的V4-V5区进行PCR扩增。用1%的琼脂糖凝胶对PCR扩增产物进行检测[12],利用AxyPrepDNA凝胶回收试剂盒将其回收并送样至上海美吉生物医药科技有限公司进行Illumian MiSeq高通量测序,测序平台为Illumian MiSeq PE300。
1.3.4 序列的拼接及质控
按照文献[13]所述的方法进行序列拼接,按照序列间的重叠关系对Illumian MiSeq PE300平台获取的数据进行拼接,在拼接过程中要求重叠区的碱基数≥10 bp或最大错配比率≤0.2,同时要求引物碱基错配数≤2 bp,且切除引物后序列的长度>50 bp,若序列不符合上述条件则予以剔除。
1.3.5 生物信息统计与分析
使用QIIME(quantitative insights into microbial ecology)平台[14]进行物种鉴定和相对含量的分析;使用PyNAST工具[15]校准并排齐序列;使用UCLUST算法[16]分别以100%和97%的相似度进行序列划分并建立操作分类单元(opera tionaltaxonomicunits,OTU),从每个OTU中选出代表性序列,分别整合核糖体数据库(ribosomal database project,RDP)[17]、Greengenes[18]和SILVA数据库[19],对OTU进行序列同源性比对和种属分类学鉴定,从而分别明确石花大曲中细菌和真菌的相关信息。利用观察物种(Observed Species)指数、超1(Chao 1)指数、香农(Shannon)指数和辛普森(Simpson)指数等评价微生物菌群的丰度和多样性[20]。
1.3.6 图像绘制与数据处理
使用Matlab2016b软件进行热图的绘制,其他图由Origin 8.5软件绘制。相关性由SAS V8软件分析。
2 结果与分析
2.1 石花大曲样品细菌序列丰富度分析
使用Illumian MiSeq高通量测序技术对4个石花大曲样品的细菌多样性进行分析,其16S rRNA测序结果及多样性指数见表1。其中Observed Species指数和Chao 1指数是估算样品中含有物种数目及所含OTU数目的指数,两者均能反映样品的物种丰度,Shannon指数和Simpson指数用来表现样品中物种的丰富度和均匀度,两者均能反映样品的物种多样性。
表1 石花酒大曲样品中细菌的测序结果及多样性指数
Table 1 Sequencing results and diversity index of bacteria in Shihua Baijiu Daqu samples
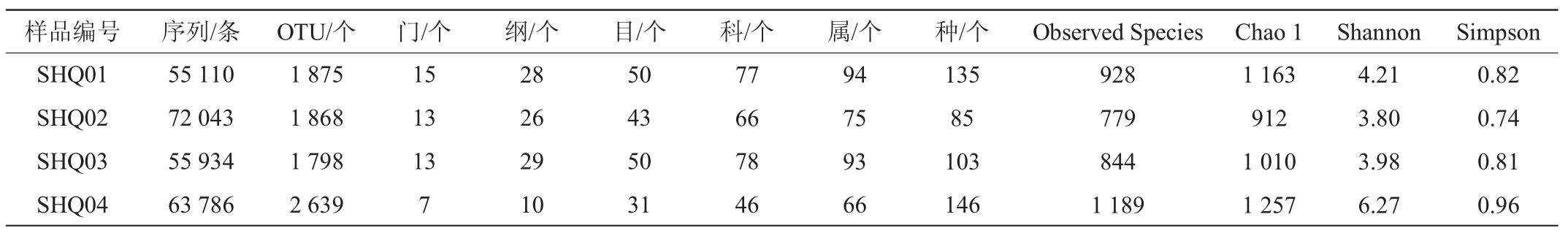
由表1可知,4个石花大曲样品中共产生246 873条细菌序列,平均每个样品产生61 718条序列。根据97%相似性归类共得到8 180个OTU,平均每个样品2 045个OTU。当测序量为54 010时,样品SHQ04的Observed Species指数、Chao 1指数、Shannon指数和Simpson指数均为各样品中的最大值,分别为1 189、1 257、6.27和0.96,表明样品SHQ04中细菌群落丰富度和多样性均为最高;样品SHQ02的Observed Species指数、Chao 1指数、Shannon指数和Simpson指数均为最小,分别为779、912、3.80和0.74,表明样品SHQ02中细菌群落丰富度和多样性均为最低。
2.2 基于门和属水平的细菌多样性分析
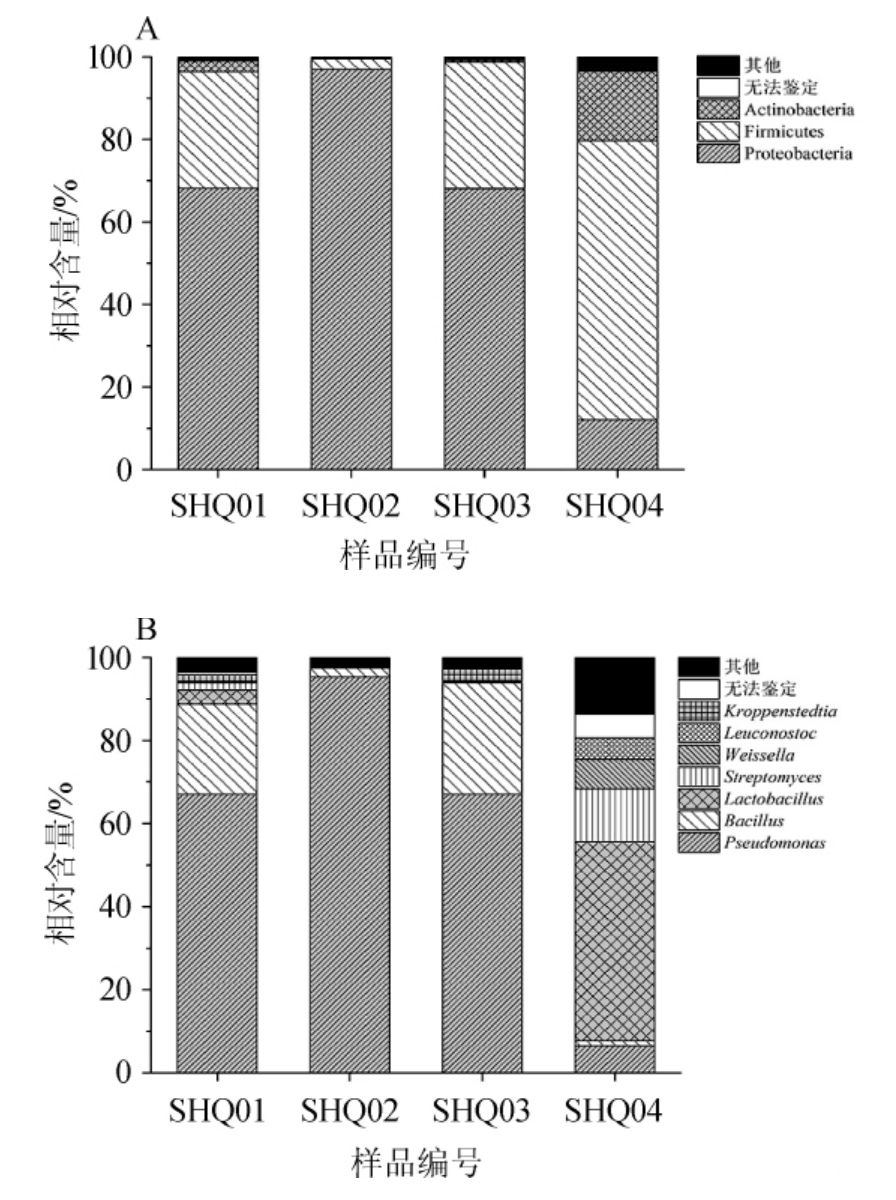
图1 基于细菌门(A)和属(B)水平石花酒大曲样品中细菌多样性分析
Fig.1 Analysis of bacteria diversity in Shihua Baijiu Daqu samples based on the phylum (A) and genus (B) level
从每个OTU中挑选一条代表性序列进行比对,然后统计其界、门、纲、目、科和属的种类和数量,其中细菌门及属的分析结果见图1,将相对含量<0.1%的细菌门或相对含量<0.1%的细菌属归为其他,相对含量>0.1%的细菌门或相对含量>1%的细菌属定义为优势细菌门或属。
由图1A可知,4个石花大曲样品中共检测出17个细菌门,其中优势细菌门为变形杆菌门(Proteobacteria)、厚壁菌门(Firmicutes)和放线菌门(Actinobacteria),平均相对含量分别为61.33%、32.25%、5.04%,且平均仅有0.1%的菌株不能鉴定到门水平。样品中变形杆菌门和厚壁菌门的累积平均相对含量高达93.58%,而放线菌门平均相对含量<5.04%。样品SHQ04中变形杆菌门的相对含量明显低于其他样品,而样品SHQ02中厚壁菌门含量亦明显低于其他样品,说明各样品中细菌门的种类和含量存在差异。
由图1B可知,4个石花大曲样品中共检测出168个细菌属,其中优势细菌属为假单胞菌属(Pseudomonas)、芽孢杆菌属(Bacillus)、乳杆菌属(Lactobacillus)、链霉菌属(Streptomyces)、魏斯氏菌属(Weissella)、明串珠菌属(Leuconostoc)和Kroppenstedtia,平均相对含量分别为58.97%、12.98%、12.86%、3.71%、1.83%、1.27%和1.25%,所有合格序列仅有1.70%不能鉴定到属水平。值得一提的是,假单胞菌属在样品SHQ04中的相对含量仅有6.51%,而在样品SHQ02中的相对含量高达95.33%;乳杆菌属在样品SHQ04中的相对含量为47.88%,而在样品SHQ01、SHQ02和SHQ03中的相对含量分别为3.37%,0.07%和0.13%;明串珠菌属仅在样品SHQ1和SHQ4中含有。由此可见,虽然石花大曲样品中含有大量细菌菌群,但这些优势细菌在各样品中并非均匀分布。陈申习等[21]采用传统分离方法和现代分子技术对清香型小曲白酒机械化生产中微生物动态变化进行了研究,发现酒醅微生物的细菌主要为乳杆菌属和芽孢杆菌属。也有研究人员采用高通量测序技术对清香型白酒发酵过程中的细菌多样性进行了解析。张双燕等[22]利用高通量测序方法对北京某清香型酒厂大曲细菌进行分析,发现厚壁菌门为优势菌门;乳杆菌属、片球菌属(Pediococcus)、乳球菌属(Factococcus)、明串珠菌属为优势菌属;王雪山等[23]利用高通量测序技术分析不同位置酒醅中微生物种群结构及演替规律,发现清香型白酒发酵过程中优势细菌种群包括乳杆菌属、魏斯氏菌属、Kroppenstedtia、假单胞菌属、明串珠菌属、芽孢杆菌属和片球菌属。上述结论中均有优势属乳杆菌属,其他属在结论中也有重复出现,结论中略微不同的原因可能是与使用原料、制作环境或制作工艺有关。
2.3 基于OTU水平的细菌多样性分析
4个石花大曲样品中的OTU数量及种类并非完全相同,其中平均相对含量>1%的OTU有9个,其在各石花大曲样品中的相对含量见图2。
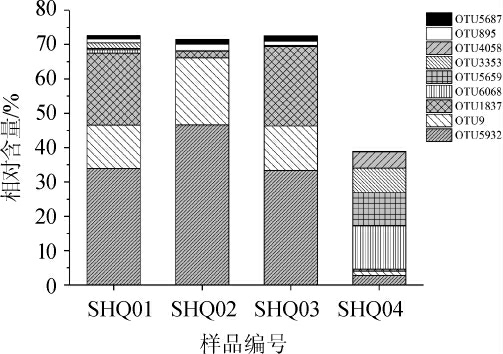
图2 石花酒大曲样品中平均相对含量>1%细菌OTU统计分析
Fig.2 Statistical analysis of OTU of bacteria with mean relative content more than 1% in Shihua Baijiu Daqu samples
由图2可知,平均相对含量>1%的OTU(平均相对含量)分别为OTU5 932(29.43%)、OTU9(11.79%)、OTU1 837(10.63%)、OTU6 068(3.49%)、OTU5 659(2.60%)、OTU3 353(2.29%)、OTU4 058(1.22%)、OTU895(1.10%)和OTU5 687(1.05%),其中OTU5 932、OTU9、OTU895和OTU5 687隶属于变形杆菌门,OTU1837、OTU6068和OTU5659隶属于厚壁菌门,OTU3353和OTU4058隶属于放线菌门,且除OTU4058,其他OTU在各样品中均存在。由图2亦可知,样品SHQ2中OTU5 932的相对含量已达65.15%,显著高于其他样品,而其OTU2 793的相对含量仅为2.09%,样品SHQ4中OTU6 068的相对含量已达32.25%,显著高于其他样品,且OTU4 058仅在样品SHQ4中出现。在每个样品中均出现的细菌门为样品的核心细菌门。由此可见,石花大曲样品中的核心细菌门为变形杆菌门、厚壁菌门和放线菌门,且各样品相对含量存在差异。
2.4 石花大曲样品真菌序列丰富度分析
使用Illumian MiSeq高通量测序技术对4个石花大曲样品的真菌多样性进行分析,其18S rRNA测序结果及多样性指数见表2。
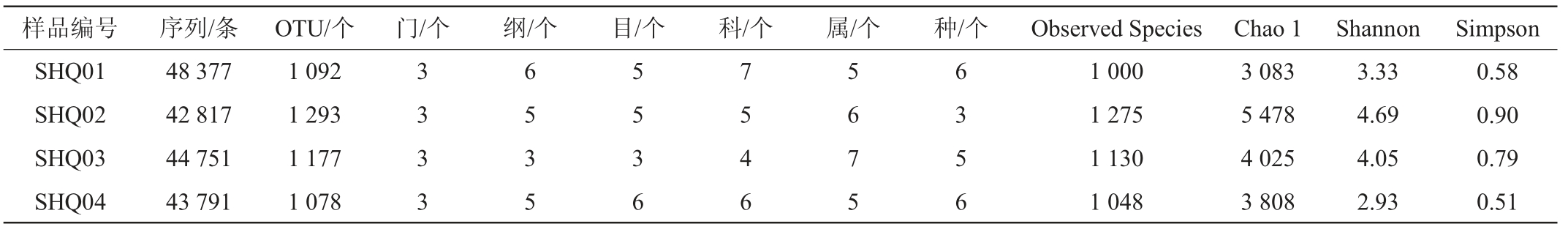
由表2可知,4个石花大曲样品中共产生179 736条真菌序列,平均每个样品产生44 934条序列。根据97%相似性归类共得到4 640个OTU,平均每个样品1 160个OTU。当测序量为42010时,样品SHQ02的ObservedSpecies指数、Chao 1指数、Shannon指数和Simpson指数均为各样品中的最大值,分别为1 275、5 478、4.69和0.90,表明样品SHQ02中真菌群落丰富度和多样性均最大;样品SHQ01的Observed Species指数和Chao 1指数最小,分别为1 000和3 083,样品SHQ04的Shannon指数和Simpson指数最小,分别为2.93和0.51。表明样品SHQ01中真菌群落丰富度最低,SHQ04多样性最低。
表2 石花酒大曲样品中真菌的测序结果及多样性指数
Table 2 Sequencing results and diversity index of fungi in Shihua Baijiu Daqu samples
2.5 基于门和属水平的真菌多样性分析
从每个OTU中挑选一条代表性序列进行比对,然后统计其界、门、纲、目、科和属的种类和数量,其中真菌门及属水平的分析结果见图3,将相对含量<0.1%的真菌门或相对含量>1%的真菌属归为其他,相对含量>0.1%的真菌门或相对含量>1%的真菌属定义为优势真菌门或属。
由图3A可知,4个石花大曲样品中主共检测出3个真菌门,其中优势真菌门为子囊菌门(Ascomycota)、其他的真菌门有担子菌门(Basidiomycota)和Mucoromycota,平均相对含量分别为99.70%、0.17%、0.12%,且平均仅有0.01%不能鉴定到门水平。样品中子囊菌门的平均相对含量高达99.70%,而担子菌门和Mucoromycota的累计平均相对含量<0.29%。样品SHQ04中担子菌门和Mucoromycota的相对含量明显低于其他样品,而样品SHQ02中子囊菌门含量低于其他样品,说明各样品中真菌门的种类和含量存在差异。
由图3B可知,4个样品中共检测出25个真菌属,其中优势真菌属为复膜孢酵母属(Saccharomycopsis)、红曲菌属(Monascus)、热子囊菌属(Thermoascus)、青霉菌属(Penicillium)、曲霉属(Aspergillus)和Leiothecium,平均相对含量分别为57.08%、17.58%、5.38%、4.76%、1.78%和1.22%,所有合格序列有8.47%不能鉴定到属水平。值得一提的是,复膜孢酵母属在样品SHQ02中的相对含量仅有21.25%,而在样品SHQ04中的相对含量高达88.39%;青霉菌属在样品SHQ02中的相对含量为18.58%,而在样品SHQ01、SHQ03和SHQ04中的相对含量分别为0.13%、0.34%和0.01%;红曲菌属在样品SHQ4中仅含有0.04%。由此可见,虽然石花大曲样品中含有大量真菌菌群,但这些优势真菌在各样品中并非均匀分布。王雪山等[23]通过高通量测序技术还发现,在清香型白酒发酵过程中,真菌种群中子囊菌门在所有酒醅中占主导地位;优势真菌属包括毕赤酵母属、假丝酵母属、曲霉属、复膜孢酵母属和Kazachstania;雷振河[24]应用高通量测序技术对清香型白酒酿造用大曲和酒醅中微生物构成进行了分析,发现大曲中优势真核微生物主要包括曲霉菌属、热子囊菌属、根霉菌属、嗜热真菌属、假丝酵母属。上述结论中的真菌优势属间均有相似之处,但结论中却没有出现一个共有的优势真菌属。出现这一结果这可能是由于原料、地域、环境、发酵工艺等因素不同造成的。
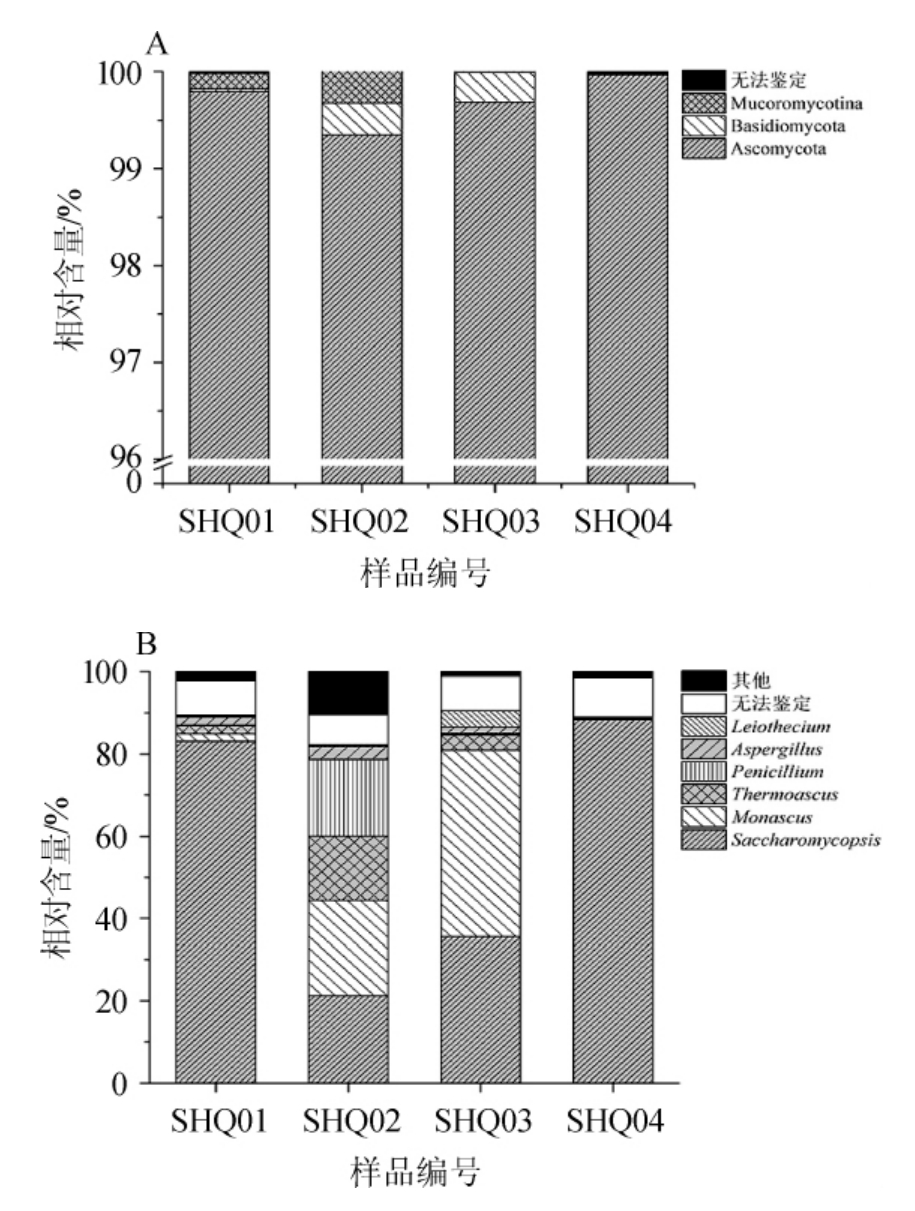
图3 基于真菌门(A)和属(B)水平石花酒大曲样品中细菌多样性分析
Fig.3 Analysis of fungal diversity in Shihua Baijiu Daqu samples based on the phylum (A) and genus (B) level
2.6 基于OTU水平的真菌多样性分析
4个石花大曲样品中的OTU数量及种类并非完全相同,其中平均相对含量>1%的OTU有7个,其在各石花大曲样品中的相对含量见图4。
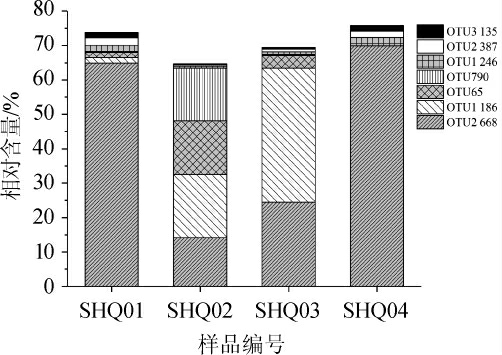
图4 石花酒大曲样品中平均相对含量>1%真菌OTU统计分析
Fig.4 Statistical analysis of OTU of fungi with mean relative abundance more than 1%in Shihua Baijiu Daqu samples
由图4可知,平均相对含量>1%的OTU(平均相对含量)分别为OTU2 668(43.97%)、OTU1 186(14.50%)、OTU65(5.04%)、OTU790(3.74%)、OTU1 246(1.42%)、OTU2 387(1.34%)和OTU3 135(1.00%),OTU均属于子囊菌门,且这些OTU在各样品中均存在。由图4亦可知,各样品中所有相对含量>1%的OTU的平均累计相对含量已达71.03%。在每个样品中均出现的真菌门为样品的核心真菌门。由此可见,石花大曲样品中的核心真菌门为子囊菌门,且各样品相对含量存在差异。
2.7 石花大曲真菌与细菌优势属的相关性分析
采用SAS V8软件对4个石花大曲样品中的真菌和细菌的优势属进行初步的相关性分析,结果见图5。
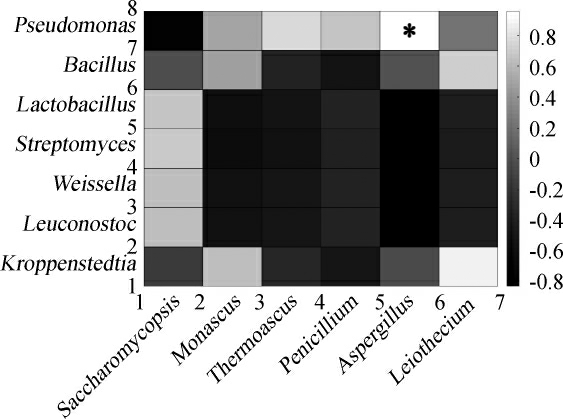
图5 石花酒大曲优势真菌属与细菌属相关性分析热图
Fig.5 Heat map of relativity analysis of dominant fungi and bacteria genera in Shihua Baijiu Daqu
“*”表示相关性显著(P<0.05)。
由图5可知,假单胞菌属和曲霉属呈现显著正相关(P<0.05),R=0.96,而其他真菌与细菌优势属之间相关性均不显著(P>0.05),且部分呈现负相关。说明假单胞菌属细菌和曲霉属真菌在石花大曲中有着相互促进生长的作用,而其他属的细菌与真菌相互促进作用不明显,有的甚至相互抑制生长。
3 结论
本研究使用IlluminaMiSeq高通量测序技术,以16SrRNA的V3-V4区和18S rRNA的V4-V5区为测序靶点,对石花大曲中的细菌和真菌微生物多样性进行分析。结果发现,石花大曲中的细菌主要由隶属于变形杆菌门(Proteobacteria)、厚壁菌门(Firmicutes)和放线菌门(Actinobacteria)的假单胞菌属(Pseudomonas)、芽孢杆菌属(Bacillus)、乳杆菌属(Lactobacillus)、链霉菌属(Streptomyces)、魏斯氏菌属(Weissella)、明串珠菌属(Leuconostoc)和Kroppenstedtia的7个属组成;石花大曲中的真菌主要由隶属于子囊菌门(Ascomycota)、担子菌门(Basidiomycota)和Mucoromycota的复膜孢酵母属(Saccharomycopsis)、红曲菌属(Monascus)、热子囊菌属(Thermoascus)、青霉菌属(Penicillium)、曲霉属(Aspergillus)和Leiothecium 6个属组成,且石花酒曲样品共有大量的核心细菌和真菌菌群。此外,本研究还发现细菌中的假单胞菌属与真菌的曲霉属有显著的正相关性(P<0.05)。该研究为石花大曲中微生物资源的开发奠定一定的理论基础。
[1]刘桂君,朱婷婷,刘红霞,等.清香大曲及酒醅中地衣芽孢杆菌的分离鉴定[J].酿酒科技,2010(1):31-35,38.
[2]李增胜.大曲清香型酒酿造中主要微生物[J].酿酒科技,1994(4):93.
[3]兰玉倩,樊丹敏,刘志磊,等.应用PCR-DGGE 指纹技术解析清香型大曲生产过程中酵母群落结构[J].中国酿造,2012,31(10):156-159.
[4]王彩虹.基于克隆文库法研究不同香型大曲微生物群落结构[D].自贡:四川理工学院,2014.
[5]LUCA C,VALENTINA A,PAOLA D,et al.Culture independent methods to assess the diversity and dynamics of microbiota during food fermentation[J].Int J Food Microbiol,2013,167(2):29-43.
[6]HU X L,DU H,REN C,et al.Illuminating anaerobic microbial community and cooccurrence patterns across a quality gradient in Chinese liquor fermentation pit muds[J].Appl Environ Microbiol,2016,82(8):2506-2515.
[7]YANG H Y,WU H,GAO L J,et al.Effects of Lactobacillus curvatus and Leuconostoc mesenteroides on Suan Cai fermentation in Northeast China[J].J Microbiol Biotechn,2016,26(12):2148-2158.
[8]沈馨,尚雪娇,董蕴,等.基于MiSeq 高通量测序技术对3 个孝感凤窝酒曲细菌多样性的评价[J].中国微生态学杂志,2018,30(5):525-530,544.
[9] POLKA J,REBECCHI A,PISACANE V,et al.Bacterial diversity in typical Italian salami at different ripening stages as revealed by highthroughput sequencing of 16S rRNA amplicons[J].Food Microbiol,2015,46:342-356.
[10] 蔡丽云,黄泽彬,须子唯,等.处理垃圾渗滤液的SBR 中微生物种群与污泥比阻[J].环境科学,2018,39(2):880-888.
[11]王丹丹,沈馨,董蕴,等.孝感凤窝酒曲真菌多样性评价[J].中国酿造,2017,36(11):38-42.
[12]刘小飞,孟建玉,赵晓超,等.棉铃虫c-Jun 氨基末端激酶基因的克隆、表达谱及对UV-A 胁迫的响应[J].昆虫学报,2019,62(4):407-417.
[13]王玉荣,沈馨,董蕴,等.鲊广椒细菌多样性评价及其对风味的影响[J].食品与机械,2018,34(4):25-30.
[14]CAPOEASO J G,KUCZYNSKI J,STOMBAUGH J,et al.QIIME allows analysis of high-throughput community sequencing data[J].Nat Meth,2010,7(5):335-336.
[15] CAPOEASO J G,BITTINGER K,BUSHMAN F D,et al.PyNAST:a flexible tool for aligning sequences to a template alignment[J].Bioinformatics,2010,26(2):266-267.
[16] ROBERT C E.Search and clustering orders of magnitude faster than BLAST[J].Bioinformatics,2010,26(19):2460-2461.
[17] COLE J R,CHAI B,FARRIS R J,et al.The ribosomal database project(RDP-II):introducing myRDP space and quality controlled public data[J].Nucleic Acids Res,2007,35(1):D169-D172.
[18] DESANTIS T Z,HUGENHOLTZ P,LARSEN N,et al.Greengenes,a chimera-checked 16S rRNA gene database and workbench compatible with ARB[J].Appl Environ Microbiol,2006,72(7):5069-5072.
[19]CHRISTIAN Q,ELMAR P,PELIN Y.The SILVA ribosomal RNA gene database project:improved data processing and web-based tools[J].Nucl Acids Res,2013,41(D1):D590-D596.
[20]孙儒泳,李庆芬,牛翠娟,等.基础生态学[M].北京:高等教育出版社,2002:160-162.
[21]陈申习,唐洁,张龙,等.清香型小曲白酒机械化生产中微生物动态变化研究[J].中国酿造,2018,37(6):68-72.
[22]张双燕,廖永红,纪南,等.基于高通量测序技术分析北京清香型大曲微生物多样性[J].中国酿造,2016,36(11):49-53.
[23]王雪山,杜海,徐岩.清香型白酒发酵过程中微生物种群空间分布[J].食品与发酵工业,2018,44(9):1-8.
[24]雷振河.采用高通量测序技术分析清香型白酒酿造微生物[J].食品与发酵工业,2015,41(9):164-167.