酸菜是酸渍大白菜的简称,是在低盐浸渍下经过微生物自然发酵而形成的蔬菜发酵制品,是我国特色传统发酵食品之一[1-2]。由于酸菜的制作环境相对开放或半开放,因而大量不同种类的微生物参与酸菜的发酵[3],且不同地域环境制作的酸菜中微生物群落结构存在显著差异。乳杆菌属(Lactobacillus)、明串珠菌属(Leuconostoc)、肠杆菌属(Enterobacter)、乳球菌属(Lactococcus)、假单胞菌(Pseudomonas)、梭菌属(Clostridium)和柠檬酸杆菌属(Citrobacter)是酸菜发酵的主要微生物菌属[4]。此外,通过传统的分离培养发现,植物乳杆菌(Lactobacillus plantarum)、短乳杆菌(Lactobacillus brevis)、罗伊氏乳杆菌(Lactobacillus reuter)、肠膜明串珠菌属(Leuconostoc mesenteroides)和米酒乳杆菌(Lactobacillus sakei)是参与酸菜发酵的主要乳酸菌[5]。
目前,传统的分离培养技术对酸菜中微生物种类的鉴定非常有限,严重低估了微生物丰度和多样性。此外,鉴于一些微生物具有不可培养的特性,故系统全面地解析酸菜微生物的组成和变化始终是一项重要的挑战。近年来,16S rRNA高通量测序技术被广泛应用于传统酿造食品中复杂微生物群落的研究,高通量测序不仅具有高通量、高覆盖、高准确率等特点,还可以全面地揭示样本微生物群落的组成及多样性等信息[6-7]。马艺荧[8]研究发现,乳杆菌属在东北酸菜的细菌菌群中占据主要地位,是东北酸菜中的主要优势菌群。LIU Z等[9]研究表明,乳杆菌属和片球菌属(Pediococcus)是酸菜中的主要微生物菌属。LIU Z等[10]研究发现,乳杆菌属、片球菌属、沙雷氏菌属(Serratia)、寡养单胞菌属(Stenotrophomonas)和魏斯氏菌属(Weissella)是自制酸菜发酵过程中的主要微生物菌属。为了解自然发酵酸菜发酵过程中细菌群落的结构组成,本研究按照东北地区传统方法进行酸菜的腌制,利用16S rRNA高通量测序技术对酸菜发酵细菌群落的动态变化进行全面解析,旨在能够全面准确地分析酸菜发酵期间的微生物动力学,并为建立酸菜微生物信息数据库和优化酸菜发酵工艺提供一定的理论基础。
1 材料与方法
1.1 材料与试剂
新鲜大白菜、食盐:市售。
E.Z.N.A.® 水样DNA 提取试剂盒:美国Omega公司;微生物高通量测序建库试剂盒:北京诺禾致源公司;Phusion HF MM高保真聚合酶链式反应(polymerase chain reaction,PCR)酶:美国BioLabs公司;MinElute® PCR Purification Kit:德国Qiagen公司;Truseq® 脱氧核糖核酸(deoxyribonucleic acid,DNA)PCR-Free Sample Preparation Kit:美国Illumina公司;细菌16S rRNA V3-V4区PCR扩增引物(338F:5'-ACTCCTACGGGAGGCAGCA-3'和806R:5'-GGACTACHVGGGTWTCTAAT-3'):北京诺禾致源公司。
1.2 仪器与设备
Illumina Miseq PE250型测序仪:美国Illumina公司;S1000型PCR仪、CFX96型荧光定量PCR仪、Gel DocTM XR+型凝胶成像系统、低温冷冻离心机:美国Bio Rad公司;DYY-8C型水平电泳槽:北京六一仪器厂;VORTEX-3型旋涡混匀器:德国IKA公司;BT25S型准微量分析天平:日本SANYO公司;BioPhotometer Plus型核酸蛋白测定仪、5810R型高速冷冻离心机:德国Eppendorf公司;NaneDrop 2000型超微量分光光度计:美国Thermo公司。
1.3 实验方法
1.3.1 酸菜的腌制
根据东北地区传统方法进行酸菜的腌制[11],具体方法如下:挑选优质的大白菜,将其在室外晾晒2~3日(晒蔫、杀菌、去除一定的水分)后,摘除黄叶、老帮并清洗干净,将整颗大白菜纵劈成两半,浸入沸水煮2 min(使酶失活、改变大白菜中大分子的结构、杀死部分蔬菜细胞、酵母和霉菌),捞出并沥干水分,冷却后,整齐摆入5 L的大缸中装满压实,用质量分数为2%的无菌食盐水填满密封。用塑料布将发酵罐口扎紧,置于20~25 ℃,相对湿度为40%~65%且避光阴凉的环境进行自然发酵,取发酵1 d、15 d、30 d、45 d、60 d的酸菜水进行微生物的提取,分别命名为SC1、SC15、SC30、SC45和SC60。为保证每次取样的一致性,在大缸同一部位取3个样本,置于-20 ℃条件下贮藏备用。
1.3.2 酸菜DNA提取及PCR建库
利用E.Z.N.A.® 水样DNA 提取试剂盒提取酸菜液样本中微生物的基因组DNA。使用引物338F和806R进行PCR扩增,PCR扩增体系(20 μL):包括模板DNA 10 ng,正向及反向引物各10 pmol,2×Phusion Master Mix 10 μL。扩增程序参考沈毅等[12]的方法,扩增产物使用2%的琼脂凝胶进行电泳检测。使用MinElute® PCR Purification Kit对PCR扩增产物进行纯化。利用Truseq® DNAPCR-FreeSamplePreparationKit制备测序文库并添加接头序列,经NaneDrop2000确定文库质量,使用Illumina Miseq PE250测序平台进行高通量测序。
1.3.3 生物信息学分析
采用FLASH软件[13]对测序的原始数据进行拼接,利用QIIME软件[14]对低质量的拼接序列进行过滤,使用UCHIME软件[15]去除嵌合体,从而获得高质量的Tags序列。根据UCLUST软件[16]在相似性97%的水平上对序列进行聚类,以所有序列数的0.005%作为阈值过滤操作分类单元(operational taxonomic unit,OTU)[17]。利用Silva 132数据库[18]对OTU进行分类学注释。使用QIIME软件[14]对样本进行多样性分析。
1.3.4 总酸度的测定
酸菜水中总酸含量采用国标GB/T 12456—2008《食品中总酸的测定》[19]方法测定。
2 结果与分析
2.1 样本测序数据处理结果
不同发酵时期酸菜样本的细菌16S rRNA V3-V4区序列的测定结果见表1。
表1 酸菜发酵期间样本测序数据处理结果统计
Table 1 Statistics of sequencing data processing results during sauerkraut fermentation
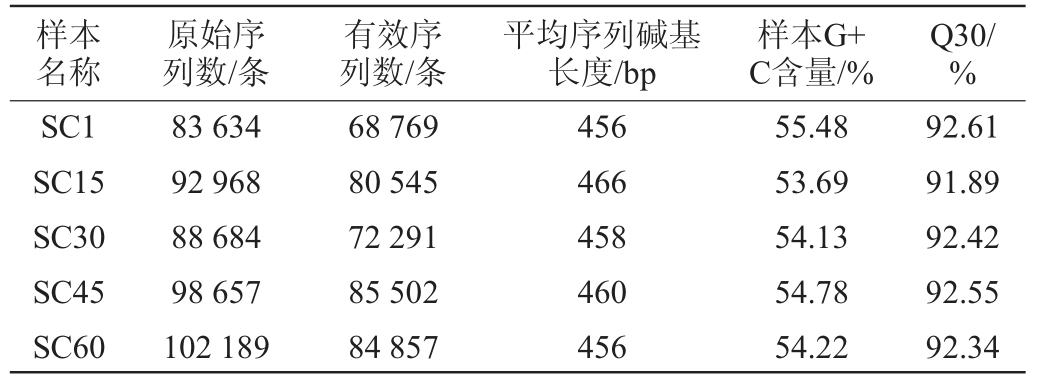
由表1可知,5个不同发酵时期的酸菜样本共检出466 132条原始序列,通过相关软件过滤后最终得到391 964条有效序列。去除条形码(barcode)和引物后,5个不同发酵时期的酸菜样本的平均碱基长度均在450 bp以上,且平均鸟嘌呤(guanine,G)和胞嘧啶(cytosine,C)所占的比率(G+C含量)为54.46%。此外,5个酸菜样本的Q30值(Q30为质量值>30的碱基占总碱基数的百分比)均高于90%,说明测序数据可靠且质量较好。
稀疏性曲线是判定样本的取样大小是否合理及确定测序深度是否可以覆盖整个群落。5个不同发酵时期的酸菜样本的稀疏性曲线的变化见图1。
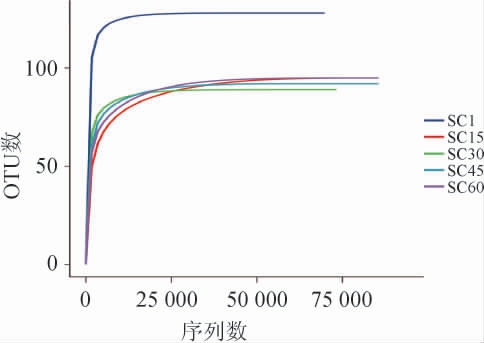
图1 酸菜发酵期间样本细菌群落的稀疏性曲线
Fig.1 Rarefaction curves of bacterial community of samples during sauerkraut fermentation
由图1可知,不同颜色的稀疏性曲线代表该发酵时期样本的稀疏平均值,随着测序深度的增加,被检测发现的OTU呈先迅速增加,后趋于平稳的趋势,说明测序深度足够的覆盖整个酸菜发酵的群落。从而进一步说明16S rRNA高通量测序技术有助于全面解析酸菜微生物群落的组成。
2.2 细菌群落的多样性分析
在97%的相似度阈值下,对391 964条有效序列进行聚类分析,根据QIIME软件进行OTU划分,并根据得到OTU进行Alpha多样性分析,结果见表2。
表2 酸菜发酵期间样本细菌Alpha多样性结果统计
Table 2 Statistics of Alpha diversity results of bacteria during sauerkraut fermentation
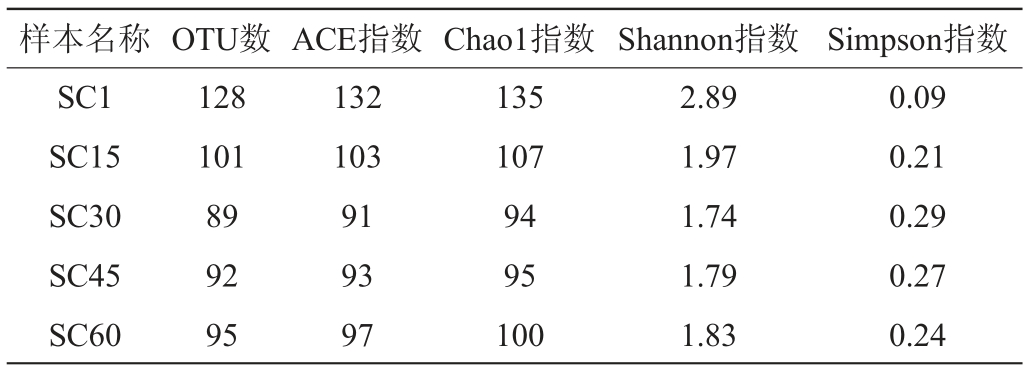
由表2可知,酸菜发酵1 d、15 d、30 d、45 d、60 d样本中细菌的OTU数量分别为128个、101个、89个、92个和95个。SC1细菌群落的ACE指数、Chao1指数和Shannon指数最大,且Simpson指数最小,表明SC1中细菌菌群的多样性最高。酸菜的发酵是由多种微生物共同调节,从而促进其发酵的不断进行。随着发酵时间的不断进行,酸菜样本中细菌群落的ACE指数、Chao1指数和Shannon指数呈先降后缓慢增加的趋势,而Simpson指数则呈相反的趋势。这与DU R等[20]的研究结果一致,推测与酸菜发酵液的环境变化有关,一些不耐酸或不耐盐的微生物在低pH且低盐环境下生长受到抑制。
进一步对不同发酵时期的酸菜样本中细菌菌群的构成进行比较分析并绘制韦恩(Venn)图,结果见图2。
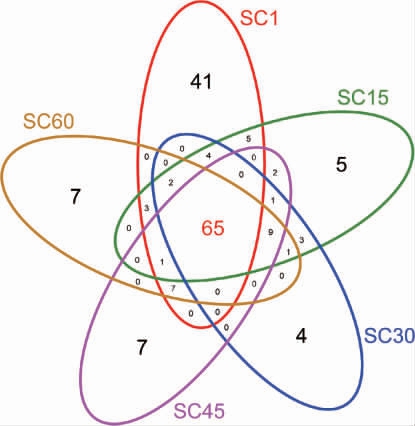
图2 不同发酵时期的酸菜发酵样本中操作分类单元(OTU)的Venn图
Fig.2 Venn diagram of operational taxonomic unit in sauerkraut fermentation samples with different fermentation period
由图2可知,5个不同发酵时期点的样本共有的OTU数为65个,而酸菜发酵期间1 d、15 d、30 d、45 d和60 d样本中细菌的OTU数量分别为41个、5个、4个、7个和7个。结合Alpha多样性分析结果,多样性最丰富的发酵SC1样本中存在41个特有的OTU,凸显酸菜原料中微生物菌群的复杂性和特殊性。
对上述得到的OTU表进行Beta多样性分析,利用非度量多维尺度(nonmetric multidimensional scaling,NMDS)分析对酸菜样本中细菌群落的演替进行分析,结果见图3。
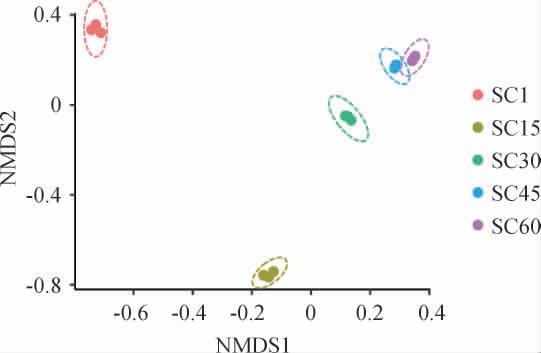
图3 基于酸菜发酵样本的细菌群落结构的非度量多维尺度法分析
Fig.3 Non-metric multidimensional scaling analysis of fermented sauerkraut samples based on the structure of bacterial community
由图3可知,NMDS的结果表明酸菜发酵期间细菌群落在NMDS1和NMDS2的分布是不同的,这就意味着在酸菜发酵过程中细菌群落存在差异。SC1样本与其他样本之间的距离最远,说明SC1样本中细菌群落结构与其他样本差异最大。而SC45和SC60这两个样本之间的距离最近,说明SC45和SC60样本的酸度变化比较稳定,且细菌群落结构相近似。此外,根据NMDS结果可知,酸菜发酵期间细菌群落的变化呈类似抛物线的趋势变化,说明某些主要的微生物影响了酸菜发酵过程,可能正是这些微生物推动酸菜的发酵。
2.3 细菌群落的结构分析
在划分OTU后,挑选不同OTU中丰度最高的序列作为该OTU的代表序列,随后与微生物参考数据库进行97%相似性比对分析,从而对酸菜发酵期间得到的每个OTU进行分类学鉴定,在分类学门水平揭示酸菜发酵期间细菌群落的结构差异,结果见图4。
由图4可知,酸菜发酵期间的细菌群落分别来自酸杆菌门(Acidobacteria)、放线菌门(Actinobacteria)、拟杆菌门(Bacteroidetes)、厚壁菌门(Firmicutes)、芽单胞菌门(Gemmatimonadetes)和变形菌门(Proteobacteria)。其中拟杆菌门、厚壁菌门和变形菌门占整个测序序列的98%左右。这与LIU Z等[9]的研究结果相一致,说明这些门水平的微生物决定酸菜的发酵性能。此外,在酸菜发酵期间,变形菌门的相对丰度随着发酵的不断进行而降低,而厚壁菌门的相对丰度则随着发酵的不断进行而增加。SC1样本中厚壁菌门的相对丰度只有9.64%,然而SC60样本中厚壁菌门的相对丰度增加至97.97%。说明这些来自厚壁菌门的微生物都具有厌氧和耐酸等生长特性。
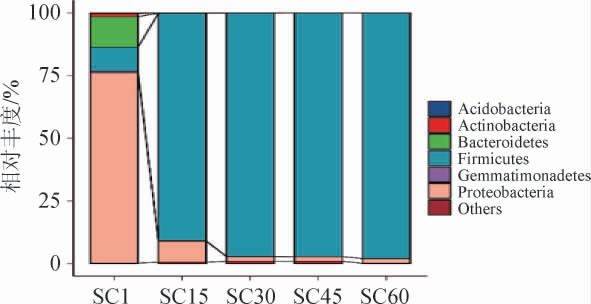
图4 基于门水平酸菜发酵样本中的细菌群落结构分析
Fig.4 Analysis of bacterial community structure in fermented sauerkraut samples based on phylum level
“Others”为未得到分类学注释的OTU之和,每个柱子代表该发酵时期样本的平均值。下同。
为了进一步准确揭示酸菜发酵期间细菌群落的结构,故从分类学的属水平探究构成酸菜发酵细菌群落的组成,并揭晓其在发酵过程中的变化规律,结果见图5。
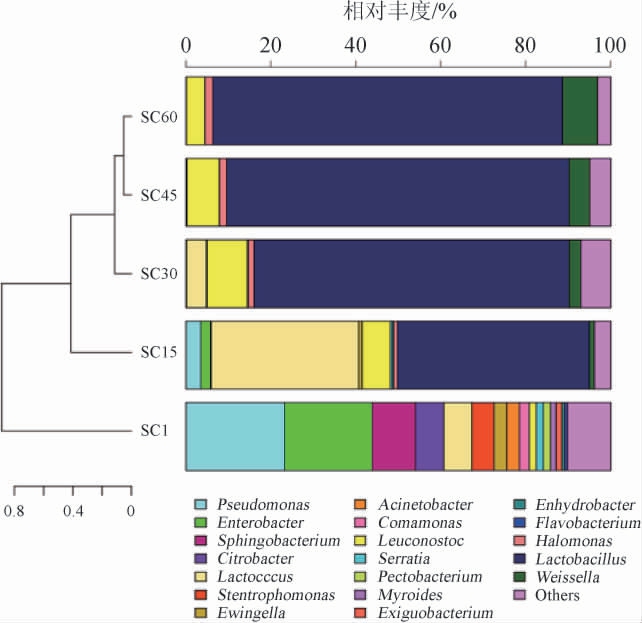
图5 基于聚类分析和属水平酸菜发酵样本中的细菌群落结构分析
Fig.5 Analysis of bacterial community structure in fermented sauerkraut samples based on cluster analysis and genus level
由图5可知,通过聚类分析发现SC1与其他样本的距离是最远的,而SC45和SC60则可以聚在同一个分支。这个结果与图3的结果是相一致,说明在酸菜发酵过程中微生物的结构发生了显著变化,从而导致功能也发生了明显变化,进而促进酸菜的发酵。此外,将相对丰度>1%的属定义为优势细菌属。SC1样本中的优势细菌属主要隶属于假单胞菌属(Pseudomonas)(23.26%)、肠杆菌属(Enterobacter)(20.60%)、鞘氨醇杆菌属(Sphingomonas)(10.21%)、柠檬酸杆菌属(Citrobacter)(6.63%)、乳球菌属(Lactocccus)(6.62%)、寡养单胞菌属(Stentrophomonas)(5.21%)、欧文氏菌属(Ewingella)(2.99%)、不动杆菌属(Acinetobacter)(2.99%)、丛毛单胞菌属(Comamonas)(2.33%)、明串珠菌属(Leuconostoc)(1.66%)、沙雷氏菌属(Leuconostoc)(1.66%)、坚固杆菌属(Pectobacterium)(1.66%)、类香味菌属(Myroides)(1.33%)和微小杆菌属(Exiguobacterium)(1.33%)。这与DU R等[20]的研究结果相似,但其在酸菜发酵初期没有检测到鞘氨醇杆菌属和柠檬酸杆菌属,且假单胞菌属的相对丰度要远高于本研究结果,而明串珠菌属的相对丰度显著低于本研究结果,说明环境的差异造就酸菜发酵初期微生物群落结构的差异。鞘氨醇杆菌属和柠檬酸杆菌属均广泛存在于我国的传统发酵食品中,如醋[21]和辣白菜[10]。假单胞菌属存在于新鲜蔬菜中,生存能力强,适应环境范围广[22]。
随着发酵的不断进行,具有耐酸且厌氧特性的微生物可在该环境中迅速生长繁殖,并占领足够多的生态位,而不耐酸的微生物的生长则受到抑制。由图5可知,SC15样本中的优势细菌属主要隶属于乳杆菌属(Lactobacillus)(45.16%)、乳球菌属(34.68%)、明串珠菌属(6.13%)、假单胞菌属(3.45%)、肠杆菌属(2.35%)和魏斯氏菌属(Weissella)(1.25%),相比SC1样本,乳杆菌属、乳球菌属、明串珠菌属和魏斯氏菌属的相对丰度显著增加,假单胞菌属和肠杆菌属则显著降低,说明乳酸菌是驱动酸菜发酵的主要微生物。其他样本中的优势细菌属主要来自明串珠菌属、乳酸杆菌属、魏斯氏菌属和盐单胞菌属(Halomonas)。说明酸菜的发酵会显著改变微生物的群落结构和组成。此外,本研究发现明串珠菌属和乳酸乳球菌属的相对丰度的变化在酸菜发酵期间呈先增后降的趋势。有趣的是,在SC60样本中不能检测出乳酸乳球菌属,而明串珠菌属的相对丰度仍还有4.40%。说明明串珠菌属能耐受更低的pH环境,这与SONG H S等[23]的研究相似,其发现在自然发酵50 d的韩国泡菜中明串珠菌属的相对丰度为35%。盐单胞菌属、魏斯氏菌属和乳酸杆菌属的相对丰度则在酸菜发酵过程中持续增加。在酸菜发酵期间,盐单胞菌属的相对丰度从0.03%增加至1.84%,而魏斯氏菌属的相对丰度则从0.01%增加至8.26%。盐单胞菌属具有耐盐的特性,能分解葡萄糖,产生多种风味物质,推测其的存在与生产酸菜“盐渍”这一步骤有关[24]。魏斯氏菌属广泛存在与含盐的发酵食品中,可参与生成醛、醇和酮等挥发性风味物质,还可使发酵食品的颜色更鲜亮和口感更醇厚[25]。SC1样本中乳酸杆菌属的相对丰度极低,仅仅只有0.01%,而仅仅发酵15 d就可达到45.16%,说明乳杆菌属可以迅速适应酸菜的发酵环境,从而快速生长繁殖。而SC60样本中的相对丰度可增加至82.32%,相比SC1样本中乳杆菌属的相对丰度增加了8 000多倍。乳酸菌是酸菜发酵最为重要的微生物之一,其在发酵期间可产生大量的有机酸(如乳酸、柠檬酸、乙酸等),导致酸菜发酵液的pH降低,进一步抑制其他杂菌的生长繁殖。此外,乳酸菌不仅具有抑制腐败微生物的生长的能力,还可以延长发酵食品的保质期,提高产品的质量安全[26]。乳酸菌产生的乳酸,是酸菜酸味的主要来源,且在发酵过程中能与醇、醛和酮类物质发生反应,产生大量的挥发性风味物质[27]。
2.5 酸菜发酵过程中酸度的变化及相关性分析
酸度是决定酸菜产品品质的重要因素之一,酸菜发酵过程中总酸含量的变化见图6。
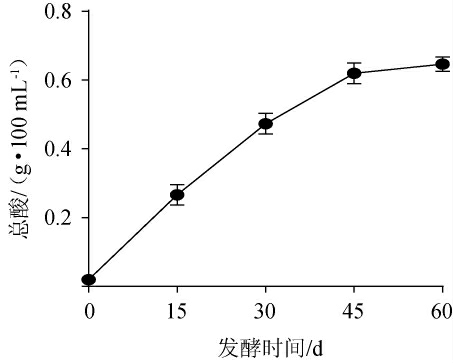
图6 酸菜发酵过程总酸含量的变化
Fig.6 Changes of total acid contents during sauerkraut fermentation period
由图6可知,酸菜发酵起始总酸含量为0.02 g/100 mL,随着发酵进行逐渐上升,于发酵45 d达到稳定,其总酸含量为0.62 g/100 mL,而发酵60 d后达到最高0.65 g/100 mL。说明酸菜发酵45 d就可达到成熟。
进一步对酸菜发酵过程中总酸和主要微生物菌属的变化进行相关性分析,为了降低错误率,只保留相关系数R>0.6和显著性(P<0.05)值结果见图7。
由图7可知,总酸与乳杆菌属、盐单胞菌属和魏斯氏菌属呈正相关,而与其他发酵过程中的主要微生物菌属呈负相关,说明乳杆菌属、盐单胞菌属和魏斯氏菌属是驱动酸菜成熟的主要微生物菌属,总酸是影响酸菜发酵过程中细菌菌群结构变化的重要因素之一。乳杆菌属、盐单胞菌属和魏斯氏菌属与假单胞菌属、肠杆菌属、柠檬酸杆菌属、欧文氏菌属、不动杆菌属、沙雷氏菌属和水栖菌属呈负相关,说明与乳杆菌属、盐单胞菌属和魏斯氏菌属呈负相关的微生物菌属大部分都是不耐酸的微生物菌属,不能在酸菜发酵环境生长繁殖。
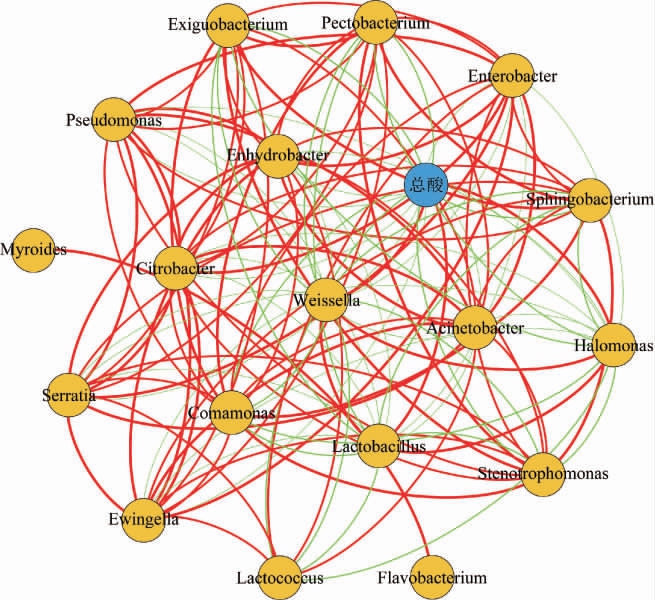
图7 酸菜发酵过程中总酸与属水平微生物之间的相关性
Fig.7 Correlation analysis between total acid and bacterial communities in genes level during sauerkraut fermentation period
红色的线表示该属水平微生物与总酸呈正相关,绿色的线表示该属水平微生物与总酸呈负相关。
3 结论
本研究利用16S rRNA高通量测序技术主要研究了酸菜发酵过程中的细菌群落结构演替规律。从5个不同发酵时期的酸菜样本中收集的有效序列的平均碱基长度均在450 bp以上,且平均GC含量为54.46%;在97%相似度水平下聚类分析得到各样本的OTU数分别为128个、101个、89个、92个和95个。不同发酵时期酸菜样本的微生物群落结构和多样性均存在一定的差异,其中发酵第1天的酸菜样本中微生物丰富度和多样性最高。但随着发酵的不断进行,微生物丰度度和多样性呈先降后缓慢增加的趋势。结果表明,细菌群落结构与发酵进程显著相关,因此不同发酵时期优势菌属不同。酸菜发酵期间的主要优势细菌属为乳酸杆菌属(Lactobacillus)、肠杆菌属(Enterobacter)、明串珠菌属(Leuconostoc)、盐单胞菌属(Halomonas)和魏斯氏菌属(Weissella)等,乳酸菌占据绝对的优势地位。本研究可为建立酸菜微生物信息数据库和优化酸菜发酵工艺提供一定的理论基础。
[1]杜书.酸菜自然发酵过程中风味及质地变化规律研究[D].沈阳:沈阳农业大学,2013.
[2]张玉龙,胡萍,湛剑龙,等.发酵酸菜的研究及其进展[J].食品安全质量检测学报,2014(12):3998-4003.
[3]荆雪娇,李艳琴,燕平梅,等.传统发酵蔬菜微生物群落结构分析[J].食品与发酵科技,2016,52(1):28-32.
[4]ZHOU Q,ZANG S,ZHAO Z,et al.Dynamic changes of bacterial communities and nitrite character during northeastern Chinese sauerkraut fermentation[J].Food Sci Biotechnol,2018,27(1):79-85.
[5]张鲁冀,孟祥晨.自然发酵东北酸菜中乳杆菌的分离与鉴定[J].东北农业大学学报,2010(11):131-137.
[6]SHOKRALLA S,SPALL J L,GBISON J F,et al.Next-generation sequencing technologies for environmental DNA research[J].Mol Ecol,2012,21(8):1794-1805.
[7]王兴春,杨致荣,王敏,等.高通量测序技术及其应用[J].中国生物工程杂志,2012,32(1):109-114.
[8]马艺荧.东北酸菜发酵过程中细菌多样性及有机酸的变化规律[D].哈尔滨:东北农业大学,2019.
[9]LIU Z,LI J,WEI B,et al.Bacterial community and composition in Jiangshui and Suan-cai revealed by high-throughput sequencing of 16S rRNA[J].Int J Food Microbiol,2019,306(10):1-9.
[10] LIU Z,PENG Z,HUANG T,et al.Comparison of bacterial diversity in traditionally homemade paocai and Chinese spicy cabbage[J].Food Microbiol,2019,83(10):141-149.
[11]韩宏娇,丛敏,李欣蔚,等.自然发酵酸菜化学成分含量和微生物数量的动态变化及其相关性分析[J].食品工业科技,2019,40(2):148-153.
[12]沈毅,陈波,王西,等.酱香型郎酒高温大曲、酒醅和窖泥中细菌群落结构分析[J].中国酿造,2020,39(2):89-93.
[13] MAGOCˇT,SALZBERG S L.FLASH:fast length adjustment of short reads to improve genome assemblies[J].Bioinformatics,2011,27(21):2957-2963.
[14]CAPORASO J G,KUCZYNSKI J,STOMBAUGH J,et al.QIIME allows analysis of high-throughput community sequencing data[J].Nat Meth,2010,7(5):335-336.
[15] EDGAR R C,HAAS B J,CLEMENTE J C,et al.UCHIME improves sensitivity and speed of chimera detection[J].Bioinformatics,2011,27(16):2194.
[16]EDGAR R C.Search and clustering orders of magnitude faster than BLAST[J].Bioinformatics,2010,26(19):2460-2461.
[17]BOKULICH N A,SUBRAMANIAN S,FAITH J J,et al.Quality-filtering vastly improves diversity estimates from Illumina amplicon sequencing[J].Nat Meth,2012,10(1):57-59.
[18]QUAST C,PRUESSE E,YILMAZ P,et al.The SILVA ribosomal RNA gene database project:improved data processing and web-based tools[J].Nucl Acid Res,2012,41(D1):D590-D596.
[19]中华人民共和国国家质量监督检验检疫总局,中国国家标准化管理委员会.GB 12456—2008 食品中总酸的测定[S].北京:中国标准出版社,2008.
[20]DU R,SONG G,ZHAO D,et al.Bacterial diversity and community structure during fermentation of Chinese sauerkraut with Lactobacillus casei 11MZ-5-1 by Illumina Miseq sequencing[J].Lett Appl Microbiol,2017,66(1):55-62.
[21]王宗敏.镇江香醋醋酸发酵阶段菌群结构变化与风味物质组成之间的相关性研究[D].无锡:江南大学,2016.
[22] NÜBLING S,SCHMIDT H,WEISS A.Variation of the Pseudomonas community structure on oak leaf lettuce during storage detected by culturedependent and -independent methods[J].Int J Food Microbiol,2016,129:95-103.
[23]SONG H S,WHON T W,KIM J,et al.Microbial niches in raw ingredients determine microbial community assembly during kimchi fermentation[J].Food Chem,2020,318(7):141-150.
[24]巢雨舟.盐渍芥菜二次发酵技术研究及其盐渍液成分分析[D].长沙:湖南农业大学,2015.
[25]李巧玉,方芳,堵国成,等.魏斯氏菌在发酵食品中的应用[J].食品与发酵工业,2017,43(10):241-247.
[26] LEMAY M J,RODRIGUE N,GARIÉPY C,et al.Adaptation of Lactobacillus alimentarius to environmental stresses[J].Int J Food Microbiol,2000,55(3):249-253.
[27] TAMANG B,TAMANG J P,SCHILLINGER U,et al.Phenotypic and genotypic identification of lactic acid bacteria isolated from ethnic fermented bamboo tender shoots of North East India[J].Int J Food Microbiol,2008,121(1):35-40.