中国传统发酵豆制品酱油、豆豉、纳豆、腐乳(sufu)在调味品中具有重要地位,而腐乳因其独特而诱人的风味被称为“中国奶酪”或“东方芝士”。腐乳可分为红方、白方、青方以及其他各种花色腐乳,白腐乳因其独特的风味和纯净的外观在华南地区及海外拥有较大的市场[1-2]。
白腐乳的生产工艺包括大豆浸泡、打浆、滤浆、煮浆、点浆、压榨成型、切块,以及接种毛霉、前酵、加盐腌制、灌汤后酵、包装等。虽然接种了纯种毛霉进行前期发酵[3],但由于固态发酵是在开放的空间进行,环境中的微生物特别是细菌仍然可以进入并参与发酵,从而影响腐乳的品质。白腐乳在后酵过程中常常出现的白点[4]、胀盖漏汁[5]等感官品质问题,均可能与后酵过程中的微生物相关。此外,也有报道显示,腐乳生产原料豆腐可能被金黄色葡萄球菌、肠杆菌等食源致病菌污染而存在安全问题[6]。因此,解析白腐乳发酵过程中出现的细菌群落变化将对腐乳生产及产品质量提升具有很好的指导作用。
研究表明,高通量测序方法可以在脱氧核糖核酸(deoxyribonucleic acid,DNA)层面比较清晰地展示发酵体系中出现的微生物[7],而传统的培养组学则可以从发酵体系中得到纯的菌种,可以进一步从菌种的角度进行深入的研究。目前,对腐乳菌群的研究主要集中在红腐乳[8-10],白腐乳菌群研究的报道较少。因此,本研究拟对白腐乳发酵过程的全过程(从前酵至后酵180 d)细菌群落的演替进行系统的研究,为进一步研究白腐乳发酵过程中的细菌群落变化对白腐乳品质的影响奠定基础。
1 材料与方法
1.1 材料与试剂
1.1.1 原料
实验原料均采自广东某腐乳厂同一批次生产的白腐乳样品,实验原料包括酸浆水(SJ)、白坯(BP)、毛坯(MP)、盐坯(YP)、后酵45 d(CG45)、后酵90 d(CG90)、后酵180 d(CG180)。所有样品均收集三份然后混匀冻存于-80 ℃冰箱备用。
1.1.2 化学试剂
核酸荧光定量染料(P7589):美国Invitrogen 公司;聚合酶链式反应(polymerase chain reaction,PCR)Mix、Marker、DNA提取试剂盒、PCR产物纯化试剂盒:广州东盛生物科技有限公司。
1.1.3 培养基
MRS肉汤培养基、M17肉汤培养基、LB固体培养基、营养琼脂(nutrient agar,NA)培养基:广州环凯生物科技有限公司。
1.2 仪器与设备
IlluminaHiSeq2500高通量测序平台:美国Illumina公司;DYY-6C电泳仪:北京六一生物科技有限公司;BG-gdsAUTO(130)凝胶成像系统:北京百晶生物技术有限公司;LDX-30KBS立式压力蒸汽灭菌器:上海申安医疗器械厂;SW-CJ-1C超净工作台苏州净化设备厂;MDF-86V50超低温冰箱:安徽中科都菱电器有限公司。
1.3 方法
1.3.1 白腐乳加工工艺流程及操作要点

操作要点:白腐乳生产工艺可分为四个阶段。第一阶段是豆腐的生产,包括泡豆、打浆、煮浆、点浆、蹲脑、挤压成型等工艺,其中用于点浆的凝固剂是酸浆水,即自然发酵的豆腐乳清。第二阶段,也称为前期发酵阶段,新鲜切好的豆腐块(白坯)接种纯种毛霉,在适当的条件下培养约48 h,待豆腐块表面覆盖着厚实的白色真菌菌丝结束(毛坯)。第三阶段,将毛坯进行腌制,降低毛坯中的水分(盐坯)。第四阶段为后熟阶段,将盐坯装罐并加入含有一定浓度的食盐和酒精的汤汁,陈放1~6个月然后包装白腐乳成品。
1.3.2 白腐乳发酵过程中细菌群落演替分析
对各个样品按照试剂盒操作说明进行总DNA提取,并通过0.8%琼脂糖凝胶电泳检测DNA提取质量,同时采用紫外分光光度计对DNA进行定量。然后,以各样本提取的总细菌基因组DNA为模板,以16S rRNA 基因V3-V4区特异引物338F(5'-ACTCCTACGGGAGGCAGCA-3')和806R(5'-GGACTACHVGGGTWTCTAAT-3')对模板进行PCR扩增,PCR扩增体系(25 μL):5×反应缓冲液(reaction buffer)5 μL,5×GC buffer 5 μL,脱氧核苷三磷酸(deoxyribonucleoside triphosphate,dNTP)(2.5 mmol/L)2 μL,正向引物(Forward primer)(10 μmol/L)1 μL,反向引物(10 μmol/L)1 μL,DNA 模板2 μL,双蒸水(ddH2O)8.75 μL,Q5 DNA聚合酶0.25 μL。PCR扩增参数:98 ℃预变性2 min;98 ℃变性15 s,55 ℃退火30 s,72 ℃延伸30 s,扩增30个循环;72 ℃末端延伸5 min。PCR扩增产物通过2%琼脂糖凝胶电泳进行检测,并对目标片段进行切胶回收。最后,送样至派诺森生物使用Illumina MiSeq平台进行高通量测序及生物信息学分析。
1.3.3 白腐乳发酵过程中可培养细菌的分离鉴定
采用传统平板法对部分可培养细菌进行分离、纯化,采用16S rDNA对分离的细菌进行鉴定。用于分菌的样品及其采集和保存方法有1.1中所有样品和后酵30 d腐乳(CG30),其采集和保存方法同1.1。
细菌的分离纯化:在超净台中取5 g腐乳于研钵中(酸浆水取5 mL),研磨后加入45 mL生理盐水混匀制备样品液,在超净台中吸取1 mL样品液于9 mL无菌生理盐水中,依次稀释至10-3、10-4、10-5倍,分别取100 μL用MRS和M17固体培养基依次倾注倒平板,分别取100 μL于LB、NA固体培养基涂布,于30 ℃的恒温培养箱中,培养72 h。挑取单菌落液体培养,经平板划线法进一步纯化直至获得单菌落。将分离得到的菌株编号并于-80 ℃冰箱甘油保藏备用。
分离菌株的鉴定:采用东盛生物细菌DNA提取试剂盒按照说明书步骤提取每个分离菌株的基因组DNA,然后使用16S rDNA通用引物(上游引物27F:5'-AGAGTTTGATCCTGGCTCAG-3'和下游引物1495R:5'-CTACGGCTACCTTGTTACGA-3')进行PCR扩增,PCR扩增体系为50 μL,包括:λDNA 1 μL,引物27F和引物1495R各2 μL,2×PCR Mix 20 μL,超纯水25 μL。PCR扩增程序:94 ℃预变性5 min;94 ℃变性30 s,42 ℃退火30 s,72 ℃延伸1 min,30次循环;72 ℃再延伸10 min。在琼脂糖凝胶中加入GoldView染料制胶,取8.0 μLPCR产物进行琼脂糖凝胶电泳,在紫外凝胶成像仪下观察。然后利用PCR产物及DNA片段纯化试剂盒按照其操作手册从PCR反应液中回收DNA片段,将回收得到的DNA片段送到生工生物工程(广州)公司进行DNA测序,将测得的DNA序列在美国国家生物技术信息中心(national center of biotechnology information,NCBI)进行基本局部比对搜索工具(basic local alignment search tool,BLAST)对比。
1.3.4 数据处理
数据分析使用Excel 2019、Origin 2018等软件。生物信息学分析:采用滑动窗口法对FASTQ格式的双端序列逐一作质量筛查,随后利用FLASH软件(v1.2.7)对通过质量初筛的双端序列根据重叠碱基进行配对连接,根据每个样本所对应的Index信息,将连接后的序列识别分配入对应样本(要求Index序列完全匹配),从而获得每个样本的有效序列。对于每个OTU的代表序列,在QIIME软件中使用默认参数,通过将OTU代表序列与对应数据库的模板序列相比对,获取每个OTU所对应的分类学信息,计算各样本的Alpha多样性。使用Origin 2018软件,对关注的特定样本在特定分类水平的组成绘制柱状图。
2 结果与分析
2.1 白腐乳发酵过程中细菌群落演替分析
对于微生物群落而言,有多种指数来反映其Alpha多样性。常用的度量指数主要包括侧重于体现群落丰富度的Chao1指数和ACE指数,以及兼顾群落均匀度的Shannon指数和Simpson指数。一般而言,Chao1或ACE指数越大,表明群落的丰富度越高;Shannon指数对群落的丰富度以及稀有OTU更敏感,而Simpson指数对均匀度和群落中的优势OTU更敏感。样品Alpha多样性指数结果见表1。由表1可知,毛坯中细菌群落的丰富度最高,白坯和后酵180 d样品中细菌群落的丰富度最低。
表1 白腐乳各样品中细菌群落测序的Alpha多样性指数
Table 1 Alpha-diversity indexes of bacterial community sequencing in each sample of white sufu
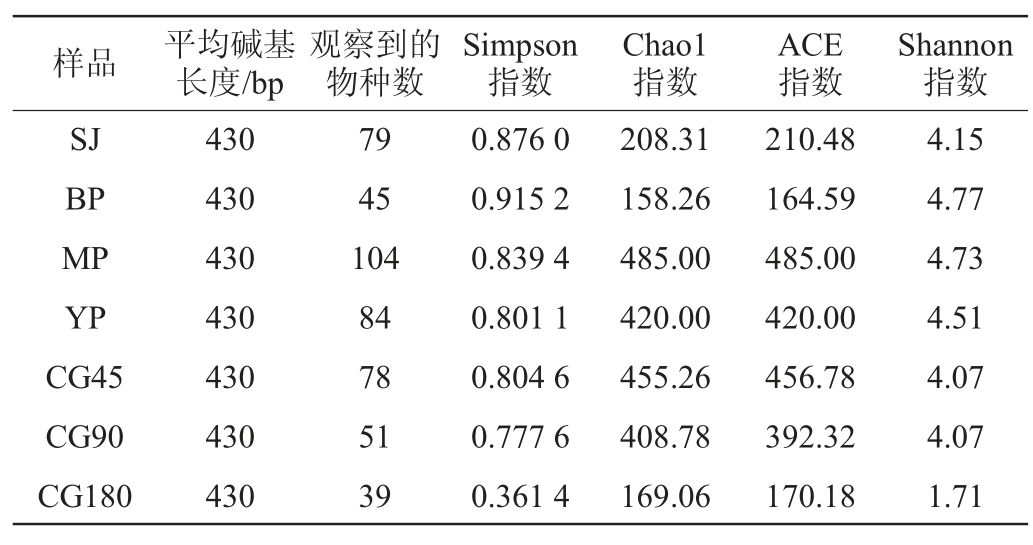
腐乳发酵过程中各样品门水平、属水平细菌群落相对丰度分布结果见图1。由图1A可知,在后酵45 d及以前样品中厚壁菌门(Firmicutes)和变形菌门(Proteobacteria)为优势菌门;在整个发酵过程中厚壁菌门均为优势菌门,尤其是在后酵45~180 d,厚壁菌门相对丰度均在92%以上。由图1B可知,在酸浆水和白坯中相对丰度较高的有乳杆菌属(Lactobacillus)、醋酸菌属(Acetobacter);毛坯中的优势菌属是库特氏菌属(Kurthia)、不动杆菌属(Acinetobacter)、芽孢杆菌属(Bacillus);盐坯中的优势菌属是库特氏菌属(Kurthia)、魏斯氏菌属(Weissella)和不动杆菌属(Acinetobacter);后酵45 d样品中的优势菌属是明串珠菌属(Leuconostoc)、乳球菌属(Lactococcus)和四联球菌属(Tetragenococcus);后酵90 d样品中的优势菌属是库特氏菌属、明串珠菌属和乳球菌属;后酵180 d样品中的优势菌属是四联球菌属、库特氏菌属和魏斯氏菌属。
图1 白腐乳发酵过程中细菌群落基于门水平(A)及属水平(B)相对丰度
Fig.1 Relative abundance of bacterial community in white sufu samples in the fermentation process based on phylum level (A) and genus level (B)
2.2 白腐乳发酵过程中可培养细菌的分离鉴定
利用DNA提取试剂盒提取分离菌株DNA,采用通用引物27F和1492R进行PCR扩增并进行琼脂糖凝胶电泳,结果见图2。由图2可知,所有条带碱基数均在1 500 bp左右,条带单一且清晰明亮,可进行PCR产物纯化并测序。

图2 部分菌株基于16S rDNA序列PCR扩增产物琼脂糖凝胶电泳图
Fig.2 Electrophoretogram of 16S rDNA PCR amplification products of some isolated strains
对PCR产物纯化并测序后,将测得的DNA序列在NCBI进行BLAST对比,相似度均在99%以上。从酸浆水、白坯、毛坯、后酵45 d、后酵90 d、后酵180 d 6个样品中用MRS、M17、LB和NA四种培养基分离、纯化和鉴定出64株细菌,结果见表2。
表2 白腐乳发酵过程中可培养细菌基于16S rDNA序列的分离鉴定结果
Table 2 Isolation and identification results of culturable bacteria in the white sufu fermentation process based on 16S rDNA sequences

注:相对丰度是指分离鉴定到的细菌菌株的属在对应样品的高通量测序结果中的相对丰度。
由表2可知,从酸浆水中主要分离鉴定出融合魏斯氏菌(Weissella confuse),其在白坯和毛坯中也大量分离到,而在白坯中还分离鉴定到了蜡样芽孢杆菌(Bacillus cereus)和肉葡萄球菌(Staphylococcus carnosus);在毛坯中还分离鉴定到乳酸乳球菌(Lactococcus lactis)和粪肠球菌(Enterococcus faecalis),这两种细菌在后酵45 d和90 d腐乳中均有分离到;除乳酸乳球菌外,后酵45 d和90 d样品中还分离出较多的肠球菌;后酵180 d样品中分离鉴定到蜡样芽孢杆菌和乳酸乳球菌。
2.3 高通量测序与培养分离方法结合分析菌群变化
酸浆水中丰度较高的细菌有乳杆菌和醋酸菌,这和课题组先前的研究一致(样品源自不同工厂)[11],本实验从酸浆水、白坯和毛坯中都分离到比较多的融合魏斯氏菌。魏斯氏菌是革兰氏阳性细菌,细胞不运动,显微镜下菌体是短杆状的,其广泛分布于食物、土壤、胃肠道和呼吸道等环境中,其中一个分支由食窦魏斯氏菌和融合魏斯氏组成,它们常分布在含盐的发酵食品中。魏斯氏菌是参与食品发酵的重要乳酸菌之一,对于发酵食品的发酵进程和风味有重要的影响和贡献[12]。
序列分析的结果显示,从毛坯开始出现乳球菌,且在后酵45~90 d样品中乳球菌属相对丰度较高,而通过培养组学分析,从白坯到后酵的各个阶段均分离到多株乳酸乳球菌,说明乳球菌是白腐乳发酵过程中的主要细菌之一。乳酸乳球菌被广泛应用于发酵乳制品中,对牛乳蛋白质的降解和酸奶风味具有重要的影响[13]。
明串珠菌属在后酵45~150 d腐乳中相对丰度较高。明串珠菌属是兼性厌氧菌,具有嗜温性,广泛存在于发酵蔬菜、肉制品等[14],但并未分离到该菌。在白坯中分离到肉葡萄球菌,该菌对发酵香肠的风味有重要作用[15]。在后酵45~90 d腐乳中还分离鉴定到较多的粪肠球菌、耐久肠球菌及未鉴定到种的肠球菌,肠球菌在其他腐乳中也有分布[9]。有报道表明,某些芽孢杆菌、乳杆菌和肠杆菌会导致辣椒酱及豆瓣酱胀盖[16-17],本研究高通量测序结果显示白腐乳发酵后期也存在一定的芽孢杆菌、乳杆菌和肠杆菌,因此后酵过程中某些活跃的芽孢杆菌、乳杆菌和肠杆菌可能对腐乳“胀盖漏汁”产生影响。在白坯及后酵180 d腐乳中均分离到蜡样芽孢杆菌,但在后酵阶段不是优势菌属。
在腐乳成熟的后期,嗜盐四联球菌的丰度越来越高。虽然四联球菌的丰度很高,一开始并未分离到该菌。后期通过文献调研,在培养基中增补了氯化钠后从后熟30 d腐乳中分离到11株嗜盐四联球菌(Tetragenococcus halophilus)。嗜盐四联球菌对盐的耐受性较强,是一种革兰氏阳性、四聚体形成、不活动、过氧化氢酶阴性、广泛存在于高盐发酵食品中的微生物[18-19],在发酵食品中具有抑制组胺产生[20]、降解黄曲霉毒素B1的作用[21],并且能够产生风味物质[22]。后酵45 d和90 d腐乳中乳球菌、明串珠菌和四联球菌等菌属相对丰度较高,后酵180 d腐乳中四联球菌、魏斯氏菌和乳杆菌等菌属相对丰度较高,可能是由于这些菌属的某些菌种对腐乳后酵体系的高盐、微氧及复杂的营养环境具有较好的适应性,并且对其他微生物产生抑制作用。
值得关注的是,在发酵的过程中还出现了不动杆菌和库特氏菌。不动杆菌属在毛坯中的相对丰度最高,在发酵各阶段都有存在,这和XU D D等[10]的研究结果一致;不动杆菌属的部分种是有感染性的[23-24],但大多数种是非致病的环境生物,不动杆菌属也存在于其他各种发酵食品中[15,22]。库特氏菌在盐坯和后酵90 d样品中丰度较高,在腐乳发酵过程中一直存在且有一定的波动,这与LIANG J J等[25]的结果相似。
对不同样品进行细菌的分离鉴定,发现能够分离鉴定出的细菌种类较少,并且纯培养出的细菌菌属不是该样品丰度最高的菌属,可能原因是培养基、培养条件不适合该样品高丰度菌属生长,或分菌时挑取菌数有限,而纯培养出来的细菌是较适合实验条件生长的,或是高通量测序方法可能检测到死亡的微生物群的完整核苷酸序列[26-27]。
3 结论
通过高通量测序及培养分离结合的方法分析了白腐乳发酵过程中细菌群落演替。白腐乳从前酵到后酵180 d的整个发酵过程中细菌群落均有较大变化,从白坯、毛坯、后熟45 d腐乳至后熟180 d腐乳中的优势菌属分别为:乳酸杆菌属、不动杆菌属、明串珠菌属和四联球菌属。从白腐乳生产及发酵过程中分离鉴定出75株细菌,包括融合魏斯氏菌(Weissella confusa)、蜡样芽孢杆菌(Bacillus cereus)、肉葡萄球菌(Staphylococcus carnosus)、乳酸乳球菌(Lactococcus lactis)、嗜盐四联球菌(Tetragenococcus halophilus)、粪肠球菌(Enterococcus faecalis)和耐久肠球菌(Enterococcus durans)等。
[1]蔡若纯.腐乳多肽的分离纯化及功能性研究[D].广州:华南理工大学,2017.
[2]许家威,李一红,叶芒,等.腐乳前酵期微生物与理化成分的动态分析[J].中国酿造,2019,38(1):84-88.
[3]魏冠棉,张军伟,周鹏.油腐乳发酵过程中的品质分析[J].食品科学,2019,40(14):138-143.
[4]YANG J,SUNW D,CUI C,et al.Gamma-glutamylation of the white particulates of sufu and simultaneous synthesis of multiple acceptor amino acids-containing gamma-glutamyl peptides:Favorable catalytic actions of glutaminase[J].LWT-Food Sci Technol,2018,96:315-321.
[5]许喜林,黄恺婷,魏萍.腐乳中产气芽孢杆菌的分离与控制研究[J].现代食品科技,2011,27(6):615-617.
[6]HAN B Z,CAO C F,ROMBOUTS F M,et al.Microbial changes during the production of Sufu-aChinesefermented soybean food[J].Food Control,2004,15(4):265-270.
[7]LO Y T,SHAW P C.Application of next-generation sequencing for the identification of herbal products[J].Biotechnol Adv,2019,37(8):107450.
[8]XIE C,ZENG H,WANG C,et al.Volatile flavour components,microbiota and their correlations in different sufu,a Chinese fermented soybean food[J].J Appl Microbiol,2018,125(6):1761-1773.
[9]HUANG X,YU S,HAN B,et al.Bacterial community succession and metabolite changes during sufu fermentation[J].LWT-Food Sci Technol,2018,97:537-545.
[10]XU D D,WANG P,ZHANG X,et al.High-throughput sequencing approach to characterize dynamic changes of the fungal and bacterial communities during the production of sufu,a traditional Chinese fermented soybean food[J].Food Microbiol,2020,86:103340.
[11]FEI Y,LI L,CHEN L,et al.High-throughput sequencing and culturebased approaches to analyze microbial diversity associated with chemical changes in naturally fermented tofu whey,a traditional Chinese tofu-coagulant[J].Food Microbiol,2018,76:69-77.
[12]李巧玉,方芳,堵国成,等.魏斯氏菌在发酵食品中的应用[J].食品与发酵工业,2017,43(10):241-247.
[13]CAVANAGH D,FITZGERALD G F,MCAULIFFE O.From field to fer-mentation:The origins of Lactococcus lactis and its domestication to the dairy environment[J].Food Microbiol,2015,47:45-61.
[14]HEMME D,FOUCAUD-SCHEUNEMANN C.Leuconostoc,characteristics,use in dairy technology and prospects in functional foods[J].Int Dairy J,2004,14(6):467-494.
[15]TJENER K,STAHNKE L H,ANDERSEN L,et al.Growth and production of volatiles by Staphylococcus carnosus in dry sausages:Influence of inoculation level and ripening time[J].Meat Sci,2004,67(3):447-452.
[16]NIU C,XUE Y,LIU C,et al.Identification of gas-forming spoilage bacteria in chili sauce and its control using nisin and salt[J].LWT-Food Sci Technol,2020,118:108658.
[17]NIU C,FAN Z,ZHENG F,et al.Isolation and identification of gas-producing spoilage microbes in fermented broad bean paste[J].Food Control,2018,84:8-16.
[18]JEONG D W,HEO S,LEE J H.Safety assessment of Tetragenococcus halophilus isolates from doenjang,a Korean high-salt-fermented soybean paste[J].Food Microbiol,2017,62(4):92-98.
[19]JEONG D W,KIM H R,JUNG G,et al.Bacterial community migration in the ripening of doenjang,a traditional Korean fermented soybean food[J].J Microbiol Biotechn,2014,24(5):648-660.
[20]KUDA T,IZAWA Y,ISHII S,et al.Suppressive effect of Tetragenococcus halophilus,isolated from fish-nukazuke,on histamine accumulation in salted and fermented fish[J].Food Chem,2012,130(3):569-574.
[21]LI J,HUANG J,JIN Y,et al.Aflatoxin B1 degradation by salt tolerant Tetragenococcus halophilus CGMCC 3792[J].Food Chem Toxicol,2018,121:430-436.
[22]UDOMSIL N,RODTONG S,CHOI Y J,et al.Use of Tetragenococcus halophilus as a starter culture for flavor improvement in fish sauce fermentation[J].J Agr Food Chem,2011,59(15):8401-8408.
[23]THAO N V,BYUN J,D'SOUZA R,et al.Adjustment of modified carbapenem inactivation method conditions for rapid detection of carbapenemase-producing Acinetobacter baumannii[J].Ann Laborat Med,2020,40(1):21-26.
[24]CARNEY R L,LABBATE M,SIBONI N,et al.Urban beaches are environmental hotspots for antibiotic resistance following rainfall[J].Water Res,2019,167:115081.
[25]LIANG J J,LI D,SHI R,et al.Effects of microbial community succession on volatile profiles and biogenic amine during sufu fermentation[J].LWTFood Sci Technol,2019,114:108379.
[26]JAGADEESAN B,GERNER-SMIDT P,ALLARD M W,et al.The use of next generation sequencing for improving food safety:Translation into practice[J].Food Microbiol,2019,79:96-115.
[27]LAGIER J,DUBOURG G,MILLION M,et al.Culturing the human microbiota and culturomics[J].Nat Rev Microbiol,2018,16(9):540-550.