中国传统固态发酵白酒的酿造微生物包括大曲、酒醅、窖泥和酿酒环境等四大微生物菌群[1]。不同菌群微生物在酿酒过程中的活动影响白酒的品质[2]。酱香型白酒在其独特的酿造环境和工艺条件下,酒曲、酒醅、窖泥中形成了丰富的微生物资源[3]。这些微生物在酱香型白酒制曲和酿造过程中,扮演十分重要的角色,它不仅仅是酿酒原料的组成部分,还参与乙醇和香味物质的形成[4]。
细菌是酱香型白酒酿造过程中重要的产香功能微生物,具有分泌代谢降解蛋白质及淀粉的水解酶类,对酒体的风味质量有着极大的贡献和关键调控作用[5]。高温大曲中,细菌占绝对优势,有90%以上的数量,以获得所需香味物质[6-8]。堆积发酵又称“二次制曲”,是网罗堆积场地和空气中的微生物进行堆积发酵,并生成多种酶类和多种酶类的相互作用下合成酒体的前驱物质及香味物质,为入窖发酵创造必要的条件,对酱香型白酒优良酒质的形成起着关键性作用[9-12]。细菌及其代谢产物在高温操作过程中强化了酒醅自身形成酱香的原动力,促进了酱香物质的进一步生成,研究最多的枯草芽孢杆菌(Bacillus subtilis)和地衣芽孢杆菌(Bacillus licheniformis)被认为是利用糖类和氨基酸促成“美拉德反应”而大量产生呈香物质的[13-14]。窖内发酵是堆积发酵的延续,将前者的代谢产物进行积累,利用窖产酸细菌(乳酸菌、醋酸菌等)成为主力,产酒精和二氧化碳的同时,也是酸类、酯类物质(乳酸、乙酸、乳酸乙酯、乙酸乙酯等)形成的基础[15]。石窖泥是酱香型白酒的特色发酵容器,窖泥中包含了大量的微生物,绝大部分为厌氧菌如厌氧菌乳杆菌科(Lactobacillaceae)、梭菌科(Clostridiaceae)等[16]可以为酿酒提供丰富的风味物质(如丁酸、己酸和己酸乙酯等),窖泥对白酒中微量香味成分的形成及其量比关系的协调起着重要的作用,极大地影响着白酒酿造的品质[17]。
本研究采用高通量测序技术,利用Illumina公司Miseq测序平台对酱香型白酒第四轮次酒酿造过程中高温大曲、酒醅(堆积酒醅、窖内酒醅)以及窖泥进行研究,分析细菌微生物多样性,群落结构特点、菌群组成的差异性以及各酿造阶段的主要优势菌群动态变化规律,为优化利用酿酒微生物和生产质量控制提供理论支撑。
1 材料与方法
1.1 材料与试剂
1.1.1 材料
酒曲、酒醅(堆积酒醅、窖内酒醅)、窖泥样品:采自贵州省仁怀市茅台镇H酒厂和J酒厂,酒醅采样轮次为第四轮次。酒曲的采样方式:以曲块对角线的中点作为中心抽样点,再于对角线上选择四个与中心样点距离相等的点作为取样点,将每次取样点样品直接混匀后装于采样袋,置于-80 ℃冰箱中备用。酒醅(堆积酒醅、窖内酒醅)采样方式:每层中心和四角收集,每100 g混合样品装于采样袋,置于-80 ℃冰箱中备用。窖泥采样方式:从窖池池壁中线位置距池底10 cm处的3个点及池底中心部位分别取10 g窖底泥,混合均匀后装于采样袋,置于-80 ℃冰箱中备用。共采集10个样品,具体样品采集信息见表1。
表1 样品采集信息表
Table 1 Sample collection information

注:H酒厂1号(新)车间使用年限为4年;H酒厂3号老车间使用年限为10年;J酒厂车间使用年限为1年。
1.1.2 试剂
脱氧核糖核酸(deoxyribonucleic acid,DNA)提取试剂盒:美国OMGEA公司;AxyPrepDNA凝胶回收试剂盒:美国AXYGEN公司。
1.2 仪器与设备
ABIGeneAmpR9700型聚合酶链反应(polymerase chain reaction,PCR)仪:美国ABI公司;Illumina Miseq测序平台:上海美吉生物医药科技有限公司;QuantiFlourTM-ST蓝色荧光定量系统:美国Promega公司。
1.3 方法
1.3.1 样品DNA提取及MiSeq测序分析
用E.Z.N.A. Soil DNA Ki(tD5625-01)进行样品提取。完成基因组DNA抽提后,利用1%琼脂糖凝胶电泳检测抽提的基因组DNA,用NanoDrop2000进行浓度的检测。使用细菌的16S rDNA 通用引物对样品所提取的DNA进行PCR扩增,16S rDNA V4可变区引物515F(5'-GTGCCAGCMGCCGCGGTAA-3')和806R(5'-GGACTACHVGGGTWTCTAAT-3')[18],每个样本3个重复,将同一样本的PCR产物混合后用2%琼脂糖凝胶电泳检测,使用AxyPrepDNA凝胶回收试剂盒切胶回收PCR产物,TrisHCl洗脱;2%琼脂糖电泳检测。参照电泳初步定量结果,将PCR产物用QuantiFluorTM-ST蓝色荧光定量系统进行检测定量,之后按照每个样本的测序量要求,进行相应比例的混合。再利用Illumina MiSeq进行测序分析。
Soil DNA Ki(tD5625-01)进行样品提取。完成基因组DNA抽提后,利用1%琼脂糖凝胶电泳检测抽提的基因组DNA,用NanoDrop2000进行浓度的检测。使用细菌的16S rDNA 通用引物对样品所提取的DNA进行PCR扩增,16S rDNA V4可变区引物515F(5'-GTGCCAGCMGCCGCGGTAA-3')和806R(5'-GGACTACHVGGGTWTCTAAT-3')[18],每个样本3个重复,将同一样本的PCR产物混合后用2%琼脂糖凝胶电泳检测,使用AxyPrepDNA凝胶回收试剂盒切胶回收PCR产物,TrisHCl洗脱;2%琼脂糖电泳检测。参照电泳初步定量结果,将PCR产物用QuantiFluorTM-ST蓝色荧光定量系统进行检测定量,之后按照每个样本的测序量要求,进行相应比例的混合。再利用Illumina MiSeq进行测序分析。
1.3.2 高通量测序数据统计分析
测序得到的序列信息,首先根据双端序列数据之间的对应关系,将成对的数据拼接成一条序列,同时对其质量和拼接效果进行质控过滤,根据序列首尾两端的序列和引物序列区分样品得到有效序列,并校正序列方向。之后利用不同软件进行数据分析:通过USEARCH[19]将标签分组成具有97%相似性的操作分类单元(operational taxonomic units,OTU)。基于Silva库对97%相似水平的OTU代表序列进行分类学注释分析、利用QIIME[20]等软件对样本进行Alpha和Beta多样性分析。
2 结果与分析
2.1 酱香型白酒酿造过程中的细菌多样性分析
2.1.1 Shannon-Wiener曲线分析
Shannon-Wiener[21]是反映样本中微生物多样性的指数,利用各样本的测序量在不同测序深度时的微生物多样性指数构建曲线,以此反映各样本在不同测序数量时的微生物多样性。当曲线趋向平坦时,说明测序数据量足够大,可以反映样本中绝大多数的微生物信息。10个样品细菌的Shannon-Wiener曲线见图1。
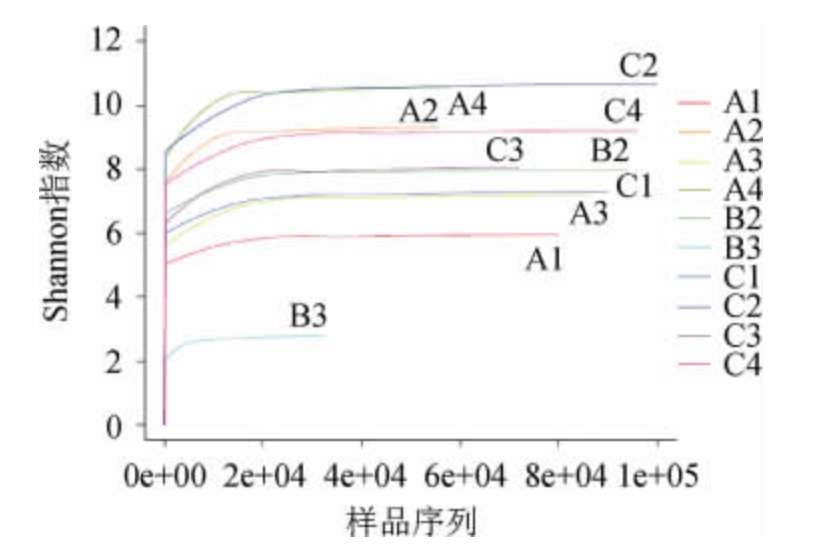
图1 10个样品细菌Shannon-Wiener曲线
Fig.1 Shannon-Wiener curve of bacteria in ten samples
由图1可知,10个样品细菌的Shannon-Wiener曲线都趋向平坦,说明测序的数据量足够大,对酒醅中微生物多样性分析基本覆盖酒醅中细菌的种类。
2.1.2 细菌群落结构分析
基于门(phylum)水平上的样品群落结构见图2。

图2 基于门水平的样品菌落组成
Fig.2 Microbial community composition of samples based on phylum level
结合图2在门(phylum)水平上分析样品群落结构组成可知,在门的水平上10个样品共检测到16种类群,其中最重要的细菌是厚壁菌门(Firmicutes),在10个样品间的平均占有百分比达35.1%,平均占有比例最高,其次是变形菌门(Proteobacteria)、拟杆菌门(Bacteroidetes)、放线菌门(Actinobacteria)等。
基于属(genus)水平上的样品群落结构见图3。
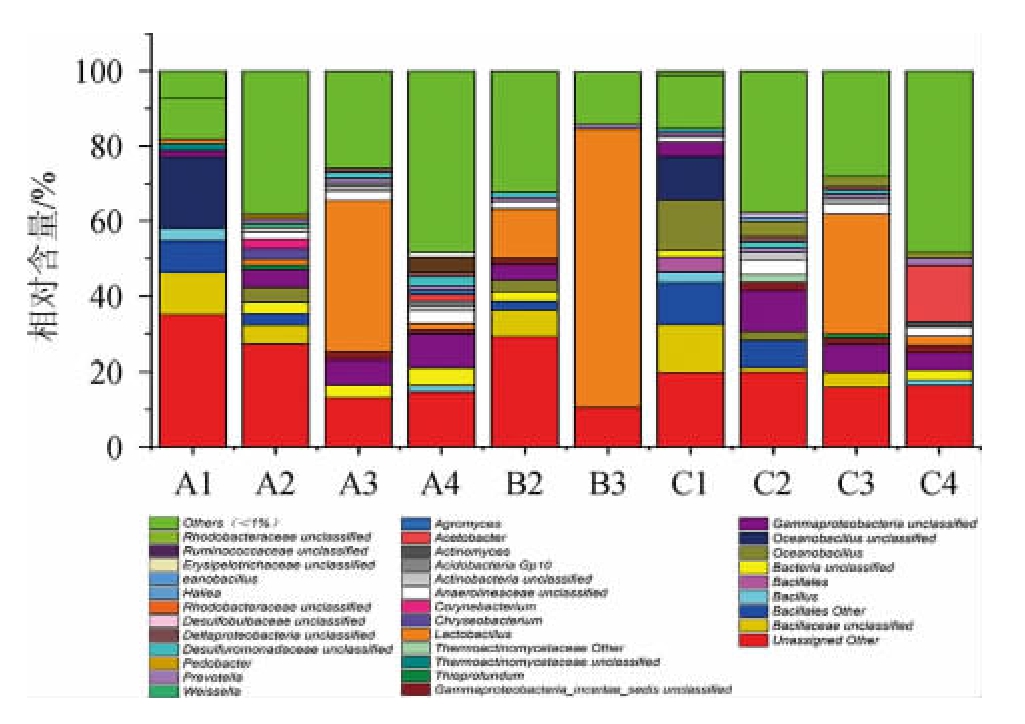
图3 基于属水平的样品菌落组成
Fig.3 Microbial community composition of samples based on genus level
由图3可知,在10个样品中主要存在34个细菌菌属,在酒曲中主要是未定义微生物菌群、芽孢杆菌科的未定义菌属、海洋芽胞杆菌属(Oceanobacillus)和芽孢杆菌目的其他菌群、堆积发酵中主要优势菌有未定义微生物菌群、芽孢杆菌科的未定义菌属、变形菌纲的未定义菌属、乳杆菌属(Lactobacillus)和金黄杆菌属(Chryseobacterium)、乳杆菌属(Lactobacillus)和普氏菌属(Prevotella);在窖内发酵中乳杆菌属(Lactobacillus)占绝对优势;窖泥中主要优势菌有未定义微生物菌群、放线菌(Actinomyces)、乳杆菌属(Lactobacillus)和醋菌属(Acetobacter)。
2.1.3 多样本聚类树
基于样本OTU即操作分类单元种类数计算样本间的weighted unifrac距离矩阵,综合考量序列数据、序列同源性、微生物构成的丰度等对各个样本进行聚类。聚类树枝间的距离反应了各样品间的微生物组成间的亲缘关系。样本相似度分析结果见图4。由图4可知,样品A1和C1归属到了一起,说明了不同酒厂A1和C1的细菌组成亲缘关系较近,说明不同酒厂间的成品曲细菌群组成相似,这可能是各酒厂之间制曲配方及工艺的相似所致。
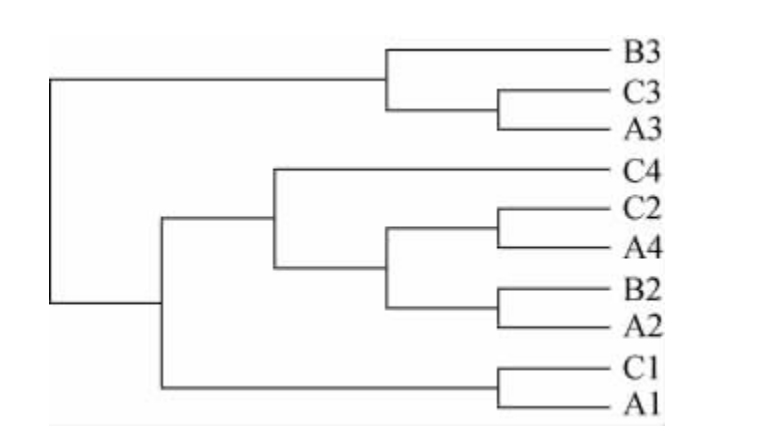
图4 样本相似度分析树状图
Fig.4 Dendrogram of sample similarity
样品A2和B2归属到了一起,但与样品C2相隔一定距离。说明同一酒厂不论是新车间还是老车间它们堆积发酵酒醅的细菌组成亲缘关系很接近,而不同酒厂堆积发酵酒醅细菌组成亲缘关系较远。不同酒厂虽然都在茅台镇赤水河边,但酒厂间地理位置有差异,发酵车间环境微生物以及来自大曲的微生物亲缘关系有较大差异。同时也说明了堆积发酵与发酵车间环境关联度较高,发酵环境微生物的多样性影响着堆积酒醅的微生物多样性。
样品A3、B3、C3都归属到了一起,说明不同酒厂以及同一酒厂新老车间窖内酒醅细菌群组成相似,其中样品A3和C3亲缘关系相对较近。依据聚类树枝最先分枝最进化的原理,从图4亦可知,样品B3中的微生物进化优先于A3和C3中的微生物,说明在窖内酿造环境中,老窖池的微生物优势菌群最突出。样品A4和C4没有归于同一归属并且与酒醅相距较远,说明不同酒厂窖泥相互之间细菌菌相组成差别较大,这可能是窖泥的来源不同所致。
2.2 酿酒过程优势细菌群落动态变化分析
2.2.1 酒曲细菌多样性分析
由图5可知,H酒厂和J酒厂成品酒曲主要优势菌群组成相似,但细菌主要优势菌群的丰度存在明显差异。未定义微生物菌群、芽孢杆菌科(Bacillaceae)、海洋芽胞杆菌属(Oceanobacillus)和芽孢杆菌目的其他菌群是H酒厂和J酒厂的成品曲中主要的优势菌群,在样品A1和C1中占有丰度分别是35.06%和20.86%,19.14%和13.56%,11.13%和15.28%,8.53%和11.12%。说明芽胞杆菌是高温大曲中的主要微生物菌群。芽孢杆菌具有耐高温的特性,能够分泌蛋白酶、淀粉酶等水解原料,产生发酵性糖和氨基酸,为发酵酿酒过程中美拉德反应初期阶段生成阿马多利化合物提供大量原料。

图5 不同车间不同酒厂酒曲样品细菌群落结构饼状图
Fig.5 Pie chart of bacterial community structure of different distilleries samples in different workshops
2.2.2 堆积酒醅细菌多样性分析
堆积发酵是将淀粉糖化的关键环节,这时需要更多的微生物代谢产生大量的水解酶类。由于高温堆积阶段要求温度达50 ℃左右[25],所以耐热微生物成为了优势菌群。堆积处于开放式的好氧、兼氧环境条件下,再次网罗空气中有益微生物,进一步为产生酱香物质和积累酱香前体物质创造条件,它是生成酱香物质的接力站,直接关系到产品的风格质量。
结合图6对比分析可得,A2中共发现450个菌属,在0.1%以上的菌属111个,其中样品A2中的优势微生物菌群为:未定义微生物菌群27.24%、芽孢杆菌科的未定义菌属5.03%、变形菌纲的未定义菌属4.74%、海洋芽胞杆菌属(Oceanobacillus)3.82%、芽孢杆菌目的其他菌群3.01%、金黄杆菌属(Chryseobacterium)2.80%、棒状杆菌属(Corynebacterium)2.48%、乳杆菌属(Lactobacillus)1.61%、魏斯氏菌属(Weissella)1.18%、普氏菌属(Prevotella)1.15%、地杆菌属(Pedobacter)1.05%等。
样品B2中共发现404个菌属,在0.1%以上的菌属93个,仍以未定义微生物菌群27.24%、乳杆菌属(Lactobacillus)13.22%、芽孢杆菌科的未定义菌属5.03%、γ-变形菌纲的未定义菌属4.74%、海洋芽胞杆菌属(Oceanobacillus)3.82%、芽孢杆菌目的其他菌群3.01%、普氏菌属(Prevotella)1.15%为主要优势菌群。样品A2和B2相比主要优势菌群组成相似,但丰度差异较大,这说明堆积发酵与发酵车间环境有关,同一酒厂,同样的制酒工艺和酒曲,发酵车间使用年限长的比新车间微生物多样性降低,但优势菌群越突出。

图6 不同车间不同酒厂堆积酒醅细菌群落结构饼状图
Fig.6 Pie chart of bacterial community structure of the fermented grain piled-up on the ground of different distilleries in different workshops
J酒厂的堆积酒醅C2中共发现437个菌属,在0.1%以上的菌属有108个。其中未定义微生物菌群27.24%、γ-变形菌纲的未定义菌属4.74%、芽孢杆菌科的未定义菌属5.03%、海洋芽胞杆菌属(Oceanobacillus)3.82%等为主要优势菌属。样品A2与C2相比主要优势菌群组成相似,但丰度和占比有较大差异。这说明不同的酒厂发酵车间之间环境微生物多样性的差异影响着堆积酒醅的微生物多样性。另外,不同酒厂的发酵工艺虽然相似,但发酵物料物态、其工艺参数及地理位置也会造成优势菌群差异。
2.2.3 窖内酒醅细菌多样性分析
样品A3中共发现400个菌属,在0.1%以上的菌属有77个,样品B3中共发现292个菌属,在0.1%以上的菌属有31个,样品C3中共发现404个菌属,在0.1%以上的菌属有83个。结合图7对比分析可知,3种窖内酒醅细菌菌群多样性均较高,主要优势细菌群组成相似结构相似,但丰度差异较大。乳杆菌属(Lactobacillus)在窖内发酵中占绝对优势,在样品A3、B3和C3中占有丰度分别达到40.37%、74.00%和32.03%。窖内发酵过程中是处于高温厌氧环境,乳酸菌的乳杆菌属是酱香型白酒窖内发酵过程中最常见的重要微生物菌群。乳杆菌属(Lactobacillus)的作用是通过代谢产生乳酸等有机酸。酸类也是主体呈味物质,起缓冲协调作用,是决定酒的香型、风格的基础要素之一。与样品B3相比,样品A3的细菌多样性较高,但样品B3的优势菌群更突出。说明同一酒厂,同样的制酒工艺和酒曲,不同使用年龄的车间和窖龄的微生物多样性的差异影响着窖内发酵酒醅细菌多样性。老窖池中细菌菌落多样性同样较高,并且主要优势细菌的种类和丰度与堆积发酵酒醅相关度高。这说明使用时间长的车间和窖池其环境微生物种群结构更稳定,优势菌群更突出。样品C3与A3相比,主要优势菌群相似,但丰度存在明显差异。说明不同的酒厂发酵车间使用年限和窖龄影响着窖内酒醅的微生物多样性。
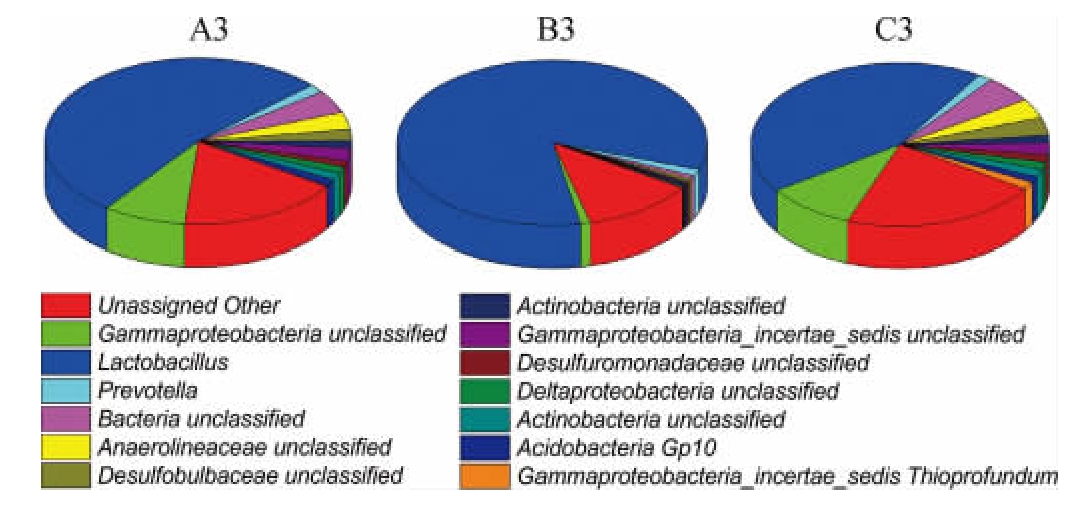
图7 不同车间不同酒厂窖内酒醅细菌群落结构饼状图
Fig.7 Pie chart of bacterial community structure of the fermented grain in pit of different distilleries in different workshops
2.2.4 窖泥细菌多样性分析
样品A4中共发现525个菌属,在0.1%以上的菌属有143个,C4样品中共发现482个菌属,在0.1%以上的菌属有114个。
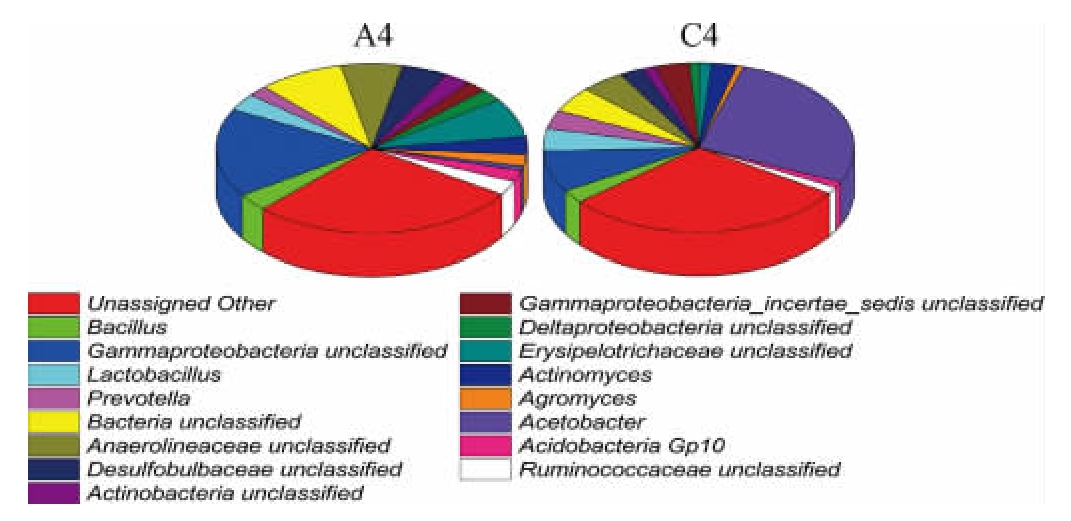
图8 不同酒厂窖泥细菌群落结构饼状图
Fig.8 Pie chart of bacterial community structure in pit mud in different distilleries
由图8可知,不同酒厂使用的窖泥中细菌组成差异明显。A4中的主要优势菌群是放线菌(Actinomyces)1.79%、乳杆菌属(Lactobacillus)1.70%。C4的主要优势菌群是醋杆菌属(Acetobacter)14.98%、乳杆菌属(Lactobacillus)2.35%,与样品A4对比主要优势菌群存在明显差异。这可能与不同酒厂的窖泥来源和使用时间、使用方法不同有关。
3 结论
本研究应用Illumina MiSeq高通量测序技术,探究酱香型白酒第四轮次酒酿造过程中细菌多样性,阐明第四轮次酒酿造过程中优势细菌微生物群落结构及其随酿造工艺动态变化。结果表明,酱香型白酒的高温大曲、堆积发酵和窖池发酵的细菌资源丰富且主要优势菌群存在动态变化。在酒曲中主要优势菌有:芽孢杆菌科的未定义菌属、海洋芽胞杆菌属(Oceanobacillus);堆积发酵中主要优势菌有芽孢杆菌科的未定义菌属、γ-变形菌纲的未定义菌属;在窖内发酵中乳杆菌属(Lactobacillus)占绝对优势;窖泥中主要优势菌有放线菌(Actinomyces)、乳杆菌属(Lactobacillus)和醋杆菌属(Acetobacter)。同一酒厂新老车间酒曲、堆积发酵、窖内发酵、窖泥之间细菌组成相似度较高,不同酒厂酒曲、堆积发酵、窖内发酵、窖泥之间细菌组成相似度亦较高,但其优势菌群丰度差异显著。发酵车间使用年限及窖龄影响着微生物多样性;时间长的车间和窖池其环境微生物种群结构更稳定,优势菌群更突出。
[1]李欣,王彦华,林静怡,等.高通量测序技术分析酱香型白酒酒醅的微生物多样性[J].福建师范大学学报(自然科学版),2017,33(1):51-59.
[2]沈才萍,李德林,敖宗华,等.酒醅微生物的分子生物学研究[J].技术与市场,2013,20(6):40-42.
[3]WU Q,CHEN L,XU Y.Yeast community associated with the solid state fermentation of traditional Chinese Maotai-flavor liquor[J].Int J Food Microbiol,2013,166(2):323-330.
[4]连宾.微生物在酱香型白酒香味物质形成中的作用[J].中国酿造,1997,16(1):13-14.
[5]吴徐建.酱香型白酒固态发酵过程中酵母与细菌群落结构变化规律的研究[D].无锡:江南大学,2013:10.
[6]崔利.形成酱香型酒风格质量的关键工艺是“四高两长,一大一多”[J].酿酒,2007,34(3):24-35.
[7]崔利,杨大金.提高大曲酱香型酒风格质量几个关键环节的探讨[J].酿酒,1988(1):2.
[8]姚粟,葛媛媛,李辉,等.利用非培养技术研究芝麻香型白酒高温大曲的细菌群落多样[J].食品与发酵工业,2012,38(6):1-6.
[9]庄名扬,孙达孟.酱香型白酒高温堆积糟醅中酵母菌分离、选育及其分类学鉴定[J].酿酒,2003,30(2):12-13.
[10]韩兴林,尚柯,王德良,等.酱香白酒堆积发酵过程中代谢风味生成规律的分析[J].酿酒,2018,45(4):43-50.
[11]戴栾杰,李宗军,田志强,等.酱香型白酒的轮次酒以及“二次制曲”过程中的风味物质分析[J].食品研究与开发,2019,40(19):24-32.
[12]张守财.堆积发酵对酱香型白酒风味形成的作用及其控制[J].福建轻纺,2006(11):17-18.
[13]赵兴秀,何义国,赵长青,等.产酱香功能菌的筛选及其风味物质研究[J].食品工业科技,2016,37(6):196-200.
[14]戴奕杰,李宗军,田志强.酱香型白酒大曲和糟醅的细菌多样性分析[J].食品科学,2019,40(4):152-159.
[15]杨国华,邱树毅,黄永光.酱香白酒生产中产香微生物研究[J].中国酿造,2011,30(4):24-27.
[16]王莉,王亚玉,王和玉,等.酱香型白酒窖底泥微生物组成分析[J].酿酒科技,2015(1):12-15.
[17]TAO Y,LI J,RUI J,et al.Prokaryotic communities in pit mud from different-aged cellars used for the production of Chinese strong flavored liquor[J].Appl Environ Microbiol,2014,80(7):2254-2260.
[18]TOJU H,TANABE A S,YAMMAMOTO S,et al.High-coverage its primers for the DNA-based identification of Ascomycetes and Basidiomycetes in environmental samples[J].PloS One,2012,7(7):e40863-e4084.
[19]EDGAR R C.UPARSE:Highly accurate OTU sequences from microbial amplicon reads[J].Nat Meth,2013,10(10):996-998.
[20]CAPORASO J G,KUCZYNSKI J,STOMBAUGHJ,et al.QIIME allows analysis of high-throughput community sequencing data[J].Nat Meth,2010,7(5):335-336.
[21]WAN Y,SHENG H F,HE Y,et al.Comparison of the levels of bacterial diversity in freshwater,intertidal wetland,and marine sediments by using millions of Illumina tags[J].Appl Environ Microbiol,2012,78(23):8264-8271.