中国白酒是世界六大蒸馏酒之一,是我国特有的一种经固态发酵而得的蒸馏酒,其独特的生产工艺造就了别具一格的产品风格[1]。大曲作为白酒固态酿造的糖化剂、发酵剂以及生香剂,是白酒固态酿造的物质基础。大曲按照其生产产品的特点,分为浓香大曲、清香大曲、酱香大曲、兼香大曲等。传统白酒大曲中主要有三系(物系、菌系和酶系),其中菌系主要由细菌、霉菌、酵母以及放线菌等微生物组成。大曲中微生物主要来源于生产环境,包括空气、制曲用水、原料、器具以及曲房环境等。传统制曲的实质是与酿酒相关微生物的扩大培养过程,即将来源于环境中的野生菌种进行自然培养,并通过翻曲和通风排潮等人为干预的方式,淘汰杂菌选育出有益菌种的过程。由于试验方法和条件的落后及局限,初期对大曲中各种微生物的功能及群落结构多样性研究时主要采用传统可培养技术。近年来,随着具有高可信度、高分辨力及快速灵敏等优点的现代分子生物学方法在研究微生物群落结构上的快速发展[2],给传统的微生物可培养技术及微生物分类鉴定等方面带来了巨大的革新,对传统可培养方法的缺陷起到了一定的弥补作用[3],因此现代分子生物学方法逐渐被引入到大曲微生物的研究当中。
虽然真菌在大曲发酵体系中具有极其重要的作用,但是相对于细菌的研究,对于真菌的研究就相对较少。基于此,该文主要阐述大曲中真菌群落结构多样性的研究技术以及研究进展,以期为研究大曲发酵过程中真菌多样性构成及变化规律,解析各种真菌的功能及相互作用,提高大曲质量以及形成对传统大曲固态发酵机理的最新认识奠定理论基础。
1 微生物群落多样性研究技术
21世纪以来,微生物群落结构及多样性研究已经成为微生物生态学以及环境科学的研究重点和热点[4],全面剖析目标样品中微生物群落结构及多样性,为揭示微生物具体代谢功能、控制和优化群落结构、强化目标微生物的代谢功能等提供理论依据[5-6]。微生物群落结构及多样性解析技术的发展主要经历了传统可培养技术、生理学指纹法及生物标记物方法、结合聚合酶链式反应(polymerase chain reaction,PCR)技术的传统分子生态学技术、高通量测序技术等四个阶段。随着微生物生态学的发展及研究技术的不断升级,中国白酒酿造微生物的群落多样性的研究手段也由传统可培养技术逐渐升级为下一代测序技术。
1.1 传统可培养技术
可培养分离技术是最早应用于研究微生物群落结构及多样性的重要手段[4]。该技术是将样品或样品稀释液接种于固体培养基中,在一定条件下培养后对培养基上生长的菌落进行统计,并通过其菌落形态、微观结构以及生理生化等特性进行微生物种属鉴定。由于该方法忽略了来源环境或发酵体系中的养分与人工培养基的差异以及不同微生物之间的相互作用,这就直接导致样品中许多可培养微生物的种类和数量大大减少。AMANN R I等[7]研究指出,自然环境中的微生物仅10%能够被人工培养分离,同时传统可培养技术操作繁琐、准确性较差,不能满足监测群落结构动态变化的需求。因此,传统可培养技术在微生物群落结构及多样性研究中使用得越来越少[8]。
尽管传统可培养技术具有上述不可避免的局限性,但是该技术到现在仍在被广泛使用,原因之一便是能够得到从样品中得到活菌,便于后续研究。研究人员通过可培养方法逐步揭示了大曲中微生物优势菌群的种属分布,在一定程度上促进了制曲技术的快速发展[9]。传统微生物分类鉴定方法(可培养技术)应用于研究大曲(清香型、浓香型和酱香型)中真菌群落结构多样性的情况见表1。
表1 可培养技术在大曲真菌研究中的应用情况
Table 1 Application of culture-dependent technology in fungi study of Daqu

1.2 现代分子生物学技术
1.2.1 磷脂脂肪酸技术
磷脂脂肪酸(phospholipid fatty acid,PLFA)指纹图谱技术已经广泛应用于研究复杂体系的微生物群落及动态变化规律[2]。PLFA是除古生菌以外其他所有微生物活细胞细胞膜的主要成分,并且环境中PLFA含量与目标环境中活性微生物生物量具有很好的相关性[18]。由于不同微生物种属的PFLA结构及种类存在一定差异,因此可以作为微生物标记物,用于研究微生物的群落结构,同时该技术也用于微生物菌株的辅助鉴定[19]。
PLFA指纹图谱技术应用于研究大曲(清香型、浓香型和酱香型)中真菌群落和多样性的具体情况见表2。
表2 磷脂脂肪酸技术在大曲真菌研究中的应用情况
Table 2 Application of phospholipid fatty acid technology in fungi study of Daqu

1.2.2 聚合酶链式反应-变性梯度凝胶电泳
表3 PCR-变性梯度凝胶电泳技术在大曲真菌研究中应用情况
Table 3 Application of PCR-denaturing gradient gel electrophoresis technology in fungi study of Daqu

聚合酶链式反应-变性梯度凝胶电泳(polymerase chain reaction-denatured gradient gel electrophoresis,PCR-DGGE)技术是一种经典的脱氧核糖核酸(deoxyribonucleic acid,DNA)指纹图谱技术,是用于研究环境微生物群落结构的重要手段[19]。通过比较DNA指纹图谱中的条带(数目及亮度)能够剖析样品中微生物的群落组成以及多样性,若对图谱中的条带进行测序分析就能进一步获得样品中微生物的系统发育地位[27]。PCR-DGGE技术已经广泛用于环境及发酵体系中微生物群落组成、多样性及动态变化的研究。与其他方法相比,PCR-DGGE是目前国内用于白酒酿造环境中微生物群落组成及多样性研究的最常用手段。PCR-DGGE技术应用于大曲真菌群落和多样性研究情况见表3。
1.2.3 聚合酶链式反应-单链构象多态性技术
聚合酶链式反应-单链构象多态性技术(polymerase chain reaction-single chain conformation polymorphism technique,PCR-SSCP)技术与PCR-DGGE技术相似,但是原理有明显的差异。SSCP技术是根据各类碱基组成的单链DNA形成的不同三维构象进行区分,当不同三维构象的单链DNA分子在非变性聚丙烯酰胺凝胶电泳(polyacrylamide gel electrophoresis,PAGE)中因迁移率不同而形成将DNA条带进行分离,从而将长度相同但DNA序列不同的片段分离开来[2]。由于样品中不同微生物种属的rDNA基因序列碱基组成存在差异,通过对PAGE上各DNA条带测序后进行分析,就能进行样品中微生物系统发育的研究[19]。因此,该方法也常用于大曲样品中微生物群落多样性的剖析。PCR-SSCP技术应用于大曲真菌群落和多样性研究情况见表4。
表4 PCR-单链构象多态性技术在大曲真菌研究中应用情况
Table 4 Application of PCR-single-strand conformation polymorphism technology in fungi study of Daqu

1.2.4 基因文库
基因克隆文库技术可剖析目标样品中各类微生物的种类及对应比例,通过该技术能够检测出不能人工分离培养的微生物。通过建立目标样品中微生物的基因文库,再测定文库中的基因序列,从而可以得到样品中所含微生物基因的种类和相对数量[2]。目前基因克隆文库技术已经广泛应用于环境微生物学及生态学的相关研究,其中最为常用的是核糖体RNA基因克隆文库的构建,包括细菌16SrDNA基因克隆文库、真菌18S rDNA基因克隆文库以及真菌ITS基因克隆文库[2]。基因克隆文库技术能准确获得目标样品中微生物的基因序列信息,从而可以检测出样品中微生物群落中的优势菌群。该技术已广泛应用多种环境及发酵体系中微生物群落组成、多样性和动态变化规律及平行样品间微生物群落差异性比价研究。基因克隆文库技术应用于大曲真菌群落和多样性研究情况见表5。
表5 基因克隆文库技术在大曲真菌研究中应用情况
Table 5 Application of gene clone library technology in fungi study of Daqu

1.2.5 高通量测序技术
高通量测序(high-throughput sequencing,HTS)技术,又称为“下一代”测序(next-generation sequencing technology,NGS)技术,是新一代的基因测序技术。该技术一次最多能并行测定几百万条DNA分子序列,并且读长较短,能够深入、细致、全貌的分析一个物种的转录组和基因组。目前常用的二代测序平台主要有Solexa、SOLiD和454等3种平台。由于高通量测序的各种优点,其越来越多的被用于研究大曲、窖泥中微生物群落结构多样性。高通量测序技术应用于大曲真菌群落和多样性研究情况见表6。
表6 高通量测序技术技术在大曲真菌研究中应用情况
Table 6 Application of high-throughput sequencing technology in fungi study of Daqu
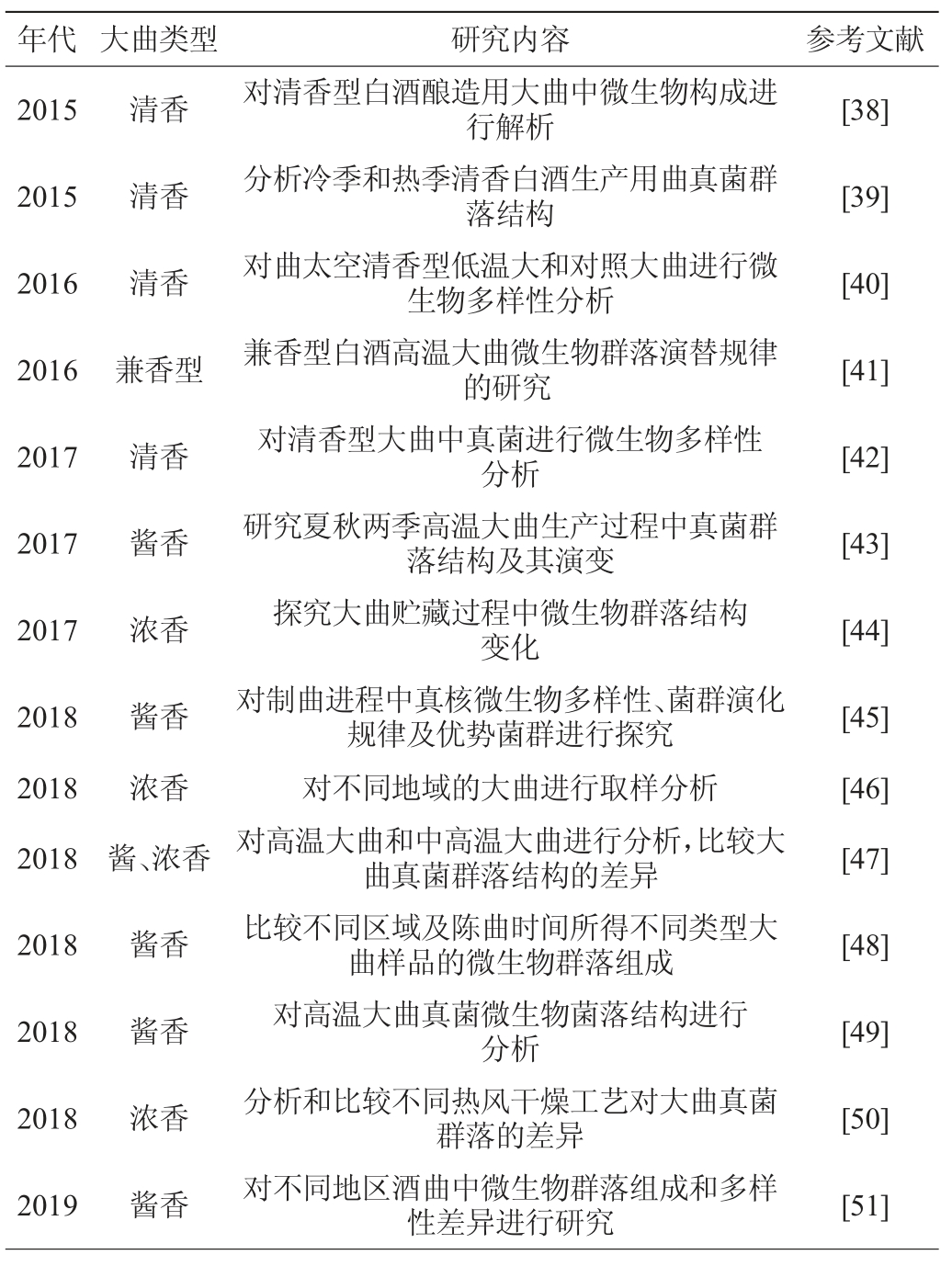
续表

1.2.6 组学技术
组学(Omics)是一项具有广阔应用前景的新兴技术,按照分析目标不同分为基因组学、转录组学、蛋白质组学和代谢组学等[54]。目前应用于复杂环境中微生物多样性研究较多且较为成熟的技术是宏基因组学技术。它是HANDELSMAN J等[55]于1998年首次提出,将宏基因组定义为环境中全部微小生物遗传物质的总和[56]。随着高通量测序技术的不断发展,宏基因组学被广泛应用于自然环境中微生物群落结构(如水、空气、土壤、海洋等)、发酵食品微生物群落结构(奶酪、浆水菜、普洱茶、泡菜等)以及肠道微生物群落结构(人体及其他动物)的研究。宏基因组学技术应用于食品微生物群落和多样性研究情况见表7。
表7 宏基因组学技术在发酵食品微生物多样性研究中的应用
Table 7 Application of metagenomic technology in microbial diversity study of fermented food
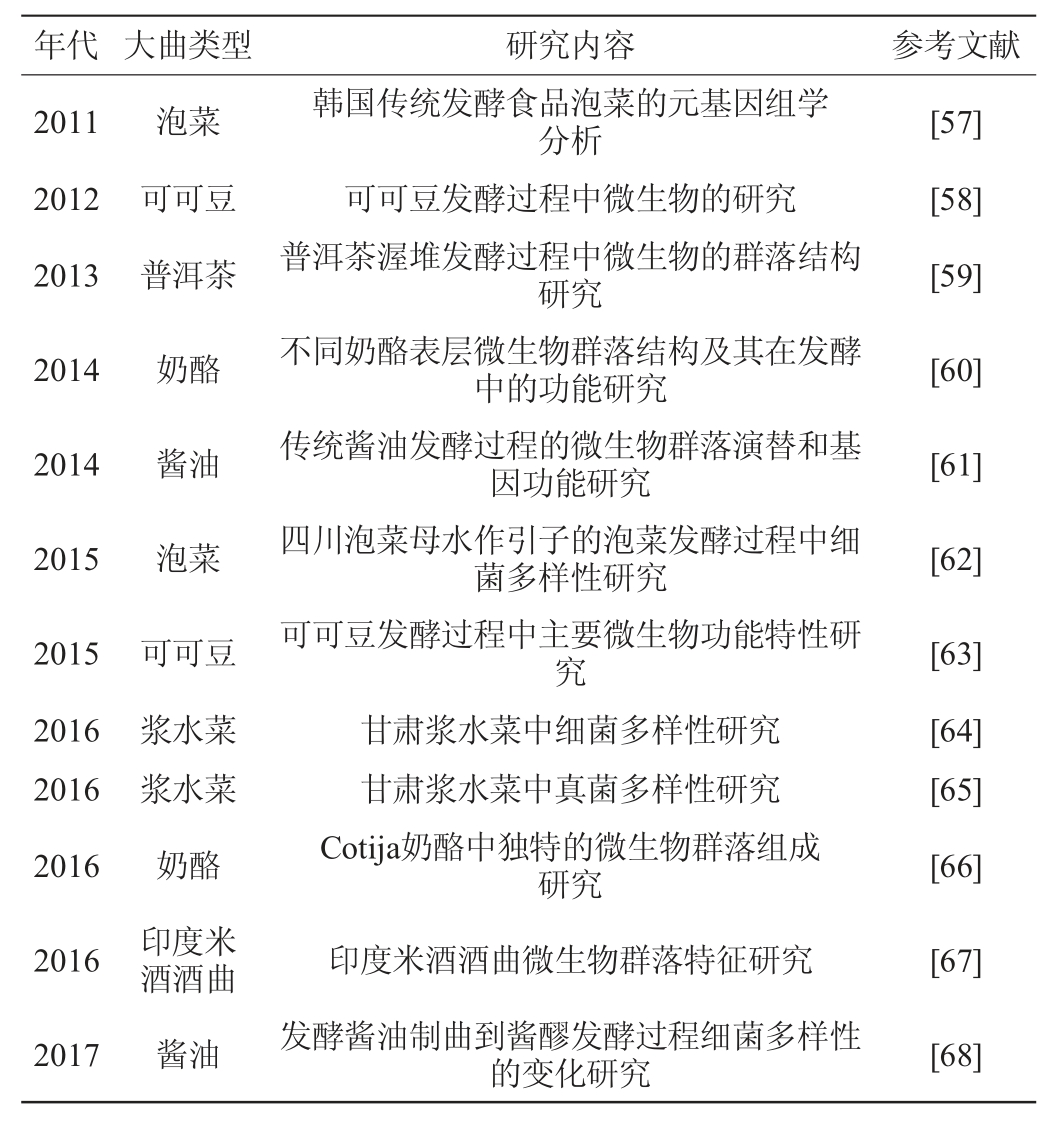
目前,宏基因组学在大曲微生物的研究中应用较少,在此简要介绍了近年来宏基因组学技术在一些发酵食品中的应用情况,以期为利用宏基因组学分析大曲微生物群落结构多样性以及发现性新的功能基因提供一定的参考依据。
1.2.7 微生物群落多样性的研究手段优缺点比较
随着各种微生物分离技术的不断发展和进步,能被分离出来的微生物越来越多,但在实验室条件下分离出来的微生物种类仍不足环境微生物的10%[7]。同时,虽然各种微生物群落和多样性的研究方法也在不断发展,但是每种方法都有其优缺点,因此对微生物群落和多样性的研究不能局限某一研究方法上,必须结合现代分子生物学的各种方法,包括传统可培养分离技术,这样才能对可培养微生物和非可培养微生物有一个系统的认识。各种微生物群落和多样性研究方法的优缺点比较见表8。
表8 微生物群落结构多样性研究方法的比较
Table 8 Comparison of research technology on microbial community structure diversity

2 大曲中真菌多样性
大曲中的主要微生物种类是霉菌、酵母菌、细菌和放线菌四大类。大曲发酵体系中的微生物菌系不断发生着复杂的动态变化,其中霉菌是丰度最高的微生物类群,其次是酵母和细菌[17]。大曲中霉菌的特殊作用(如为酿造提供糖化力、液化力、蛋白质分解能力以及多种有机酸等物质)决定着大曲各方面的质量特性,霉菌菌群的功能分化,对大曲的质量起着关键的作用[69]。酵母菌为大曲提供发酵力,作为大曲微生物区系中最重要的类群之一,不仅主导了酒精的产生,而且大量产生酯类、醇类、酮类、烯类、酚类等挥发性化合物,对大曲和大曲酒的品质有着决定性的影响[70]。
不同类型大曲中分离出的霉菌和酵母菌种类见表9。从表9中可知,各类大曲中霉菌和酵母菌多样性差异较大,且浓香型大曲>清香大曲>酱香大曲,同时大曲中霉菌多样性少于酵母菌[58]。各类大曲中均有的真菌主要有根霉属、曲霉属、毛霉属、横梗霉属、嗜热真菌属以及对大曲及大曲酒酿造有害的青霉属等;三类大曲中共同的酵母菌主要有酵母属、毕赤酵母属、假丝酵母属、汉逊酵母属、复膜孢酵母属、伊萨酵母属等。
表9 不同类型大曲中霉菌和酵母菌对比
Table 9 Comparison of mold and yeast in different types of Daqu

3 展望
近年来,随着各种现代分子生物学新技术的不断完善与发展,对大曲中真菌群落结构的研究不再受传统微生物纯培养技术的限制,同时使用这些新兴的高新技术可以从不同的维度来研究大曲中真菌群落结构的多样性,为大曲中真菌群落结构及多样性解析奠定了坚实的基础。需要特别指出的是,由于各种方法均具有一定的局限性,因此为了获得更为全面地解析大曲中真菌群落结构多样性的信息,在研究过程中应当根据研究目的,选择合适的一种或多种方法结合起来使用,形成优势互补,才能更有效地挖掘和利用大曲中真菌多样性信息。目前对于大曲中真菌群落结构多样性的研究是多方面的,通过对其多样性的研究必能为大曲质量以及曲酒质量的提升提供理论基础。
[1]傅国城.中国白酒科技创新步入分子生物学时代[J].酿酒,2017,44(2):9-12.
[2]周庆伍,曹润洁,何宏魁,等.现代分子生物学技术对白酒酿造微生物的研究进展[J].酿酒科技,2017(6):95-102.
[3]王晓丹,谢晓莉,胡宝东,等.现代微生物分类鉴定技术在白酒酿造中的应用[J].中国酿造,2015,34(7):5-9.
[4]车玉伶,王慧,胡洪营,等.微生物群落结构和多样性解析技术研究进展[J].生态环境,2005(1):127-133.
[5]SRADNICK A,MURUGAN R,OLTMANNS M,et al.Changes in functional diversity of the soil microbial community in a heterogeneous sandy soil after long-term fertilization with cattle manure and mineral fertilizer[J].Appl Soil Ecol,2013,63(1):23-28.
[6]LENNON J T,AANDERUD Z T,LEHMKUHL B,et al.Mapping the niche space of soil microorganisms using taxonomy and traits[J].Ecology,2012,93(8):1867-1879.
[7]AMANN R I,LUDWIG W,SCHLEIFER K H.Phylogenetic identification and in situ detection of individual microbial cells without cultivation[J].Microbiol Rev,1995,59(1):143-169.
[8]胡婵娟,刘国华,吴雅琼.土壤微生物生物量及多样性测定方法评述[J].生态环境学报,2011,20(6-7):1161-1167.
[9]杨代永,范光先,汪地强,等.高温大曲中的微生物研究[J].酿酒科技,2007(5):37-41.
[10]李健容,蔡爱群.民间传统酒曲主要微生物的分离及鉴定[J].酿酒科技,2007(5):111-115.
[11]惠丰立,褚学英,冯金荣,等.大曲中可培养霉菌多样性的分子分析[J].食品与生物技术学报,2007,26(2):76-79.
[12]明红梅,董瑞丽,许德富,等.浓香型大曲中优势菌的分离及初步鉴定[J].酿酒科技,2013(12):57-60.
[13]李绍亮,李学思,侯小歌,等.宋河酒曲中主要霉菌的鉴定及其产酶特性的研究[J].酿酒,2016,43(6):24-29.
[14]黄晓宁,黄晶晶,李兆杰,等.浓香型和酱香型大曲微生物多样性分析[J].中国酿造,2016,35(9):33-37.
[15]樊建辉,侯建光,郭福祥,等.仰韶陶融型白酒大曲可培养微生物多样性研究[J].中国酿造,2017,36(7):71-75.
[16]朱婷婷.牛栏山大曲可培养微生物多样性分析[J].酿酒科技,2018(5):75-79.
[17]向慧平,林宜锦,关统伟,等.四川浓香型大曲生产中酵母菌、芽孢杆菌与工艺指标的关联性分析[J].食品科学,2020,41(2):196-201.
[18]FROSTEGÅRD Å,TUNLID A,BÅÅTH E.Use and misuse of PLFA measurements in soils[J].Soil Biol Biochem,2011,43(8):1621-1625.
[19]胡晓龙.浓香型白酒窖泥中梭菌群落多样性与窖泥质量关联性研究[D].无锡:江南大学,2015.
[20]秦臻,郑佳,彭昱雯,等.生物标记法剖析传统酿造用大曲微生物群落结构[J].食品科学,2011,32(11):165-170.
[21]赵金松,郑佳,吴重德,等.基于磷脂脂肪酸技术研究酱香大曲微生物群落结构[J].应用与环境生物学报,2014,20(4):558-563.
[22]ZHANG H X,ZHANG X H,ZHAO J L,et al.Analysis of the microbial communities of three kinds of Fen-Daqu by PLFAs[J].J I Brewing,2016,122:34-41.
[23]曹宇,翟磊,信春晖,等.芝麻香型白酒高温大曲制曲过程中微生物群落结构特征的磷脂脂肪酸(PLFA)分析[J].酿酒科技,2016(3):33-36.
[24]赵金松,朱开宪,邓波,等.大曲挥发性组分与其微生物群落相关性的分析[J].基因组学与应用生物学,2017,36(2):702-706.
[25]赵金松,郑佳,沈才洪,等.基于磷脂脂肪酸分析技术的大曲微生物群落结构多样性研究[J].食品工业科技,2017,38(1):160-164.
[26]赵金松,张良,孙啸涛,等.酱香型大曲微生物群落特征与理化指标的相关性研究[J].基因组学与应用生物学,2019,38(1):199-204.
[27]王海燕.PCR-DGGE 技术对清香型汾酒微生物群落结构演变规律的研究[D].无锡:江南大学,2014.
[28]叶光斌,李丹宇,杨跃寰,等.PCR-DGGE 解析浓香型大曲发酵、储藏过程真菌群落的演替规律[J].四川理工学院学报(自然科学版),2013,26(4):5-9.
[29]徐占成,唐清兰,刘孟华,等.剑南春大曲曲药真菌群落结构的分析[J].酿酒,2015,42(4):13-16.
[30]唐贤华,王思思,隋明,等.不同原料大曲中微生物的群落结构研究[J].食品科技,2019,44(3):21-26.
[31]罗惠波,黄治国,李浩,等.浓香型大曲真核微生物群落的PCR-SSCP解析[J].中国酿造,2009,28(8):42-45.
[32]周森,李艳敏,胡佳音,等.清香型大曲酿酒酵母株系分类及其可挥发代谢产物分析[J].中国酿造,2017,36(9):137-141.
[33]窦晓,杨建刚,曹新志,等.基于26S rRNA D1/D2 区序列对不同时期大曲中酵母菌的分离与鉴定[J].现代食品科技,2018,34(2):97-101.
[34]罗惠波,王彩虹,甄攀,等.基于ITS 基因文库法研究清香型大曲真菌群落结构[J].酿酒科技,2014(5):31-35.
[35]王彩虹.基于克隆文库法研究不同香型大曲微生物群落结构[D].自贡:四川理工学院,2014.
[36]王彩虹,罗惠波,卓毓崇,等.基于ITS 基因文库法研究中高温大曲和超高温大曲真菌群落结构[J].食品科技,2014,39(7):41-46.
[37]苏畅,窦晓,叶新,马莹莹,等.基于ITS4/5 rRNA 区序列对不同时期大曲中霉菌的分离与鉴定[J].现代食品科技,2018,34(3):54-58.
[38]雷振河.采用高通量测序技术分析清香型白酒酿造微生物[J].食品与发酵工业,2015,41(9):164-167.
[39]乔晓梅,赵景龙,杜小威,等.高通量测序法对清香大曲真菌群落结构的分析[J].酿酒科技,2015(4):28-31.
[40]周森,胡佳音,赵卫鹏,等.高通量测序技术在航天大曲微生物多样性中的应用[J].酿酒科技,2016(9):76-78.
[41]李申奥.兼香型白酒高温大曲微生物群落演替规律的研究[D].武汉:华中农业大学,2016.
[42]郭敏,黄永光,邱树毅,等.高通量测序在酱香白酒微生态多样性研究中的应用[J].中国酿造,2017,36(5):146-151.
[43]朱文优,聂尧,徐岩.不同季节大曲生产过程中真菌群落结构的演变[J].食品与机械,2017,33(10):16-22.
[44]施思,彭智辅,乔宗伟,等.浓香型大曲贮藏过程中糖化力发酵力变化及真菌多样性分析[J].食品与发酵工业,2017,43(5):76-79.
[45]蒋英丽,邓皖玉,王亚军,等.酱香高温大曲微生物菌群演化规律研究[J].酿酒科技,2018(12):33-38.
[46]谭崇尧,徐军.高通量测序法对不同地域浓香型大曲微生物结构的分析[J].酿酒科技,2018(9):118-122.
[47]李静心,王艳丽,何宏魁,等.基于高通量测序技术解析高温大曲和中高温大曲的真菌群落结构[J].食品与发酵工业,2018,44(12):52-59.
[48]李登勇,黄钧,丁晓菲,等.酱香大曲间微生物群落结构时空特征的表征[J].食品工业科技,2018,39(23):139-145.
[49]戴奕杰,李宗军,田志强.酱香型白酒大曲和糟醅的真菌多样性分析[J].现代食品科技,2018,34(7):97-104.
[50]夏玙,罗惠波,周平,等.不同处理方式的大曲真菌群落差异分析[J].食品科学,2018,39(22):166-172.
[51]母应春,姜丽,苏伟.应用Illumina 高通量测序技术分析3 种酒曲中微生物多样性[J].食品科学,2019,40(14):115-122.
[52]王勇,周森,魏金旺.基于高通量测序技术分析牛栏山大曲微生物多样性[J].中国酿造,2019,38(2):58-61.
[53]吴树坤,谢军,卫春会,等.四川不同地区浓香型大曲微生物群落结构比较[J].食品科学,2019(14):144-152.
[54]刘迪,陈佳伟,张聪,等.组学技术在人参皂苷活性研究中的应用进展[J].食品工业,2019,40(10):285-288.
[55]HANDELSMAN J,RONDON M R,BRADY S F,et al.Molecular biological access to the chemistry of unknown soil microbes:a new frontier for natural products[J].Chem Biol,1998,5(10):245-249.
[56]RIESENFELD C S,SCHLOSS P D,HANDELSMAN J et al.Genomic analysis of microbial communities[J].Annurevgenet,2004,38(1):525-552.
[57]JUNG J Y,LEE S H,KIM J M,et al.Metagenomic analysis of kimchi,a traditional Korean fermented food[J].Appl Environ Microbiol,2011,77(7):2264-2274.
[58]ILLEGHEMS K,DE VUYST L,PAPALEXANDRATOU Z,et al.Phylogenetic analysis of a spontaneous cocoa bean fermentation metagenome reveals new insights into its bacterial and fungal community diversity[J].Plos One,2012,7(5):38040.
[59]LÜ C Y,CHEN C Y,GE F,et al.A preliminary metagenomic study of puer tea during pile fermentation[J].J Sci Food Agr,2013,93(11):3165-3174.
[60]WOLFE B E,BUTTON J E,SANTARELLI M,et al.Cheese rind communities provide tractable systems for in situ and in vitro studies of microbial diversity[J].Cell,2014,158:422-33.
[61]SULAIMAN J,GAN H M,YIN W F,et al.Microbial succession and the functional potential during the fermentation of Chinese soy sauce brine[J].Front Microbiol,2014,5:556.
[62]佟婷婷,田丰伟,王刚,等.基于宏基因组分析四川泡菜母水作引子的泡菜发酵过程中细菌多样性变化[J].食品工业科技,2015,36(21):173-177.
[63]ILLEGHEMS K,WECKX S,DE VUYST L.Applying meta-pathway analyses through metagenomics to identify the functional properties of the major bacterial communities of a single spontaneous cocoa bean fermentation process sample[J].Food Microbiol,2015,50:54-63.
[64]CHEN P,WU Z G,ZHAO Y,et al.Cultivation-independent comprehensive investigations on bacterial communities in serofluid dish,a traditional Chinese fermented food[J].Genomics Data,2016,7:127-128.
[65]CHEN P,ZHAO Y,WU Z G,et al.Metagenomic data of fungal internal transcribed spacer from serofluid dish,a traditional Chinese fermented food[J].Genomics Data,2016,7:134-136.
[66]ESCOBAR-ZEPEDA A,SANCHEZ-FLORES A,BARUCH M Q.Metagenomic analysis of a Mexican ripened cheese reveals a unique complex microbiota[J].Food Microbiol,2016,57:116-127.
[67]BORA S S,KEOT J,DAS S,et al.Metagenomics analysis of microbial communities associated with a traditional rice wine starter culture (Xajpitha)of Assam,India[J].3 Biotech,2016,6(2):153.
[68]WANG H,WEI Q,GUI S,et al.Metagenomic profiling of the bacterial community changes from koji mash stage in the brewing of soy sauce[J].Pol J Microbiol,2047,66(4):537-541.
[69]章克昌.酒精与蒸馏酒工艺学[M].北京:中国轻工业出版社,2004:79-90.
[70]梁金辉,李安军,李兰,等.基于Biolog ECO 技术分离鉴定古井贡酒大曲微生物[J].酿酒,2017,44(1):28-32.